牛樟芝不同发育阶段菌丝体实时荧光定量PCR中内参基因的筛选
李晶 王泽辉 陈莉 刘艳玲 夏舒宁 林占熺


摘 要:实时荧光定量PCR(quantitative Real-time PCR,qRT-PCR)技术具有较高的敏感性、准确性和特异性,被广泛应用于基因表达分析。在基因表达分析中,选择合适的内参基因是衡量样品表达量和准确性的重要前提和保障。本研究以液体发酵7、21、35 d的牛樟芝菌丝体为样本,采用qRT-PCR技术检测牛樟芝菌丝体内Actin、TPK、Floxuridine、CAP-Gly、α-Amylase、Phosphatase、HA2-helicase、MAGT1等8个候选内参基因在3组样本中的表达水平,采用geNorm、NormFinder、BestKeeper 3個软件对其分别进行稳定性比较和评价。结果表明,geNorm软件计算得出Floxuri?dine、HA2-helicase、TPK、CAP-Gly在牛樟芝菌丝体中具有更高的稳定性;NormFinder软件计算得出TPK、CAP-Gly、Floxuridine、HA2-helicase在牛樟芝菌丝体中具有较高的稳定性;BestKeeper软件计算得出HA2-helicase的表达水平最好,其次是TPK和MAGT1。综合分析结果认为TPK和HA2-helicase比其他内参基因更稳定,更适合作为牛樟芝菌丝体研究的内参基因。可见,通过牛樟芝菌丝体转录组数据来筛选和挖掘稳定表达的内参基因具有可靠性、高效性和可行性,为牛樟芝菌丝体基因表达分析提供了可靠的内参基因。
关键词:牛樟芝;内参基因;实时荧光定量PCR中图分类号:S567.3;R282 文献标识码:A
Reference Genes Selection for Quantitative Real-time PCR inAntrodia cinnamomea Mycelium of Different Development Stages
LI Jing1, WANG Zehui1,2, CHEN Li2, LIU Yanling1, XIA Shuning2, LIN Zhanxi1*
1. China National Engineering Research Center of JUNCAO Technology, Fujian Agriculture and Forestry University, Fuzhou, Fujian 350002, China; 2. College of Life Science, Fujian Agriculture and Forestry University, Fuzhou, Fujian 350002, China
Abstract: Quantitative Real-time PCR (qRT-PCR) technology is widely used in the analysis of gene expression because of its high sensit-ivity, high accuracy and high specificity. Therefore, it is important to select appropriate reference genes to measure the expression and accuracy of the sample. In this study, the expressions of 18 candidate internal reference genes ofActin,TPK,Floxuridine,CAP-Gly,α-Amylase,Phosphatase,HA2- helicaseandMAGT1were detected by qRT-PCR withA. cinnamomeaMycelium under 7, 21 and 35 d liquid fermentation time. The stability of the internal reference genes were analyzed using softwares of geNorm, NormFinder and BestKeeper.Floxuridine/HA2-helicase,TPK,CAP-Glygenes were found more stable with geNorm.TPK,CAP-Gly,Floxuridine,HA2-helicasewere found more stable with NormFinder.HA2-helicasewas found to be best expressed with BestKeeper, followed byTPKandMAGT1. The comprehensive analysis showed thatTPKandHA2-helicasehad the best stability and were more suitable as the internal reference genes forA. camphorata. The results of this study would have a practical guiding role in the analysis of the selection of internal reference genes in the gene expression ofA. cinnamomeaby qRT-PCR.
Keywords: Antrodia cinnamomea; reference gene; quantitative Real-time PCR
DOI: 10.3969/j.issn.1000-2561.2020.06.013
随着转录组技术在复杂生物学过程、代谢途径及逆境生理胁迫等相关研究中的广泛应用,实时荧光定量PCR技术(Quantitative Real-time PCR,qRT-PCR)成为生物学中研究基因定量表达最常见、简单、快速的方式之一,具有高灵敏性和特异性,被广泛应用于分子医学、食品安全检测和病虫害防治[1-2]。qRT-PCR對样本的RNA和cDNA的质量、完整性、引物特异性、扩增效率以及内参基因的选择具有较严格的要求。内参基因(reference genes)是指在细胞中能够稳定表达的一类基因,是细胞基本转录组的成分之一,它参与维持细胞的基本功能。内参基因的基本功能是消除不同样品间RNA的提取质量、反转录效率等对定量表达分析造成的偏差,起到校正的作用[3]。因此,随着基因表达的深入研究,选择合适的内参基因是保障实验分析结果准确性的重要前提[4]。目前,在真菌中常用的内参基因包括肌动蛋白(actin)、泛素(ubiquitin,UBQ)、18S核糖体RNA(18S rRNA)、微管蛋白(tubulin beta,TUB)和翻译延长因子(translation elongation factor,TEF)等。然而,内参基因的转录水平随着生物体发育阶段和实验条件的变化而有所差异[5-6],在研究过程中,若选择不能稳定表达的内参基因,将对实验结果的准确性产生巨大影响。
牛樟芝(Antrodia cinnamomea)为台湾地区常见药用菌,又名樟芝、樟菇、牛樟菇、红樟芝,属担子菌门(Basidiomycota)、薄孔菌属(Basi?diomycetes)[7]。牛樟芝仅生在中国台湾保育树种牛樟树(Cinnamomum kanehiraiHay)中空的树干内部或腐烂的树干表面[8]。近年来,随着对牛樟芝分子生物学研究的不断深入,qRT-PCR技术成为重要研究手段之一,而选择表达高效、稳定的内参基因是开展qRT-PCR的关键。目前,对于牛樟芝内参基因的研究报道较少,本研究选取3种不同阶段牛樟芝菌丝体,利用qRT-PCR技术筛选来自牛樟芝转录组数据的8个候选内参基因,采用geNorm[9]、NormFinder[10]和BestKeeper[11]软件进行基因的稳定性评估,从而选择稳定表达的内参基因,为其后续基因表达研究、转录组数据分析提供更可靠的参考和校正依据。
1 材料与方法
1.1材料
牛樟芝菌株由福建农林大学国家菌草工程技术研究中心菌种保藏室提供。将菌种接种到PDA培养基上进行活化,然后转移至牛樟芝液体培养基,置于120 r/min摇床,在28 ℃黑暗培养7、21、35 d,然后收集菌丝体备用[12]。
1.2方法
1.2.1 牛樟芝总RNA提取及cDNA合成 参照全式金生物公司的Trizol试剂盒说明书步骤提取牛樟芝菌丝体的总RNA,每个样品设置3次重复,采用NanoDrop 2000对提取的总RNA浓度和质量进行检测,合格后置于-80 ℃保存备用。
参照艾德莱生物科技公司的TRUEscript RT Master Mix试剂盒说明书步骤进行cDNA合成,每20 μL体系中含有500 ng总RNA样品。将合成后的cDNA置于?20 ℃保存备用。
1.2.2 内参基因的选择与引物设计 候选内参基因序列是根据转录组数据中的序列E-value<1e-50进行筛选获得的[12]。利用Primer 5.0软件设计引物(表1),引物退火温度为50~60 ℃,设计的引物由铂尚生物技术(上海)有限公司合成。
1.2.3 实时荧光定量PCR 按照全式金生物公司的SYBR TransStart实时荧光定量PCR试剂盒说明书进行反应,反应在Bio Rad CFX96实时荧光定量PCR仪上进行。反应总体系为20 μL:总cDNA模板1 μL,正反引物各1 μL(10 μmol/L),2×SYBR qPCR Mix 10 μL,ddH2O补足至20 μL,每个样品3次重复。qRT-PCR扩增程序为:95 ℃预变性5 min;95 ℃变性5 s,52~55 ℃退火20 s,40个循环,在延伸阶段采集荧光信号,反应结束后增加60~95 ℃熔解曲线分析。
1.2.4 扩增效率分析与标准曲线绘制 取3个cDNA样品等量混合后,按10倍依次稀释6个梯度作为模板,每个反应3个重复。熔解温度通过PCR仪计算得到,根据所得的Ct值绘制标准曲线,然后计算标准曲线方程,从而得出斜率(k)、线性相关系数(R2)、扩增效率(E)。
1.3数据处理
利用PCR仪统计Ct值,根据公式Q=2-ΔΔCt计算各样品的相对表达量Q,其中ΔCt=Ct(样品)?Ct(min),Ct(min)为所有样品中最低的Ct值。根据8个内参基因在牛樟芝菌丝体3个发育阶段的相对表达量,采用geNorm、NormFinder、BestKeeper软件分别计算内参基因的稳定值,比较并确定适宜的内参基因。
2 结果与分析
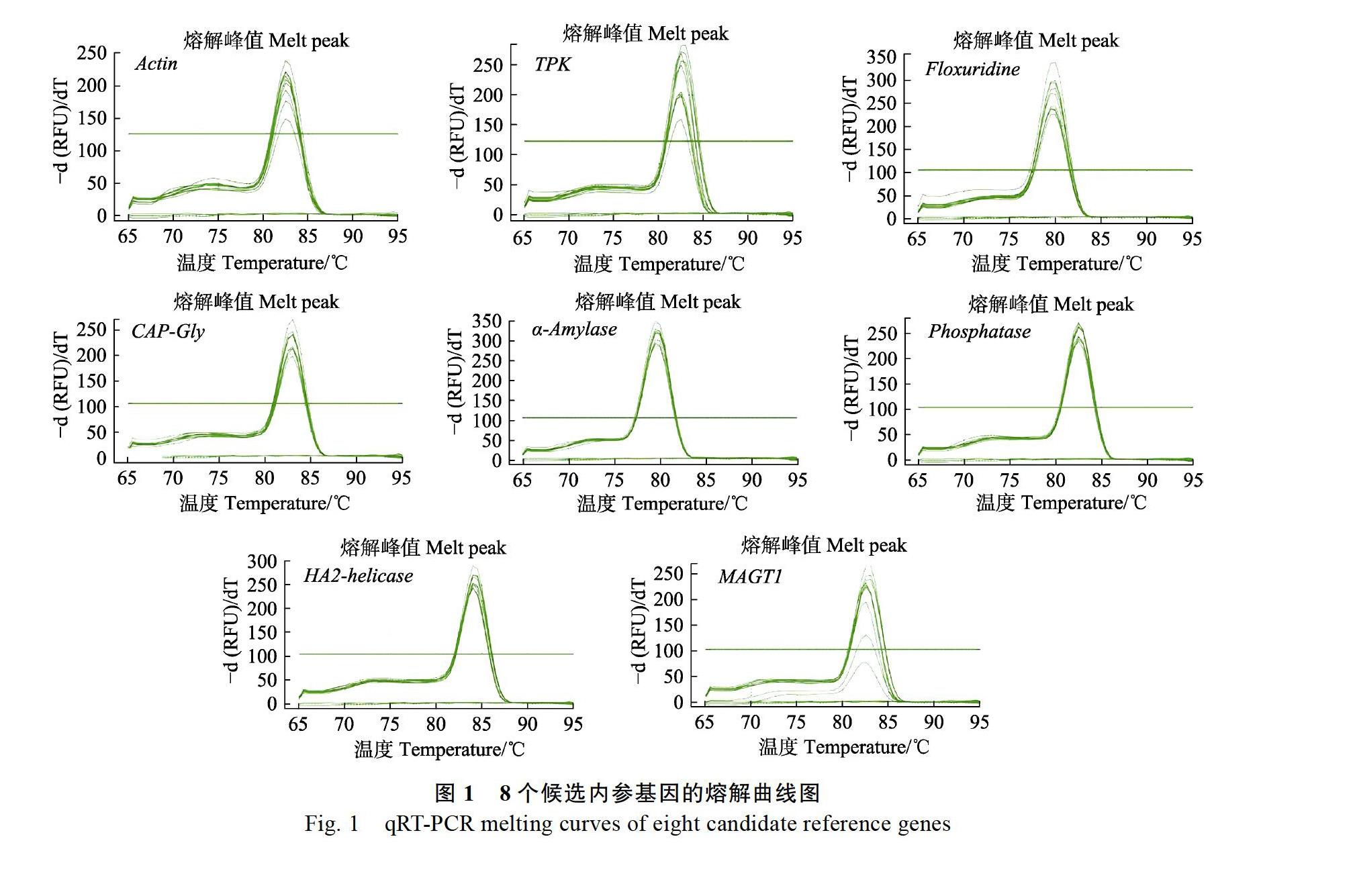
2.1引物扩增效果及特异性分析
以等量梯度稀释的cDNA为模板进行PCR扩增,绘制每个内参基因的标准曲线,得到引物的相关数据(表2)。分析结果表明,候选内参基因具有良好的扩增效果和线性相关性(表2、图1、图2)。
2.2内参基因表达丰度分析
由于Ct值的变化与内参基因的表达水平有密切关系。Ct值越高,其表达量越低,其表达丰度越低;反之,表达丰度越高。对8个内参基因在发酵时期进行表达丰度分析,发现随着培养时间的增长,Actin和CAP-Gly表达丰度不随培养时间的增长而增加,而其他候选内参基因均随培养时间的增长表达丰度变高(图3)。
2.3geNorm软件对内参基因的稳定性评价
geNorm软件常用于实时荧光定量PCR中筛选及确定内参基因。其分析结果以候选基因表达稳定度M值(average expression stability values)进行排序,以M=0.5为取舍值,M值越高,稳定性越差,相反M值越低则稳定性越高[9]。采用geNorm软件分析8个候选内参基因,结果显示最稳定的基因是二氢尿嘧啶核苷和HA2解旋酶(图4)。
根据geNorm软件计算结果,不同培养时期牛樟芝菌丝体中8个内参基因的表达稳定性排序前3位依次为Floxuridine/HA2-helicase>TPK>CAP-Gly,其中Floxuridine、HA2-helicase、TPK的M值都小于软件推荐的0.5,可以作为实时荧光定量PCR分析中的内参基因。同时,geNorm软件根据配对变异系数来确定最佳内参基因数目,即Vn/(n+1)<0.15说明该条件下最佳内参基因的数目为n个。如图5所示,只有V2/3时,变异系数低于0.15,则在牛樟芝液体发酵时期最好使用两个内参基因,即H2A解旋酶和二氢尿嘧啶核苷作为内参基因(图5)。
2.4NormFinder软件对候选内参基因的稳定性评价
NormFinder软件是由Claus等[10]开发的、用于筛选稳定的内参基因的一种程序,其计算原理与geNorm软件相似,即结合组内方差与组间方差计算各内参基因的稳定值。通过先获得的内参基因表达稳定值,然后根据稳定值大小筛选出最合适的内参基因,判定标准为表达稳定值最小的内参基因为最合适的内参基因;反之越不稳定[13]。表3结果显示,筛选的内参基因稳定性依次是TPK>CAP-Gly>Floxuridine>HA2-helicase>Actin。其他内参基因Phosphatase、α-Amylase、MAGT1的穩定值>1不适合作为内参基因[10]。
2.5 BestKeeper软件对候选内参基因的稳定性评价
BestKeeper软件根据Q-PCR的Ct值计算各个基因的相关系数(r)、标准偏差(SD)、变异系数(CV)等,通过比较值的大小来分析内参基因的表达水平;标准偏差(SD)>1,则内参基因的稳定性差,不可以用作内参基因;相关系数(r)值越低,其稳定性越高。软件分析得到的结果如表4所示,根据BestKeeper软件计算原理可知HA2-helicase的表达水平最好,其次是TPK和MAGT1。
3 讨论
随着生物技术的高速发展和推广应用,qRT- PCR技术由于其特异性强、灵敏度高和重复性好已成为各行业、各领域广泛用于基因表达研究的技术之一[14]。内参基因在qRT-PCR技术应用过程
中作为参考、对照基因具有重要作用,选择合适的内参基因是检测基因表达水平变化时的重要参照物。理想的内参基因所具有的特点包括,应在所有组织和细胞类型中都能稳定表达,且不受外界环境及组织形态的影响[15],其作用是矫正了qRT-PCR操作过程中的操作误差,从而保证了实验结果的稳定性和准确性。本研究通过前期实验结果,系统筛选和分析了Actin、TPK、Floxu?ridine、CAP-Gly、α-Amylase、Phosphatase、HA2- helicase和MAGT1等8个候选内参基因在牛樟芝不同培养阶段牛樟芝菌丝体中的表达结果,经geNorm、NormFinder、BestKeeper等常用内参基因分析软件对结果进行评估,其中geNorm软件筛选得到的最适合的内参基因为Floxuridine、HA2-heli?case、TPK;NormFinder软件筛选得到的最适合的内参基因是TPK、CAP-Gly、Floxuridine、TPK;BestKeeper软件筛选得到的最适合的内参基因是HA2-helicase、TPK、MAGT1。由于3种软件的分析计算方法不同,其结果也略有不同[15-16],该现象也广泛存在于其他物种的内参基因研究中[17-20]。综合分析各评估结果,认为TPK和HA2- helicase适合作为牛樟芝菌丝体qRT-PCR研究中的内参基因。酪氨酸激酶(PTKs)基因家族是生物体信号网络的核心组分,是在细胞通讯、细胞黏附、细胞增殖和信号转导过程中起到关键作用的一类生长因子,具有能促进生长因子信号转导、参与细胞生长、增殖、分化等功能。在目前已报道的研究中,并未发现该基因作为内参基因的报道,但在研究其基因家族差异进化的过程中,发现PTK经历了更显著的规模化扩张和成员基因间的功能分化,组织表达分化更明显,催化更特异的底物发生磷酸化,参与特异的差异进化作用[21]。通常在多数作物中,Actin和tubulin作为常见的内参基因,韩晓雪等[22]利用geNorm和Norm?Finder软件分析得到Actin可以作为番茄基因表达中的内参基因;在对日本沼虾的研究中发现,在缺氧条件下筛选得到的β-act是最稳定的内参基因[23];Gopalam等[24]研究认为在盐胁迫条件下,Actin、EF1α、18S等在红树中能稳定表达。在本研究中,采用3项软件进行评估,结果表明Actin在牛樟芝菌丝体中的表达呈一定的波动性和不稳定性,不能作为最适合的内参基因。由此可见,内参基因的表达相对稳定性存在于特定物种和特定细胞中,因此,需要根据不同实验条件和样品状态筛选合适的内参基因。
目前,常用的内参基因评价软件包括ge?Norm、NormFinder和BestKeeper[25],本研究利用这3种评价软件所分析的结果趋势基本相同,略有差异,因为3种评价软件对于内参基因的选择都是依据Ct值设计的,但计算原理和方法均有不同,结果也会有一定差异[16]。geNorm软件在对内参基因的评价过程中不用考虑其他内参基因结果来判定合适的内参基因[9, 26],而NormFinder软件需要考虑其他内参基因再选择合适的内参基因[10];BestKeeper软件不但能分析内参基因的稳定性,还能比较目的基因的表达水平[11]。因此,在实际应用过程中,需要充分考虑程序的适用性和特点,确保内参基因筛选结果的准确性,降低实验误差。
参考文献
- Yoo W G, Kim T I, Li S, et al. Reference genes for quantitative analysis on clonorchis sinensis gene expression by real- time PCR[J]. Parasitology Research, 2009, 104(2): 321-328.
- Lovdal T, Lillo C. Reference gene selection for quantitative real-time PCR normalization in tomato subjected to nitrogen, cold, and light stress[J]. Analytical Biochemistry, 2009, 387(2): 238-242.
- Derveaux S, Vandesompele J, Hellemans J. How to do successful gene expression analysis using real-time PCR[J]. Methods, 2010, 50(4): 227-230.
- Gu C S, Liu L Q, Xu C, et al. Reference gene selection for quantitative real-time RT-PCR normalization in Iris. lactea var. chinensis roots under cadmium, lead, and salt stress conditions[J]. The Scientific World Journal, 2014, 2014: 532713.
- Long X Y, Wang J R, Thérèse O, et al. Genome-wide identification and evaluation of novel internal control genes for q-PCR based transcript normalization in wheat[J]. Plant Molecular Biology, 2010, 74(3): 307-311.
- Tang R Y, Dodd A, Lai D, et al. Validation of zebrafish (Danio rerio) reference genes for quantitative real-time RT-PCR normalization[J]. Acta Biochimica et Biophysica Sinica, 2007(5): 384-390.
- Chang T T, Chou W N. Antrodia cinnamomea sp. Nov. on Cinnamomum kanehirai in Taiwan[J]. Mycological Research, 1995, 99(6): 756-758.
- Lu M C, El-Shazly M, Wu T Y, et al. Recent research and development of Antrodia cinnamomea[J]. Pharmacology and Therapeutics, 2013, 139(2): 124-156.
- Vandesompele J, De Preter K, Pattyn F, et al. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes[J]. Genome Biology, 2002, 3(7): research0034.
- Andersen C L, Jensen J L, ?rntoft T F. Normalization of real-time quantitative reverse transcription-PCR data: a model-based variance estimation approach to identify genes suited for normalization, applied to bladder and colon cancer data sets[J]. Cancer Research, 2004, 64(15): 5245-5250.
- Pfaffl M W, Tichopad A, Prgomet C, et al. Determination of stable housekeeping genes, differentially regulated target genes and sample integrity: Bestkeeper – excel-based tool using pair-wise correlations[J]. Biotechnology Letters, 2004, 26(6): 509-515.
- 李 晶. 牛樟芝MVA途徑中基因克隆及转录组、代谢组研究[D]. 福州: 福建农林大学, 2016.
- 吴建阳, 何 冰, 杜玉洁, 等. 利用genorm、normfinder和bestkeeper软件进行内参基因稳定性分析的方法[J]. 现代农业科技, 2017(5): 278-281.
- Udvardi M K, Czechowski T, Scheible W R. Eleven golden rules of quantitative RT-PCR[J]. Plant Cell, 2008, 20(7): 1736-1737.
- Bustin S A, Benes V, Nolan T, et al. Quantitative real-time RT-PCR – a perspective[J]. Journal of Molecular Endocrinology, 2005, 34(3): 597-601.
- Hu R, Qi G, Kong Y, et al. Comprehensive analysis of NAC domain transcription factor gene family in Populus trichocarpa[J]. Bmc Plant Biology, 2010, 10(1): 145.
- Hong S Y, Seo P J, Yang M S, et al. Exploring valid reference genes for gene expression studies in brachypodium distachyonby real-time PCR[J]. Bmc Plant Biology, 2008, 8(1): 112.
- Wan H, Zhao Z, Qian C, et al. Selection of appropriate reference genes for gene expression studies by quantitative real-time polymerase chain reaction in cucumber[J]. Analytical Biochemistry, 2010, 399(2): 257-261.
- Huis R, Hawkins S, Neutelings G. Selection of reference genes for quantitative gene expression normalization in flax (Linum usitatissimum L.)[J]. Bmc Plant Biology, 2010, 10(1): 71.
- Mafra V, Kubo K S, Alves-Ferreira M, et al. Reference genes for accurate transcript normalization in citrus genotypes under different experimental conditions[J]. PloS One, 2012, 7(2): e31263.
- 刘当云, 谷 迅. 脊椎动物酪氨酸激酶基因家族差异进化研究[J]. 基因组学与应用生物学, 2018, 37(8): 3339-3353.
- 韩晓雪, 韩佳轩, 姜 晶. 番茄在非生物胁迫下实时定量RT-PCR中内参基因的筛选[J]. 分子植物育种, 2015, 13(4): 822-831.
- Mallona I, Lischewski S, Weiss J, et al. Validation of reference genes for quantitative real-time PCR during leaf and flower development in Petunia hybrida[J]. BMC Plant Bio?logy, 2010, 10(1): 4.
- Gopalam R, Rupwate S D, Tumaney A W. Selection and validation of appropriate reference genes for quantitative real-time PCR analysis in Salvia hispanica[J]. PloS One, 2017, 12(11): e0186978.
- 张玉芳, 赵丽娟, 曾幼玲. 基因表达研究中内参基因的选择与应用[J]. 植物生理学报, 2014, 50(8): 1119-1125.
- Infante C, Matsuoka M P, Asensio E, et al. Selection of housekeeping genes for gene expression studies in larvae from flatfish using real-time PCR[J]. BMC Molecular Biology, 2008, 9(1): 28.

