基于RT-PCR与平板计数法对比分析发酵香肠发酵过程中的乳酸菌数
杨明阳 田建军 赵丽华 张开屏 景智波 李权威 靳 烨*
(1 内蒙古农业大学食品科学与工程学院 呼和浩特010018 2 内蒙古商贸职业学院食品工程系 呼和浩特010070)
乳酸菌是一类革兰氏阳性、过氧化氢酶阴性、无芽孢细菌的总称[1]。乳酸菌作为发酵剂在肉制品发酵中可以缩短产品加工周期,改善产品品质和风味,提高产品质量稳定性,并在抑制腐败微生物方面也有一定的作用[2-4]。发酵肉制品中乳酸菌的数量是衡量肉制品质量好坏的重要指标[5]。
目前对发酵香肠中乳酸菌数的测定一般采用传统的平板计数法,这种方法操作误差大,用时长、灵敏度低,无法准确、迅速地反映发酵过程中菌群的动态变化[6]。所使用的选择性培养基无法对虽有代谢活力但难以培养的菌体细胞进行计数。
近年来国内外研究人员也开始利用分子生物学手段检测乳酸菌,应用较为广泛的为实时荧光定量PCR (real-time polymerase chain reaction,RT-PCR),该方法不仅具有传统PCR 的检测速度快、灵敏度高的特点,还具有无污染、特异性强、重复性好、检测时间短等特点,在对食品中优势菌和有害菌的检测上得到一定的应用[7-9]。Quijada 等[10]使用PMA-qPCR 方法检测西班牙发酵香肠中人腺病毒-2 和门式病毒,结果发现该方法能快速检测出在现有技术下不能培养出的病毒。Alves 等[11]首次使用多重实时荧光定量PCR 检测鸡肉中的沙门氏菌。Furet 等[12]研究发现实时荧光定量与传统平板计数相比,对发酵乳制品的检测更为精确。汪冬冬等[13]使用实时荧光定量PCR 对泡菜中的植物乳杆菌、葡萄球菌属、芽孢菌属和大肠埃希菌进行快速鉴定。
为了快速定量分析乳酸菌,本试验通过将植物乳杆菌F16 作为发酵剂加入发酵香肠的发酵过程中,分别用RT-PCR 技术和平板计数检测发酵剂组和自然发酵组发酵过程中乳酸菌数的动态变化,构建两者间的线性回归方程,以期寻找一条快速途径对乳酸菌进行定量。
1 材料与方法
1.1 材料
1.1.1 材料与试剂 羊后腿肉和羊尾肥膘,内蒙古蒙羊食品有限公司;天然羊肠衣(15mm),润泰(河北)肠衣有限公司。
2xSG Fast qPCR Master Mix,大连宝生物工程有限公司;PCR 引物,上海桑尼生物科技有限公司;PCR Master Mix,上海生工;QIAamp DNA Stool Mini Kit,德国;无菌水、DNA 提取试剂盒,北京天根生化;核酸燃料,索莱宝;MRS 培养基所用试剂以及具体的配制方法参考文献[14]。
植物乳杆菌 (Lactobacillus plantarum)F16 分离自传统发酵乳制品中;植物乳杆菌CICC:6238,由内蒙古农业大学肉品科学技术团队提供。
1.1.2 引物 引物序列设计由上海生工生物工程有限公司合成,具体序列及来源如表1所示。

表1 目标菌及其RT-PCR 扩增引物Table 1 Dominant bacteria and RT-PCR amplification primes
1.1.3 仪器与设备 ZHJH-C1112C 超净台,上海智城分析仪器制造有限公司;普通PCR 仪,美国Applied Biosystems;CR3i 高速冷冻离心机,美国赛默飞公司;CFX96TM Real-Time PCR 仪、凝胶成像系统、电泳仪,美国Bio-rad;恒温恒湿箱,上海智诚分析仪器有限公司。
1.2 方法
1.2.1 发酵香肠制作与取样 配方:瘦肉70%,肥肉20%,食盐2.5%,葡萄糖0.5%,抗坏血酸0.05%,亚硝酸钠/硝酸钠0.01%,黑胡椒粉0.5%,白酒2.0%,发酵剂107CFU/g。

表2 发酵香肠加工条件Table 2 The processing conditions of fermented sausage
按照以上配方和发酵条件,以自然发酵为对照,以植物乳杆菌F16 为发酵剂制作发酵香肠。
取样:分别在0,2,4,6,12 d 和24 d 取样(发酵香肠)分析,这些对应的时间点分别为发酵香肠腌制后、发酵、成熟、干燥和储藏期。每次取样5 g,置真空袋中-80℃密封保存。
1.2.2 样品乳酸菌的计数 参照GB4789.35-2016《食品安全国家标准》测定样品的乳酸菌数。
1.2.3 样品总DNA 的提取 参照试剂盒 ( QIAamp DNA Stool Mini Kit)的方法并作适当修改来提取香肠发酵过程中样品的DNA,于-20 ℃密封保存。具体操作步骤如下:
1)称取180~220 mg 样品于2 mL 离心管中,在冰上操作。
2)加入1.4 mL 缓冲液ASL 涡旋1 min,95 ℃处理5 min,涡旋15 s,12 000 r/min 离心2 min。
3)吸取1.2 mL 上清液于2 mL 离心管中,加入1 个抑制片到样品中,立刻涡旋1 min,室温孵育1 min 使抑制片充分吸附抑制物。
4)12 000 r/min 离心6 min,将所有上清液转移到1.5 mL 离心管中,弃沉淀,12 000 r/min 离心6 min。
5)吸取第4 步中的200 μL 上清液,置于含有15 μL 蛋白酶K 的1.5 mL 离心管中,加入200 μL 缓冲液AL,涡旋15 s。
6)70 ℃孵育10 min,加入200 μL 无水乙醇(96%~100%),涡旋混匀。
7)将上一步中混匀的溶解液小心转移到柱子中,12 000 r/min 离心2 min。
8)打开柱子,加入500 μL 缓冲液AW1,12 000 r/min 离心2 min。
9)加入500 μL 缓冲液AW2,12 000 r/min 离心6 min,弃含离心液的收集管,12 000 r/min 离心2 min。
10)把柱子转移到1 个1.5 mL 离心管中,加入200 μL AE,12 000 r/min 离心2 min,离心管中的溶液即DNA。
最终得到的DNA 使用1%的琼脂糖电泳和紫外分光光度计检测其质量和浓度,置于-20 ℃冰箱中保存备用。
1.2.4 标准菌株的活化及DNA 的提取 用MRS培养基对植物乳杆菌活化培养3 代后(菌液浓度为108CFU/mL),按照天根试剂盒说明书进行细菌基因组DNA 提取,用溶菌酶破壁处理标准菌株,用125 μL 缓冲液TE 将DNA 溶出。用ND-1000型微量紫外分光光度计测定其质量浓度及光密度值(OD260nm/280nm),提取的DNA 于-20 ℃保存备用。
1.2.5 标准品的制作 将1.2.3 节中测得的DNA原液的质量浓度稀释至100 ng/μL,以此为扩增模板,用表1中的特异性引物对其所对应的标准菌株进行特异性PCR 扩增,所得目标片段约340 bp,扩增体系为:PCR MIX 25 μL,正、反向引物各2 μL,2 μL DNA 模板,无菌水补充至50 μL;反应程序:95 ℃预变性7 min;95 ℃变性30 s,58 ℃退火1 min,72 ℃延伸30 s,共35 个循环;72 ℃延伸10 min,4 ℃保存。扩增产物经1%琼脂糖凝胶电泳检测后,用无菌手术刀切取目的DNA 条带,采用DNA 纯化试剂盒对目的DNA 片段回收、纯化,用ND-1000 型微量紫外分光光度计测定其质量浓度及光密度值(OD260nm/280nm),于-20 ℃保存备用。
1.2.6 RT-PCR 扩增 植物乳杆菌RT-PCR 反应体系见表3。

表3 RT-PCR 反应体系Table 3 RT-PCR reaction system
1.2.7 发酵香肠中特异性菌株的定量PCR 分析将1.2.4 节中的植物乳杆菌DNA 质量浓度用无菌超纯水进行10 倍稀释,以10-1~10-6的DNA 稀释液为模板,用表1中的特异性引物对其对应的标准菌株稀释液模板进行RT-PCR 扩增,扩增程序:95 ℃预变性30 s;95 ℃变性5 s,58 ℃退火30 s,72℃延伸30 s,共35 个循环;72 ℃延伸10 min。以其浓度的对数值为横坐标,扩增循环数(Ct)值为纵坐标绘制标准曲线。
以上述提取的发酵香肠样品宏基因组DNA为样品模板,以无菌无酶水为阴性对照模板,分别用表1中的特异性引物,对发酵香肠样品中植物乳杆菌进行定量分析。
1.2.8 熔解曲线制作 当1.2.5 节扩增程序中35个循环结束后,在72 ℃每5 s 升高0.5 ℃,直至95℃的每个阶段收集荧光信号。以温度为横坐标,荧光值变化速度为纵坐标绘制熔解曲线。
1.2.9 线性方程的建立以及试验验证 根据RTPCR 和平板计数结果,建立两种方法间的线性关系。任取1 批样品进行DNA 的提取和平板计数,比较通过线性关系和平板计数所得活菌数的吻合度。
1.3 数据处理
采用spass18 和Excel(Version 2013)软件进行数据处理与分析。
2 结果与分析
2.1 RT-PCR 方法的建立
2.1.1 标准菌株PCR 产物电泳结果 以提取的植物乳杆菌DNA 为模板,使用特异性引物lap F和lap R 进行PCR 扩增后,对产物进行纯化。纯化的PCR 产物条带清晰、明亮,无拖带现象,并且条带在340bp 左右,符合试验要求。
2.1.2 标准曲线的建立 不同浓度梯度的植物乳杆菌DNA 模板的PCR 扩增荧光曲线如图2所示。植物乳杆菌在85.5 ℃出现单一的熔解峰,说明在扩增过程中没有产生非特异性扩增产物和二聚体,扩增产物单一,特异性强。植物乳杆菌OD260nm/280nm值在1.8~2.0 范围,植物乳杆菌原DNA质量浓度为138.7 μg/mL,以相对浓度的对数值为横坐标,以Ct 值为纵坐标,得到菌株的标准曲线为y=-3.553x+2.467(R2=0.993)。扩增效率为E=91.2%,标准质粒的扩增效率在90%~110%范围,R2≧0.99,符合RT-PCR 的基本要求[16]。
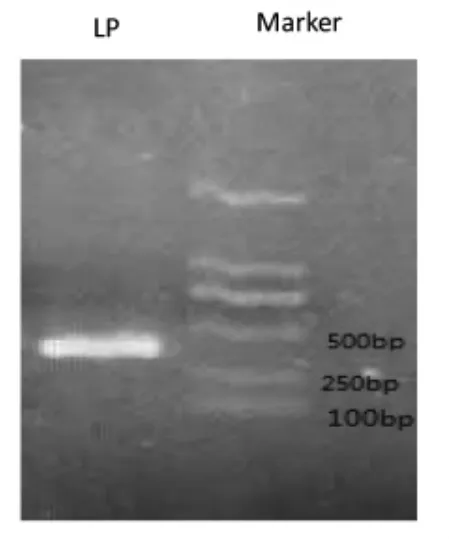
图1 植物乳杆菌PCR 纯化产物凝胶电泳图Fig.1 Gel electrophoresis diagram of PCR purified product of Lactobacillus plantarum

图2 植物乳杆菌实时荧光定量PCR 的熔解曲线Fig.2 Quantitative PCR melting curves for Lactobacillus plantarum
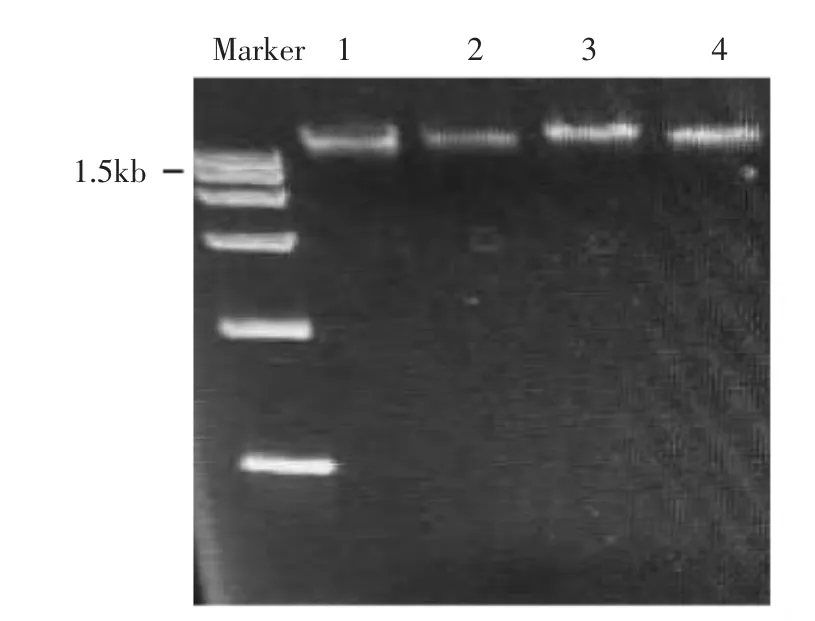
图3 样品宏基因组DNA 凝胶电泳图Fig.3 Gel electrophoresis of sample metagenomic DNA
2.2 RT-PCR 检测发酵香肠发酵过程中乳酸菌数的动态变化
2.2.1 发酵香肠样品宏基因组DNA 的提取结果 采用试剂盒法提取12 份发酵香肠样品的宏基因组DNA,用微量分光光度计检测其质量浓度和OD260nm/280nm。发酵香肠样品宏基因组DNA 的质量浓度在50~300 μg/mL 之间,OD260nm/280nm值在1.8~2.0之间。部分样品用1.0%琼脂糖凝胶电泳后,条带清晰、明亮,无拖带现象,条带大小15 000 bp,说明提取样品的总DNA 较好。
2.2.2 发酵香肠发酵过程中乳酸菌数的动态变化 本试验方法符合RT-PCR 定量的基本条件,可用于定量分析。以标准曲线为依据,采用RTPCR 方法对发酵香肠中乳酸菌数计数,其对数值变化趋势见图4所示。F16 组和对照组在第0 天的拷贝数分别为6.46×107copies/g 和5.13×106copies/g,发酵香肠在发酵过程中乳酸菌数呈先上升后下降趋势,在发酵前期呈上升趋势,F16 组在第4 天达到峰值2.57×109copies/g,对照组在第6 天达到峰值3.89×107copies/g。
2.3 RT-PCR 与平板计数法检测发酵香肠发酵过程中乳酸菌数的对比
如图5所示,采用平板计数法对香肠发酵中乳酸菌进行计数,其数值变化趋势与RT-PCR 一致。F16 组和对照组的乳酸菌数在第6 天达到最大值,分别为7.41×107CFU/g 和6.46×106CFU/g,与Matamgas M 等[17]研究结果一致。发酵香肠中的乳酸菌数在48~72 h 迅速上升,在成熟后期,F16组乳酸菌数约为108CFU/g,表明乳酸菌是这一过程结束时的优势菌群,随着发酵的进行,在24 d时达到较低值[18]。发酵剂组的乳酸菌数显著高于对照组,说明加入的植物乳杆菌为发酵香肠发酵过程中的优势菌群。
由图4和图5可知,在每个取样点,平板计数结果都少于RT-PCR 计数1 lg(CFU/g)左右。在发酵过程中,随着发酵的进行,荧光定量所得结果高于平板计数1 lg(CFU/g)左右,这可能是由于实时荧光定量PCR 以定量细菌DNA 为基础,人们普遍认为DNA 水平与细胞活力无关,因为死亡细胞也可能保留DNA 信号,所以实时荧光定量无法区分死菌和活菌,仅是直接对DNA 的拷贝数进行分析[19]。Fujimoto 等[20]在一项对人类志愿者的研究中发现,RT-PCR 过高地估计了粪便中短乳杆菌的数量,并将这一差异归因于死细胞的存在或选择性培养基的使用,低估的数据与本研究相似,约为1 个对数CFU/g。CHO G S 等[21]在使用RT-PCR对植物乳杆菌进行定量分析时发现,RT-PCR 定量结果高于平板计数约1 lgCFU。

图4 RT-PCR 检测发酵香肠发酵过程中乳酸菌数的动态变化Fig.4 Dynamic changes of lactic acid bacteria in the fermentation process of fermented sausage by RT-PCR

图5 平板计数检测发酵香肠发酵过程中乳酸菌数的动态变化Fig.5 Dynamic changes of lactic acid bacteria in the fermentation process of fermented sausage by plate counting
2.4 线性回归方程的建立
结合两种方法检测两组发酵香肠中乳酸菌的动态变化,应用matlab 软件得到线性回归方程y=0.8116x+0.4254(图6)。式中,x 代表拷贝数的对数值,y 代表平板计数所得活菌数的对数值。
2.5 数学模型的验证
选取4 个时期除本试验外的另一组发酵香肠样品进行分析,同时进行DNA 的提取、RT-PCR和平板计数试验。通过RT-PCR 可得到拷贝数x,利用拷贝数x,通过线性回归方程y=0.8116x +0.4254 计算得到活菌数值y,结果发现y 值与平板计数所得活菌数的相似度均在94%以上,二者相差不到1 个数量级,可认为y 值与平板计数所代表的数量级一致,说明本试验所构建的线性方程可替代平板计数方法。

图6 乳酸菌线性回归方程分析Fig.6 Analysis of linear regression equation of lactic acid bacteria

表4 不同发酵时期发酵香肠的模型验证结果Table 4 Results of Model validation of fermented sausages during different fermentation periods
3 讨论
采用实时荧光定量法和平板计数对比分析发酵香肠发酵过程中乳酸菌数的动态变化,所得标准曲线为y=-3.553x+2.467 (R2=0.993 E=91.2%)。Fredlund 等[22]研究发现,当标准曲线的斜率在-3.1 和-3.6 之间,扩增效率在80%~110%之间时,相关系数R2≥0.99,满足上述条件的标准曲线,可用于菌株的定量分析。熔解曲线也只出现单一的熔解峰,无非特异性二聚体产生。采用这一方法对乳酸菌进行定量,与平板计数相比,变化趋势一致,均呈先上升后下降的趋势。F16 组的峰值分别为2.57×109copies/g 和7.41×107CFU/g,对照组的峰值分别为3.89×107copies/g 和6.46×106CFU/g,说明加入的乳酸菌能快速成为发酵过程中的优势菌。荧光定量法所得数值显著高于平板计数1lg(CFU/g)左右,是由于平板计数只对活菌计数,而在发酵香肠发酵过程中存在未被分解的死亡微生物的DNA,或者会形成可存活但受损的非可培养细胞,在扩增阶段,死细胞或损伤细胞的DNA 可作为RT-PCR 模板,可能会导致假阳性结果,因此,试验结果数值存在一定的差异[13]。有研究发现脱氧胆酸钠(SD)和单叠丙酸丙啶(PMA)与RTPCR 技术相结合,可有效消除死亡细胞和损伤细胞的影响,对食品中的活菌细胞快速定量[23]。这可作为下步研究的切入点。
经验证,线性回归方程所得活菌数与平板计数结果的相似度在94%以上,说明可用y=0.8116x+0.4254 预测发酵肉制品的活菌数。
综上所述,应用RT-PCR 技术对乳酸菌定量是可行的。传统的微生物培养方法用时长、灵敏度低,不能准确检测传统发酵食品环境中微生物的多样性分析[24-25]。实时荧光定量操作省时、简单并有较强的特异性和灵敏度,所得结果与平板计数差异较小,优势明显,可作为研究肉类发酵过程中乳酸菌动态变化的工具。

