基于SNP芯片的云南玉米自交系遗传多样性和群体遗传结构分析
张鹏 管俊娇 黄清梅 杨晓洪 张建华 康祝科

摘要:【目的】对107个云南玉米自交系进行遗传多样性和群体遗传结构分析,为云南省玉米种质创新、遗传改良、品种管理等提供理論依据,也为今后深入挖掘优良性状相关基因打下基础。【方法】以云南当地推广的107个优良玉米自交系为供试材料,以45个我国常用玉米骨干自交系作为杂种优势群划分的参照,在Axiom? Maize56K SNP Array平台上利用玉米SNP芯片(56K)进行玉米全基因组扫描,并使用Treebest的NJ-tree模型构建系统发育进化树,利用GCTA(全基因组复杂性状分析)工具进行主成分分析,揭示其遗传多样性与群体遗传结构。【结果】从107个云南玉米自交系中检出5533个均匀分布的高质量SNP分子标记位点。基于这些SNP分子标记位点分析结果可知,107个云南玉米自交系的Neis基因多样性指数(H)为0.2981~0.5000,平均为0.4832;多态信息含量(PIC)为0.2536~0.3750,平均为0.3662;最小等位基因频率为0.5000~0.8178,平均为0.5744。群体遗传结构分析结果显示,K=6时△K最大即供试自交系可划分为六大类群,分别为塘四平头血缘类群、PB血缘类群、335母本血缘类群、自330和旅大红骨血缘类群及2个未知类群,无自交系划分到其他杂交优势群,其中,2个未知类群共37个云南玉米自交系,未能与我国目前已知的10个杂种优势群归在一类。主成分分析结果显示,107个云南玉米自交系与45个我国常用玉米骨干自交系能明显区分,大部分云南玉米自交系集中在我国常用玉米骨干自交系附近,但少数云南玉米自交系与我国常用玉米骨干自交系距离较远。【结论】云南地区玉米种质资源遗传多样性较丰富,含有多个杂种优势群,育种亲本遗传基础丰富,与我国常用玉米骨干自交系能明显区分,且部分与骨干自交系遗传距离较远,可创建新的杂交优势群,具有良好的应用潜力。
关键词: 玉米;SNP芯片;群体遗传结构;遗传多样性;主成分分析
中图分类号: S513.024 文献标志码: A 文章编号:2095-1191(2020)09-2082-08
Genetic diversity and genetic structure of maize inbred lines from Yunnan revealed by SNP chips
ZHANG Peng1, GUAN Jun-jiao1, HUANG Qing-mei1, YANG Xiao-hong1,
ZHANG Jian-hua1*, KANG Zhu-ke2
(1Quality Standard and Testing Technology Research Institute,Yunnan Academy of Agricultural Sciences, Kunming 650205, China; 2Agriculture and rural Bureau of Suijiang County, Zhaotong, Yunnan 657700, China)
Abstract:【Objective】Analyzed the genetic diversity and population genetic structure of 107 inbred lines of maize in Yunnan, in order to provide technical support for maize germplasm innovation, genetic improvement of germplasm resources, variety management, and lay a solid foundation for fine traits related genes mining in the future. 【Method】The 107 maize inbred lines generalized in Yunnan were selected,45 backbone inbred lines commonly used in China were used as reference for heterotic group classification. On Axiom? Maize 56K SNP Array platform, maize SNP chip(56K) was used to scan the whole maize genome, and the NJ-tree model of Treebest was used to construct phylogenetic tree, principal component analysis(PCA) was conducted by GCTA(genome-wide complex trait analysis) to reveal the genetic diversity and population genetic structure. 【Result】In 107 Yunnan local inbred lines, 5533 uniformly distributed high-quality SNP marker sites were finally detected. Based on the analysis of these SNP marker sites, Neis gene diversity index(H) of 107 maize germplasm genes was 0.2981-0.5000 with average value being 0.4832, polymorphism information content(PIC) value was 0.2536-0.3750 with average value being 0.3662. The minimum allele frequency value was 0.5000-0.8178 with average value being 0.5744. The analysis of population genetic structure showed that when K=6, the maximum value of △K was the maximum, which meaned that the inbred lines used in this study could be divided into six groups. They were Tangsi Pingtou blood relationship group, PB blood relationship group, 335 female blood relationship group, Zi 330 and the Lüda Honggu blood relationship group, unknown group 1 and unknown group 2. No inbred lines were divided into other heterosis groups. Among them, 37 inbred lines from 2 unknown groups could not be classified into the same group as 10 known heterosis groups in China. The results of principal component analysis showed that 107 maize inbred lines generalized in Yunnan could be clearly distinguished from the backbone maize inbred lines commonly used in China. Most of the maize inbred lines in Yunnan were concentrated near the reference backbone inbred lines. But some Yunnan inbred lines were far away from the reference inbred lines commonly used in China. 【Conclusion】The genetic diversity of maize germplasm resources in Yunnan is abundant, including multiple heterosis groups, and the genetic basis of breeding parents is abundant, too. It can be clearly distinguished from the backbone inbred lines commonly used in China, and some of them have a long genetic distance from the backbone inbred lines. The resources which have good application potential can be used to create new heterotic groups.
Key words: maize; SNP chips; group genetic structure; genetic diversity; principal component analysis
Foundation item: Yunnan Youth Project of Applied Basic Research(2019FD061)
0 引言
【研究意义】玉米是我国第一大粮食作物,栽培历史悠久,其种质资源不断推陈出新,目前我国审定品种数量已达6000余个(杨杨等,2014;王凤格等,2017),主要种植于在东北、华北和西南地区。云南地处我国西南部,独特的气候生态多样性造就了特色且多样化的玉米种质资源(何永坤等,2016)。对云南玉米遗传种质进行遗传结构类群、进化格局及群体遗传结构分析,梳理云南玉米自交系中的骨干自交系和杂交优势群体,明确玉米品种亲缘关系(吕学高等,2014),对选育优良品种及规范云南省玉米品种市场管理具有重要意义。【前人研究进展】SNP是在RAPD、RFLP、AFLP、SSR等分子标记技术基础上发展起来的第三代最具优势的分子标记技术(宋伟等,2016;赵久然等,2018;董艳辉等,2019),已广泛应用于作物的遗传多样性分析、QTL定位、指纹图谱构建、群体结构分析、种质资源分析及分子标记辅助选择等研究领域(Elisabetta et al.,2013;路明等,2016)。陈广凤等(2015)以205份我国冬麦区小麦品种(系)为材料,利用分布于小麦全基因组的24355个SNP分子标记对株高相关性状进行关联分析,结果发现38个与株高相关性状极显著关联的SNP分子标记。路明等(2016)基于SNP分子标记对玉米品种吉单50的亲本及10个骨干自交系进行遗传关系分析。蔡露等(2018)利用SNP分子标记对92份烟草种质资源进行遗传多样性和遗传结构分析。黄飘飘等(2018)研究了SNP如何通过影响miRNA和靶基因对的互补模式从而控制miRNA相关的性状,且基于3000份水稻基因组数据对编码miRNA的基因进行全基因组SNP分析。赵久然等(2018)以344个具有广泛代表性和时效性的玉米自交系为材料,利用SNP芯片对其进行遗传多样性与群体遗传结构分析。崔德周等(2019)基于90K芯片SNP分子标记构建小麦遗传图谱,并对抗纹枯病性状进行QTL定位。么大轩等(2019)利用SNP和SSR分子标记分析23个甜玉米自交系的遗传多样性。【本研究切入点】至今,有关利用SNP分子标记对云南地区玉米自交系进行群体遗传结构分析的研究鲜见报道。【拟解决的关键问题】以云南当地推广的107个优良玉米自交系为材料,在Axiom? Maize56K SNP Array平台上采用玉米SNP芯片(56K)进行全基因组扫描,采用SNP分子标记对其群体遗传结构和遗传多样性进行分析,以期了解云南地方自交系的血缘关系、进化类群、杂交优势群体及群体遗传结构,为云南省玉米种质创新、遗传改良、品种管理等提供理论依据,也为今后深入挖掘优良性状相关基因打下基础。
1 材料与方法
1. 1 试验材料
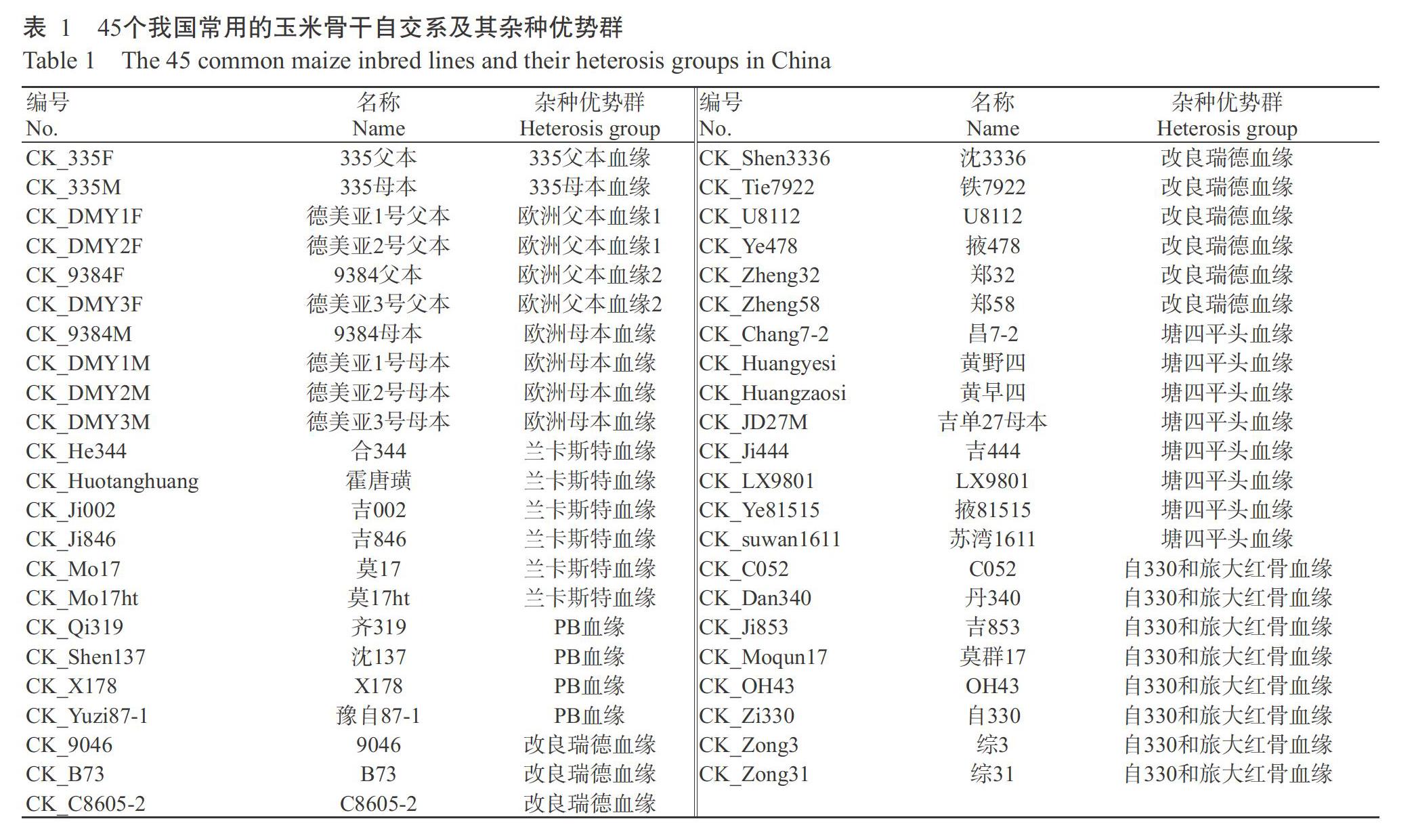
供试材料为107个云南当地玉米自交系(编号ZBS001~ZBS107),以45个我国常用玉米骨干自交系(表1)作为杂种优势群划分的参照(CK),含我国目前已知的10个杂种优势群,以上材料均由云南大天种业有限公司提供。玉米SNP芯片(56K)购自Affymetrix芯片公司,采用独特光蚀刻原位合成技术合成,可检测56000个SNPs位点,标记检出率在97%以上,芯片为384制式芯片。
1. 2 SNP分子标记分析
春播种植玉米自交系,种植密度为60000株/ha,于苗期3叶1心时采集新叶,每材料取5片幼嫩叶片于液氮下混合研磨,采用CTAB法提取其DNA,以紫外分光光度计检测DNA浓度,将DNA纯化处理后备用。在Axiom? Maize56K SNP Array平台上采用玉米SNP芯片(56K)进行全基因组扫描。样品准备工作在Backman自动工作站上完成,杂交、洗涤、扫描等在GeneTitan上完成。
1. 3 統计分析
使用Treebest的NJ-tree模型构建系统发育进化树,利用GCTA(全基因组复杂性状分析)工具进行主成分分析。基于检出的5533个均匀分布的高质量SNP分子标记,利用Structure 2.2进行供试材料的群体遗传结构分析。参照Evanno等(2005)的K变化下△K的折线图模型,K预设为1~10,估计最佳群体组群数K,每个K值均独立运算10次(管俊娇等,2018)。利用PowerMarker v3.25计算多态信息含量(PIC)、最小等位基因频率和Neis基因多样性指数(H)等(Liu and Muse,2005)。
2 结果与分析
2. 1 遗传多样性和群体遗传结构分析结果
群体遗传结构是指遗传变异在物种或群体中的一种非随机分布。可按照地理分布或其他标准将一个群体分为若干亚群,处于同一亚群内的不同个体亲缘关系较高,而亚群与亚群间的亲缘关系稍远。群体结构分析有助于理解进化过程,且可通过基因型和表型的关联性确定个体所属的亚群。107个云南玉米自交系群体遗传结构分析结果如图1所示。以浅蓝色为主有33个供试自交系,为塘四平头血缘;以蓝色为主有13个供试自交系,为PB血缘;以浅绿色为主有22个供试自交系,为335母本血缘;以绿色为主有26个供试自交系,以粉色为主有11个供试自交系,因绿色和粉色分类中无参照自交系,故划分为未知类群;最后以红色为主有2个供试自交系,为自330和旅大红骨血缘。
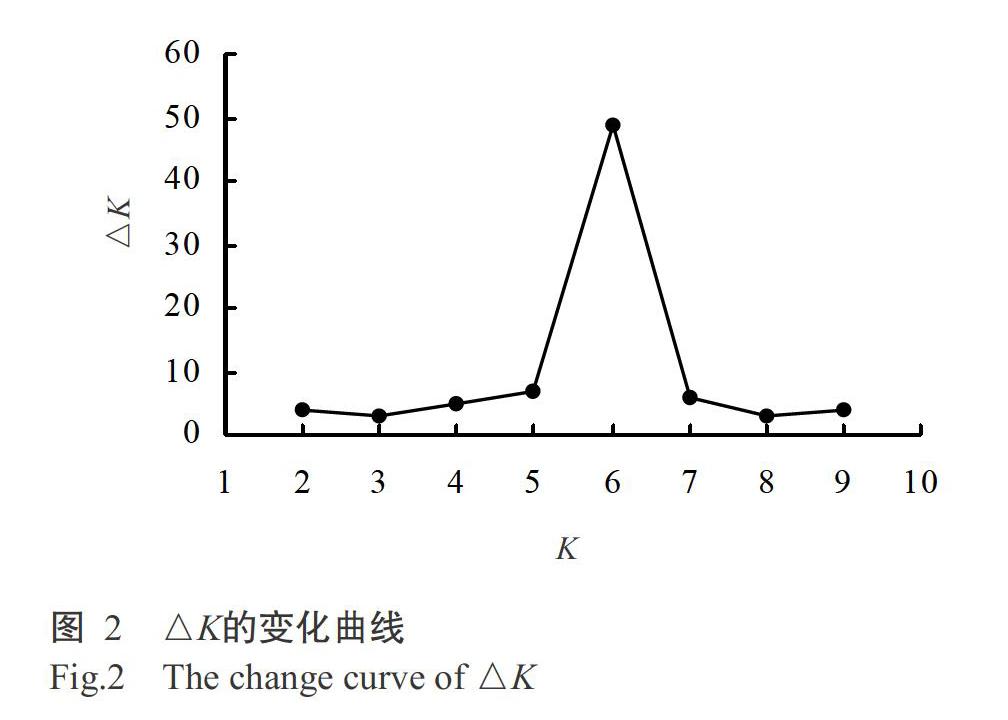
利用Structure 2.2对107个云南玉米自交系的群体遗传结构进行系统解析,结果(图2)显示,当类群数(K)为1~10时,ΔK在K=6时达最大值,表明107个供试自交系和45个参照自交系可划分为六大类群。结合图1可知,其可分为塘四平头血缘类群、PB血缘类群、335母本血缘类群、自330和旅大红骨血缘类群及两个未知类群,无自交系划分到335父本血缘、欧洲父本血缘1、欧洲父本血缘2、欧洲母本血缘、兰卡斯特血缘和改良瑞德血缘类群等,其中2个未知类群共有37个供试自交系未能与我国目前已知的杂种优势群划分为一类。
从107个云南玉米自交系中检出5533个均匀分布的高质量SNP分子标记位点,基于这些SNP分子标记位点分析结果可知,107个供试自交系的H为0.2981~0.5000,平均为0.4832;PIC为0.2536~0.3750,平均为0.3662;最小等位基因频率为0.5000~0.8178,平均为0.5744(表2),表明供试自交系整体遗传多样性较丰富。六大类群的H和PIC均较高,但不同类群存在明显差异,其中,塘四平头血缘类群、PB血缘类群、335母本血缘类群和未知类群1的PIC相对较高,分别为0.3432、0.2983、0.2905和0.3258,而未知类群2和自330和旅大红骨血缘的PIC相对较低,分别为0.2414和0.1938。
2. 2 系统发育进化分析结果
以45个我国常用玉米骨干自交系作为杂种优势群划分的参照,各优势群均能很好的区分,如图3所示。所有材料明显分为三大群,其中,塘四平头血缘为Ⅰ类群,该类群不包含供试自交系;兰卡斯特血缘和欧洲父本血缘为Ⅱ类群,该类群也不包含供试自交系;其余材料归为Ⅲ类群,该类群包含107个供试自交系,Ⅲ类群又分成两个亚群(Ⅲ-a和Ⅲ-b)。Ⅲ-a亚群中335父本血缘、335母本血缘、欧洲母本血缘为一小类群,PB血缘为一小类群,自330和旅大红骨血缘与改良瑞德血缘为一小类群;Ⅲ-b亚群中72个供试自交系与苏湾1611聚到一类,推测这72个自交系为热带种质材料。Ⅲ类群中,有31个云南当地玉米自交系的血缘构成不在我国目前已知的10个杂种优势群中,占比28.97%,与群体遗传结构分析结果存在差异,推测这31个自交系为新的杂种优势群。
结合上述群体遗传结构分析可知,107个云南玉米自交系中,以PB血缘为主构成的材料数量最多,为32个,占29.91%;以335父本、改良瑞德血缘及自330和旅大红骨血缘为主构成的材料数量最少,各为3个,各占2.80%。
2. 3 主成分分析结果
基于个体基因型SNP,使用主成分分析方法将不同基因型品种聚类成不同亚群,以明确107个云南玉米自交系的遗传结构。主成分分析图的前三个维度能解释所有材料变异情况,也是遗传变异最多的3个维度(Yang et al.,2011)。本研究分别将维度1v2、1v3和2v3提取出来制成二维图,结果发现PCA1v2最能代表所有材料的遗传变异,PCA1v3和PCA2v3次之。由图4可知,107个云南玉米自交系与45个我国常用玉米骨干自交系能明显区分,大部分云南玉米自交系集中在我国常用玉米骨干自交系附近,但有少数云南玉米自交系与我国常用玉米骨干自交系距离较远,其中,与欧洲母本血缘、欧洲父本血缘1和欧洲父本血缘2等3个杂种优势群距离相对较近的自交系极少,还有部分自交系与10个杂种优势群距离均较远,未与我国常用玉米骨干自交系归为一类,与群体遗传结构和系统发育进化树分析结果相似。
3 讨论
本研究选用45个我国常用玉米骨干自交系作为杂种优势群划分的参照自交系(CK)分析107个云南当地玉米自交系的群体遗传结构,这些参照自交系是我国当前或历史上玉米生产中应用的骨干亲本资源,涵盖了10个杂种优势类群。通过构建系统发育进化树,按群体遗传特征的共同点或差异可推断出个体间的亲缘关系(梁凤萍等,2018),分支分离的越早代表亲缘关系越远(Vilella et al.,2009)。同时,结合主成分分析法可应用到植物表型、资源评价、遗传分析等研究及其他方法进行相互验证(张鹏等,2018;赵勇等,2019)。本研究采用主成分分析与群体遗传结构分析相结合,将107个云南当地玉米自交系和45个参照骨干自交系分为六大类群,分别为塘四平头血缘类群、PB血缘类群、335母本血缘类群、自330和旅大红骨血缘类群及2个未知类群,无自交系划分到兰卡斯特血缘和改良瑞德血缘类群等,其中,2个未知类群共含37个云南玉米自交系,未能与我国目前已知的杂种优势群归在一类,而根据系统发育进化树可知,亚群III-b中共72个品种与参照自交系苏湾1611相近,划分为一类群,推测该未知类群品种含有热带血缘,为热带种质材料,但具体原因还应结合血缘同源(Identity by descent,IBD)遗传背景比例进行综合分析。这与前人研究结果(王懿波等,1998;赵久然等,1999,2018)相近,均发现传统的杂种优势群如塘四平头血缘、PB血缘与335母本血缘能明显区分,但也存在差异,其原因可能是供试材料数量或来源地不同。
玉米品种的遗传改良与优良种质的发掘或引进密切相关,其中,引入自交系和商业杂交种选系起重要作用,将外来优异的种质优良基因导入国内种质能有效增加遗传变异性,从而产生强杂种优势群(赵久然等,2018)。云南地区有关引入玉米外来种质资源最早的报道为1976年5月通过中国科学院遗传与发育生物学研究所引入玉米自交系SSE232(汪小香,1984),该自交系被作为亲本广泛应用于品种选育。随后,有许多学者通过外引品种以获得较强杂种优势,其中最多的是引入熱带和亚热带玉米自交系(李勇成等,2009;段智利等,2017),研究表明热带、亚热带玉米种质与经热带种质改良后的温带玉米种质间具有较强的杂种优势,且导入热带种质的温带系与热带和亚热带供体系的杂种优势仍存在,其所属的杂种优势群并未改变(陈洪梅等,2011)。由此可见,外来优质种质资源通过改良后可与地区品种资源形成优势互补(赵久然等,2018),外引材料在保持原有优势的基础上还具有较强的杂种优势,能在品种选育中发挥重要作用。目前,热带种质的引进及创新利用,拓宽我国玉米育种的遗传基础已成为我国玉米育种的一个重要方向。因此,今后应充分利用本研究发现的新杂交优势群,与我国现有优势群进行杂交组配,以提升我国玉米育种创新能力。
4 结论
云南地区玉米种质资源遗传多样性较丰富,含有多个杂种优势群,育种亲本遗传基础丰富,与我国常用玉米骨干自交系能明显区分,且部分与骨干自交系遗传距离较远,可创建新的杂交优势群,具有良好的应用潜力,亟需充分利用当地杂交优势群进行杂种组配,加快产生新的遗传类群。
参考文献:
蔡露,杨欢,王勇,李廷轩,陈光登. 2018. 利用GBS技术开发烟草SNP标记及遗传多样性分析[J]. 中国烟草科学,39(5):17-24. [Cai L,Yang H,Wang Y,Li T X,Chen G D. 2018. Analysis of genetic diversity of tobacco germplasm resources based on SNP markers via genotyping-by-sequencing technology[J]. Chinese Tobacco Science,39(5):17-24.]
陈广凤,陈建省,田纪春. 2015. 小麦株高相关性状与SNP标记全基因组关联分析[J]. 作物学报,41(10):1500-1509. [Chen G F,Chen J S,Tian J C. 2015. Genome-wide asso-ciation analysis between SNP markers and plant height related traits in wheat[J]. Acta Agronomica Sinica,41(10):1500-1509.]
陈洪梅,汪燕芬,姚文华,罗黎明,李佳莉,徐春霞,番兴明,郭华春. 2011. 导入热带种质的温带玉米自交系的利用潜力[J]. 作物学报,37(10):1785-1793. [Chen H M,Wang Y F,Yao W H,Luo L M,Li J L,Xu C X,Fan X M,Guo H C. 2011. Utilization potential of the temperate maize inbreds integrated with tropical germplasm[J]. Acta Agronomica Sinica,37(10):1785-1793.]
崔德周,李永波,樊庆琦,隋新霞,黄承彦,楚秀生. 2019. 基于90K芯片SNP标记的小麦遗传图谱构建及抗纹枯病QTL定位[J]. 山东农业科学,51(2):13-17. [Cui D Z,Li Y B,Fan Q Q,Sui X X,Huang C Y,Chu X S. 2019. Construction of wheat genetic linkage map based on 90K SNP array and mapping QTLs for sharp eyespot resistance[J]. Shandong Agricultural Sciences,51(2):13-17.]
董艳辉,刘龙龙,温鑫,于宇凤,杨方,刘根科,崔林,曹秋芬,秦永軍. 2019. 基于基因分型技术的燕麦SNP标记研究[J]. 华北农学报,34(1):97-106. [Dong Y H,Liu L L,Wen X,Yu Y F,Yang F,Liu G K,Cui L,Cao Q F,Qin Y J. 2019. Study on oat SNP markers based on genoty-ping technology[J]. Acta Agriculturae Boreali-Sinica,34(1):97-106.]
段智利,杨晓婷,陈洪梅,希从芳,汪燕芬,王晶,李建宾. 2017. 热带、亚热带玉米自交系与温带玉米自交系的配合力和遗传参数分析[J]. 西南农业学报,30(3):497-504. [Duan Z L,Yang X T,Chen H M,Xi C F,Wang Y F,Wang J,Li J B. 2017. Study on combining ability and genetic parameters between tropical,subtropical maize inbred lines and temperate inbred lines[J]. Southwest China Journal of Agricultural Sciences,30(3):497-504.]
管俊娇,杨晓洪,张建华,王江民,张鹏,李彦刚. 2018. 云南粳稻遗传多样性及群体结构分析[J]. 生物技术通报,34(1):90-96. [Guan J J,Yang X H,Zhang J H,Wang J M,Zhang P,Li Y G. 2018. Genetic diversity and population structure analysis of japonica rice varieties from Yunnan Province[J]. Biotechnology Bulletin,34(1):90-96.]
何永坤,唐余学,范莉,阳园燕. 2016. 近50年西南地区玉米干旱变化规律研究[J]. 西南师范大学学报(自然科学版),41(1):34-42. [He Y K,Tang Y X,Fan L,Yang Y Y. 2016. Study on the variation of drought for maize in southwestern China in the recent 50 years[J]. Journal of Southwest University(Natural Science Edition),41(1):34-42.]
黄飘飘,李俊彦,张大兵. 2018. 水稻中介导基因沉默相关microRNA的SNP分析[J]. 基因组学与应用生物学,37(3):1280-1292. [Huang P P,Li J Y,Zhang D B. 2018. SNP analysis of microRNAs related to gene silencing media-ted in rice[J]. Genomics and Applied Biology,37(3):1280-1292.]
李勇成,番兴明,胡云,乐自祥,谭静,陈洪梅,张云波,邵思全,王增明. 2009. 外来热带、亚热带玉米自交系与云南地方玉米自交系的配合力研究[J]. 玉米科学,17(3):45-50. [Li Y C,Fan X M,Hu Y,Le Z X,Tan J,Chen H M,Zhang Y B,Shao S Q,Wang Z M. 2009. Study on combining ability between exotic tropical,subtropical maize inbred lines and Yunnan local maize inbred lines[J]. Journal of Maize Sciences,17(3):45-50.]
梁鳳萍,文祥宁,高赫一,张颖. 2018. 菊科植物叶绿体基因组特征分析[J]. 基因组学与应用生物学,37(12):5437-5447. [Liang F P,Wen X N,Gao H Y,Zhang Y. 2018. Analysis of chloroplast genome characteristics of Compositae plants[J]. Genomics and Applied Biology,37(12):5437-5447.]
路明,张志军,郑淑波,岳尧海,刘宏伟,王敏,刘文国. 2016. 基于SNP标记分析玉米品种吉单50的遗传基础[J]. 玉米科学,24(6):1-7. [Lu M,Zhang Z J,Zheng S B,Yue Y H,Liu H W,Wang M,Liu W G. 2016. Genetic analysis on Jidan 50 based on SNP marker in maize[J]. Journal of Maize Sciences,24(6):1-7.]
吕学高,赵军华,楼肖成,朱正梅. 2014. 三十份特异玉米自交系的SSR聚类分析[J]. 浙江农业学报,26(2):274-278. [Lü X G,Zhao J H,Lou X C,Zhu Z M. 2014. Clus-tering analysis of 30 specific maize inbred lines by SSR markers[J]. Acta Agriculturae Zhejiangensis,26(2):274-278.]
宋伟,赵久然,王凤格,田红丽,葛健镕,王元东,赵衍鑫. 2016. SSR和SNP标记在玉米分子标记辅助背景选择中的应用比较[J] . 玉米科学,24(3):57-61. [Song W,Zhao J R,Wang F G,Tian H L,Ge J R,Wang Y D,Zhao Y X. 2016. Comparison of SSR and SNP markers in maize molecular marker assisted background selection[J]. Journal of Maize Sciences,24(3):57-61.]
汪小香. 1984. 墨西哥玉米自交系SSE_(232)在云南被广泛利用[J]. 作物品种资源,(2):12-18. [Wang X X. 1984. Mexican maize inbred line SSE_(232) is widely used in Yunnan Province[J]. Crop Variety Resources,(2):12-18.]
王凤格,杨扬,易红梅,赵久然,任洁,王璐,葛建镕,江彬,张宪晨,田红丽,侯振华. 2017. 中国玉米审定品种标准SSR指纹库的构建[J]. 中国农业科学,50(1):1-14. [Wang F G,Yang Y,Yi H M,Zhao J R,Ren J,Wang L,Ge J R,Jiang B,Zhang X C,Tian H L,Hou Z H. 2017. Construction of an SSR-based standard fingerprint database for corn variety authorized in China[J]. Scientia Agricultura Sinica,50(1):1-14.]
王懿波,王振华,王永普,张新,陆利行. 1998. 中国玉米主要种质杂种优势群的划分及其改良利用[J]. 华北农学报,(1):75-81. [Wang Y B,Wang Z H,Wang Y P,Zhang X,Lu L X. 1998. Division utilization and the improvement of main germplasm heterosis of maize in China[J]. Acta Agriculturae Boreali-Sinica,13(1):74-81.]
么大轩,张彬,刘松涛,Zenda Tinashe,段会军. 2019. 基于SNP和SSR对甜玉米种质遗传多样性的评价[J]. 江苏农业科学,47(7):45-50. [Yao D X,Zhang B,Liu S T,Tinashe Z,Duan H J. 2019. Evaluation of genetic diversity of sweet corn germplasms based on SNP and SSR[J]. Jiangsu Agricultural Sciences,47(7):45-50.]
杨扬,王凤格,赵久然,刘亚维. 2014. 中国玉米品种审定现状分析[J]. 中国农业科学,47(22):436-437. [Yang Y,Wang F G,Zhao J R,Liu Y W. 2014. Analysis of the current situation of accredited maize varieties in China[J]. Scientia Agricultura Sinica,47(22):436-437.]
张鹏,王江民,管俊娇,刘艳芳,杨晓洪,马芙荣,张建华. 2018. 蝴蝶兰品种数量性状与分组性状的DUS判定[J]. 西北农林科技大学学报(自然科学版),46(11):81-88. [Zhang P,Wang J M,Guan J J,Liu Y F,Yang X H,Ma F R,Zhang J H. 2018. DUS testing evaluation of quantitative and grouping characters of Phalaenopsis[J]. Journal of Northwest A & F University(Natural Science edition),46(11):81-88.]
趙久然,郭景伦,郭强,尉德铭,孔艳芳. 1999. 应用RAPD分子标记技术对我国骨干玉米自交系进行类群划分[J]. 华北农学报,14(1):32-37. [Zhao J R,Guo J L,Guo Q,Wei D M,Kong Y F. 1999. Heterotic grouping of 25 maize inbreds with RAPD markers[J]. Acta Agriculturae Boreali-Sinica,14(1):32-37.]
赵久然,李春辉,宋伟,王元东,张如养,王继东,王凤格,田红丽,王蕊. 2018. 基于SNP芯片揭示中国玉米育种种质的遗传多样性与群体遗传结构[J]. 中国农业科学,51(4):626-644. [Zhao J R,Li C H,Song W,Wang Y D,Zhang R Y,Wang J D,Wang F G,Tian H L,Wang R. 2018. Genetic diversity and population structure of important Chinese maize breeding germplasm revealed by SNP-Chips[J]. Scientia Agricultura Sinica,51(4):626-644.]
赵勇,赵培方,胡鑫,赵俊,昝逢刚,姚丽,赵丽萍,杨昆,覃伟,夏红明,刘家勇. 2019. 基于农艺性状分级对317份甘蔗种质资源的评价[J]. 中国农业科学,52(4):602-615. [Zhao Y,Zhao P F,Hu X,Zhao J,Zan F G,Yao L,Zhao L P,Yang K,Qin W,Xia H M,Liu J Y. 2019. Evaluation of 317 sugarcane germplasm based on agronomic traits rating data[J]. Scientia Agricultura Sinica,52(4):602-615.]
Elisabetta F,Tobias A S,Albrecht E. 2013. Melchinger genetic diversity analysis of elite European maize(Zea mays L.) inbred lines using AFLP,SSR,and SNP markers reveals ascertainment bias for a subset of SNPs[J]. Theoretical and Applied Genetics,126(1):133-141.
Evanno G,Regnaut S,Goudet J. 2005. Detecting the number of clusters of individuals using the software STRUCTURE:Asimulation study[J]. Molecular Ecology,14:2611-2620.
Liu K J,Muse S V. 2005. PowerMarker:An integrated analysis environment for genetic marker analysis[J]. Bioinformatics,21:2128-2129.
Vilella A J,Severin J,Ureta-vidal A,Heng L,Durbin R,Birney E. 2009. EnsemblCompara GeneTrees:Complete,duplication-aware phylogenetic trees in vertebrates[J]. Genome Research,19(2):327-335.
Yang J,Lee S H,Goddard M E,Visscher P M. 2011. GCTA:A tool for genome-wide complex trait analysis[J]. American Journal of Human Genetics,88(1):76-82.
(责任编辑 陈 燕)

