烯酰还原酶基因的替换对裂殖壶菌合成二十碳五烯酸的影响
杨瑞雄,郑鑫,陆涛,赵誉泽,杨庆华,卢英华,2,3,何宁,2,凌雪萍,2,3
(1 厦门大学化学化工学院,福建厦门361005; 2 福建省海洋生物资源开发利用协同创新中心,福建厦门361005;3 福建省化学生物学重点实验室(厦门大学),福建厦门361005)
引 言
ω-3型多不饱和脂肪酸对健康的重要性已在临床和流行病学研究中得到充分证实,其中备受关注的有二十碳五烯酸(EPA,C20:5)和二十二碳六烯酸(DHA,C22:6)[1]。作为脂质的重要组成部分[2],EPA可以调控生物膜的结构与功能,并通过降低血纤维蛋白水平和低密度脂蛋白水平来预防心血管疾病[3],例如脑血栓、动脉粥样硬化[4]。深海鱼油是目前多不饱和脂肪酸(polyunsaturated fatty acids,PUFAs)工业化生产的主要来源,但自然资源的匮乏以及较高的分离成本导致其供不应求[5]。因为鱼油中丰富的PUFAs 通过食物链积累而来,所以藻类、低级真菌以及海洋细菌等微生物才是PUFAs 的初始来源[6]。El Razak 等[7]从地中海、红海等地筛选了250 株海洋细菌,通过响应面等方法进行培养基优化后EPA 的最大产量达到45 mg/L。Zhang 等[8]通过优化希瓦氏菌的培养条件以及培养基成分等使得EPA 的 产 量 达 到30 mg/g。Chen 等[9]对 菱 形 藻(Nitzschia laevis)进行高密度培养,在最大细胞干重达 到22.1 g/L 的 基 础 上,EPA 产 量 达695 mg/L。Wang 等[10]研究的破囊壶菌基因工程菌株通过发酵罐补料分批发酵后EPA 产量达到2.7 g/L。由此可见,利用微生物发酵法生产EPA 将成为未来的主要发展趋势。
近年来,研究者们不断探索PUFAs 的合成机理,PKS 途径被认为是主要用来合成PUFAs 的途径[11],进而研究通过替换不同微生物中PKS 基因簇上的基因来增加PUFAs 的含量。Hayas hi 等[12]将Photobacterium profundum的PKS 基因簇上部分基因与Aureispira marina的PKS 基因簇上对应基因相互置换,发现ORFC和ORFB上的脱氢酶(dehydratase,DH)基因在花生四烯酸(arachidonic acid,ARA)的合成中起关键作用。Ren 等[13]通过在Schizochytriumsp. HX-308 中敲入Shewanella的酰基转移酶(Acyltransferase,AT)基因,EPA 占比提高3.7 倍。Matsuda 等[14]敲除破囊壶菌中的Δ12 脱饱和酶基因后,传统脂肪酸合成途径(fatty acid synthase,FAS)受阻,而PKS 途径合成的PUFAs 含量增加。以上研究表明基因工程调控脂肪酸合成相关途径对改善油脂的组成是可行的,可以进一步提高PUFAs 的产量。
裂殖壶菌具有生长繁殖快、耐受机械搅拌和可异养培养等特点,非常适宜发酵罐大规模培养。裂殖壶菌的油脂含量可达细胞干重的40%~70%,PUFAs占比达30%~50%,其中DHA占比30%~40%,但EPA 含量较低[15]。研究表明,裂殖壶菌的脂肪酸合成主要通过两个途径,分别是需氧的FAS 途径和厌氧的PKS 途径[16]。Wallis 等[17]从裂殖壶菌PKS 基因簇中分离了三个阅读表达框(ORF A-C),其中包含8个功能不同的基因(图1)。其中ER基因所表达的烯酰还原酶主要催化不饱和C====C 键还原成饱和的C—C 键,使底物烯酰-ACP 还原成酰基-ACP,是从头合成脂肪酸的最后一步[18]。Heath 等[19]首次提出ER主要作用于大肠杆菌中的反式-烯酰-ACP,将其还原为酰基-ACP。ER基因在PKS 基因簇中包含两个部分,分别位于ORFB上(ORFB-ER)和ORFC上(ORFC-ER)(图1)[19]。Ling 等[20]利用基因工程手段对两个ER基因的功能进行了验证,结果表明ORFB-ER基 因 在Schizochytrium limacinumSR21 的PUFAs 合成中起着关键作用,而ORFC-ER基因则更多地和饱和脂肪酸(SFAs)的合成及细胞生长相关;此外,研究还发现ORFC-ER基因转录水平的降低有利于Schizochytrium limacinumSR21 细胞内油脂的积累。Lee 等[21]将希瓦氏菌属的Shewanellasp.SCRC2738 菌株中完整的PKS 基因簇重组表达在大肠杆菌中,在提取的总脂肪酸中成功检测到了EPA。Metz 等[22]提出希瓦氏菌PKS 基因簇中的ER基因与肺炎链球菌FAS 合成途径的FabI基因具有高度同源性。因此,ER基因可能是PKS 途径和FAS途径的重要中枢基因,在PUFAs 的合成中发挥重要作用。
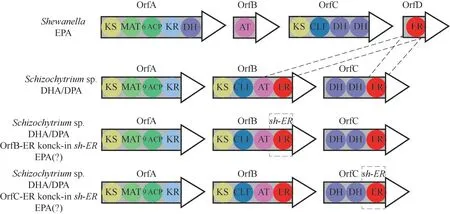
图1 希瓦氏菌、裂殖壶菌SR21、B-sh-ER菌株和C-sh-ER菌株中的PKS基因簇Fig.1 PKS gene clusters of Shewanella sp.SCRC2738,Schizochytrium limacinum SR21,B-sh-ER strain and C-sh-ER strain
希瓦氏菌Shewanella在低温、高压条件下生长时,EPA 作为细胞内唯一的PUFA,含量可达20%左右。因此,希瓦氏菌中的PKS 基因簇(图1)被认为主要催化合成EPA[6]。ER基因存在于希瓦氏菌PKS基因簇的ORFD结构域上,表达的烯酰还原酶对反式-2-烯基-ACP 中间体的双键进行NADPH 依赖的还原,该反应是合成EPA 的关键步骤[1]。目前已有研究学者将希瓦氏菌属的Shewanellasp. SCRC 2738、MR-1、MAC1 等菌株中完整的EPA 基因簇导入至大肠杆菌中表达,成功地通过PKS 途径合成了EPA[23]。其中,Lee 等[24]在大肠杆菌中异源表达希瓦氏菌(Shewanella oneidensisMR-1)EPA 合成基因簇,EPA 产量能达到总油脂的0.689%。美国杜邦公司在解脂酵母(Yarrowia lipolytica)中完整构建了FAS途径的十八碳二烯酸至二十碳五烯酸合成通路,使PUFAs占总油脂的70%以上[25]。通过在EPA生物合成途径中过表达不同的去饱和酶和延长酶,Yarrowia lipolytica的EPA 在总油脂中占比超过25%[26]。 在 聚 球 藻Synechococcus中 异 源 表 达Shewanella的PUFAs 合成基因簇,EPA 的产量也明显提高[27]。对于PUFAs的合成来说,大肠杆菌、酵母以及微藻等微生物菌种相比于裂殖壶菌不具备天然的优势,而且单一菌株插入多个外源基因在稳定性上也不尽人意。因此,本研究将SchizochytriumlimacinumSR21 的高油脂含量与Shewanellasp.SCRC2738 较强的PKS 基因簇相结合,利用同源重组技术将Shewanellasp. SCRC2738 的ER基因分别敲入Schizochytrium limacinumSR21 的ORFB-ER基因和ORFC-ER基因中,使Shewanellasp.SCRC2738的ER基因代替原宿主的ER基因发挥作用,对重组菌株脂肪酸成分以及相关基因的转录水平进行分析,探究sh-ER基因在裂殖壶菌PUFAs 合成中的功能。
1 实验材料和方法
1.1 工具酶与试剂
大肠杆菌Trans110感受态细胞、EasyScript First-Strand cDNA Synthesis SuperMix 试剂盒和TransStart Top Green QPCR 试剂盒购买于北京全式金生物有限公司。质粒小提试剂盒、普通DNA 产物纯化试剂盒、琼脂糖凝胶DNA 回收试剂盒、SuperMix MiniBEST 通用RNA 提取试剂盒和限制性内切酶购自宝日医生物技术有限公司。十七烷酸甲酯、博来霉素和脂肪酸混合标准品购自Sigma 公司。正己烷和三氟化硼乙醚购自阿拉丁试剂有限公司。
1.2 菌种和培养基
本研究从美国典型培养物保藏中心(ATCC,USA)获 取Schizochytrium limacinumSR21 菌 株。pTEF1/Zeo 载体和pMD19T-easey 载体购自英骏公司(Invitrogen,USA)。
LB 培养基(氨苄)用于大肠杆菌培养及转化子筛选。裂殖壶菌种子培养基用于SchizochytriumlimacinumSR21 常规培养[20]。含博来霉素种子培养基用于裂殖壶菌转化子筛选[28]。发酵培养基用于Schizochytrium limacinumSR21发酵生产脂肪酸[29]。
1.3 发酵培养
种子液制备:取裂殖壶菌种子在固体种子培养基上划线培养,在28℃条件下培养36 h。在已培养好的固体种子培养基上挑选形态饱满的单菌体,置于50 ml 种子培养基中,在28℃,250 r/min 条件下培养24 h。
摇瓶发酵:从二级种子培养基中,按照6%的接种量,转移一定量种子培养液至50 ml 发酵培养基中,在28℃,250 r/min条件下培养120 h。
发酵罐扩大培养:本实验采用补料分批发酵的方法,采用5 L 发酵罐(Winpact,USA)进行扩大培养,初始葡萄糖浓度为60 g/L,转速为300 r/min,通气量为4 vvm,培养温度保持为28℃,pH 维持7.5。初始溶氧设定为100%,根据发酵过程中溶氧情况逐级提高,最大至700 r/min。
1.4 菌株构建
如图2 所示构建替换Schizochytrium limacinumSR21 中ORFB-ER和ORFC-ER基因的敲入载体。将合成的希瓦氏菌ER基因(sh-ER)表达框(TEF1启动子、sh-ER基因、CYC1 终止子)克隆到pMD19T Easy Vector 中进行扩增,并命名为pMD-sh-ER。使用BamHI 和PstI 限 制 酶 对 pMD-sh-ER和pBlueZEO-B 载体双酶切后使用T4 连接酶在16 ℃过夜连接,产生重组敲入载体pBlueZEO-B-sh-ER。使用相同的方法构建pBlueZEO-C-sh-ER载体。
根据Ling 等[20]报道的方法,通过电转化将构建的两个载体线性化后转化并重组到裂殖壶菌基因组上。制备好的裂殖壶菌感受态细胞经过电穿孔后,在含有1 mol/L 山梨糖醇的种子培养基中于28℃培养2~3 h。将回收的细胞在含有30 mg/L博来霉素的固体培养基中培养3~5 d。最后,将在含博来霉素平板上生长出的阳性敲入菌株在28℃,250 r/min 下培养,并进行PCR验证和发酵实验。
1.5 生物量和总油脂测定
生物量通过重量法测定法[28]。将1 ml发酵液转移至预先称重的离心管中,然后以10000g离心2 min。沉淀用去离子水洗涤两次,在-50℃冻干至恒重约24 h,最后称量干细胞重量。
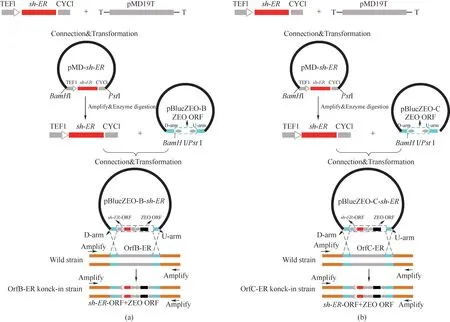
图2 B-sh-ER载体构建(a);C-sh-ER载体构建(b)Fig.2 Construction of B-sh-ER vector(a);Construction of C-sh-ER vector(b)
脂质提取方法为正己烷提取法[28]。将3 ml发酵液转移到离心管中,加入4 ml 12 mol/L 浓盐酸混合,然后将混合溶液在65℃水浴中反应50 min。最后将反应后的混合物用4 ml 正己烷萃取数次直至上清无色,收集上清后氮气吹干至恒重,65℃烘箱干燥2 h后称量总油脂重量。
1.6 脂肪酸组分分析
根据文献[30]报道的方法制备脂肪酸甲酯并进行了一些修改。首先,5 ml 0.5 mol/L 氢氧化钾-甲醇溶液溶解提取的总油脂,65℃的水浴中皂化10 min后加入三氟化硼乙醚甲酯化。冷却至室温后,以十七烷酸甲酯作为内标,将脂肪酸甲酯样品于配备有100 m × 0.25 mm 毛细管柱(SP-2560,USA)的气相色谱仪(Agilent GC 7890,USA)中进行分析。
1.7 葡萄糖浓度测定
将1 ml 发酵液10000g离心2 min,收集上清液以测量葡萄糖浓度。葡萄糖浓度通过3,5-二硝基水杨酸(DNS)方法测定[31]。
1.8 实时定量PCR分析
使用MiniBEST 通用RNA 提取试剂盒提取菌体中总RNA 后,通过Easy-Script First-Strand cDNA Synthesis SuperMix 试剂盒以总RNA 为模板反转录制备cDNA。将获得的cDNA 稀释至100 ng/µl,使用Trans-Start Top Green QPCR SuperMix 配置RT-PCR反应体系并通过Step-One 实时PCR 系统(Applied Biosystems,China)检测基因的转录水平。无cDNA模板的反应用作阴性对照,肌动蛋白(ACT)基因作为参照对mRNA 的表达水平进行标准化,并使用2-ΔΔCT法计算相对基因表达水平[32]。表A1 列出用于RT-qPCR的引物。
1.9 统计学分析
通过t值检验显示重组敲入菌株和野生型菌株之间的统计学显著性差异。P< 0.05(a)表示显著性差异;P< 0.01(b)表示极显著性差异。每个实验均设有三组生物学重复,两组技术重复以保证数据的准确性,数值均表示为平均值±标准偏差的形式。
2 实验结果与讨论
2.1 S.limacinum SR21 sh-ER基因敲入菌株构建
重组敲入质粒如图2 所示,包含sh-ER基因的上下游同源臂,博来霉素表达框(TEF1启动子,博来霉素基因和CYC1终止子)和sh-ER表达框(TEF1启动子,sh-ER基因和CYC1 终止子)。利用博来霉素基因作为筛选标记,将敲入质粒线性化后分别电转进入Schizochytrium limacinumSR21 中。如图3 所示,在含有30 mg/L 博来霉素的平板上分别挑取Bsh-ER敲入(sh-ER基因敲入ORFB-ER)和C-sh-ER敲入(sh-ER基因敲入ORFC-ER)的阳性重组菌株。在两种阳性重组菌株的基因组中均成功检测到与部分博来霉素抗性表达框相对应的649 bp 片段,而在野生型菌株中未检测到(图4)。这表明sh-ER基因已成功敲入Schizochytrium limacinumSR21 的ORFB-ER和ORFC-ER基因中。
2.2 重组菌株的生物量和总油脂含量分析
如图5(a)所示,对于B-sh-ER敲入菌株,细胞前期生长缓慢,72 h后生长加快。在发酵144 h前生物量均低于野生型菌株,而发酵后期(144 h之后)细胞生物量与野生型菌株接近。分析认为sh-ER基因片段与ORFB-ER基因片段大小接近,但由于异源基因在细胞内稳定表达需要一定的适应期[33],所以Bsh-ER敲入菌株的细胞生长周期较野生型菌株明显延长。如图5(b)、(c)所示,B-sh-ER敲入菌株总油脂含量在发酵中前期低于野生型菌株,而在发酵168 h时,总油脂基本与野生型菌株持平,而重组菌株的油脂含量高于野生型菌株9.38%(P< 0.05)。上述结果表明,sh-ER基因的敲入虽然降低了菌体生长速率,但最终补偿了ORFB-ER基因的功能,使得Bsh-ER敲入菌株的生物量与总油脂恢复至野生型菌株的水平,同时提高了菌体合成油脂的能力。

图3 含博来霉素抗性平板上生长的野生型菌株(a),B-sh-ER 菌株(b)和C-sh-ER菌株(c)Fig.3 Wild strains(a),B-sh-ER strains(b)and C-sh-ER strains(c)grown on media containing zeocin
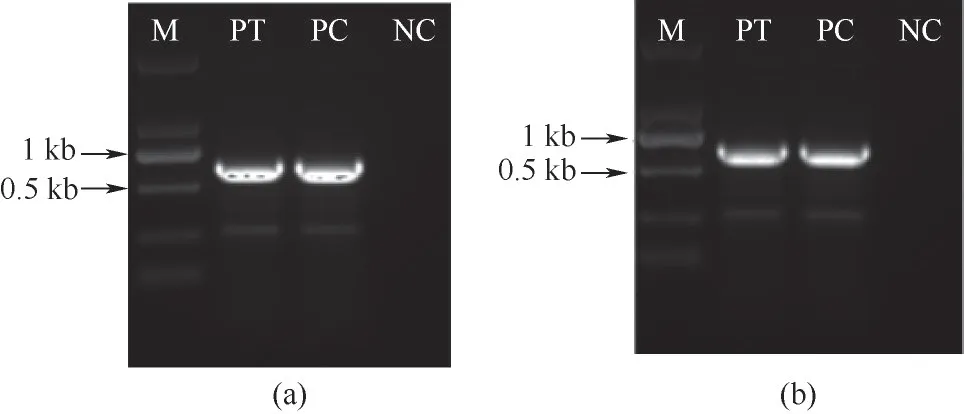
图4 B-sh-ER菌株中博来霉素表达框的基因组PCR(a);C-sh-ER菌株中博来霉素表达框的基因组PCR(b)Fig.4 Genomic PCR of zeocin expression cassette in the B-sh-ER strains(a);Genomic PCR of zeocin expression cassette in the C-sh-ER strains(b)
如图5(d)所示,C-sh-ER敲入菌株生物量相对于野生型菌株降低18.82%(P<0.05),与Ling等[20]构建的ORFC-ER敲除菌株的细胞生长相似,表明敲入sh-ER基因替换原有的ORFC-ER并没有补偿ORFC-ER基因对菌体生长的影响,导致敲入菌株生物量没有恢复到正常水平。如图5(e)所示,C-sh-ER敲入菌株总油脂产量较野生型菌株降低34.51%,但由于C-sh-ER敲入菌株生物量的降低,导致单位细胞油脂产量[图5(f)]与野生型菌株一致。而Ling等[20]报道的ORFC-ER敲除菌株总油脂产量在发酵后期与野生型菌株基本持平,并且单位细胞油脂含量较野生型菌株高19.70%。这说明sh-ER基因在该位置敲入不能发挥ORFC-ER基因对菌体生长的作用,同时对油脂合成产生了一定的抑制。Ren等[34]发现在Schizochytriumsp.中异源表达ω-3 去饱和酶和过表达乙酰辅酶A 合成酶后,生物量都明显降低,这可能与引入外源基因后细胞负担增加有关。本研究结果与上述结果类似,sh-ER基因片段较ORFC-ER基因片段长一些,可能增大了菌株的蛋白表达负荷[35]。除此之外,ORFC-ER基因更多地与细胞生长和饱和脂肪酸(saturated fatty acids,SFAs)合成相关,而sh-ER基因则更多地与PUFAs尤其是EPA 的合成相关[20],因此导致sh-ER基因的敲入不能补偿ORFC-ER基因的功能。
2.3 重组菌株脂肪酸成分分析
如表1 所示,B-sh-ER敲入菌株的单不饱和脂肪酸(monounsaturated fatty acid, MUFAs)如C14∶1含量相比于野生型菌株增加了95.02%(P< 0.05),文献报道MUFAs 的碳碳双键能够提高细胞膜的流动性[36],因此MUFAs 的增加是细胞在敲入基因的压力下为了维持细胞膜的流动性造成的。对于PUFAs 来说,B-sh-ER敲入菌株的DHA 含量虽然由29.76%下降到27.63%,但是EPA 含量提高了85.71%(P<0.01),达到0.78%;EPA/DHA 的百分含量增加了77.02%(P<0.01)。这些结果表明,B-sh-ER敲入菌促进了EPA 的合成通路,提高了EPA 的合成能力。分析认为sh-ER主要倾向于合成EPA[37],而裂殖壶菌的ORFB-ER在细胞内主要用于合成PUFAs[38]。因此,接下来主要研究B-sh-ER敲入菌的相关代谢变化。
而对于C-sh-ER敲入菌株,SFAs 的占比增加,而包括DHA、DPA 和EPA 在内的PUFAs 的含量降低,EPA/DHA 的百分含量基本保持不变,甚至略有降低。文献报道,PUFAs 主要是通过PKS 途径合成的,而SFAs 主要是通过FAS 途径合成的[38],两者都需要以乙酰辅酶A 为底物,以ATP 为能量,以NADPH 为 还 原 力[39],这 表 明sh-ER敲 入 替 换ORFC-ER使 更 多 的ATP 和NADPH 流 向SFAs 合成,与Ling 等[20]对ORFC-ER基因结构域功能鉴定结果一致。
2.4 B-sh-ER敲入菌株相关基因转录水平分析
如 图6(a)、(b)所 示,B-sh-ER敲 入 菌 株 中ORFB-ER基因在发酵过程中相比于野生型菌株均下调表达,60 h 时表达水平降低78.73%(P<0.05),同时敲入的sh-ER基因均呈现上调表达趋势,60 h时上调表达接近30 倍(P< 0.01),这进一步验证了B-sh-ER重组敲入菌株构建的准确性,为后续实验奠定了基础。如图6(c)所示,重组菌中ORFC-ER基因的转录水平整体呈现上调趋势,Ling 等[20]通过敲除Schizochytrium limacinumSR21 基因组中ORFCER基因的方式来验证其功能发现该基因与SFAs 的合成以及菌体生长密切相关,因此B-sh-ER生物量的增加以及C16:0 和C18:0 等饱和脂肪酸含量提高与ORFC-ER基因上调表达相关。如图6(d)所示,重组菌中AT 基因转录水平在84 h 提高接近20 倍(P<0.01)。AT 是一类酰基转移酶,能够为PKS 途径合成PUFAs 提供底物[29],Ren 等[13]针对AT 基因的替换研究发现酰基转移酶对其他脂肪酸组分影响不显著,但EPA的含量却明显增加,这说明了sh-ER敲入对ORFB-ER的替换促进了AT 基因的上调表达,进一步促进了EPA的合成。
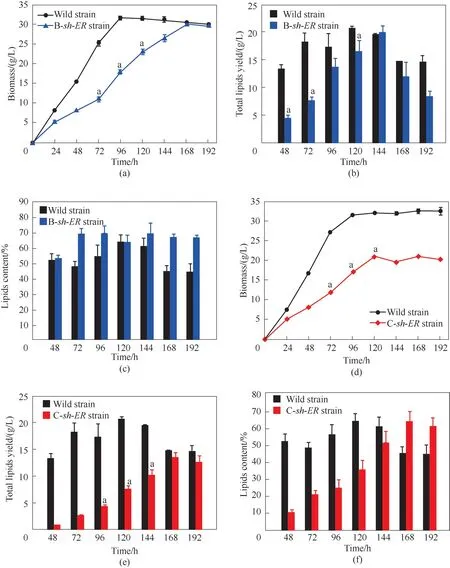
图5 野生型菌株和B-sh-ER菌株中生物量(a),总油脂量(b)和油脂含量(c);野生型菌株和C-sh-ER菌株生物量(d)、总油脂量(e)和油脂含量(f)Fig.5 Biomass(a),total lipids yield(b)and lipids content(c)in the wild strain and B-sh-ER strains;Biomass(d),total lipids yield(e)and lipids content(f)in the wild strain and C-sh-ER strains
2.5 B-sh-ER敲入菌株的补料分批发酵
通过以上生物量、总脂质和脂肪酸组成的分析,可以发现B-sh-ER敲入菌株在维持正常生物量的同时,使得总油脂和EPA 含量均增加。因此,将B-sh-ER敲入菌株在5 L 发酵罐中进行补料分批发酵。
如图7 所示,B-sh-ER敲入菌株显示在保持与野生型菌株生长速率一致的前提下细胞生长周期明显延长,最终生物量达到159.70 g/L,高于野生型菌株42.46%[图8(a)]。同时,在B-sh-ER敲入菌株中,葡萄糖显示出更快的消耗速率,总油脂和EPA产量分别增加了28.70%和71.6%,达到73.24 g/L 和732.40 mg/L[图8(b)、(c)]。B-sh-ER敲入菌株的EPA/DHA 的百分含量在整个补料分批发酵过程中均高于野生型菌株,并且在发酵终点提高了76.1%[图8(e)],而B-sh-ER敲入菌株的最终DHA 产量与野生型菌株相比变化不明显[图8(d)]。脂肪酸组成的进一步分析如表2 所示,B-sh-ER敲入菌株与野生型菌株相比C16:0 含量从49.54%增加到57.40%,同时,PUFAs、DHA 和DPA 含量分别降低了24.4%、24.4% 和29.3%。但是,B-sh-ER敲入菌株中的EPA 比例(占总脂质的1.0%)比野生型菌株高出33.3%。
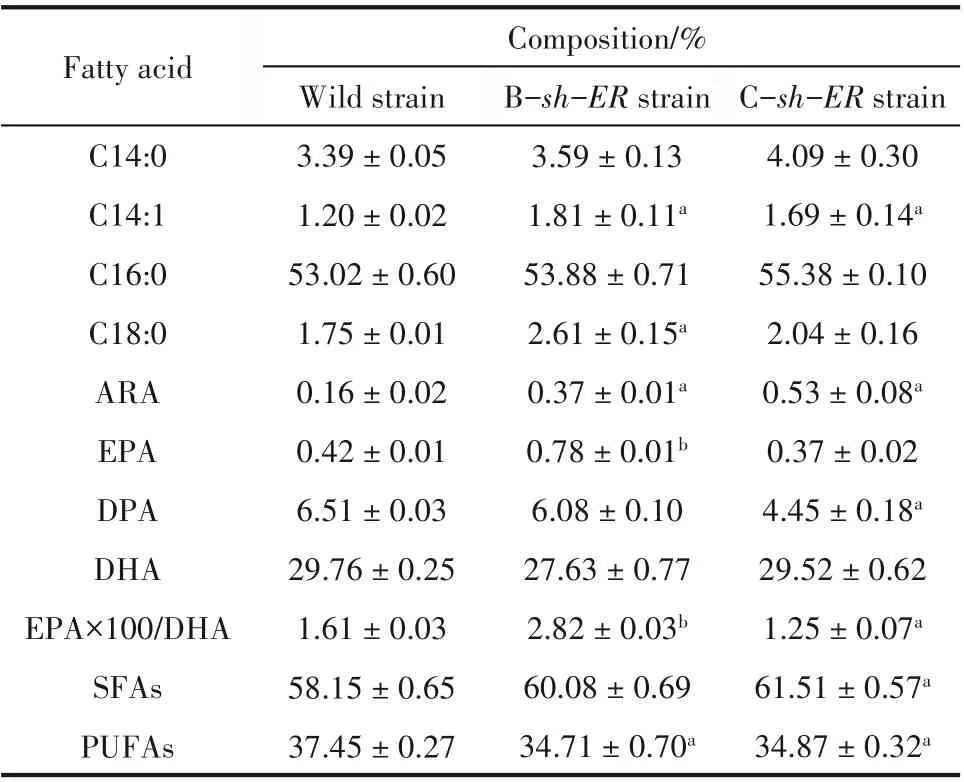
表1 野生型菌株、B-sh-ER菌株和C-sh-ER菌株脂肪酸组成Table 1 Fatty acid composition of wild strains,C-sh-ER strains and B-sh-ER strains
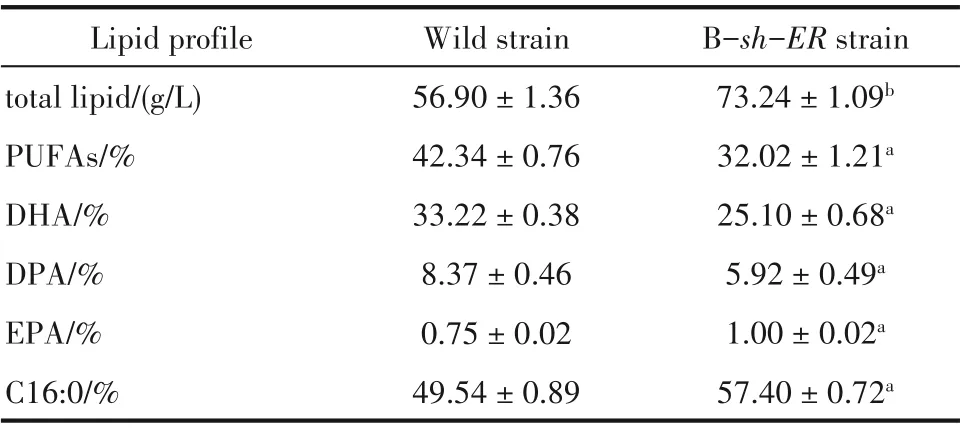
表2 补料分批发酵野生型菌株和B-sh-ER菌株的脂肪酸组成Table 2 Fatty acid composition of the wild strain and B-sh-ER strain in fed-batch fermentation
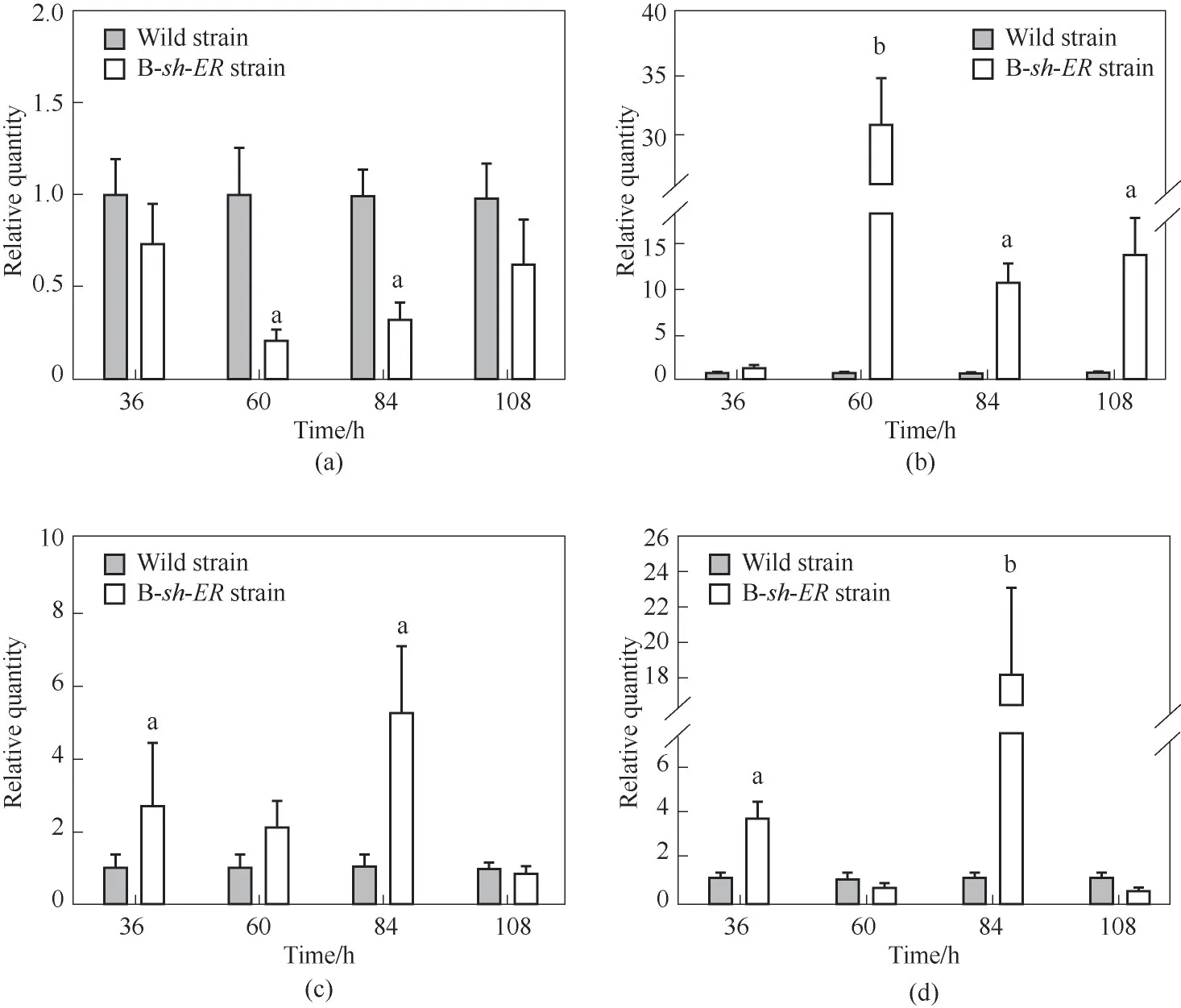
图6 B-sh-ER菌株相关基因转录水平分析(a)裂殖壶菌烯酰还原酶基因ORFB-ER;(b)希瓦氏菌烯酰还原酶基因sh-ER;(c)裂殖壶菌烯酰还原酶基因ORFC-ER;(d)酰基转移酶(AT)基因Fig.6 The transcription level of related gene in B-sh-ER strain(a)Schizochytrium limacinum SR21 enoyl-reductase gene ORFB-ER;(b)Shewanella sp.SCRC2738 enoyl-reductase gene sh-ER;(c)Schizochytrium limacinum SR21 enoyl-reductase gene ORFC-ER;(d)acyl transferase(AT)gene
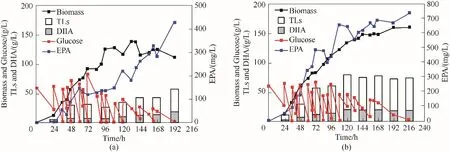
图7 野生型菌株(a)和B-sh-ER菌株(b)在5 L发酵罐中分批补料发酵生长曲线Fig.7 Growth curves of fed-batch fermentation in 5 L fermentator by wild strain(a)and B-sh-ER strain(b)
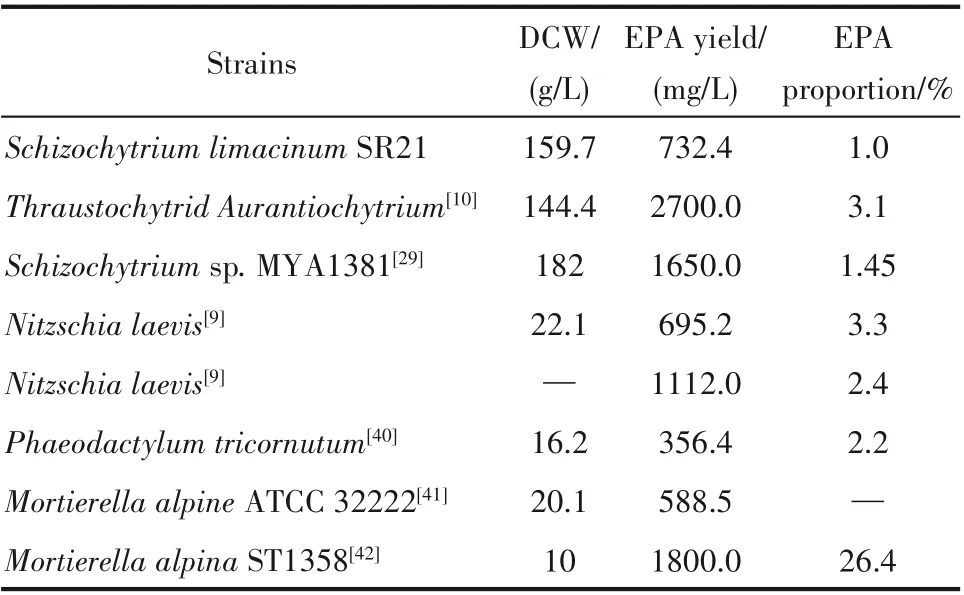
表3 相关菌株EPA产量及占比Table 3 EPA yield and proportion of related strains
表3中列出了目前用于EPA 生产菌株的产量以及总脂肪酸中EPA 的占比,主要有破囊壶菌属、微藻以及高山被孢霉。破囊壶菌属由于具有高生物量以及高油脂含量的特点,因此在PUFAs 合成方面潜力巨大。目前针对破囊壶菌属调控PUFAs 合成的主要方式以基因工程和代谢工程为主,但是在获得较高EPA 占比的条件下细胞的生长会受抑制。Cui 等[43]指出过表达六磷酸葡萄糖脱氢酶(G6PDH)导致破囊壶菌PUFAs占比增加10.60%,但是细胞生长和油脂合成受到严重影响。微藻的油脂中也含有丰富的PUFAs,光合作用菌株紫球藻(Porphyridium cruentum)中EPA 占总脂肪酸的41%[44];而根据栽培条件不同,三角褐指藻(Phaeodactylum tricornutum) 和 微 绿 球 藻(Nannochloropsissp.)中EPA 的积累占总脂肪酸的比例可分别高达57%[45]和68%[46]。产油微藻的培养方式以异养为主,因此影响微藻PUFAs 积累的主要因素是培养条件和培养基成分。高山被孢霉油脂中PUFAs 的占比较大,但由于生物量的限制,无法进行工业化生产。Shi 等[41]指出Δ6 去饱和酶的过表达使得Mortierella alpinaATCC 32222 在培养基中添加前体的情况下EPA 产量增加26.2 倍,达到588.5 mg/L。相比于以上所述的基因工程菌株,本研究构建的B-sh-ER敲入菌株在提高生物量同时,EPA 的占比也大幅度提升,对于EPA 的工业化生产有一定的借鉴意义。在后续的研究中,将结合菌株培养条件以及培养基成分的优化进一步提高EPA 的产量。
3 结 论
本研究根据前期对ER 基因功能的鉴定,选择了将Shewanellasp.SCRC2738PKS基因簇上的ER基因和具有高产PUFAs 潜力的Schizochytrium limacinumSR21 的ER基因相结合进行研究:在Schizochytrium limacinumSR21 的ORFC-ER基 因 和ORFB-ER基因的位置分别敲入sh-ER基因进行替换,进一步研究ER基因功能的同时,提高EPA 的产量。B-sh-ER敲入菌株相比于野生型菌株油脂产量提高9.38%(P< 0.05),其中EPA 占比增加最为显著,提高了85.71%(P<0.01),达到0.78%;而C-sh-ER敲入菌株单位细胞油脂含量可以恢复到野生型菌株的水平,但由于细胞生长受到严重抑制,因此菌株总油脂产量明显降低。B-sh-ER敲入菌株的发酵罐补料分批发酵结果表明B-sh-ER敲入菌株最大生物量高于野生型菌株42.46%,总油脂和EPA产量分别增加了28.70%和71.6%,达到73.24 g/L 和732.40 mg/L。根据相关基因转录水平的测定发现,在裂殖壶菌的ORFB-ER基因上敲入sh-ER基因有利于协同PKS 途径基因的表达,促进PUFAs 尤其是EPA的合成。B-sh-ER敲入菌株的构建为PUFAs合成的调控提供了新的思路,为EPA 的工业化生产奠定了基础。
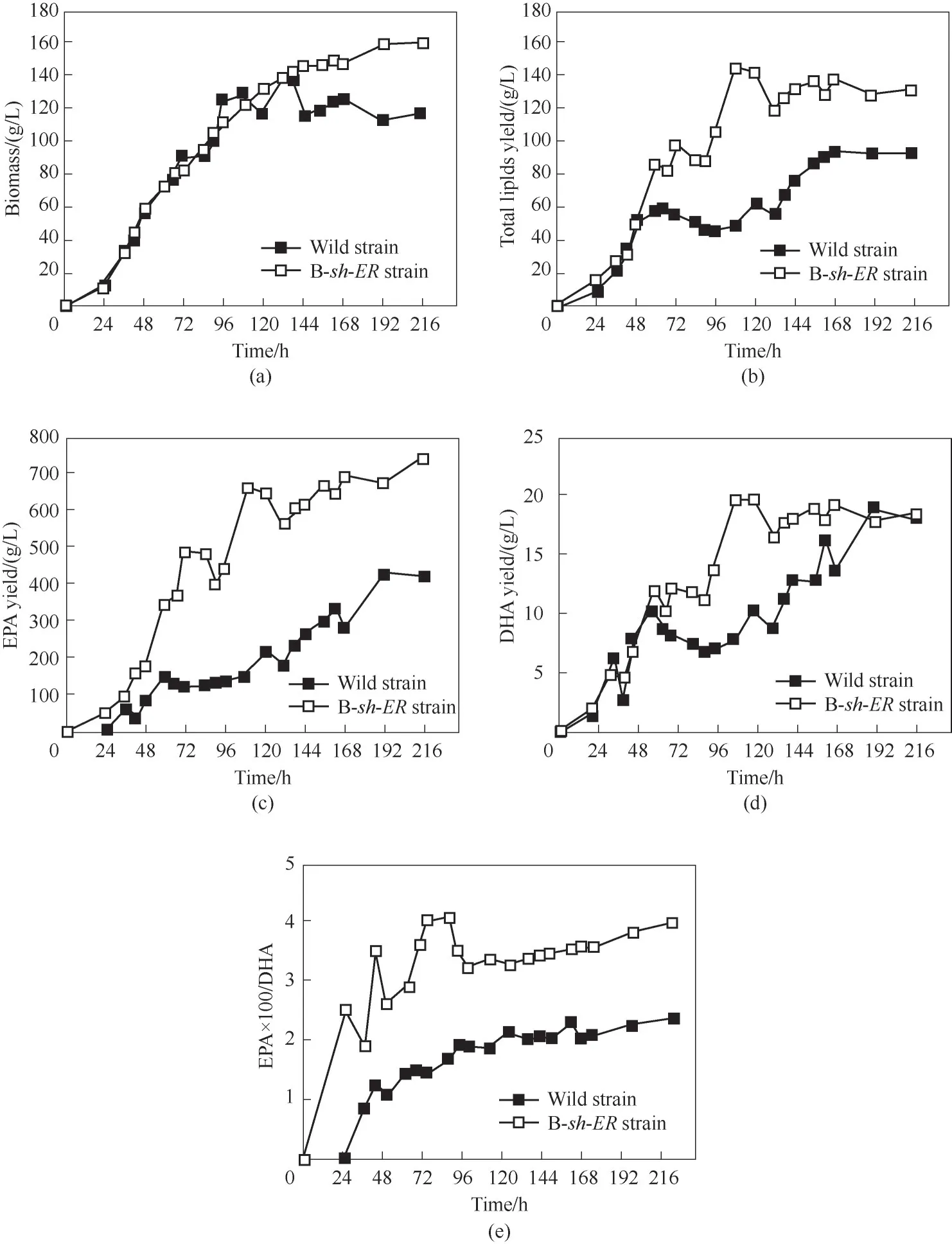
图8 野生菌株和B-sh-ER菌株的补料分批发酵参数
(a)生物量;(b)总油脂量;(c)EPA产量;(d)DHA产量;(e)EPA×100/DHA
Fig.8 Fed-batch fermentation parameters of wild strain and B-sh-ERstrain
[3] Oppedisano F, Macrì R, Gliozzi M, et al. The anti-inflammatory and antioxidant properties of n-3 PUFAs: their role in cardiovascular protection[J].Biomedicines,2020,8(9):306.
[4] Amigó N, Akinkuolie A O, Chiuve S E, et al. Habitual fish consumption, n-3 fatty acids, and nuclear magnetic resonance lipoprotein subfractions in women[J]. Journal of the American Heart Association,2020,9(5):e014963.
[5] 夏尧干,王宇,邹雯燕,等.鱼油脂肪酸EPA 和DHA 的分离影响研究[J].淮阴工学院学报,2020,29(5):49-52.Xia Y G, Wang Y, Zou W Y, et al. Study on the separation effect of EPA and DHA from fish oil fatty acids[J]. Journal of Huaiyin Institute of Technology,2020,29(5):49-52.
[6] Yazawa K. Production of eicosapentaenoic acid from marine bacteria[J].Lipids,1996,31(1):S297-S300.
[7] El Razak A A,Ward A C,Glassey J.Screening of marine bacterial producers of polyunsaturated fatty acids and optimisation of production[J].Microbial Ecology,2014,67(2):454-464.
[8] Zhang J W, Burgess J G. Enhanced eicosapentaenoic acid production by a new deep-sea marine bacteriumShewanella electrodiphilaMAR441T[J].PLoS One,2017,12(11):e0188081.
[9] Wen Z Y, Chen F. Heterotrophic production of eicosapentaenoic acid by microalgae[J]. Biotechnology Advances, 2003, 21(4):273-294.
[10] Wang S, Lan C Z, Wang Z J, et al. Optimizing eicosapentaenoic acid production by grafting a heterologous polyketide synthase pathway in theThraustochytrid aurantiochytrium[J]. Journal of Agricultural and Food Chemistry,2020,68(40):11253-11260.
[11] Sayanova O, Napier J A. Metabolic engineering of microalgae for sustainable production of omega-3 long chain polyunsaturated fatty acids[J].Current Biotechnology,2016,5(3):198-212.
[12] Hayashi S,Satoh Y,Ogasawara Y,et al.Control mechanism forcisdouble-bond formation by polyunsaturated fatty-acid synthases[J].Angewandte Chemie International Edition,2019,58(8):2326-2330.
[13] Ren L J, Chen S L, Geng L J, et al. Exploring the function of acyltransferase and domain replacement in order to change the polyunsaturated fatty acid profile ofSchizochytriumsp[J]. Algal Research,2018,29:193-201.
[14] Matsuda T, Sakaguchi K, Hamaguchi R, et al. Analysis of Δ12-fatty acid desaturase function revealed that two distinct pathways are active for the synthesis of PUFAs inT. aureumATCC 34304[J].Journal of Lipid Research,2012,53(6):1210-1222.
[15] Aasen I M,Ertesvåg H,Heggeset T M B,et al.Thraustochytrids as production organisms for docosahexaenoic acid (DHA), squalene,and carotenoids[J]. Applied Microbiology and Biotechnology,2016,100(10):4309-4321.
[16] Yaguchi T, Tanaka S, Yokochi T, et al. Production of high yields of docosahexaenoic acid bySchizochytriumsp. strain SR21[J].Journal of the American Oil Chemists ' Society, 1997, 74(11):1431-1434.
[17] Wallis J G, Watts J L, Browse J. Polyunsaturated fatty acid synthesis: what will they think of next?[J]. Trends in Biochemical Sciences,2002,27(9):467-473.
[18] Marrakchi H, Dewolf W E, Quinn C, et al. Characterization ofStreptococcus pneumoniaeenoyl-(acyl-carrier protein) reductase(FabK)[J].The Biochemical Journal,2003,370(Pt 3):1055-1062.
[19] Heath R J, Rock C O. Enoyl-acyl carrier protein reductase (fabI)plays a determinant role in completing cycles of fatty acid elongation inEscherichia coli[J]. Journal of Biological Chemistry,1995,270(44):26538-26542.
[20] Ling X P, Zhou H, Yang Q H, et al. Functions of enyolreductase(ER) domains of PKS cluster in lipid synthesis and enhancement of PUFAs accumulation inSchizochytrium limacinumSR21 using triclosan as a regulator of ER[J].Microorganisms,2020,8(2):300.
[21] Lee S J, Seo P S, Kim C H, et al. Isolation and characterization of the eicosapentaenoic acid biosynthesis gene cluster fromShewanellasp.BR-2[J]. Journal of Microbiology and Biotechnology,2009,19(9):881-887.
[22] Metz J G, Roessler P, Facciotti D, et al. Production of polyunsaturated fatty acids by polyketide synthases in both prokaryotes and eukaryotes[J]. Science, 2001, 293(5528):290-293.
[23] Peng Y F, Chen W C, Xiao K, et al. DHA production inEscherichia coliby expressing reconstituted key genes of polyketide synthase pathway from marine bacteria[J]. PLoS One,2016,11(9):e0162861.
[24] Lee S J, Kim C H, Seo P S, et al. Enhancement of heterologous production of eicosapentaenoic acid inEscherichia coliby substitution of promoter sequences within the biosynthesis gene cluster[J].Biotechnology Letters,2008,30(12):2139-2142.
[25] Gemperlein K, Dietrich D, Kohlstedt M, et al. Polyunsaturated fatty acid production byYarrowia lipolyticaemploying designed myxobacterial PUFA synthases[J]. Nature Communications, 2019,10(1):4055.
[26] Xue Z X, Sharpe P L, Hong S P, et al. Production of omega-3 eicosapentaenoic acid by metabolic engineering ofYarrowia lipolytica[J].Nature Biotechnology,2013,31(8):734-740.
[27] Takeyama H, Takeda D, Yazawa K, et al. Expression of the eicosapentaenoic acid synthesis gene cluster fromShewanellasp.in a transgenic marine cyanobacterium,Synechococcussp[J].Microbiology,1997,143(Pt 8):2725-2731.
[28] Ling X P, Guo J, Liu X T, et al. Impact of carbon and nitrogen feeding strategy on high production of biomass and docosahexaenoic acid (DHA) bySchizochytriumsp.LU310[J].Bioresource Technology,2015,184:139-147.
[29] Li Z P, Meng T, Ling X P, et al. Overexpression of malonyl-CoA:ACP transacylase inSchizochytriumsp. to improve polyunsaturated fatty acid production[J]. Journal of Agricultural and Food Chemistry,2018,66(21):5382-5391.
[30] Li Z, Ling X, Zhou H, et al. Screening chemical modulators of benzoic acid derivatives to improve lipid accumulation inSchizochytrium limacinumSR21 with metabolomics analysis[J].Biotechnology for Biofuels,2019,12:209.
[31] Pan X S, Wang B B, Duan R, et al. Enhancing astaxanthin accumulation inXanthophyllomyces dendrorhousby a phytohormone: metabolomic and gene expression profiles[J].Microbial Biotechnology,2020,13(5):1446-1460.
[32] Li J P, Wei Y X, Li J C, et al. A novel duplex SYBR Green realtime PCR with melting curve analysis method for beef adulteration detection[J].Food Chemistry,2021,338:127932.
[33] Ren L J, Zhuang X Y, Chen S L, et al. Introduction of ω-3 desaturase obviously changed the fatty acid profile and sterol content ofSchizochytriumsp[J]. Journal of Agricultural and Food Chemistry,2015,63(44):9770-9776.
[34] Ren L J, Huang H, Xiao A H, et al. Enhanced docosahexaenoic acid production by reinforcing acetyl-CoA and NADPH supply inSchizochytriumsp. HX-308[J]. Bioprocess and Biosystems Engineering,2009,32(6):837-843.
[35] Snoep J L, Yomano L P, Westerhoff H V, et al. Protein burden inZymomonas mobilis: negative flux and growth control due to overproduction of glycolytic enzymes[J]. Microbiology, 1995, 141(9):2329-2337.
[36] Nishida I, Murata N. CHILLING SENSITIVITY IN PLANTS AND CYANOBACTERIA: the crucial contribution of membrane lipids[J]. Annual Review of Plant Physiology and Plant Molecular Biology,1996,47:541-568.
[37] Venkateswaran K, Moser D P, Dollhopf M E, et al. Polyphasic taxonomy of the genusShewanellaand description ofShewanella oneidensissp. nov[J]. International Journal of Systematic Bacteriology,1999,49 Pt 2:705-724.
[38] Lippmeier J C, Crawford K S, Owen C B, et al. Characterization of both polyunsaturated fatty acid biosynthetic pathways inSchizochytriumsp.[J].Lipids,2009,44(7):621-630.
[39] Hauvermale A, Kuner J, Rosenzweig B, et al. Fatty acid production inSchizochytriumsp.: involvement of a polyunsaturated fatty acid synthase and a type I fatty acid synthase[J].Lipids,2006,41(8):739-747.
[40] Cerón Garcí M C,Fernández Sevilla J M,Acién Fernández F G,et al. Mixotrophic growth ofPhaeodactylum tricornutumon glycerol:growth rate and fatty acid profile[J]. Journal of Applied Phycology,2000,12(3/4/5):239-248.
[41] Shi H S, Chen H Q, Gu Z N, et al. Application of a delta-6 desaturase with α-linolenic acid preference on eicosapentaenoic acid production inMortierella alpina[J]. Microbial Cell Factories,2016,15(1):117.
[42] Okuda T, Ando A, Negoro H, et al. Eicosapentaenoic acid (EPA)production by an oleaginous fungusMortierella alpinaexpressing heterologous the Δ17-desaturase gene under ordinary temperature[J]. European Journal of Lipid Science and Technology, 2015, 117(12):1919-1927.
[43] Cui G Z, Ma Z X, Liu Y J, et al. Overexpression of glucose-6-phosphate dehydrogenase enhanced the polyunsaturated fatty acid composition ofAurantiochytriumsp. SD116[J]. Algal Research,2016,19:138-145.
[44] Khozin-Goldberg I, Yu H Z, Adlerstein D, et al. Triacylglycerols of the red microalgaPorphyridium cruentumcan contribute to the biosynthesis of eukaryotic galactolipids[J]. Lipids, 2000, 35(8):881-889.
[45] Fernández F G A, Pérez J A S, Sevilla J M F, et al. Modeling of eicosapentaenoic acid (EPA) production fromPhaeodactylum tricornutumcultures in tubular photobioreactors. Effects of dilution rate, tube diameter, and solar irradiance[J]. Biotechnology and Bioengineering,2000,68(2):173-183.
[46] Huerlimann R, de Nys R, Heimann K. Growth, lipid content,productivity, and fatty acid composition of tropical microalgae for scale-up production[J]. Biotechnology and Bioengineering, 2010,107(2):245-257.

