尾细桉生长和木材密度关联SNP挖掘与候选基因定位
朱显亮,周长品,贾翠蓉,翁启杰,李发根
(中国林业科学研究院热带林业研究所, 热带林业研究国家林业和草原局重点实验室,广东 广州 510520)
桉树起源于澳大利亚及其附近岛屿,是桃金娘科(Myrtaceae)桉属(Eucalyptus)、杯果木属(Angophora)和伞房属(Corymbia)的总称[1-2]。我国自1890年引种桉树,至今已有100余年历史[3]。据全国第9次森林资源清查,我国桉树人工林面积已达546万 hm2,占全国人工林总面积6.8%。桉树是我国重要的工业用材树种,其生长和材性性状是重要的经济性状,直接影响人工林产量及纸浆产量和质量,对于桉树生长和材性的遗传改良一直备受育种者重视。在桉树中,对巨桉(E.grandis)已完成全基因组测序[4]。同时,研究者在多个桉树群体的种源、家系及无性系间进行了生长及材性性状遗传分析,但不同群体的分析结果不尽相同[5-8]。目前为止,对于桉树中许多重要经济性状的遗传基础及调控机理都知之甚少。
单核苷酸多态性(single nucleotide polymorphism, SNP)是指基因组水平上由单个核苷酸变异所引起的DNA序列多态性[9]。SNP具有分布密度高、遗传稳定性强、易于高通量检测和分型的特点[10]。利用SNP标记研究桉树复杂性状遗传变异规律,对于揭示桉树重要性状的分子调控机制具有重要意义。关联分析是进行复杂性状解析的一种有力工具,利用关联分析可获得与目标性状紧密相关的分子标记或候选基因[11]。目前,利用关联分析已在桉树生长[12-13]、材性[14-16]、抗病性[17-18]等方面开展研究,挖掘了一些与生长和材性相关联的位点和候选基因。但林木的生长与木材的形成受复杂多层次网络调控和微效多基因控制,可能有大量的基因参与其中[19],仍需加大力度开展桉树重要经济性状候选基因的发掘。
尾叶桉(E.urophylla)和细叶桉(E.tereticornis)杂种具有生长迅速、抗风性强、产量高的特点,在华南沿海地区广泛种植[20]。本研究以尾细桉F1全同胞子代试验林为研究对象,对各F1子代的树高、胸径和木材基本密度进行测定及表型遗传分析;筛选这3个性状的极端表型个体,利用测序分型(genotyping by sequencing, GBS)方法开发SNP位点;通过关联分析挖掘与尾细桉生长和木材基本密度显著相关的位点,为桉树分子标记辅助育种选择提供标记资源。
1 材料与方法
1.1 植物材料
研究群体包含320个尾细桉F1子代,来源于母本尾叶桉(x-30)和父本细叶桉(13443-05)的杂交子代扦插后营建的无性系试验林[21]。试验林位于广东省鹤山市共和镇,于2006年6月造林,采用随机完全区组设计,每个无性系设置4次重复,单株小区,株行距为2 m×3 m。
1.2 表型性状分析
2014年1月对试验林全部植株进行表型测定。树高和胸径测量分别使用超声波测高仪(VertexIII,瑞典)和测树胸径尺测定。木材基本密度通过生长锥法钻取树木1.3 m处南北向的木芯,参考国家标准GB/T 1933—2009以排水法测定。性状统计及相关性分析利用SAS V6.3。
1.3 极端表型个体选择
在试验林320个F1子代中筛选树高(height,H)、胸径(diameter,D)和木材基本密度(basic density,ρ)的极高和极低表型各6个开展关联分析。由于表型性状间的极端个体有部分重叠,最终共选择了21个个体。极端表型个体与群体间的差异显著性采用独立样本t检验。采集极端表型个体树干中部、南向的健康叶片,以塑封袋密封后于干冰中临时保存,之后保存于-80 ℃冰箱用于DNA提取。
1.4 GBS文库构建及SNP标记开发
DNA提取采用改良CTAB法[22],GBS文库构建主要参照Poland等[23]的建库流程,QubitTM3.0测定文库浓度后使用微流控毛细管电泳系统(LabChip GX Touch 24,美国)检测文库质量。双端测序(PE 150)由北京贝瑞基因的高通量测序平台Illumina HiSeq 2500完成。
SNP开发主要流程为:采用Stacks V2.5[24]进行下机数据的拆分并过滤低质量序列,利用Bowtie2 V2.2.2.9[25]将过滤后的序列比对到巨桉参考基因组(https://phytozome.jgi.doe.gov/pz/portal.html)。利用GATK V3.8[26]开发SNP后通过VCFtools V0.1.13[27]进行过滤,SNP过滤标准为:SNP支持的reads数目4条以上,数据缺失率小于30%,次要等位基因频率(MAF)>0.05。剔除缺失率大于50%的位点,保留高质量的SNP。
1.5 标记-性状关联分析及候选基因定位
采用单因素方差分析法[28]开展关联分析,以P<0.001为阈值筛选与目标性状显著关联的SNP,并根据Bonferroni(α=0.05)多重检验校正后的阈值确定极显著关联SNP。利用巨桉参考基因组信息定位关联SNP的候选基因,利用Blast2GO[29]进行候选基因功能注释与分类。根据基因注释信息,以及结合相应文献报道,初步了解候选基因的基本生物学功能。
2 结果与分析
2.1 尾细桉F1代的表型分析
研究群体中尾细桉F1子代的树高、胸径和木材基本密度均呈现连续分布(图1),且偏度、峰度较小(表1),表型值近似符合正态分布。其中树高和木材基本密度的偏度均接近-1,整体分布偏向高值,表明该试验林长势优,树高及木材基本密度呈中上水平的子代居多。所选树高、胸径、木材基本密度的极端个体与群体间的差异均显著(P<0.001)。树高、胸径的变异系数分别为17.38%和26.19%,而木材基本密度仅为7.51%,表明生长性状比木材密度的遗传变异大。
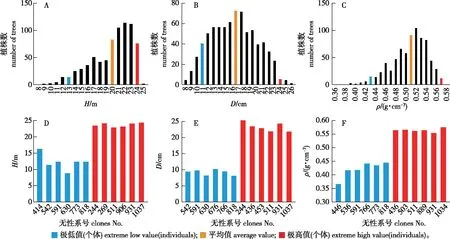
图1 尾细桉试验林的生长性状和木材密度分布及所选极端表型个体

表1 试验群体表型数据参数
2.2 尾细桉生长性状与木材密度相关性分析
生长性状与木材基本密度间均呈显著正相关(P<0.01)(表2)。其中,树高与胸径间的相关性最高,为0.792;树高与木材基本密度次之,为0.559;而木材基本密度与胸径间的相关性最低,为0.480。表明生长性状之间的相关性高于生长性状与木材材性间的相关性。

表2 生长性状与木材基本密度相关性
2.3 GBS测序质量及SNP开发
通过高通量测序,在21个尾细桉极端表型个体中获得的原始序列数为5 568 416~14 002 206,经过滤低质量的序列后,得到的高质量序列数为4 966 451~12 404 893(表3)。质控后,高质量序列的Q30值介于85.6%~92.3%,平均GC含量为41.6%,与巨桉参考基因组的比对率为96.2%~97.8%,表明测序质量较高。过滤低质量位点后,最终保留15 185个高质量SNP用于后续分析。
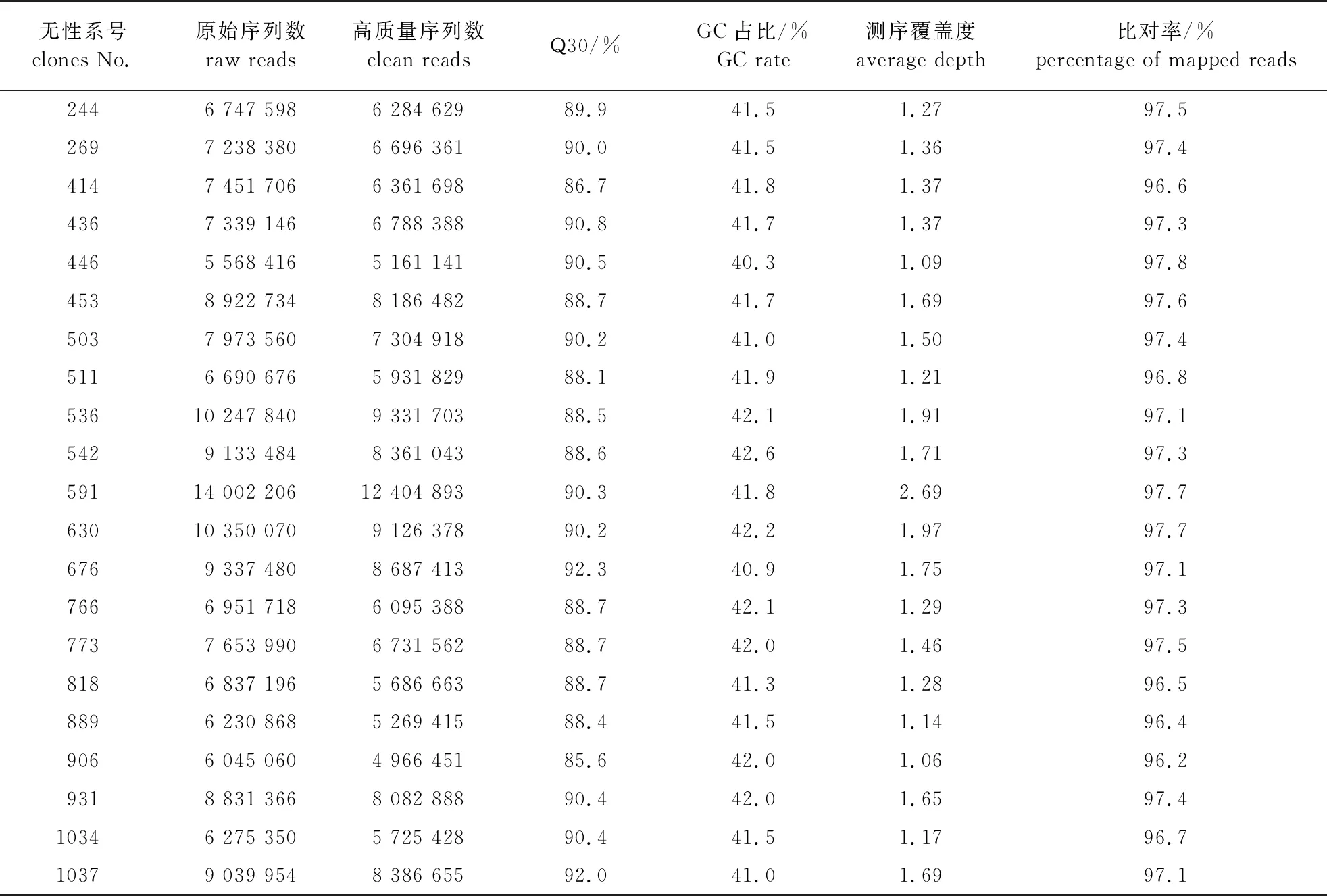
表3 各样品的GBS测序数据统计
2.4 SNP标记与表型性状关联分析
剔除缺失率大于50%的标记后,最终用于关联分析的标记分别为10 432 (H)、10 849 (D)和10 495 (ρ)个。关联分析获得与树高、胸径及木材基本密度显著关联的标记分别为66、31和22个(图2、表4)。通过比较3个性状间的关联SNP,在树高与胸径间发现6个多效性SNP,在树高与木材基本密度间发现1个多效性SNP,在胸径与木材基本密度间发现1个多效性SNP(图3A)。经Bonferroni多重检验校正,共检测到3个SNP与树高极显著关联,其中1、5、6号染色体各分布1个;13个与胸径极显著关联,其中2号染色体分布7个,7号染色体分布1个,11号染色体分布2个以及3个未锚定至巨桉参考基因组;3个与木材基本密度极显著关联,其中4号和7号染色体各分布1个以及1个未锚定巨桉参考基因组(表5)。这些位点与目标性状间存在强烈的关联信号,可能为调控该性状的主效位点。
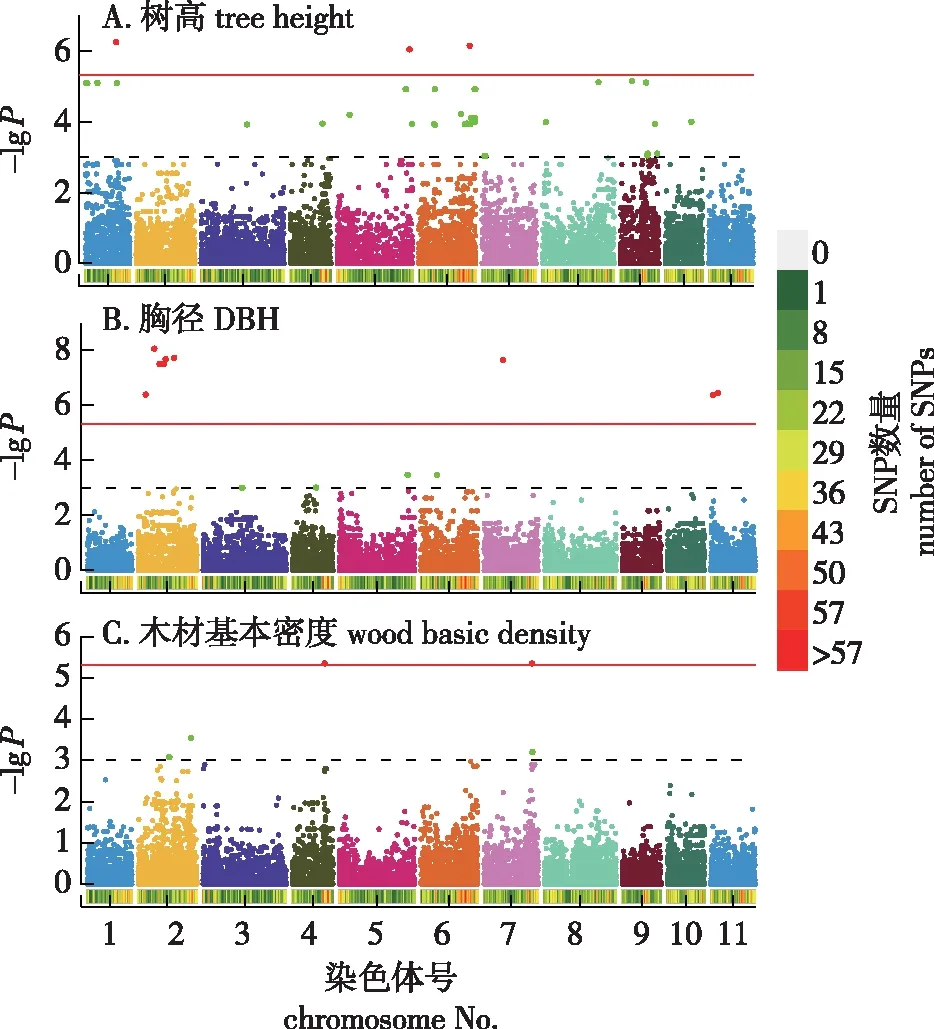
红线表示Bonferroni(α=0.05)校正的阈值,绿线表示-lg P =3.0的阈值。Red line indicates the Bonferroni-corrected threshold with an experimental type I error rate at α=0.05 and green line represents the threshold of -lg P =3.0.
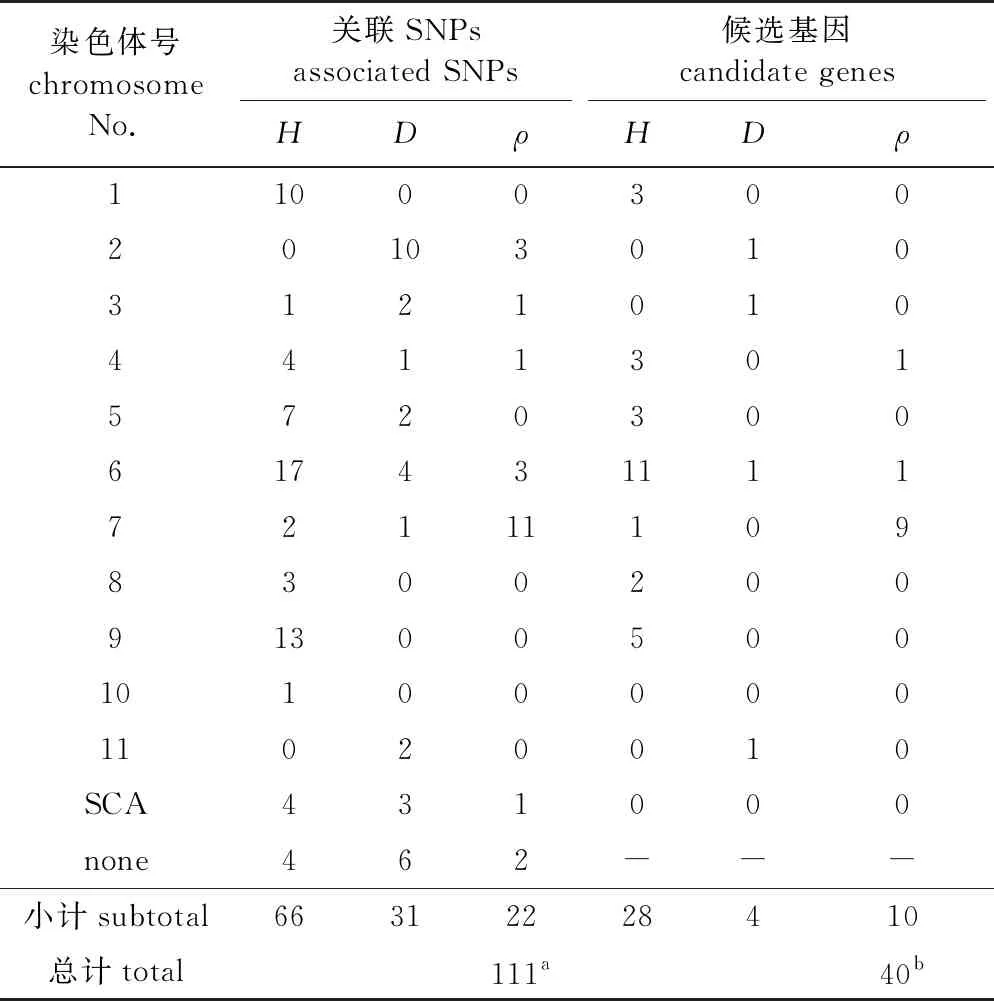
表4 关联标记及候选基因分布情况

图3 全部关联标记(A)及候选基因(B)韦恩图

表5 各性状极显著关联标记信息
2.5 候选基因定位及GO功能分类
利用SNP位点的物理位置在巨桉参考基因组上进行基因查找,共发掘候选基因40个(表4),其中,树高28个,23个功能已知;胸径4个,3个功能已知;木材基本密度10个,9个功能已知。树高与胸径、树高与木材基本密度中各定位到1个共有的候选基因(图3B)。GO功能分类主要将生长和木材基本密度的相关候选基因归为生物过程、分子功能和细胞成分3大类的52个GO terms(图4)。在生物过程中,候选基因大部分富集在有机物代谢过程、主要代谢过程、细胞代谢过程和氮化合物代谢过程。在分子功能中,候选基因主要富集在杂环化合物结合和有机环化合物结合。在细胞组成中,候选基因主要位于细胞器、细胞内细胞器和膜固有成分。
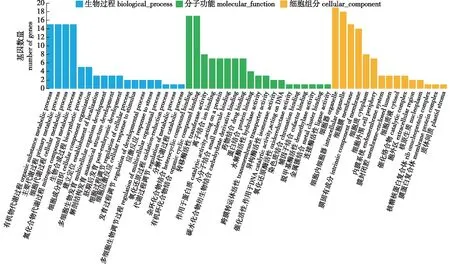
图4 候选基因GO功能分类
3 讨 论
关联分析可以在群体水平上分析性状变异,准确定位与目标性状关联的基因或基因片段[30]。早先在赤桉(E.camaldulensis)[12]、蓝桉(E.globulus)[15]、大花序桉(E.cloeziana)[16]和柠檬桉(C.citriodora)[17]等桉树中开展的关联研究多使用直接来源于天然群体的种质资源。这些种质资源中富含大量的等位变异,有利于提高关联位点和基因地检测,然而不足之处在于天然群体中复杂的群体结构和亲缘关系会影响检测结果。尽管可以通过一些统计模型解决部分问题[31],但更重要的是在未驯化的天然群体中获得的关联标记还需要面临如何转化为有用育种信息的难题。而在人工选育的优质育种群体中获得的关联位点,可以更直接有效地应用于实际的育种工作中[32]。基于该理论基础,最近两个研究报道了经人工选育后的桉树杂种育种群体的关联分析[13,18]。与先前的研究[13, 18]相比,尽管本研究也使用了人工构建的尾细桉杂种群体,但笔者重点关注了其中一些具有极端表型的个体。通过对极端表型个体进行关联分析,在生长和木材密度中共获得111个显著关联的SNP标记,进一步丰富了桉树现有的基因组资源。同时也表明,基于人工育种群体的极端表型个体筛选相关性状关联位点是一种快速、经济和有效的策略。但值得注意的是,这种策略可能降低了对于生长和木材形成中具有微效作用的基因位点的检测能力,为保证关联分析的检测效力和精度,应提高检测样本的数量。此外,关联分析的结果还需要验证,如使用不同亲本群体、标记及相关的生理生化实验验证等。
基于Bonferroni多重检验校正方法以确定极显著关联的标记,3、13和3个SNP分别与树高、胸径和木材基本密度极显著关联,这些SNP所在的区域可能存在调控桉树生长和木材性状的主效基因位点。其中,在2号染色体上检测到了生长性状(胸径)的强烈关联信号。基于全基因组的关联位点扫描,先前的研究[13,18]鉴定了桉树2号染色体22.0 Mb和36.4 Mb的临近位置存在生长性状的关联位点,与本研究的结果一致。此外,笔者在1、4、5、6、7和11号染色体的部分区域也检测到了与目标性状间的强烈关联信号。与以往研究中[33-35]利用QTL定位鉴定的生长和木材密度相关位点的位置部分一致。桉树的生长和木材密度均受众多基因位点调控,但由于研究群体的遗传背景不同,以及表型受基因型与环境的影响等,本研究鉴定的关联区域与先前研究鉴定的QTL区域有所差异。另一方面,这些差异的位点可能为本研究在尾细桉中发现的与生长和木材密度相关的新标记位点。
通过对关联SNP进行注释初步定位了40个与生长和木材密度相关的候选基因,共富集在52个GO terms。基因功能注释分析表明其功能主要与植物抗逆性、生物与非生物胁迫、转录因子家族等相关。胸径(Eucgr.C02139)和树高(Eucgr.L00646)中均发现与DNA拓扑异构酶I (topoisomerase I, TOP I)合成相关的候选基因。TOP I通过催化DNA链的瞬时断裂和连接,从而控制DNA的复制、重组、转录等过程,在植物整个生长发育过程都发挥重要作用[36-37]。研究显示,TOP I对于拟南芥(Arabidopsisthaliana)的茎及花中的干细胞维持十分重要[38],推测TOP I可能在桉树中也发挥着类似的作用。一些与抗逆性相关的基因可能在尾细桉的生长和木材形成中发挥重要作用。如,树高(Eucgr.F04201)和木材基本密度(Eucgr.G02820)中均发现与蛋白激酶(protein kinases, PK)合成相关的候选基因。PK是植物体内一类重要的与抗逆相关的调节因子,植物通过膜受体PK感知外界胁迫信号,从而启动相应的生理生化反应来抵抗危害[39]。类似功能的还有树高候选基因Eucgr.G00235,其与叶绿体热激蛋白70(heat shock protein 70,HSP70)合成有关。HSP是植物细胞受到高温或其他胁迫时产生的一类功能性蛋白[40],其一旦产生,将极大增强植物的抗逆性。在烟草(Nicotianatabacum)[41]和拟南芥[42]中转HSP70基因,可显著提高其耐热性和耐旱性。基于上述研究,笔者推测与抗逆性相关的候选基因主要通过调节植物的应激能力和抵抗能力,进而对生长及木材合成造成间接影响。此外,先前的研究表明多效性基因在调控桉树生长及材性性状方面具有重要作用[34]。本研究对树高、胸径和木材基本密度分别进行了关联分析,其中8个SNP和2个候选基因可能在调控生长和材性性状方面具有多效性作用。尽管这些多效性基因位点的鉴定可能受表型性状间本身的相关性,以及在取材上的部分重叠影响。但控制林木数量性状的基因普遍存在一因多效现象[43],多效性基因可能同时参与不同遗传调控,可应用于育种中多个性状改良,是今后的研究重点。后期将开展QTL精细定位进一步解析尾细桉生长及材性变异规律。
参考文献(reference):
[1]HILL K,JOHNSON L.Systematic studies in the eucalypts.7.A revision of the bloodwoods,genusCorymbia(Myrtaceae)[J].Telopea,1995,6(2/3):185-504.DOI:10.7751/telopea19953017.
[2]TURNBULL J W.Eucalyptplantations[J]. New Forests, 1999, 17(1/3): 37-52. DOI:10.1023/a:1006524911242.
[3]王豁然.桉树生物学概论[M].北京:科学出版社,2010.WANG H R. Chinese appreciation of eucalyptus[M].Beijing:Science Press,2010.
[4]MYBURG A A,GRATTAPAGLIA D,TUSKAN G A,et al.The genome ofEucalyptusgrandis[J].Nature,2014,510(7505):356-362.DOI:10.1038/nature13308.
[5]STACKPOLE D J,VAILLANCOURT R E,AGUIGAR M,et al.Age trends in genetic parameters for growth and wood density inEucalyptusglobulus[J].Tree Genet Genomes,2010,6(2):179-193.DOI:10.1007/s11295-009-0239-4.
[6]李昌荣,陈健波,郭东强,等.锯材大花序桉生长和材性的综合指数选择[J].南京林业大学学报(自然科学版),2019,43(1):1-8.LI C R,CHEN J B,GUO D Q,et al.Comprehensive index selection on superior growth and wood properties ofEucalyptuscloezianafor saw timber[J].J Nanjing For Univ (Nat Sci Ed),2019,43(1):1-8.DOI:10.3969/j.issn.1000-2006.201805018.
[7]朱显亮,兰俊,王建忠,等.中大径材尾细桉杂种无性系选择研究[J].南京林业大学学报(自然科学版),2020,44(2):43-50.ZHU X L,LAN J,WANG J Z,et al.Clonal selection of middle/large diameter timber ofEucalyptusurophylla×E.tereticornishybrid clones[J].J Nanjing For Univ (Nat Sci Ed),2020,44(2):43-50.DOI:10.3969/j.issn.1000-2006.201904005.
[8]YANG H Y,WENG Q J,LI F G,et al.Genotypic variation and genotype-by-environment interactions in growth and wood properties in a clonedEucalyptusurophylla×E.tereticornisfamily in southern China[J].For Sci,2018,64(3):225-232.DOI:10.1093/forsci/fxx011.
[9]LANDER E S.The new genomics:global views of biology[J].Science,1996,274(5287):536-539.DOI:10.1126/science.274.5287.536.
[10]周长品,翁启杰,甘四明,等.应用SNaPshot技术对桉树SNP的检测[J].南京林业大学学报(自然科学版),2018,42(4):83-88.ZHOU C P,WENG Q J,GAN S M,et al.Application of SNaPshot to detect SNP markers inEucalyptus[J].J Nanjing For Univ (Nat Sci Ed),2018,42(4):83-88.DOI: 10.3969/issn.1000-2006.2017.05055.
[11]NEALE D B,SAVOLAINEN O.Association genetics of complex traits in conifers[J].Trends Plant Sci,2004,9(7):325-330.DOI:10.1016/j.tplants.2004.05.006.
[12]尚秀华,张沛健,谢耀坚,等.赤桉抗风和生长性状的SSR关联分析[J].南京林业大学学报(自然科学版),2018,42(4):97-105.SHANG X H,ZHANG P J,XIE Y J,et al.SSR association analysis ofEucalyptuscamaldulensiswind resistance and growth traits[J].J Nanjing For Univ (Nat Sci Ed),2018,42(4):97-105.DOI:10.3969/j.issn.1000-2006.201711019.
[13]MÜLLER B S F,DE ALMEIDA FILHO J E,LIMA B M,et al.Independent and Joint-GWAS for growth traits inEucalyptusby assembling genome-wide data for 3373 individuals across four breeding populations[J].New Phytol,2019,221(2):818-833.DOI:10.1111/nph.15449.
[14]THUMMA B R,NOLAN M F,EVANS R,et al.Polymorphisms in cinnamoyl CoA reductase (CCR) are associated with variation in microfibril angle inEucalyptusspp.[J].Genetics,2005,171(3):1257-1265.DOI:10.1534/genetics.105.042028.
[15]CAPPA E P,EL-KASSABY Y A,GARCIA M N,et al.Impacts of population structure and analytical models in genome-wide asso-ciation studies of complex traits in forest trees:a case study inEucalyptusglobulus[J].PLoS One,2013,8(11):e81267.DOI:10.1371/journal.pone.0081267.
[16]李昌荣.大花序桉生长和材性遗传变异及SSR关联分析[D].北京:中国林业科学研究院,2017. LI C R. Gere Variation and SSR association analyses in growth and wood properties inEucalyptuscloeziana[D]. Beijing: Chinese Academy of Forestry, 2017.
[17]DILLON S K,BRAWNER J T,MEDER R,et al.Association genetics inCorymbiacitriodorasubsp.variegataidentifies single nucleotide polymorphisms affecting wood growth and cellulosic pulp yield[J].New Phytol,2012,195(3):596-608.DOI:10.1111/j.1469-8137.2012.04200.x.
[18]RESENDE R T,RESENDE M D,SILVA F F,et al.Regional heritability mapping and genome-wide association identify loci for complex growth,wood and disease resistance traits inEucalyptus[J].New Phytol,2017,213(3):1287-1300.DOI:10.1111/nph.14266.
[19]GRATTAPAGLIA D,PLOMION C,KIRST M,et al.Genomics of growth traits in forest trees[J].Curr Opin Plant Biol,2009,12(2):148-156.DOI:10.1016/j.pbi.2008.12.008.
[20]彭仕尧,徐建民,李光友,等.尾细桉无性系在雷州半岛的生长与遗传分析[J].中南林业科技大学学报,2013,33(4):23-27.PENG S Y,XU J M,LI G Y,et al.Growth and genetic analysis of 42Eucalyptusurophylla×E.tereticornisclones in Leizhou Peninsula of China[J].J Central South Univ For Technol,2013,33(4):23-27.DOI:10.14067/j.cnki.1673-923x.2013.04.018.
[21]甘四明,李梅,李发根,等.尾叶桉×细叶桉杂种无性系扦插生根和生长性状的研究[J].林业科学研究,2006,19(2):135-140.GAN S M,LI M,LI F G,et al.Analysis on cutting and growth traits of clones ofEucalyptusurophylla×E.tereticornis[J].For Res,2006,19(2):135-140.DOI:10.3321/j.issn:1001-1498.2006.02.002.
[22]GAN S M,SHI J S,LI M,et al.Moderate-density molecular maps ofEucalyptusurophyllaS.T.Blake andE.tereticornisSmith genomes based on RAPD markers[J].Genetica,2003,118(1):59-67.DOI:10.1023/a:1022966018079.
[23]POLAND J A,BROWN P J,SORRELLS M E,et al.Development of high-density genetic maps for barley and wheat using a novel two-enzyme genotyping-by-sequencing approach[J].PLoS One,2012,7(2):e32253.DOI:10.1371/journal.pone.0032253.
[24]CATCHEN J M,AMORES A,HOHENLOHE P,et al.Stacks:building and genotyping Loci de novo from short-read sequences[J].G3 (Bethesda),2011,1(3):171-182.DOI:10.1534/g3.111.000240.
[25]LANGMEAD B,SALZBERG S L.Fast gapped-read alignment with Bowtie 2[J].Nat Methods,2012,9(4):357-359.DOI:10.1038/nmeth.1923.
[26]MCKENNA A,HANNA M,BANKS E,et al.The genome analysis toolkit:a MapReduce framework for analyzing next-generation DNA sequencing data[J].Genome Res,2010,20(9):1297-1303.DOI:10.1101/gr.107524.110.
[27]DANECEK P,AUTON A,ABECASIS G,et al.The variant call format and VCFtools[J].Bioinformatics,2011,27(15):2156-2158.DOI:10.1093/bioinformatics/btr330.
[28]高美玲,梁晓雪,刘秀杰,等.基于极端个体GBS测序初步定位西瓜果形基因[J].分子植物育种,2020,18(10),18:3164-3171.GAO M L,LIANG X X,LIU X J,et al.Mapping gene for fruit shape in watermelon based on extreme individuals genotyping-by-sequencing(GBS)[J].Mol Plant Breed,2020,18(10),18:3164-3171.DOI:10.13271/j.mpb.018.003164.
秦铁崖听乔十二郎介绍自己是他表姐夫的好朋友,既感到滑稽,也感到温暖,随即对老太医说:“不用客气,饮茶清谈,也很不错。”
[30]CONTRERAS-SOTO R I,MORA F,DE OLIVEIRA M A,et al.A genome-wide association study for agronomic traits in soybean using SNP markers and SNP-based haplotype analysis[J].PLoS One,2017,12(2):e0171105.DOI:10.1371/journal.pone.0171105.
[31]BRADBURY P J,ZHANG Z W,KROON D E,et al.TASSEL:software for association mapping of complex traits in diverse samples[J].Bioinformatics,2007,23(19):2633-2635.DOI:10.1093/bioinformatics/btm308.
[32]BEGUM H,SPINDEL J E,LALUSIN A,et al.Genome-wide association mapping for yield and other agronomic traits in an elite breeding population of tropical rice (Oryzasativa)[J].PLoS One,2015,10(3):e0119873.DOI:10.1371/journal.pone.0119873.
[33]FREEMAN J S,WHITTOCK S P,POTTS B M,et al.QTL influencing growth and wood properties inEucalyptusglobulus[J].Tree Genet Genomes,2009,5(4):713-722.DOI:10.1007/s11295-009-0222-0.
[34]GION J M,CAROUCHÉ A,DEWEER S,et al.Comprehensive genetic dissection of wood properties in a widely-grown tropical tree:Eucalyptus[J].BMC Genomics,2011,12:301.DOI:10.1186/1471-2164-12-301.
[35]KULLAN A R,VAN DYK M M,HEFER C A,et al.Genetic dissection of growth,wood basic density and gene expression in interspecific backcrosses ofEucalyptusgrandisandE.urophylla[J].BMC Genet,2012,13:60.DOI:10.1186/1471-2156-13-60.
[37]LIU X,GAO L,DINH T T,et al.DNA topoisomerase I affects polycomb group protein-mediated epigenetic regulation and plant development by altering nucleosome distribution inArabidopsis[J].Plant Cell,2014,26(7):2803-2817.DOI:10.1105/tpc.114.124941.
[38]ZHANG Y H,ZHENG L L,HONG J H,et al.TOPOISOMERASE1α Acts through two distinct mechanisms to regulate stele andColumellastem cell maintenance[J].Plant Physiol,2016,171(1):483-493.DOI:10.1104/pp.15.01754.
[39]裴丽丽,郭玉华,徐兆师,等.植物逆境胁迫相关蛋白激酶的研究进展[J].西北植物学报,2012,32(5):1052-1061.PEI L L,GUO Y H,XU Z S,et al.Research progress on stress-related protein kinases in plants[J].Acta Bot Boreali-Occidentalia Sin,2012,32(5):1052-1061.DOI:10.3969/j.issn.1000-4025.2012.05.032.
[40]LIN B L,WANG J S,LIU H C,et al.Genomic analysis of the Hsp70 superfamily inArabidopsisthaliana[J].Cell Stress Chape-rones,2001,6(3):201-208.DOI:10.1379/1466-1268(2001)0060201:gaoths>2.0.co;2.
[41]CHO E K,CHOI Y J.A nuclear-localized HSP70 confers thermoprotective activity and drought-stress tolerance on plants[J].Biotechnol Lett,2009,31(4):597-606.DOI:10.1007/s10529-008-9880-5.
[42]MONTERO-BARRIENTOS M,HERMOSA R,CARDOZA R E, et al.Transgenic expression of theTrichodermaharzianumhsp70gene increasesArabidopsisresistance to heat and other abiotic stresses[J].J Plant Physiol,2010,167(8):659-665.DOI:10.1016/j.jplph.2009.11.012.
[43]THUMMA B R,SOUTHERTON S G,BELL J C,et al.Quantitative trait locus (QTL) analysis of wood quality traits inEucalyptusnitens[J].Tree Genet Genomes,2010,6(2):305-317.DOI:10.1007/s11295-009-0250-9.

