在中国大豆品种上创建ALSV诱导的基因沉默体系
董永鑫,卫其巍,2,洪浩,黄莹,赵延晓,冯明峰,窦道龙,徐毅,陶小荣
在中国大豆品种上创建ALSV诱导的基因沉默体系
董永鑫1,卫其巍1,2,洪浩1,黄莹1,赵延晓1,冯明峰1,窦道龙1,徐毅1,陶小荣1
1南京农业大学植物保护学院/农业农村部大豆病虫害防控重点实验室,南京 210095;2四川省达州市农业科学研究院,四川达州 635000
【】在中国大豆品种上建立以苹果潜隐球形病毒(apple latent spherical virus,ALSV)为载体的基因沉默体系,为中国大豆品种的基因功能和遗传育种提供一种简便、省时、易操作的技术体系。构建以农杆菌介导接种的ALSV病毒侵染性克隆载体。从大豆品种威廉姆斯82(Williams 82)中特异扩增327 bp的八氢番茄红素脱氢酶()基因cDNA片段,插入病毒载体pALSV2。通过农杆菌浸润法将病毒载体导入模式植物本氏烟(),17 d后富集病毒粒子,摩擦接种大豆第一轮真叶,以接种病毒空载作为对照组,持续观察测试大豆植株系统叶表型,并结合逆转录-聚合酶链式反应(RT-PCR)或荧光定量PCR(qRT-PCR)检测ALSV衣壳蛋白基因()和的表达水平。ALSV﹕GmPDS接种大豆品种威廉姆斯82和南农1138-2,20 d后,前者系统叶未见白化表型,而后者系统叶出现明显的白化表型;qRT-PCR检测结果表明,发生白化的南农1138-2植株中的表达水平显著降低,未出现白化表型的威廉姆斯82中的表达水平没有出现显著变化。在此基础上,采用相同的方法,测试了ALSV在其他9种大豆品种中诱导的沉默效率,发现在南农47、安豆203、祥斗4号、中黄13、山宁29、齐黄34等大豆品种上接种ALSV﹕GmPDS后植株系统叶均产生白化表型,而菏豆12、中黄311和山宁16等3个品种的均不能诱导有效沉默。构建了农杆菌介导的ALSV病毒载体,利用本氏烟扩繁富集ALSV病毒,将提纯的病毒粒体摩擦接种大豆真叶,在多个中国大豆品种上成功建立了基因沉默体系。
病毒诱导的基因沉默;大豆;苹果潜隐球形病毒;大豆八氢番茄红素脱氢酶基因
0 引言
【研究意义】大豆((L.) Merr.)起源于中国,已有五千年栽培历史,是中国重要粮食作物之一,也是世界上最重要的豆类作物。大豆含有丰富的植物蛋白质,是植物油和蛋白的重要来源,在世界各地广泛种植。中国既是大豆原产地,又曾是全球大豆贸易最大出口国,然而由于国内对于大豆的需求与日俱增,中国的大豆产量难以满足国内日益增多的消费,超过八成的大豆消费依赖进口,目前,全球超过一半的大豆进口来自中国[1-2]。大豆遗传转化体系难度大,周期长,是阻碍大豆基因功能研究的主要瓶颈之一[3];此外,中国大豆品种多,在不同品种大豆上建立快速鉴定基因功能的手段和工具,对于进一步挖掘中国大豆重要农艺性状基因,研究大豆品种中与产量和抗病相关的基因功能,提高中国大豆产量和质量,促进大豆产业发展具有重要的理论科学意义[4-5]。【前人研究进展】病毒诱导的基因沉默(virus-induced gene silencing,VIGS)是一种研究基因功能的重要方法,通过将插入植物cDNA片段的重组病毒载体接种植物,利用病毒产生的双链RNA被植物中的RNA沉默元件识别,切割产生大量针对植物目的基因的小干扰RNA(small interference RNA),这些小干扰siRNA靶向植物内源目的基因mRNA,通过目的基因表达量降低后的植物表型或者生理指标的变化可以鉴定该基因的功能[6-7]。VIGS具有操作简单、周期短、同时沉默多个靶标基因及不依赖植物遗传转化体系等优点,被广泛用于不同植物基因功能的研究[8-10]。在大豆上的VIGS载体有菜豆荚斑驳病毒(bean pod mottle virus,BPMV)为基础构建的沉默载体,但该病毒需要用粒子轰击的方法将BPMV载体导入大豆的子叶中,或用体外转录的方法进行接种,方法复杂且BPMV侵染会在大豆上产生如褪绿、坏死等症状,影响表型观察[11-12]。苹果潜隐球形病毒(apple latent spherical virus,ALSV)载体也被报道用于沉默大豆基因[13-14]。ALSV属于樱桃锉叶病毒属病毒(),具有直径约25 nm的等轴形病毒颗粒,病毒粒体内包含2条单链基因组RNA(RNA1和RNA2)[15]。与其他报道的病毒载体相比,ALSV作为VIGS载体有几个优点:第一,ALSV作为一种潜隐性病毒,在大部分寄主上不会产生严重的症状[13];第二,ALSV载体具有较高的稳定性,可以在被接种植物的整个生长周期持续存在[16];第三,ALSV的寄主范围广,除了模式植物拟南芥()[17]、本氏烟()[18]外,还可应用于辣椒()[19]、菎诺藜()[20]、豇豆()、黄瓜()、葡萄()和柑橘()等多个不同科植物的基因沉默[21-24]。【本研究切入点】目前,ALSV介导的VIGS体系只在国外多个大豆品种中进行了VIGS的测试[14],发现在国外不同大豆品种中ALSV沉默靶标基因的效率存在显著差异,只有部分品种可以诱导高效的基因沉默。中国是大豆的起源地,大豆品种种类多,但目前还没有利用ALSV诱导的基因沉默体系对中国大豆品种进行沉默效率的评估,到底哪些品种能够利用该病毒载体进行高效的靶标基因沉默仍不清楚。【拟解决的关键问题】本研究基于ALSV病毒载体能够侵染大豆诱导基因沉默的能力,拟利用ALSV病毒载体在中国大豆品种中建立有效的基因沉默体系,通过构建农杆菌介导法接种的ALSV载体,在本氏烟上扩繁病毒,快速分离高浓度的病毒粒子,建立稳定的病毒接种大豆方法,在中国多个大豆品种上建立高效的基因沉默体系,为进一步研究中国大豆品种的基因功能和重要农艺性状基因挖掘提供重要的研究工具。
1 材料与方法
1.1 植物材料和生长环境
大豆种子:南农1138-2由南京农业大学大豆工程改良中心李凯教授提供,齐黄34、安豆203、南农47、祥斗4号、中黄13、山宁29、威廉姆斯82(Williams 82)、山宁16、菏豆12和中黄311等10种大豆品种由南京农业大学植物保护学院叶文武副教授惠赠。
本氏烟种子:由南京农业大学分子植物病毒学实验室保存。
本氏烟和大豆的生长环境均为温度25℃,日长16 h/黑暗8 h的隔离温室。本氏烟基因沉默生长环境为温度22℃,日长8 h/黑暗16 h的隔离温室。
1.2 序列和引物设计
运用primer premier 5.0软件设计引物(表1)。

表1 本研究所使用的引物
划线部分序列为载体pALSV2上的序列The underlined sequence from pALSV2
1.3 ALSV-VIGS载体的构建
1.3.1 农杆菌介导的ALSV载体
参考Huang等[17]方法构建ALSV RNA1和RNA2的侵染性克隆载体pALSV1、pALSV2(图1)和pALSV2-CH42(Chlorata42),pALSV2-CH42用于指示ALSV病毒在本氏烟中的侵染情况。
1.3.2 pALSV2-GmPDS载体构建
1.3.2.1 GmPDS沉默片段克隆 用NCBI(http://www. ncbi.nlm.nih.gov)已公布的拟南芥PDS基因(GenBank登录号为NM_001340908.1)序列进行大豆序列blast,选取大豆PDS基因的3条同源基因,序列号分别为M64704.1、XM_003538489.4和NM_001249840.2。3个同源大豆PDS基因用DNAMAN软件进行多序列比对(图1),选取编码区保守区域1 520—1 846 nt为目的片段。选用(GenBank登录号AW350943.1)作为内参基因。以Williams 82大豆品种的叶片为植物材料,利用天根生化科技(北京)有限公司的总RNA抽提试剂盒提取RNA,具体操作方法依据产品说明书进行。采用Promega反转录试剂盒合成大豆cDNA。以大豆cDNA为模板,按照Phanta Max Super-Fidelity DNA Polymerase反应体系,用特异性引物(表1)进行片段扩增(图1)。扩增反应条件为95℃ 3 min;95℃ 15 s,55℃ 15 s,72℃ 30 s,35个循环;72℃ 5 min。
1.3.2.2 pALSV2-GmPDS重组载体的构建 pALSV2载体经Ⅰ酶切后,用琼脂糖电泳检测(图1),参考生工生物工程(上海)股份有限公司的DnaSpin柱式DNA胶回收试剂盒步骤进行纯化回收,将GmPDS RT-PCR产物按照同样的纯化方法纯化后,通过南京诺唯赞生物科技有限公司的同源重组试剂盒与Ⅰ酶切的pALSV2载体进行同源重组,重组产物转化大肠杆菌DH,挑取阳性克隆,经菌落PCR检测后,进行测序鉴定,验证正确的克隆提取质粒保存。
1.4 农杆菌浸润
将pALSV1、pALSV2、pALSV2-CH42、pALSV2- GmPDS以及病毒基因沉默抑制子P19(tomato bushy stunt virus P19)等载体通过电击法分别转入农杆菌GV3101中,在含有25 mg·L-1利福平和50 mg·L-1卡那霉素的YEP固体培养基上培养,挑取单菌落,在YEP液体培养基中28℃过夜培养,5 000 r/min离心8 min,弃上清液,加入适量的农杆菌浸润缓冲液(10 mmol·L-1MES、10 mmol·L-1MgCl2和150 μmol·L-1乙酰丁香酮)重新悬浮菌体,在28℃恒温培养箱避光放置3 h后,调菌液浓度至OD600=1,将携带pALSV1与pALSV2、pALSV2- GmPDS、pALSV2-CH42的农杆菌等比例混合,同时每个组合中加入等比例的含有P19的农杆菌,注射6—8叶期的本氏烟。由pALSV1和pALSV2构成的病毒命名为ALSV,由pALSV1、pALSV2-AtCH42构成的病毒命名为ALSV﹕CH42,由pALSV1、pALSV2-GmPDS构成的病毒命名为ALSV﹕GmPDS。
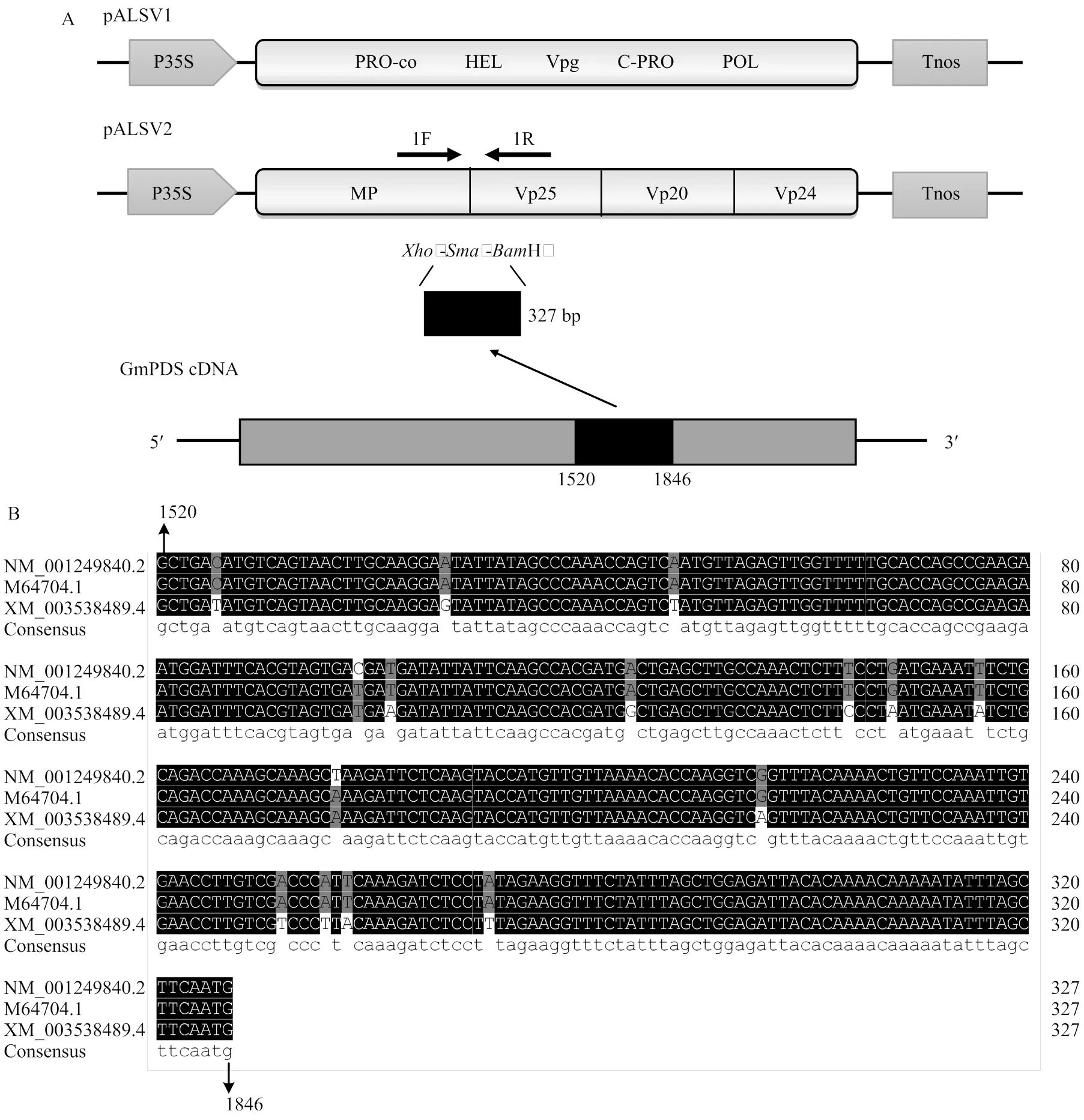
A:pALSV2-GmPDS载体构建示意图。P35S:35S启动子;Tnos:终止子;PRO-co:蛋白酶辅因子;HEL:NTP结合解旋酶;C-PRO:半胱氨酸蛋白酶;POL:RNA聚合酶;MP:42K运动蛋白;Vp25、Vp20和Vp24:衣壳蛋白。B:GmPDS cDNA扩增区域序列比对
1.5 RT-PCR验证ALSV、ALSV﹕CH42、ALSV﹕GmPDS侵染本氏烟
分别对表达pALSV1、pALSV2和P19,pALSV1、pALSV2-CH42和P19,pALSV1、pALSV2-GmPDS和P19的本氏烟提取植物系统叶总RNA,反转录为cDNA,用表1中的相应引物进行PCR扩增,PCR产物经1%琼脂糖电泳检测。
1.6 病毒粒子富集
利用侵染ALSV空载和ALSV﹕GmPDS的本氏烟系统侵染叶进行病毒粒子富集。具体步骤如下(图2):
1)农杆菌浸润17 d后,称取0.4 g系统侵染叶片,在研钵中加入液氮充分研磨成粉末,加入800 μL 0.01 mol·L-1pH=7.5的PB缓冲液,混匀后置冰上孵育30 min;
2)4℃,8 500 r/min离心10 min后取上清,加入相应体积的40% PEG/NaCl溶液(每100 μL上清液加入23 μL 40% PEG/NaCl),混匀后置冰上孵育1 h;
3)4℃,13 000 r/min离心15 min后弃上清,用100 μL 0.01 mol·L-1pH=7.0的PB缓冲液悬浮沉淀,即得到富集的病毒粒子。通过OD260测得病毒粒子浓度,将病毒终浓度调至2 μg·μL-1,4℃保存备用。
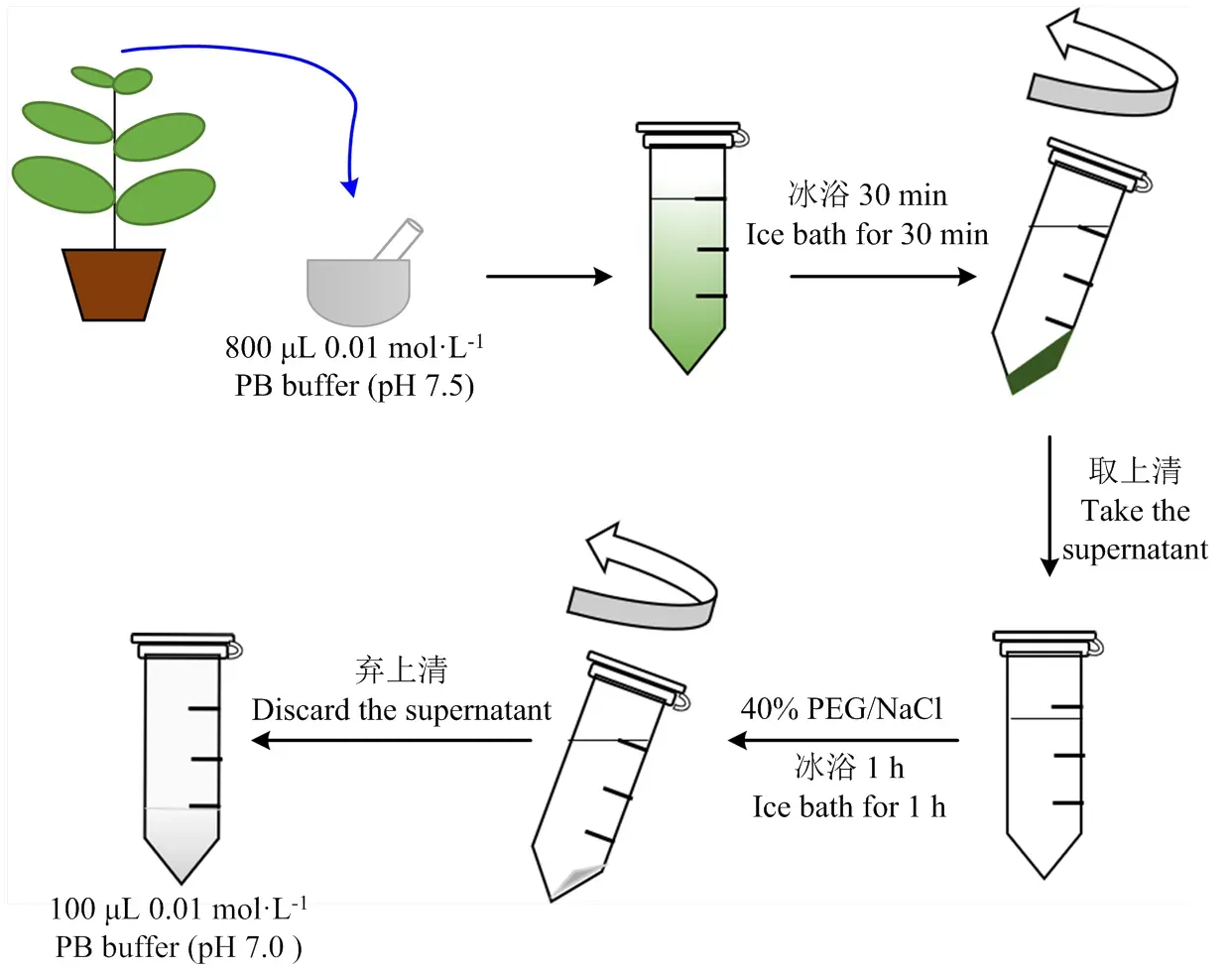
图2 本氏烟中ALSV病毒粒子的提取和富集流程
1.7 ALSV病毒粒子摩擦接种大豆
选择8—10日龄大豆幼苗,将金刚砂均匀撒在大豆叶片上,用移液枪吸取20 μL提取的病毒粒子,用一次性手套均匀涂抹在大豆真叶叶片上,每片真叶摩擦40 μg病毒。
1.8 qRT-PCR检测大豆GmPDS的沉默效率
当接种ALSV﹕GmPDS的大豆系统叶出现白化表型时,采集大豆顶部白化叶提取总RNA,同时用摩擦接种ALSV空载体的大豆系统叶作为对照,按照南京诺唯赞生物科技有限公司的快速反转录试剂盒合成cDNA,再用ChamQ Universal SYBR qPCR Master Mix定量试剂盒进行定量PCR,用作为内参基因,按照表1中的相应引物检测的沉默效率。
2 结果
2.1 农杆菌介导的ALSV侵染性克隆构建及通过侵染本氏烟扩繁病毒
ALSV载体在初始感染阶段病毒RNA积累量少,需要接种到菎诺藜上进行扩增繁殖[18, 25]。为了能够在实验室常用病毒寄主本氏烟中进行ALSV的扩增繁殖,人工合成了ALSV基因组RNA1和RNA2的全长cDNA,将病毒2条基因组分别克隆到pCB301-2x35S- RZ-NOS,构建了农杆菌介导的pALSV1和pALSV2的侵染性克隆载体(图1)[17]。将含有pALSV1、pALSV2和P19载体的农杆菌悬浮液等比例混合后浸润本氏烟,同时用含空载体的农杆菌浸润本氏烟作为阴性对照。在农杆菌接种后17 d,受感染的植株没有出现明显的病毒症状(图3-A);RT-PCR检测结果显示,pALSV1、pALSV2和P19共处理的本氏烟的系统叶可检测到ALSV病毒的表达,表明通过农杆菌介导的ALSV载体不诱导任何病症但可成功侵染本氏烟(图3-B)。
为了测试农杆菌介导的ALSV载体是否能够在本氏烟上诱导基因沉默,首先选取了(),该基因编码叶绿素产生所必需的镁螯合酶复合物的组分之一,作为指示基因被广泛应用于病毒诱导的基因沉默体系研究[26]。在pALSV2载体上插入了的cDNA片段,构建成pALSV2-CH42载体[17]。将携带pALSV1、pALSV2-CH42和P19的农杆菌等比例混合后注射本氏烟,浸润17 d后,pALSV1、pALSV2-CH42和P19共表达的植株系统叶片出现黄化表型,结果表明,构建的ALSV载体能够在本氏烟上诱导有效的基因沉默(图3-C)。进一步测试不加沉默抑制子P19时,将携带pALSV1和pALSV2-CH42农杆菌等比例混合后注射本氏烟能否实现系统侵染。结果显示,在不加入P19时,系统叶并没有出现黄化的表型(图3-C),表明不加基因沉默抑制子P19,ALSV载体不能有效侵染本氏烟。以上研究结果表明构建的ALSV载体可通过农杆菌浸润的方式侵染本氏烟,并能够诱导高效的基因沉默,基因沉默抑制子P19对于农杆菌介导的ALSV侵染本氏烟是必需的。
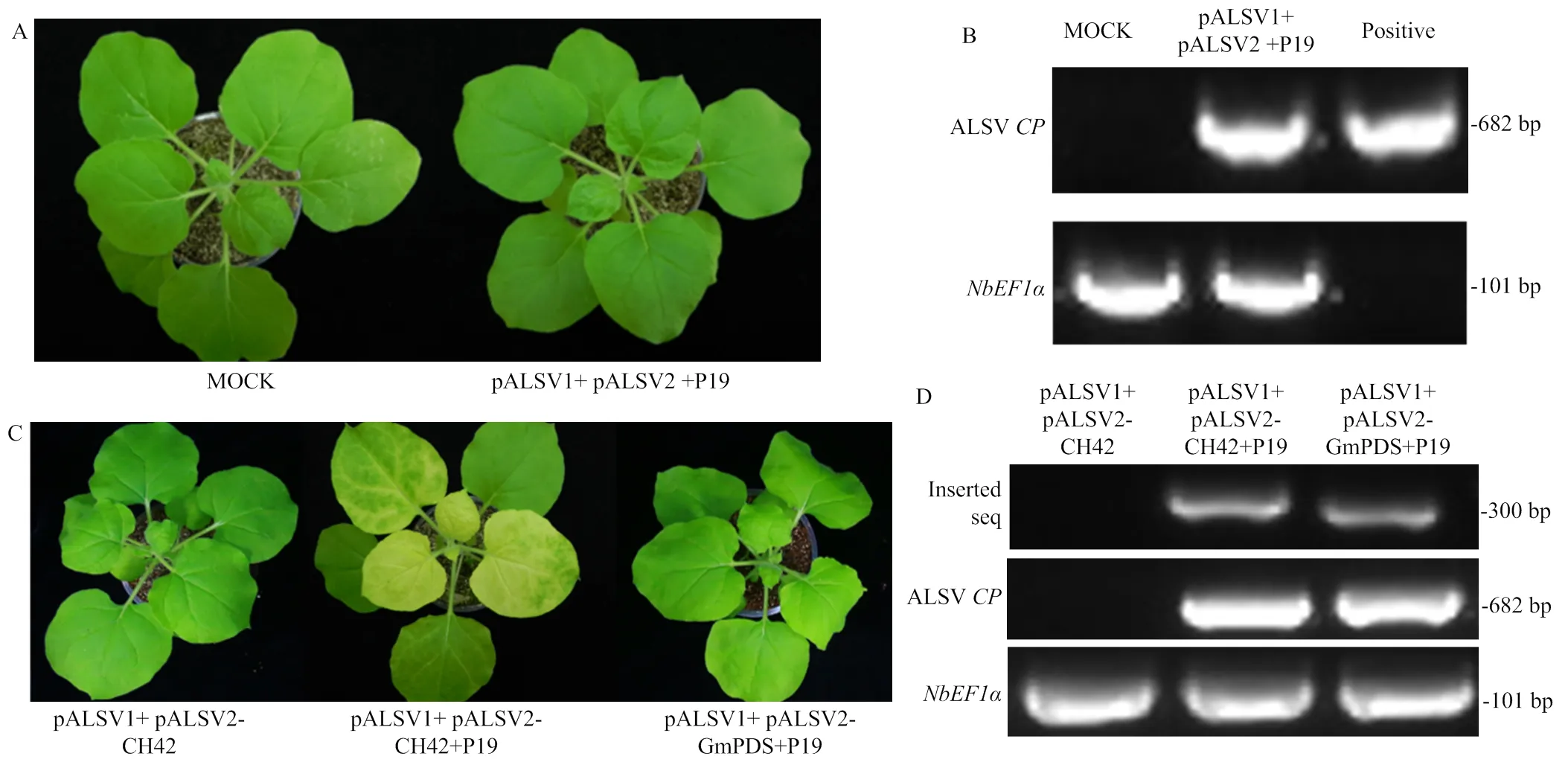
A:农杆菌介导ALSV侵染本氏烟。将含有pALSV1和pALSV2空载体、pALSV2-CH42、pALSV2-GmPDS和P19的农杆菌1﹕1﹕1混合后注射本氏烟,在侵染17 d后,对侵染植株的表型进行拍照。MOCK:空载体对照;P19:基因沉默抑制子;B:RT-PCR检测本氏烟系统叶ALSV CP积累量;C:农杆菌介导的ALSV﹕GmPDS侵染本氏烟以及P19对ALSV侵染本氏烟的作用测定,CH42:叶绿素合成相关基因;D:RT-PCR检测C图ALSV插入片段CH42、GmPDS和CP在本氏烟系统叶中的表达
随后,将327 bp大豆的cDNA片段插入pALSV2,构建pALSV2-GmPDS(图1)。通过pALSV2-GmPDS、pALSV1和P19农杆菌悬浮液等比例混合后注射本氏烟,以pALSV1、pALSV2-CH42和P19农杆菌悬浮液浸润的植株作为阳性对照。待浸润ALSV﹕CH42的植株系统叶黄化后,采集ALSV﹕GmPDS侵染本氏烟植株的系统叶,经RT-PCR验证,ALSV﹕GmPDS本氏烟成功实现系统侵染(图3-D)。
2.2 ALSV载体在南农1138-2大豆品种上的基因沉默体系的建立
为了在大豆上建立基因沉默的体系,通过一种快速提纯病毒的方法,对ALSV空载体和ALSV2﹕GmPDS的病毒粒子进行富集(图2)。将富集后的病毒粒子,摩擦接种到南农1138-2和威廉姆斯82大豆品种的第一轮真叶上,每片真叶摩擦40 μg提纯的病毒粒体。
以提纯的ALSV空载体病毒粒子摩擦接种的大豆为对照组,以ALSV﹕GmPDS病毒粒子摩擦接种的大豆为试验组。结果显示,在南农1138-2大豆品种上接种ALSV﹕GmPDS病毒粒子20 d后,在接种植株第二轮和第三轮以上的真叶上均出现了完全白化的表型,而接种ALSV空载病毒粒子的对照组大豆则没有出现白化(图4-A—图4-C),接种的6棵植物中全部出现白化表型(表2)。为明确ALSV﹕GmPDS在1138-2大豆品种中诱导的沉默效率,采集ALSV空载体大豆系统叶片和ALSV﹕GmPDS诱导白化的叶片,提取总RNA,qRT-PCR检测的表达水平。结果显示,在ALSV﹕GmPDS处理的南农1138-2大豆品种中,与对照组相比,出现白化表型的表达水平显著降低(<0.05)(图4-D)。在威廉姆斯82大豆品种,接种的8棵威廉姆斯82中都没有出现白化表型,qRT-PCR显示并没有被沉默(图4-E和图4-F)。对未出现沉默的植株进行RT-PCR检测,发现ALSV病毒在威廉姆斯82中的病毒积累量非常低或不能侵染该大豆品种(图5)。结果表明,利用ALSV在南农1138-2大豆品种上成功建立了基因沉默的体系(图4)。
2.3 ALSV介导的基因沉默在中国其他9种大豆品种的沉默效率测定
利用ALSV在南农1138-2品种建立基因沉默体系的基础上,进一步选择了国内其他9种大豆品种包括齐黄34、安豆203、南农47、祥斗4号、中黄13、山宁29等进行了沉默效率的测定。通过前面在南农1138-2诱导基因沉默相同的策略对供试大豆进行了ALSV﹕GmPDS病毒接种。结果显示,ALSV﹕GmPDS侵染2周后,在齐黄34、安豆203、南农47、祥斗4号、中黄13、山宁29等大豆品种中均诱导产生了白化的表型(图6),齐黄34中8棵接种植株有3棵出现了白化,安豆203中8棵接种的植株有3棵出现白化,南农47中接种8棵植株有3棵出现白化,祥斗4号的8棵接种植株有4棵出现白化,中黄13中6棵接种植株有4棵出现白化,山宁29中6棵接种植株有4棵出现白化(表2)。经RT-PCR检测,在接种的8棵齐黄34中,3棵出现白化表型的植株中都能检测到病毒,而在其余未出现PDS沉默的植株中,并没有检测到病毒侵染,表明未出现PDS沉默的植株是ALSV病毒未能够成功侵染引起的(图7-A)。此外,在ALSV﹕GmPDS接种的国内大豆品种菏豆12、中黄311、山宁16上,病毒接种后均没有产生白化表型(图6)。经RT-PCR检测后发现ALSV可以在这些大豆品种产生系统侵染,表明ALSV在这些大豆品种中不足以诱导PDS的沉默(图7-B—图7-D)。这些研究结果表明,ALSV介导的基因沉默体系在中国不同大豆品种的上诱导的基因沉默表型有差异,ALSV在一些大豆品种可以实现系统侵染,但是沉默效率存在差异。
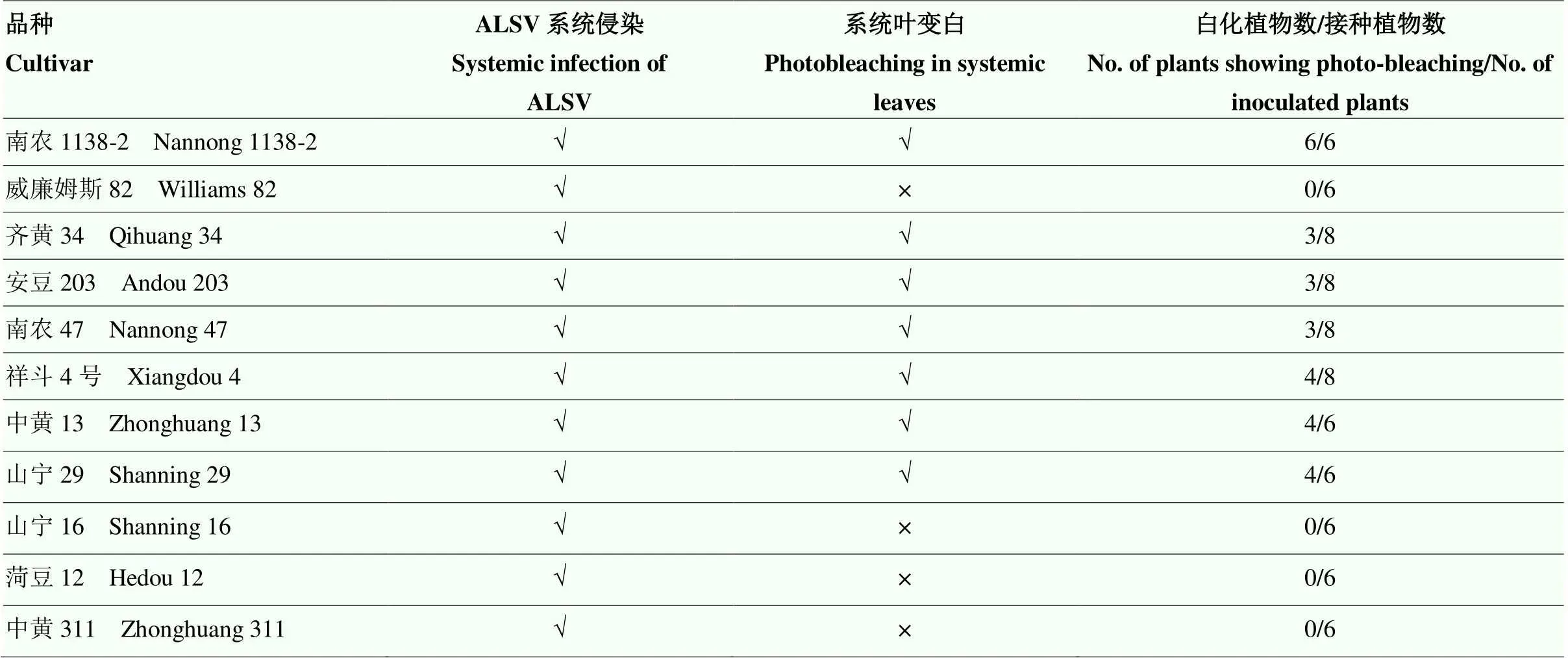
表2 ALSV诱导的基因沉默在11个大豆品种上的沉默效率
√:ALSV能够系统侵染/ALSV﹕GmPDS侵染后能够诱导系统叶变白;×:ALSV不能系统侵染/ALSV﹕GmPDS侵染后不能够导致系统叶变白
√: ALSV can infect systematically/ALSV:GmPDS can cause photo-bleaching; ×: ALSV can’t infect systematically/ALSV:GmPDS can’t cause photo-bleaching

A:ALSV空载体病毒粒体接种南农1138-2 20 d后的表型;B:ALSV﹕GmPDS病毒粒体接种南农1138-2 20 d后的表型;C:A、B图中对应南农1138-2系统叶GmPDS沉默表型,左边为接种ALSV空载对照,右边为接种ALSV﹕GmPDS上部白化叶片;D:qRT-PCR检测ALSV空载体和ALSV﹕GmPDS病毒粒体处理的南农1138-2大豆品种系统叶中PDS基因的沉默效率(n=3,**:p<0.01);E:ALSV﹕GmPDS接种威廉姆斯82大豆品种20 d后的表型;F:qRT-PCR检测ALSV空载体和ALSV﹕GmPDS病毒粒体处理的威廉姆斯82大豆品种系统叶中PDS基因的沉默效率(n=3,ns:p>0.05)
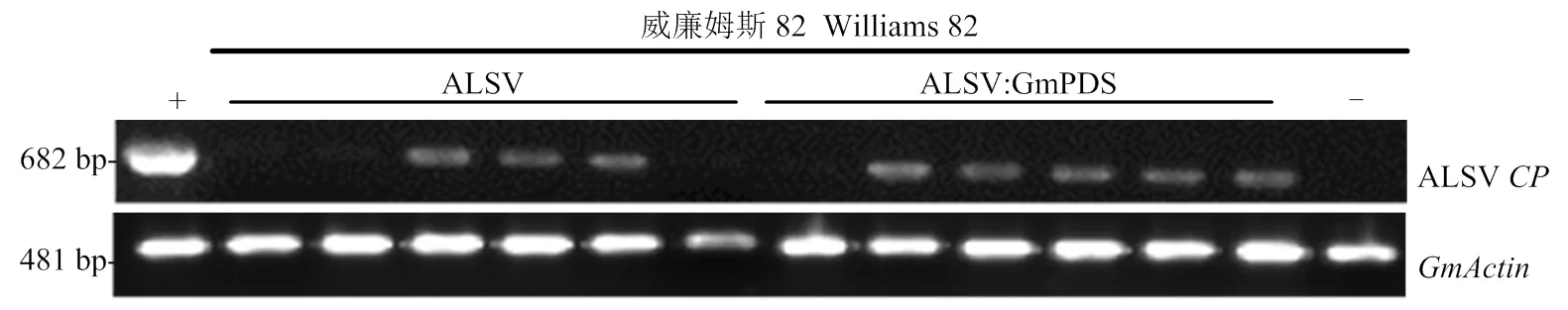
+:用已白化的南农1138-2系统叶cDNA为阳性对照;—:健康大豆cDNA为阴性对照

ALSV﹕GmPDS病毒纯化粒体接种国内其他九种不同大豆品种20 d后的表型。ALSV﹕GmPDS在齐黄34、南农47、祥斗4号、中黄13、山宁29以及安豆203系统叶诱导白化表型;山宁16、菏豆12以及中黄311系统叶未能诱导出现白化表型
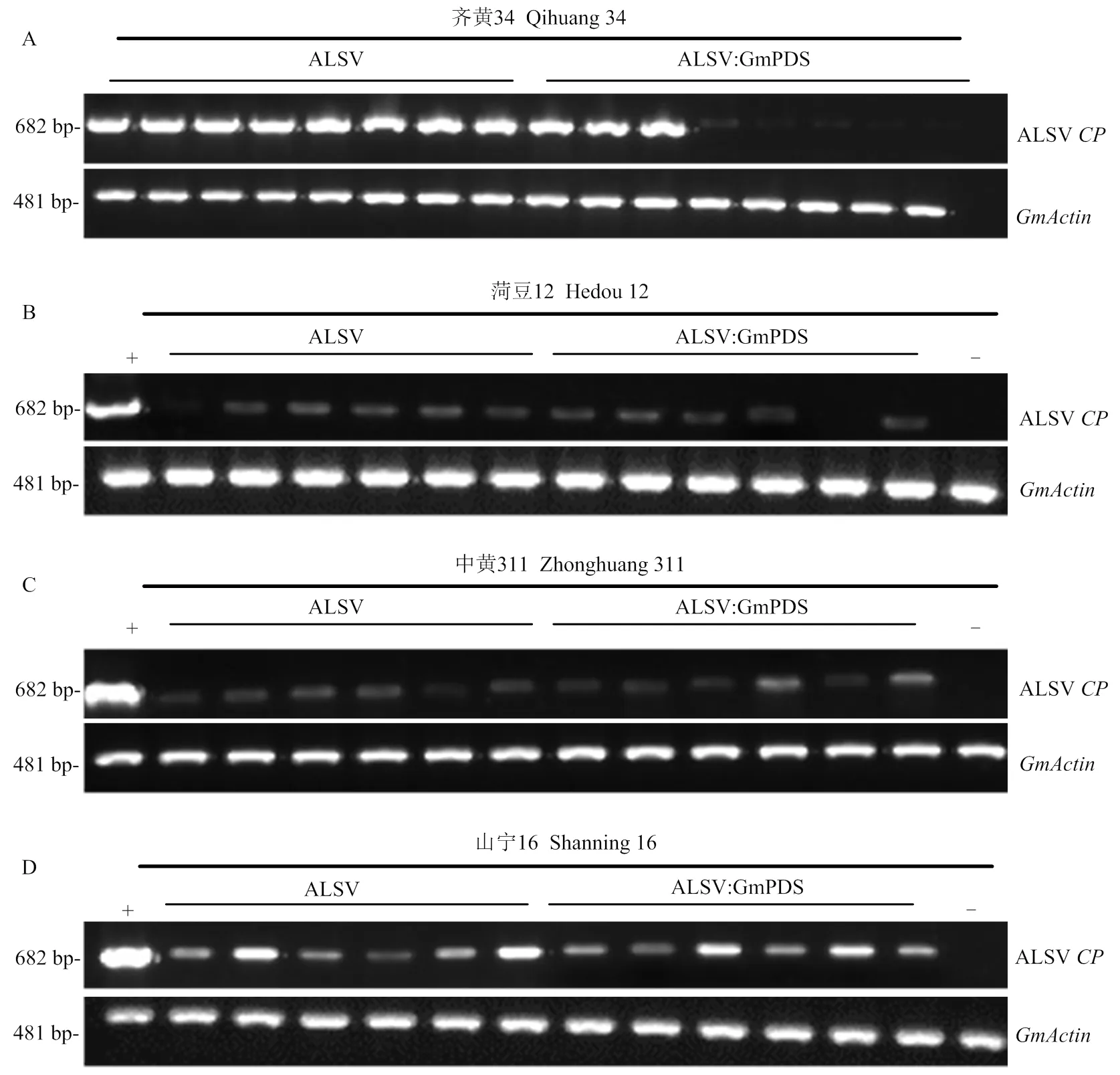
A:接种ALSV空载体的齐黄34在8棵植物系统叶都能够检测到病毒CP RNA的积累,接种ALSV﹕GmPDS的齐黄34在8棵植物有3棵发生沉默的植物系统叶能检测到CP;B:ALSV空载体和ALSV﹕GmPDS接种的菏豆12检测不到或只能检测到很低的CP RNA积累量;C:ALSV空载体和ALSV﹕GmPDS接种的中黄311只能检测到很低的CP积累量;D:ALSV空载体和ALSV﹕GmPDS接种的山宁16只能检测到很低的CP积累量。+:用已白化的南农1138-2系统叶cDNA为阳性对照,—:健康大豆cDNA为阴性对照
3 讨论
3.1 利用ALSV载体在中国多种大豆品种中成功建立有效的基因沉默体系
目前广泛应用于大豆基因沉默的载体主要是BPMV,该载体在大豆上应用方法较为复杂且会导致植物产生症状[27-29]。原始的ALSV需摩擦到菎诺藜上,并且要进行基因枪轰击到大豆上进行基因沉默,这一操作对一般的实验室较难熟练掌握。本研究将ALSV改造为农杆菌介导的病毒载体,极大地方便了病毒诱导基因沉默的操作。基于ALSV病毒在植物中积累量少的特点,创立了富集本氏烟中扩繁的ALSV病毒粒子的方法,用富集的病毒粒子接种大豆。改进的方法在大豆上诱导的基因沉默效果稳定,可重复性好。这些改进显著简化了ALSV用于大豆基因沉默和基因功能的过程。本研究发现使用基因沉默抑制子P19对于农杆菌介导的ALSV在本氏烟上的侵染是必需的,ALSV在植物中的积累量较低,基因沉默抑制子P19的加入显著促进了ALSV在本氏烟的侵染和积累,这为大量富集病毒粒子用于后续有效接种大豆创造了有利条件。基于此,本研究利用ALSV载体在南农1138-2、安豆203、齐黄34等中国多种大豆品种中建立了有效的基因沉默体系。
3.2 ALSV在不同大豆品种中诱导基因的沉默效率存在差异
发现ALSV-VIGS载体在不同大豆品种中沉默基因的效率存在显著差异,这种差异可能是由于大豆的基因型决定的。对于Williams 82、菏豆12、中黄311、山宁16这4个品种,ALSV虽然可以系统侵染,但是没有出现白化表型,RT-PCR结果表明,这些大豆品种系统叶中,病毒的积累量与对照组(1138-2品种)相比显著较低,这暗示ALSV并不能很好地侵染一些大豆品种,或ALSV在这些品种中较难诱导基因沉默。之前报道中,ALSV在不同的国外大豆品种上诱导基因沉默效率也有较大的差异[14]。此外,为了排除PDS在不同大豆品种中沉默效率存在差异是由沉默片段的保守性差异造成的,本研究扩增了11个大豆品种的沉默区域进行测序,测序结果显示不同大豆品种之间并无明显差异。对于能够产生白化表型的大豆品种,沉默效率也有差异,其中比较敏感的南农1138-2品种,沉默效率可以达到100%,而对于其他沉默效率稍低的大豆品种,未来可以对这个体系进行进一步优化,例如通过提高病毒接种浓度和接种量、尝试不同的沉默片段长度或调整植物生长温度[30],以提高ALSV在这些大豆品种的基因沉默效率。因此,利用该病毒在新的大豆品种中进行基因沉默和功能研究时,应先对供试大豆品种进行基因沉默效率的测定。
4 结论
将ALSV载体改造为农杆菌浸润的病毒载体,以常用的本氏烟作为繁殖寄主,根据ALSV病毒在植物中积累量少的特点,创造性地采用了富集本氏烟中扩繁的ALSV病毒粒子的方法,创新了基于ALSV载体诱导的大豆基因沉默体系,并应用该体系在中国10种大豆品种上进行了沉默测试,明确了该病毒载体在国内不同大豆品种上的沉默效率。
[1] 吴汶珊. 中国大豆对外贸易的发展现状及对策. 大陆桥视野, 2021(6): 88-89.
WU W S. Development status and countermeasures of Chinese soybean foreign trade. New Silk Road Horizon, 2021(6): 88-89. (in Chinese)
[2] 吴曰程, 王玉斌. 中国转基因大豆进口及其影响分析. 大豆科学, 2019, 38(4): 635-643.
WU Y C, WANG Y B. Import of transgenic soybean in China and its impact analysis. Soybean Science, 2019, 38(4): 635-643. (in Chinese)
[3] 刘晓彬, 刘娜, 李福宽, 吴立柱, 张洁, 王冬梅. TRV介导的大豆基因瞬时沉默体系的建立. 中国农业科学, 2015, 48(12): 2479-2486.
LIU X B, LIU N, LI F K, WU L Z, ZHANG J, WANG D M. Establishment of TRV-mediated transient gene-silencing system in soybean.Scientia Agricultura Sinica, 2015, 48(12): 2479-2486. (in Chinese)
[4] 王爽, 郭兵福, 郭勇, 张丽娟, 金龙国, 杨慧, 邱丽娟. 病毒诱导的基因沉默(VIGS)技术及其在大豆基因功能研究和育种中的应用潜力. 大豆科学, 2016, 35(4): 536-540.
WANG S, GUO B F, GUO Y, ZHANG L J, JIN L G, YANG H, QIU L J. Virus-induced gene silencing and its usage in soybean functional genomics. Soybean Science, 2016, 35(4): 536-540. (in Chinese)
[5] 李敏, 关博文, 杨学, 陈庆山, 张睿, 武小霞, 金慧, 吴玉娥. 大豆种质资源遗传多样性分析. 农业科技通讯, 2021(11): 4-8.
LI M, GUAN B W, YANG X, CHEN Q S, ZHANG R, WU X X, JIN H, WU Y E. Genetic diversity analysis of soybean germplasm resources. Bulletin of Agricultural Science and Technology, 2021(11):4-8. (in Chinese)
[6] LANGE M, YELLINA A L, ORASHAKOVA S, BECKER A. Virus-induced gene silencing (VIGS) in plants: an overview of target species and the virus-derived vector systems. Methods in Molecular Biology, 2013, 975: 1-14.
[7] RAMEGOWDA V, MYSORE K S, SENTHIL-KUMAR M. Virus-induced gene silencing is a versatile tool for unraveling the functional relevance of multiple abiotic-stress-responsive genes in crop plants. Frontiers of Plant Science, 2014, 5: 323.
[8] BURCH-SMITH T M, ANDERSON J C, MARTIN G B, DINESH-KUMAR S P. Application and advantages of virus-induced gene silencing for gene function studies in plants. The Plant Journal, 2004, 39: 734-746.
[9] KANAZAWA A, INABA J I, SHIMURA H, OTAGAKI S, TSUKAHARA S, MATSUZAWA A, KIM B M, GOTO K, MASUTA C. Virus-mediated efficient induction of epigenetic modifications of endogenous genes with phenotypic changes in plants. The Plant Journal, 2011, 65(1): 156-168.
[10] LU R, MARTIN-HERNANDEZ A M, PEART J R, MALCUIT I, BAULCOMBE D C. Virus-induced gene silencing in plants. Methods, 2003, 30(4): 296-303.
[11] ZHANG C, GHABRIAL S A. Development of bean pod mottle virus-based vectors for stable protein expression and sequence- specific virus-induced gene silencing in soybean. Virology, 2006, 344(2): 401-411.
[12] KACHROO A, GHABRIAL S. Virus-induced gene silencing in soybean. Methods in Molecular Biology, 2012, 894: 287-297.
[13] IGARASHI A, YAMAGATA K, SUGAI T, TAKAHASHI Y, SUGAWARA E, TAMURA A, YAEGASHI H, YAMAGISHI N, TAKAHASHI T, ISOGAI M, TAKAHASHI H YOSHIKAWA N. Apple latent spherical virus vectors for reliable and effective virus-induced gene silencing among a broad range of plants including tobacco, tomato,, cucurbits, and legumes. Virology, 2009, 386(2): 407-416.
[14] GEDLING C R, ALI E M, GUNADI A, FINER J J, XIE K, LIU Y, YOSHIKAWA N, QU F DORRANCE A E. Improved apple latent spherical virus-induced gene silencing in multiple soybean genotypes through direct inoculation of agro-infiltratedextract. Plant Methods, 2018, 14: 19.
[15] LI C, YOSHIKAWA N, TAKAHASHI T, ITO T, YOSHIDA K, KOGANEZAWA H. Nucleotide sequence and genome organization of apple latent spherical virus: a new virus classified into the family comoviridae. Journal of General Virology, 2000, 81(2): 541-547.
[16] YAMAGISHI N, SASAKI S, YAMAGATA K, KOMORI S, NAGASE M, WADA M, YAMAMOTO T, YOSHIKAWA N. Promotion of flowering and reduction of a generation time in apple seedlings by ectopical expression of thegene using the apple latent spherical virus vector. Plant Molecular Biology, 2011, 75(1/2): 193-204.
[17] HUANG Y, HONG H, XU M, YAN J, DAI J, WU J, FENG Z, ZHU M, ZHANG Z, YUAN X, DING X, TAO X. Developmentally regulatedsusceptibility to tomato spotted wilt virus infection. Molecular Plant Pathology, 2020, 21(7): 985-998.
[18] KON T, YOSHIKAWA N. Induction and maintenance of DNA methylation in plant promoter sequences by apple latent spherical virus-induced transcriptional gene silencing. Frontiers in Microbiology, 2014, 5: 595.
[19] LI C, HIRANO H, KASAJIMA I, YAMAGISHI N, YOSHIKAWA N. Virus-induced gene silencing in chili pepper by apple latent spherical virus vector. Journal of Virological Methods, 2019, 273: 113711.
[20] OGATA T, TOYOSHIMA M, YAMAMIZO-ODA C, KOBAYASHI Y, FUJII K, TANAKA K, TANAKA T, MIZUKOSHI H, YASUI Y, NAGATOSHI Y, YOSHIKAWA N, FUJITA Y. Virus-mediated transient expression techniques enable functional genomics studies and modulations of betalain biosynthesis and plant height in Quinoa. Frontiers of Plant Science, 2021, 12: 643499.
[21] YAMAGISHI N, YOSHIKAWA N. Highly efficient virus-induced gene silencing in apple and soybean by apple latent spherical virus vector and biolistic inoculation. Methods in Molecular Biology, 2013, 975: 167-181.
[22] 高鹏飞, 席飞虎, 张泽宇, 胡凯强, 陈凯, 魏文桃, 丁家治, 顾连峰. 植物VIGS技术及其在林业科学中的研究进展. 生物技术通报, 2021, 37(5): 141-153.
GAO P F, XI F H, ZHANG Z Y, HU K Q, CHEN K, WEI W T, DING J Z, GU L F. Research progress of plant VIGS technology and its application in forestry science. Biotechnology Bulletin, 2021, 37(5): 141-153. (in Chinese)
[23] WERNER R C, DUGÉ DE BERNONVILLE T, GLÉVAREC G, LANOUE A, OUDIN A, PICHON O, ST-PIERRE B, COURDAVAULT V, BESSEAU S. ALSV-based virus-induced gene silencing in apple tree (L.). Methods in Molecular Biology, 2020, 2172: 183-197.
[24] 周彦. 果树病毒载体研究进展. 中国农业科学, 2014, 47(6): 1119-1127.
ZHOU Y. Progresses in study of virus-based vectors of fruit trees. Scientia Agricultura Sinica, 2014, 47(6): 1119-1127. (in Chinese)
[25] YAEGASHI H, YAMATSUTA T, TAKAHASHI T, LI C, ISOGAI M, KOBORI T, OHKI S, YOSHIKAWA N. Characterization of virus-induced gene silencing in tobacco plants infected with apple latent spherical virus. Archives of Virology, 2007, 152(10): 1839-1849.
[26] BURCH-SMITH T M, SCHIFF M, LIU Y, DINESH-KUMAR S P. Efficient virus-induced gene silencing in. Plant Physiology, 2006, 142(1): 21-27.
[27] XU H Y, ZHANG C, LI Z C, WANG Z R, JIANG X X, SHI Y F, TIAN S N, BRAUN E, MEI Y, QIU W L, LI S, WANG B, XU J, NAVARRE D, REN D, CHENG N, NAKATA P A, GRAHAM M A, WHITHAM S A, LIU J Z. The MAPK kinase kinaseregulates cell death and defense responses.Plant Physiology, 2018, 178(2): 907-922.
[28] TIAN S N, LIU D D, ZHONG C L, XU H Y, YANG S, FANG Y, RAN J, LIU J Z. Silencingenhances the susceptibility of soybean to bacterial pathogen through attenuating the activation ofsignaling pathway. Plant Science, 2020, 292: 110386.
[29] LI C, LI K, ZHENG M, LIU X, DING X, GAI J, YANG S., a cytosolic 6-phosphogluconate dehydrogenase, enhanced tolerance to phosphate starvation by improving root system development and modifying the antioxidant system in soybean. Frontiers in Plant Science, 2021, 12: 704983.
[30] KIM K H, LIM S, KANG Y J, YOON M Y, NAM M, JUN T H, SEO M J, BAEK S B, LEE J H, MOON J K, LEE S H, LEE S H, LIM H S, MOON J S, PARK C H. Optimization of a virus-induced gene silencing system with soybean yellow common mosaic virus for gene function studies in soybeans. Plant Pathology Journal, 2016, 32(2): 112-122.
Establishment of ALSV-Induced Gene Silencing in Chinese Soybean Cultivars
DONG YongXin1, WEI QiWei1,2, HONG Hao1, HUANG Ying1, ZHAO YanXiao1, FENG MingFeng1, DOU DaoLong1, XU Yi1, TAO XiaoRong1
1College of Plant Protection, Nanjing Agricultural University/Key Laboratory of Soybean Disease and Pest Control of Ministry of Agriculture and Rural Affairs, Nanjing 210095;2Dazhou Academy of Agricultural Sciences in Sichuan Province, Dazhou 635000, Sichuan
【】 The establishment of apple latent spherical virus (ALSV)-induced gene silencing on domestic soybean cultivars will offer a simple, time-saving and operable system for gene function and genetic breeding of soybean cultivars in China.【】The infectious clones of ALSV which was used to infect plant through agro-infiltration was constructed. A 327 bp cDNA fragment of phytoene desaturase () cDNA was amplified from soybean cultivar Williams 82 and inserted into the pALSV2 vector.The pALSV1 and pALSV2-were used to co-infectplant throughAgroinfiltration, and the ALSV virus particle was purified from systemic infected leaves ofplants at 17 days post infiltration (dpi). The purified virions were mechanically inoculated onto the first-round true leaves of soybean and photobleaching phenotype on the upper non-inoculated leaves was monitored from 10 to 30 dpi. Using the empty ALSV vector as a control, the gene expression levels of ALSV coat protein () gene andin ALSV:GmPDS infected soybean were examined by RT-PCR and quantitative RT-PCR analysis, respectively. 【】At 20 dpi, soybean cultivar Nannong 1138-2 inoculated with ALSV:GmPDS showed photobleaching phenotype in the systemic infected leaves, however Williams 82 did not show gene silencing phenotype of PDS in the systemic infected leaves. The results of qRT-PCR confirmed that the RNA expression level ofin Nannong 1138-2 was significantly reduced but ALSV:was unable to silencein Williams 82. By using the same method, the gene silencing efficiency of other 9 domestic soybean cultivars mediated by ALSV:was examined. Soybean cultivars including Nannong 47, Andou 203, Xiangdou 4, Zhonghuang 13, Shanning 29 and Qihuang 34 all displayed photobleaching phenotype in plant systemic infected leaves after inoculation with ALSV:GmPDS, whilewas not silenced in Hedou 12, Zhonghuang 311 and Shanning 16.【】In this study, an-mediated ALSV viral VIGS vector was constructed. ALSV virions was propagated and purified fromplant. The purified virions were rub inoculated onto the first-round true leaves of soybean. Finally, an efficient gene silencing system was established on a number of domestic soybean cultivars in China.
virus-induced gene silencing (VIGS); soybean; apple latent spherical virus; phytoene desaturase ()

10.3864/j.issn.0578-1752.2022.09.002
2021-11-23;
2022-01-10
国家自然科学基金杰出青年基金(31925032)、海南省重大科技计划(ZDKJ2021007)
董永鑫,E-mail:2019102035@njau.edu.cn。卫其巍,E-mail:675815483@qq.com。董永鑫与卫其巍为同等贡献作者。通信作者陶小荣,E-mail:taoxiaorong@njau.edu.cn。通信作者徐毅,E-mail:xuyiqdpd@njau.edu.cn。通信作者窦道龙,E-mail:ddou@njau.edu.cn
(责任编辑 李莉)

