微生物天然产物生物及化学信息学最新进展
王梦源 常珊珊 解云英
(中国医学科学院 北京协和医学院 医药生物技术研究所 药物合成生物学重点实验室,北京 100050)
微生物天然产物一直是药物,尤其是抗感染药物开发的重要来源[1]。但随着微生物天然产物绝对数量的增多,重复发现越来越严重,如何去除重复、更高效地发现新结构天然产物成为微生物天然产物研究的一个关键问题。
通过基因组测序发现微生物还蕴藏着大量“隐形”生物合成基因簇[2],这表明其仍是新结构天然产物的重要来源。得益于测序成本的快速下降、各类分析仪器的普及以及人工智能的应用,天然产物研究领域进入了一个全新的模式。基于生物信息学[3]和化学信息学[4]而建立起的基因组挖掘技术正在成为微生物研究的主要方法[5-6]。过去10年间,天然产物相关信息学研究一直处于加速发展阶段,每年都会发布大量数据库、算法及工具[7-9],及时了解并使用这些数据库和工具对于微生物天然产物研究来说至关重要,鉴于此,本文对近两年来天然产物研究领域,新开发或更新的各种生物信息学及化学信息学工具进行了综述,以便研究者能够及时了解并选择性应用这些工具,以提高新化合物的发现效率。
1 生物信息学工具及数据库
自从天蓝色链霉菌中发现编码天然产物合成酶的基因成簇排列以来[10],生物信息学对于微生物天然产物研究就变得越来越重要,早期的天然产物生物信息学主要侧重于生物合成基因簇的识别,随着基因组数据指数级的增长,逐渐转向多个基因簇的比较分析。同时,各种类型的生物合成基因簇数据库建立,进一步提高了比较分析的效率。
1.1 生物合成基因簇识别工具
从基因组中识别次级代谢产物生物合成基因簇是天然产物基因组挖掘的基础。AntiSMASH是目前微生物天然产物研究领域使用率最高的次级代谢产物生物合成基因簇(BGCs)分析工具,它是一种基于分布型隐马尔可夫模型(pHMM)数据库的BCGs识别算法。自2011年发布以来,antiSMASH不断进行更新[11-16],目前已经更新到6.1版本,能够预测细菌、真菌和植物基因组中50余类别的生物合成基因簇,并可在基因簇水平上,通过内置的ClusterBlast算法与“生物合成基因簇最小信息”(MiBIG) 数据库[17]及AntiSMASH-DB[18]数据库中的基因簇进行比较,分析基因簇的功能及新颖性。
PRISM4[19]是另一个基因簇识别和产物结构预测工具,它在产物结构预测方面要强于antiSMASH,而且还具有活性预测功能,但其只能分析细菌基因簇。以上2种工具都是基于蛋白相似性来识别生物合成基因簇,因此它们都不能预测pHMM数据库中不存在的、非经典的生物合成基因簇。为了弥补这一不足,近来还开发了基于进化的BGCs识别算法,如针对古菌和细菌的EvoMining算法[20],针对真菌的CO-OCCUR算法[21],基于机器学习和模式识别预测核糖体肽(RiPP)BGCs的RRE-finder[22]和DecRippter[23]算法。除此之外,基于耐药基因的活性靶向基因簇分析工具抗生素耐药靶标搜寻器(ARTS)近来也进行了更新[24],将分析范围从原来的放线菌门扩展到整个细菌界以及宏基因组数据。
1.2 生物合成基因簇比较工具
随着基因组测序成本的大幅下降,人们可以轻易获得大量基因组数据,为了能够比较成千上百个生物合成基因簇的异同,科研人员开发了生物合成基因簇分析比较工具。BiSCAPE/COROSON是第一个可以对非公开的、内部基因组数据进行生物合成基因簇相似性分析的工具[25],它以antiSMASH的分析结果为输入文件,根据基因簇的相似性将基因簇聚合为不同的家族(GCFs),进一步通过与MiBIG数据库比较分析基因簇或基因簇家族的新颖性,并通过内置的COROSON算法进行家族内基因簇多样性分析。2021年BiSCAPE/COROSON开发团队又发布了一个适合百万级别BGCs相似性分析的算法BiGSLICE[26],并在此基础上建立生物合成基因簇家族数据库BiG-FAM[27],该数据库目前包括120余万个BGCs同源比较结果,而且BiG-FAM数据库提供了在线浏览和搜索功能,不但可以浏览特定类型的BGC在不同微生物中的分布,还可以快速地将用户提供的BGCs在数据库中进行定位,以分析其新颖性或与其他生物合成基因簇的关系。
1.3 生物合成基因簇数据库
“生物合成基因簇最小信息”数据库(MiBIG)是目前微生物天然产物研究中应用最广泛的数据库之一,它主要收录经实验验证的生物合成基因簇数据,目前已更新到第二版,包括2050个生物合成基因簇及其相关信息[17]。MiBIG数据不但可以提供在线检索功能,而且还提供了多种格式的下载版本,可以方便地将其整合入其他微生物天然产物分析流程中,目前MiBIG已整合入antiSMASH、BiG-SCAPE等多种天然产物分析工具中。AntiSMASH-DB是antiSMASH团队发布的一个高质量预测合成基因簇数据库,最新发布的3.0版本包括来源于388个古菌、25236个细菌以及177个真菌基因组的147517个BGCs[18]。综合生物合成基因簇合集(IMG-ABC)是联合基因组研究所基于其微生物基因组平台而建立的综合生物合成基因簇数据库,近来发布了5.0版本,不但包括基于antiSMASH V5预测的30余万个BGCs,而且还加入了1285个实验验证的BGCs[28]。Prospect是2021年新发布的一个专门针对真菌生物合成基因簇的数据库,包含来自1037株真菌基因组的3万余个BGCs,为真菌来源天然产物的基因组挖掘提供了便利[29]。
2 化学信息学工具及数据库
从复杂代谢产物中快速鉴定目标分子结构一直是天然产物发现过程中极具挑战性的工作,近年来随着各类分析仪器的普及以及与之相应的数据分析处理工具和各类数据库的不断开发和建立,天然产物的鉴定效率得到了极大的提高。
2.1 基于质谱的代谢产物分析工具
质谱因其高灵敏度及较高的普及率已成为研究复杂代谢产物的主要方法,近来质谱数据的处理和分析方法发展非常迅速。首先是质谱数据处理软件MZmine进行了升级,发布了3.0版[30],与2.0版相比在批处理能力上有了很大提升,而且针对不同类型质谱仪采集的数据提供了相应的默认参数,使数据处理过程更加友好。其次是目前天然产物研究领域最流行的质谱分析平台—全球天然产物社交分子网络(GNPS)[31]平台更新和整合了多个质谱排重和注释工具。GNPS主要功能是分子网络分析,原理是结构相似的化合物可以产生相似的质谱碎片离子,分子网络分析算法可以将其聚集成簇,同时,因整合了实验及理论质谱数据库,在分析的同时可以部分实现化合物的排重及分类。除经典分子网络之外,GNPS平台近来还发布了:①特征分子网络(FBMN)分析流程,不但可以进行定量分析,而且可以区分经典分子网络无法分辨的同分异构体[32];②Moldiscovery分析流程,通过理论质谱库搜索可以对2000 Da以下的各类结构分子进行排重和注释[33],Moldiscovery算法可以看做是之前Dereplicator+算法的升级;③CycloNovo分析流程,CycloNovo是一种基于德布莱英图(de Bruijn graphs)的环肽从头解析算法[34],可以从复杂质谱数据中特异识别环肽类化合物的质谱,并进一步利用分子网络进行相似性分析,或利用Dereplicator/VarQuest进行排重分析;④SIRIUS分析流程,SIRIUS是一个致力于质谱从头解析的软件[35],通过高分辨质谱同位素分布以及“碎片树”预测分子结构,不但可以进行理论质谱库搜索,而且还可以预测数据库中不存在的新分子结构[36]或结构类别[37]。目前SIRIUS分析流程已整合入GNPS平台,可以基于GNPS平台进行分析,也可以独立使用。除此之外,非核糖体肽分析平台(NORINE)近来发布了一个专门针对肽类化合物的在线排重工具NRPro[38],经实测,它是目前准确度最高的理论质谱搜索工具,但只能接受单个化合物的MS/MS数据,为了便于从LC-MS/MS数据中提取单个化合物的数据,本实验室开发了一个在线工具MS/MS Extraction(http://www.npba-xielab.com:8501/),可以批量提取单个目标化合物的MS/MS数据。
2.2 基于核磁共振(NMR)的代谢产物分析工具
NMR分析一直是新结构天然产物确证的金标准,最近在NMR图谱自动分析和数据库建设方面也有了一些突破性进展。SMART 2.1是一个基于卷积神经网络训练的NMR注释算法,可以由1H-13C HSQC图谱自动生成可能的化学结构[39]。DP4-AI可以自动处理和注释13C 和1H NMR原始数据[40]。天然产物核磁共振数据库(NP-MRD)[41]是NIH资助建立的一个开源天然产物数据,自2020年建立来,快速成为世界最大的天然产物核磁数据库,目前已有超过4万个天然产物的NMR数据,超过817000个核磁共振谱(包括实验、模拟及预测数据),支持浏览、检索、下载和上传。
2.3 微生物天然产物数据库
天然产物数据库对天然产物发现和排重至关重要。因此,天然产物数据库的建设一直伴随着天然产物的整个研究过程。据统计,自2000年以来共建立了120余个各种类型的天然产物数据库[42]。其中,含有微生物天然产物数据的有11个[7],近来新建立或更新的有4个,即NP Atlas[43]、Streptome-DB[44]、NORINE[45]和COCONUT[46]。NP Atlas全称the Natural Product Atlas,是2019年新建立的专门针对微生物天然产物的数据库,并于2021年发布了2.0版,包括3万余个化合物,更新后的数据库添加了产生菌完整的分类单元描述,可以非常方便地检索和浏览不同分类地位微生物的天然产物产生情况;Streptome-DB是一个专门收集链霉菌来源天然产物的数据库,目前发布了3.0版本,包括约2500个化合物;NORINE数据库是一个专门的非核糖体肽类化合物数据库,更新后的数据库包括1739个化合物[45];COCONUT全称the COlleCtion of Open Natural ProdUcTs[46],是汇总目前所有开源、可用的天然产物数据库而建立的一个非冗余、可检索的在线数据库,它也是使用MongoDB作为存储管理系统的第一个大型化学数据库,目前包括动植物、真菌、细菌等来源的40余万个天然产物化学结构。以上所综述的近两年发布或更新的微生物天然产物生物和化学信息学工具汇总于表1中。
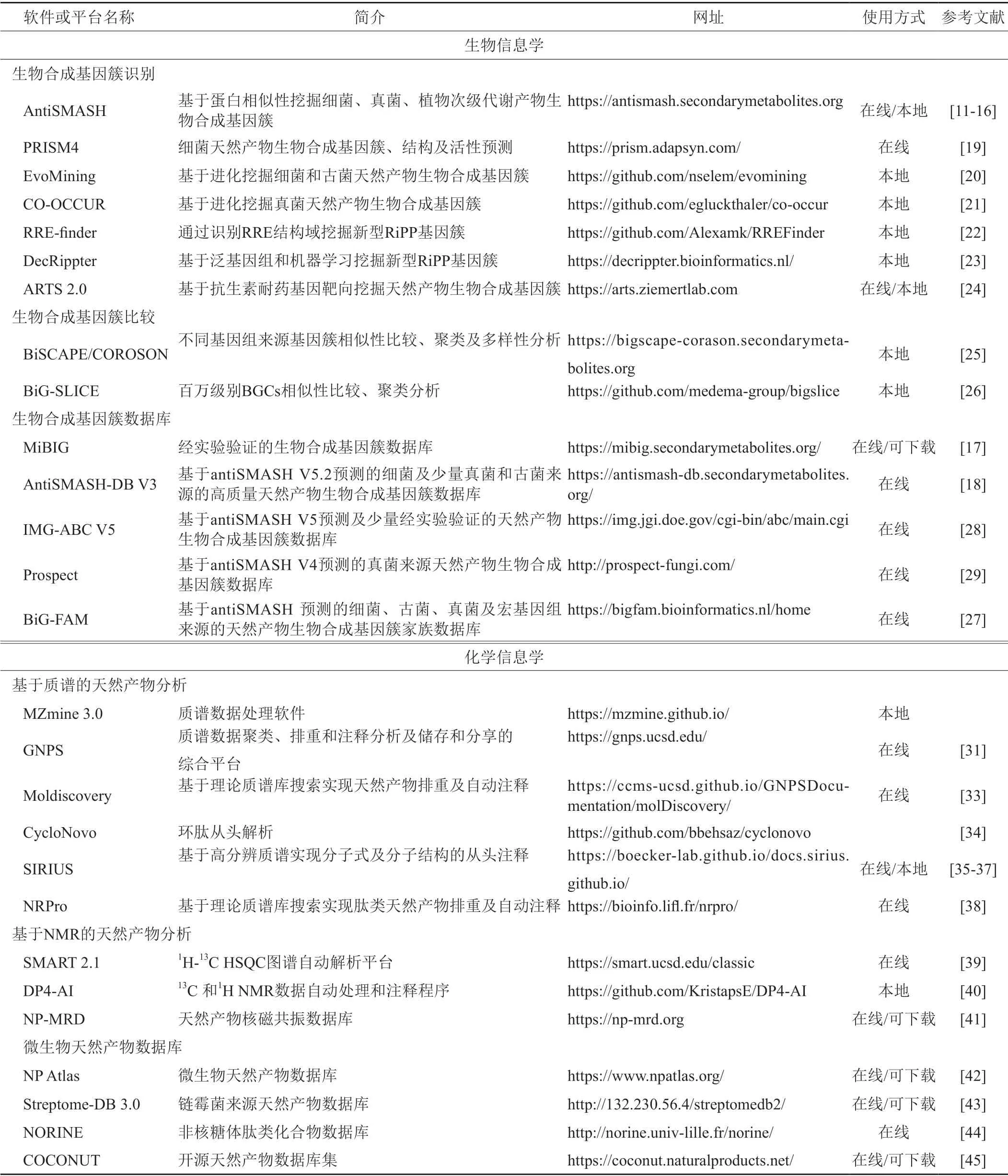
表1 近两年发布或更新的微生物天然产物生物、化学信息学开源工具和数据库Tab.1 Open microbial natural product related bioinformatics and cheminformatics tools and databases released or updated in recent two years
3 展望
基因组学和代谢组学技术的不断进步,使得微生物天然产物研究方法发展了革命性的变革,研究者越来越依赖基因组、代谢组等大数据及与之相应的生物信息学、化学信息学分析方法来提高新化合物的发现效率。天然产物相关信息学研究正处于快速发展阶段,以分析基因组数据为主的生物信息学和以分析代谢组数据为主的化学信息学各自都有了很大的进展,大大提高了科研工作者的工作效率。在此基础上,如果能综合利用基因组和代谢组数据,新型天然产物的发现效率会得到进一步的提高,虽然目前还没有开发出特别有效的多组学分析工具或平台,但信息学家已经向这方面努力,开始建立多组学数据平台,如2021年建立的配对组学数据平台(PoDP)将同一来源的基因组数据和代谢组学数据连接起来[47];微生物天然产物数据库NP Atlas与生物合成基因簇数据库MiBIG及质谱数据库GNPS进行了关联[43,48];生物合成基因簇预测平台antiSMASH与肽类化合物数据库NORINE进行了关联[11]。多组学数据必将进一步促进多组学算法的开发,提高信息学对微生物天然产物发现的指导作用。天然产物化学家一生致力于化合物的分离与鉴定的日子已经不复存在[49],新的发展趋势下,要求化学工作者不仅要擅长分离和结构鉴定,而且还要能够熟练应用各种信息学工具,甚至进一步开发新方法,以实现天然产物的理性、高效发现。

