宏基因组学在食品科学领域的应用研究进展
张维潇,李 键,骞 宇,索化夷,*
(1.西南大学食品科学学院,重庆 400715;2.西南大学 国家食品科学与工程实验教学中心,重庆 400715;3.西南民族大学生命科学与技术学院,四川 成都 610041;4.重庆教育学院生物与化学工程学院,重庆 400067)
宏基因组学在食品科学领域的应用研究进展
张维潇1,2,李 键3,骞 宇4,索化夷1,2,*
(1.西南大学食品科学学院,重庆 400715;2.西南大学 国家食品科学与工程实验教学中心,重庆 400715;3.西南民族大学生命科学与技术学院,四川 成都 610041;4.重庆教育学院生物与化学工程学院,重庆 400067)
宏基因组学是一种免培养,能够直接从环境中提取全部微生物基因组DNA,通过构建宏基因组文库并适当筛选,得到目的基因及生物活化物质的新型研究方法,它可以最大限度地挖掘微生物资源,现在愈渐成为微生物研究和开发最重要的课题之一。本文综述了宏基因组学的研究方法及其近几年来在食品科学领域的应用,着重介绍其在酶制剂开发、食品安全卫生、食品发酵群落、生态演化,食品营养和发现新型物质等领域的应用。同时探讨宏基因组学在食品科学领域未来的应用前景。
宏基因组学;食品科学;研究进展
数10年来,自然环境中微生物的鉴定识别的唯一途径就是用传统的方法进行培养[1],这不但阻碍了人们认识微生物世界的视野,还限制了生物资源的开发和利用。新兴的宏基因组学技术克服了相关培养技术的困难和限制,跳过传统培养而直接从环境样品中提取总DNA,通过构建宏基因组文库、筛选,最后表达,来获得新的功能基因和生物活性物质,文库基本上囊括了所有的微生物遗传信息。因此,宏基因组学的兴起,突破了以往限制的尴尬局面,为解决微生物限制资源问题提供了解决途径,增加了获得更多新生物活性物质的机会[2]。
1 宏基因组学概念
1998年,科学家Handelsman等[3]首次提出宏基因组这一新的概念,可以被用于在复杂环境中确定细菌群落。而宏基因组学就是利用现代各种基因组的分子技术,在微生物学和生物技术的基础上,针对宏基因组进行系统全面的研究,其基本策略流程为如图1所示。
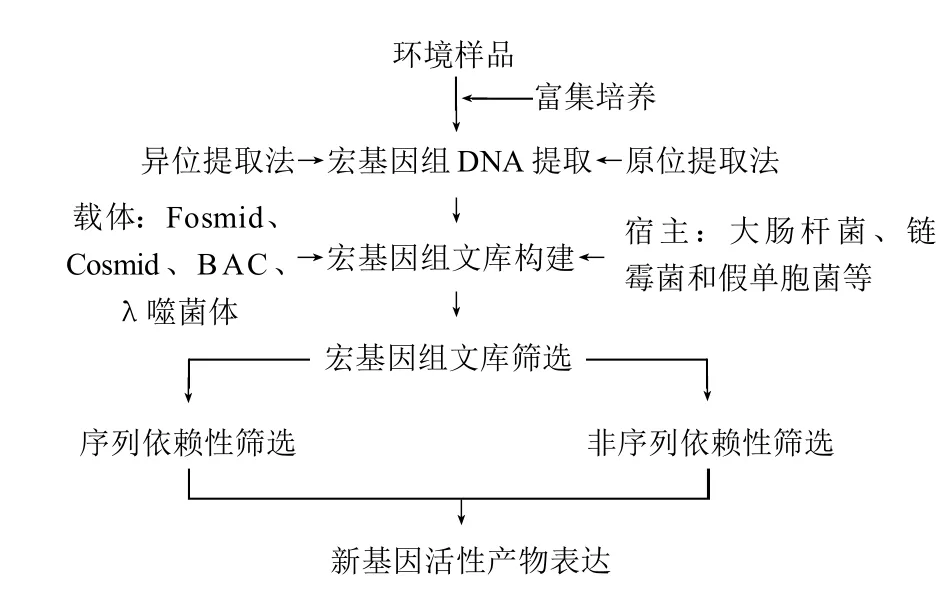
图1 环境微生物宏基因组学研究策略[4]Fig.1 Research strategies for environmental microbial metagenomics[4]
2 宏基因组学研究策略
2.1 文库构建
2.1.1 DNA的提取
DNA的提取与其提取质量与搜集DNA的大小、克隆效率、文库大小和宏基因文库的同源性紧密相关。提取方法可分为原位裂解法(直接提取法)和异位裂解法(间接提取法)。直接提取法就是将样品悬浮于裂解缓冲液中处理,然后抽提纯化。此法特点是易操作,成本低、DNA提取率高,但所提DNA片段较小(10~50kb),且腐殖酸类物质也难以彻底去除[5]。间接提取法是先采用某种物理方法将微生物细胞从原样分离,继而采用较温和的方法抽提DNA,此法可获大片段DNA(20~500kb),特点基本与原位提取法相反:所提DNA纯度较高,但操作繁琐,成本高,仍有些微生物在分离过程中可能会丢失[6]。所以,在实验过程中采用哪一种实验方法具体取决于实验所需要的目的基因大小和研究目的。
2.1.2 载体的选择
宏基因组文库的构建选择适宜的克隆载体,要考虑的方面有:研究目的及所提DNA的质量,期望插入目的片段大小、需要的载体拷贝数、宿主以及筛选方法等[7]。常用于DNA克隆的载体主要包括质粒、黏粒、细菌人工染色体和柯斯质粒等。小插入量文库载体如质粒和噬菌体载体(插入量少于15kb),用于单个基因和小型操纵子的筛选,可筛选出酯酶、淀粉酶、磷酸酶、双加氧酶、蛋白酶、木聚糖酶、氧合酶芳香烃分解酶、氧化多元醇、乙醇氧化还原酶、酰胺酶、β-内酰胺酶等。大插入量文库载体如细菌人工染色体(高于40kb,一般100kb以上甚至达到200kb)、黏粒、柯斯质粒(均可达到40kb),更常用来重新恢复期望基因组或大型DNA片段的复杂提取途径。细菌人工染色体广泛用于淀粉酶、抗菌物质等大片段基因插入,后两者都可筛选出雌二醇双氧酶、抗博来霉素、酰基高丝氨酸等。
2.1.3 宿主的选择
宿主菌株的选择主要要考虑:转化效率、载体在宿主细胞稳定性,宿主为相关基因提供必需的转录表达体系的能力,对异源表达基因产物的相容性,目标功能性状及缺陷型等因素[8]。目前,构建宏基因组文库最常用的宿主就是操作简单、繁殖迅速、培养代谢易于控制的大肠杆菌(Escherichia coli)。E. coli不仅适用于淀粉酶、磷酸酶等常用物质,还成功表达了羟基丁酸、羰基产物和抗菌产物等[9]。其次,变铅青链霉菌(Streptomyces lividans)和恶臭假单胞杆菌(Pseudomonasputida)主要用于抗生素的鉴定。另外,根癌土壤杆菌、黄单胞菌(表达天然色素)[10]、根瘤菌(酒精乙醛脱氢酶)、伯克氏菌(抗菌性基因)克隆的噬菌体宏基因组DNA正被研究用于由大肠杆菌作宿主的标准菌株,来抵抗宿主限制性内切酶和穿孔素、溶菌酶等致死因子[11]。同时还进行着对更高效适宜宿主菌株的探索。
2.2 目标基因克隆的筛选
根据研究目的基因组文库、宏基因组文库的筛选主要有基于功能筛选和基于序列的筛选。
功能筛选法基于生物活性基因,挖掘新的基因编码酶和药物,不用再进行序列分析,对全长基因及功能基因产物有选择性。用此方法除筛选出抗菌素耐药性基因[12]、酯酶、脂肪酶[13]、膜蛋白质[14]、几丁质酶[15]、4-羟基丁酸的基因编码酶[16]等常用常见物质外,还合成生物素[17],此方法用于抗生素、重金属等有毒化合物的降解。此法限制性在于许多基因不仅在宿主菌株(如大肠杆菌)里表达不明显,所以更新的宿主和更高的表达效率还有待开发。
序列筛选法包括完整的基因序列,通过保守的rRNA基因序列用于探索生物多样性,或指导评估鸟枪法导出序列库,有可能筛选到某一类结构中的新基因片段。现已筛选出聚酮合成酶和缩氨聚合酶[18],用于复杂的抗生素。另外还有1,2-苯甲酸盐双氧化酶、硫化氧合还原酶、烷烃羟化酶、4-氯苯甲酰基辅酶A脱卤素酶、聚酮合成酶、亚硝酸还原酶、一氧化二氮还原酶、甲基卤化转移酶、氧化还原酶[19]。虽然新的高通量序列技术使相当多的微生物多样变得可行,但组合文库基因的能力很容易受到复杂的微生物群体的制约。所以在实验之前必须对相关基因序列进行一定了解。
另外,化合物结构水平筛选通过比较基因筛选,其产物不一定具有生物活性;底物诱导基因表达筛选是利用底物诱导克隆子分解代谢基因,可用于活性酶的筛选等[20]。结合了稳定同位素标记法的宏基因组学能提高发现酶的基因探测频率,显著促进了完整基因鸟枪法序列的群体复杂性的发展。此方法中,被标记的C13基质被放到环境中。细菌可以利用有C13合成DNA,与C12区别,再被梯度离心出来。污染的水体中发现包含31kb黏粒运载的芳香环羟基双氧酶的阳性基因[21]。此外,这个研究证明了完整基因鸟枪法宏基因组学结合稳定同位素标记法,能用于重建微生物种群的基因和生物代谢途径。
3 宏基因组学在食品科学领域的应用
微生物在食品科学领域占有极其重要的地位,用宏基因组学的方法来研究食品中可能带有的各种微生物,应用有生物系统多样性的描述,新基因生物体的表征,新生化途径或能量转换的基本代谢的说明,地质储集层的耐环境污染物的识别,酶的发现,二次代谢物和其他生物活性小分子的发现,高分子聚合物的发现[22]。可以发现有利于新食品技术开发的新型微生物活性物质(现阶段主要目标为新型生物催化剂、新型微生物种类、基因和酶、抗生物质、产甲烷生物等的开发),或对食品质量有影响的食品新微生物,也深入了对人体营养的探究,对危害食品安全的各个途径的分析,与从各方面降低安全风险。另外,传统的细菌培养耗费人力物力大,将宏基因组学方法运用到食品检测和食品研究中会带来更多便捷。
3.1 宏基因组学在酶制剂开发的应用
宏基因组学的方法可以提取更多更新更高效的酶类。目前,多糖修饰酶也是食品工业相当重要的一类酶,如:α-1淀粉酶、α-2淀粉酶、葡萄糖淀粉酶和淀粉脱支酶,它们可用于转化一些可利用的原材料[23]。更多的淀粉酶、水合酶、脂肪酶、蛋白酶、腈水解酶、糖苷酶和肌醇六磷酸酶等[24]已经被成功商业化。如漆酶[25]作为食品饮料、食品成分测定及食品组分功能改进中重要的作用物质,也通过宏基因组学得到了进一步的研究和发展。到目前为止,研究人员已经利用以功能和序列为基础的方法,通过宏基因组搜寻,还鉴定了核酸酶、葡糖酸还原酶[26]、4-羟基丁酸脱氢酶、L-氨基酸氧化酶[27]、脂肽、脂蛋白、具有表面活性的脂多糖等多个这样的新型生物催化剂。欧敏功等[28]通过实验筛选了甲基丙二酸半醛脱氢酶(MMSDH)。Suenaga等[29]构建的Fosmid文库用邻苯二酚作为基质筛选出雌二醇加双氧酶,产出91个雌二醇加双氧酶。杨键等[30]成功富集宏基因组DNA中克隆到一种广泛应用于食品行业的α淀粉酶。
研究人员已通过构建湖羊瘤胃未培养微生物的宏基因组文库,筛选其中新的木聚糖酶基因,建立一个瘤胃来源的木聚糖酶库,构建高效分解木聚糖的工程菌,降低木聚糖酶的生产成本,提高其应用适应性[31]。从水牛瘤胃未培养微生物宏基因组文库[32]中克隆并鉴定出1个β-葡萄糖苷酶基因,在同步糖化共发酵工艺生产酒精中有潜在的应用价值。这些新型的酶都在食品科学领域具有极大的潜力和应用价值。
3.2 宏基因组学在食品安全卫生的应用
近年随着人类的发展,生态系统发生了巨大的变化,并从各个方面作用于人们的生活中。如工业废水污染河流、土壤,污染物及其代谢产物以不同形式随作物生长经生物富集作用逐渐渗透入人体内,产生不同的食品安全和卫生问题。最新资料显示,更多的发展和应用逐步建立在微生物生态群落这个重要的环节。
张建坤等[33]提到要应用宏基因组的新技术对酰胺类除草剂甲草胺进行生物降解。赵宇等[34]根据有机磷降解酶基因保守区设计引物,从宏基因组中扩增有机磷降解酶基因片段。宏基因组技术还被应用到对多环芳烃的降解[35],从而降低了农作物或者水体动物从环境中吸收有害因素进而危害人体的可能性。楚雍烈等[36]建立宏基因文库筛选出利用甲醇做碳源的微生物种群,为酒工业的安全生产提供了一些启发。目前还有研究宏基因组学方法筛选卤醇脱卤酶[37],催化环境中有害的有机卤化物分解。牛泽等[38]探索出高效重金属污染土壤混合基因组的提取纯化方法。颜庆云等[39]提出用宏基因PCR-DGGE指纹来反映不同湖区的重金属污染程度,建立DNA多态性与环境因子的关系。
Martin等[40]构建了宏基因文库解密微生物群落的生态和代谢功能,包含加强生物磷酸盐降解系统。宏基因通过整体群落基因扩增(WCGA)和多重取代扩增(MDA)提升了低生物量环境的宏基因可行性和效力,保证了群落微生物基因的代表性[41]。MDA已成为宏基因组学研究的重要手段,如在污水处理系统中,作为预PCR富集步骤减轻联合提取腐植酸和胞外多糖的困难[42]。
3.3 宏基因组学在生态群落的应用
研究人员逐渐深入宏基因组学的目的之一——在于研究可用特定群落的动态关系和特殊环境各种物质的相互作用。用宏基因组学的方法获取完整微生物群落基因,使在生态群落、食品发酵群落构建基因文库这个计划更易得到有效实行。
宏基因可以直接从食品生态系统经过群落PCR锁定16S rRNA基因。PCR结果池扩增随后由变性梯度凝胶电泳(DGGE)或温度梯度凝胶电泳(TGGE)分离,随后可以提取、净化,并在特定的DNA片段序列产生生态系统指纹,可应用到已知的发酵食品生态系统中[43]。
发酵食品生态系统主要由乳酸菌、醋酸菌和一些革兰氏阳性菌、革兰氏阴性菌所构成。所谓的“基因探索微列阵”可以用于在12d的韩国泡菜发酵中,监控乳酸菌的群落动态和生物活性,从而使用10个相关时间的宏基因和宏转录[44]。直接从发酵的克非尔(俄国一种酸牛奶酒)液体和鼓舞样品中提出26个包含16S rDNA片段的细菌宏基因,可识别不同细菌种类的存在[45]。此外,宏基因技术分析还在发酵对虾、韩国泡菜、德国泡菜3种发酵食品的细菌和病毒群体进行研究[46]。通过实验获得309445个病毒宏基因序列,表现出与细菌基因序列的强烈相似性。还从发酵蔬菜食品(韩国泡菜和德国泡菜)中获取了明显不同于发酵对虾的细菌群体。近期研究显示,宏基因序列可以检测病毒和宿主群落的组成。有趣地是,很大一部分数量的宏基因序列片段可以整合为几千个不同集合模式的重叠群[47]。随着对食品乳酸菌外部影响研究的深入,微生物群的宏基因文库微列阵日后会越渐容易[48]。
黄祖新[49]还提出借助宏基因组技术分离纯化大曲酒微生物分泌的功能酶,发现大曲酒发酵生产用微生物的新的功能酶类。筛选出的脂肪酶通过减少不饱和脂肪酸来达到加强茶深度发酵的目的[50]。而由超速454-焦磷酸测序宏基因组法成功分析了发酵罐中沼气微生物群落评判其结构、基因容量、代谢能力和特定沼气生物作用[51]。
具有潜在工业应用性的多功能糖基水解酶从奶牛瘤胃中筛选出来[52]。琥珀酸死杆菌、瘤胃球白菌等被揭示在沼气反应中表现出强大的生物降解木质纤维素的作用[53]。近期,宏基因组学被应用在消化生态系统,发掘在瘤胃细菌的新型水解酶基因和克隆到小鼠大肠的β-葡聚糖酶[54]。一项研究[55]首次揭示人类G1段微生物组在粪便样本中1299个基因型。高通量宏基因技术产出大量的环境基因序列,因此,复杂环境微生物群落的系统组成陆续得到解密[56]。
3.4 宏基因组学在食品营养的应用
近来还提出用宏基因组学的方法深入研究人体肠道内生态系统即在黏膜建立食物-微生物群体系的运作[57]。人类的肠道微生物群系是一个复杂的生态系统,它们包含了1013~1014数量的微生物,而这些微生物所包含的基因至少是人类基因的100倍。Gill等[58]将来自两个健康成人的粪便的DNA扩增,率先发现肠道微生物极大丰富着各种聚糖、氨基酸和一些外来物质的代谢,还有甲烷和2-甲基-D-赤藓糖醇4-磷酸途径介导的维生素和异戊二烯合成等。Qin Junjie等[59]发现最小的肠道宏基因都含有相当多的功能基因片段,它们虽然很罕见,但是可能对肠道起着至关重要的作用。在研究肠道微生物对膳食纤维的分解代谢起的作用时,Tasse等[60]用多步骤功能性手段引导人体肠道宏基因的特定区域的进一步焦磷酸测序来编码消化活性碳水化合物(包括膳食纤维素)的酶,首次用高通量功能测序筛选出26个高效降解植物多糖原料的与非富余宏基因相关的克隆子,覆盖了35个不同源族的73种活性碳水化合物酶类。这相当于人体肠道宏基因随机测序的5倍目的基因富集量。此发现为人类肠道功能营养链提供了新的研究途径。
而人群膳食与人体肠道微生物菌群结构紧密相关。Preidis等[61]将一组由10个人体肠道菌群基因序列构成的群落引入大鼠体内,观测到其反应表达的变化。一组对成人和断奶儿童的结肠微生物宏基因分析显示,膳食因素和人体各种状态,包括先天免疫和适应性免疫、对感染的相对敏感性、免疫耐受、营养素的生物利用度、肠屏障功能可能会改变的肠道微生物群落的组成和功能。针对此状况,可用抗生素、益生菌的合理调配来改善肠道不良状况。肠道中的共生细菌代谢可作为能量来源的食品组分,而这些组分也能影响肠道细菌群落的组成[62]。如十字花科食物中的硫化合物[63]、多酚等植物化学物质和多不饱和脂肪酸,它们的生物利用度依赖于肠道菌群的利用,但又会对肠道聚群产生一定影响。
3.5 宏基因组学在发现新型物质的应用
近期的报道称,宏基因组学在对新型物质的发现上起着至关重要的作用。Coetzee等[64]用此方法发现对葡萄藤有害的新型病毒上。宏基因组学还聚焦于与2,5-二酮酸-D-葡萄糖酸相关的以葡萄糖为基质的新型VC的生产研究[65]。赵越等[66]报道正在研究通过宏基因组学方法筛选具有高产核黄素能力的微生物。
4 结 语
宏基因组学克服了传统培养方法限制微生物资源开发利用的局限性,更大程度揭示了自然群体的生物多样性。到目前为止,宏基因组克隆技术已为研究者们筛选获得大量新型基因和活性物质,从中还可以提取更多可以进行高产量的食品工业原料所需的生物活性物质。如上所总结的,已经有一些在食品行业被利用起来,这些成果对于食品微生物研究、食品快速检测及食品安全系统分析等有着长远的利用价值,还有很多将来会被应用于食品科学之中。环境中存在的各类微生物通过不同的方式进入到食品之中,相信通过宏基因组学的方法,能确定对人类有影响而还未确定的致病因子。但宏基因组学技术作为一项年轻的生物技术手段还有许多亟待解决的问题,如文库的筛选方法有待进一步朝更敏感、更高通量方向完善;更新、更合适的载体还有待继续搜寻和选择;以质粒为载体的文库受限于所能包容的插入尺寸,所以捕获染色体上项目多基因是很不容易的。文库表达的宿主会影响筛选结果。由于异源基因不能被宿主的转录机制识别,筛选时就不能表达进而不能富集。但可以试设想一个具有多重兼容的转录机制的平台微生物[67]来解决这个问题。宏基因组学技术将继续在食品科学领域开发一片崭新、光明的天地。
[2] SIMON C, DANIEL R. Achivements and new knowledge unraveled by metagenomic approaches[J]. Applied Microbiology and Biotechnology,2009, 85(2): 265-276.
[3] HANDELSMAN J, RONDON M R, BRADY S F, et al. Molecular biological access to the chemistry of unkown soil microbes: a new frontier for natural products[J]. Chemistry & Biology, 1998, 5(10): 245-249.
[4] 黄循柳, 黄仕杰, 郭丽琼. 宏基因组学研究进展[J]. 微生物学通报,2009, 36(7): 1058-1066.
[5] 马莉莉, 宗浩, 宋培勇. 宏基因组学: 研究环境微生物的钥匙[J]. 安徽农业科学, 2009, 37(20): 9377-9379.
[6] 阎冰, 洪葵, 许云. 宏基因组克隆: 微生物活性物质筛选的新途径[J].微生物学通报, 2005, 32(1): 113-117.
[7] 宋培勇, 马莉莉, 王庆容. 宏基因组技术及其应用研究进展[J]. 贵州农业科学, 2009, 37(10): 14-18.
[8] DESAI C, PATHAK H, MADAMWAR D. Advances in molecular andtechnologies to gauge microbial communities and bioremediation at xenobiotic/anthropogen contaminated sites[J].Bioresource Technology, 2010, 101(6): 1558-1569.
[9] SINGH B K, MACDONALD C A. Drug discovery from uncultivable microorganisms[J]. Drug Discovery Today, 2010, 15(17/18): 792-799.
[10] TAUPP M , MEWIS K, HALLAM S J. The art and design of functional metagenomic screens[J]. Current Opinion in Biotechnology, 2011, 22(3): 465-472.
[11] RAJENDHRAN J, GUNASEKARAN P. Strategies for accessing soil metagenome for desired applications[J]. Biotechnology Advances, 2008,26(6): 576-590.
[12] DIAZ-TORRES M L, MCNAB R, SPRATT D A, et al. Novel tetracycline resistance determinant from the oral metagenome[J]. Antimicrob Agents Chemother, 2003, 47(4): 1430-1432.
[13] HENNE A, SCHMITZ R A, BOMEKE M, et al. Screening of environmental DNA libraries for the presence of genes conferring lipolytic activity on Escherichia coli[J]. Applied and Environmental Microbiology,2000, 66(7): 3113-3116.
[14] MAJERNIK A, GOTTSCHALK G, DANIEL R. Screening of environmental DNA libraries for the presence of genes conferring Na+(Li+)/H+antiporter activity on Escherichia coli: characterization of therecovered genes and the corresponding gene products[J]. Journal of Bacteriology,2001, 183(22): 6645-6653.
[15] COTTRELL M T, MOORE J A, KIRCHMAN D L. Chitinases from unculturedmarine microorganisms[J]. Applied and Environmental Microbiology, 1999, 65(6): 2553-2557.
[16] HENNE A, DANIEL R, SCHMITZ R A, et al. Construction of environmental DNA libraries in Escherichia coli and screening for the presence of genes conferring utilization of 4-hydroxybutyrate[J]. Applied and Environmental Microbiology, 1999, 65(9): 3901-3907.
[17] ENTCHEVA P, LIEBL W, JOHANN A, et al. Direct cloning from enrichment cultures, a reliable strategy for isolation of complete operons and genes from microbial consortia[J]. Applied and Environmental Microbiology, 2001, 67(1): 89-99.
[18] SCHLOSS P D, HANDELSMAN J. Biotechnological prospects from metagenomics[J]. Current Opinion in Biotechnology, 2003, 14(3): 303-310.
[19] RAES J, FOERSTNER K U, BORK P. Get the most out of your metagenome: computational analysis of environmental sequence data[J].Current Opinion in Microbiology, 2007, 10(5): 490-498.
[20] 张辉, 崔焕忠. 宏基因组学及其研究进展[J]. 中国畜牧兽医, 2007,10(5): 490-498.
[21] SUL W J, PARK J, JOHN F, et al. DNA-stable isotope probing integrated with metagenomics for retrieval of biphenyl dioxygenase genes from polychlorinated biphenyl-contaminated river sediment[J]. Applied and Environmental Microbiology, 2009, 75(17): 5501-5506.
[22] HANDELSMAN J. Metagenomics: application of genomics to uncultured microorganisms[J]. Microbiology and Molecular Biology Reviews,2004, 68(4): 669-685.
[23] 钱莉莉, 史炳照. 宏基因组学在新型生物催化剂开发中的研究进展[J]. 微生物学杂志, 2006, 26(4): 68-71.
[24] BRADY S F, CLARDY J. Long-chain N-acetyl amino acid antibiotics isolated from heterologously expressed environmental DNA[J]. Journal of the American Chemical Society, 2000, 122(51): 12903-12904.
[25] 曹治云, 郑腾, 谢必峰, 等. 漆酶工业应用的研究进展[J]. 生物技术通报, 2004, 15(4): 414-416.
[26] 赵勇, 孙晓红, 韩丽, 等. 微生物分子生态学技术及其在食品产业中的应用前景[J]. 上海水产大学学报, 2007, 16(4): 381-388.
[27] 强慧妮, 田宝玉, 江贤章, 等. 宏基因组学在发现新基因方面的应用[J]. 生物技术, 2009, 19(4): 82-85.
[28] 欧敏功, 崔晓龙, 李一青, 等. 宏基因组学在未培养微生物研究中的应用[J]. 微生物学杂志, 2007, 27(2): 88-91.
[29] SUENAGA H, OHNUKI T, MIYAZAKI K. Functional screening of a metagenomic library for genes involved in microbial degradation of aromatic compounds[J]. Environmental Microbiology, 2007, 9(9): 2289-2297.
[30] 杨键, 曾丽娟, 廖思明, 等. 富集宏基因组DNA中α-淀粉酶全长基因的克隆及重组表达[J]. 中国生物工程杂志, 2010, 30(3): 56-60.
[31] KNIETSCH A, BOWIEN S, WHITED G, et al. Identification and characterization of coenzyme B12-dependent glycerol dehydratase-and diol dehydratase-encoding genes from metagenomic DNA libraries derived from enrichment cultures[J]. Applied and Environmental Microbiology, 2003, 69(6): 3048-3060.
[32] 郭鸿, 封毅, 莫新春, 等. 水牛瘤胃宏基因组的一个新的β-葡萄糖苷酶基因umcel3G的克隆、表达及其表达产物的酶学特性[J].生物工程学报, 2008, 24(2): 232-238.
[33] 张建坤, 赵晓亚, 赵晖, 等. 酰胺类除草剂甲草胺的微生物降解研究进展[J]. 化学与生物工程, 2009, 26(6): 19-23.
[34] 赵宇, 许丽, 田健, 等. 有机磷污染土壤和活性泥中有机磷降解酶基因和微生物多样性研究[J]. 中国农业科技导报, 2009, 11(6): 63-68.[35] 张薇, 高洪文, 张化永, 等. 宏基因组技术及其在环境保护和污染修复中的应用[J]. 生态环境, 2008, l7(4): 1696-1701.
[36] 楚雍烈, 杨娥. 宏基因组学及其技术的研究进展[J]. 西安交通大学学报: 医学版, 2008, 29(6): 601-608.
[37] 聂洪丽, 汤丽霞. 基于宏基因组学的卤醇脱卤酶筛选[J]. 生物产业技术, 2009(增刊1): 158-161.
[38] 牛泽, 曾艳, 王敏, 等. 北京污灌区重金属污染土壤DNA提取及宏基因组文库构建[C]//中国农业生物技术学会第三届会员代表大会暨学术交流会论文摘要集. 北京: 中国农业生物技术学会, 2006.
[39] 颜庆云, 余育和, 冯伟松, 等. 武汉东湖浮游生物宏基因组与环境重金属的关系[J]. 中国环境科学, 2010, 30(增刊1): 52-56.
[40] MARTIN H G, IVANOVA N, KUNIN V, et al. Metagenomic analysis of two enhanced biological phosphorus removal (EBPR) sludge communities[J]. Nature Biotechnology, 2006, 24(10): 1263-1269.
[41] GALVAO T C, MOHN W W, LORENZO V D, et al. Explring the microbial biodegradation and biotransformation gene pool[J]. Trends in Biotechnology, 2005, 23(10): 497-506.
[42] GONZALEZ J M, PORTILLO M C, SAIZ-JIMENEZ C. Multiple displacement amplification as a pre-polymerase chain reaction (pre-PCR)to process difficult to amplify samples and low copy number sequences from natural environments[J]. Environmental Microbiology, 2005, 7(7):1024-1028.
[43] ROH S W, KIM K H, NAM Y D, et al. Investigation of archaeal and bacterial diversity in fermented seafood using barcoded pyrosequencing[J]. The ISME Journal, 2010, 4(1): 1-16.
[44] NAM Y D, CHANG H W, KIM K H, et al. Metatranscriptome analysis of lactic acid bacteria during kimchi fermentation with genome-probing microarrays[J]. International Journal of Food Microbiology, 2009, 130(2): 140-146.
[46] CLAESSON M J, O,SULLIVAN O, WANG Qiong, et al. Comparative analysis of pyrosequencing and a phylogenetic microarray for exploring microbial community structures in the human distal intestine[J]. PLoS One, 2009, 4(8): 66-69.
[47] PARK E J, KIM K H, ABELL G C J, et al. Metagenomic analysis of the viral communities in fermented foods[J]. Applied and Environmental Microbiology, 2011, 77(4): 1284-1291.
[48] VAUGHAN E E, HEILIG H G, BEN-AMOR K, et al. Diversity, vitality and activities of intestinal lactic acid bacteria and bifidobacteria assessed by molecular approaches[J]. FEMS Microbiology Reviews, 2005, 29(3): 477-490.
[49] 黄祖新. 宏基因组学及其在大曲酒微生物研究中的应用[J] . 酿酒科技, 2009(10): 17-21.
[50] HASAN F, SHAH A A, HAMEED A. Industrial applicationgs of microbial lipases[J]. Enayme and Microbial Technology, 2006, 39(2): 235-251.
[51] SCHLUTER A, BEKEL T, DIAZ N N, et al. The metagenome of a biogas-producing microbial community of a production-scale biogas plant fermenter analysed by the 454-pyrosequencing technology[J]. Journal of Biotechnology, 2008, 136(1/2): 77-90.
[52] PALACKAL N, LYON C S, ZAIDI S, et al. A multifunctional hybrid glycosyl hydrolase discovered in an uncultured microbial consortium from ruminant gut[J]. Applied and Micobiology Biotechnology, 2007,74(1): 113-124.
[53] LISSENS G, VERSTRAETE W, ALBRECHT T, et al. Advanced anaerobic bioconversion of lignocellulosic waste for bioregenera-tive life support following thermal water treatment and biodegradation by Fibrobacter succinogenes[J]. Biodegradation, 2004, 15(3): 173-183.
[54] FERRER M, GOLYSHINA O V, CHERNIKOVA T N, et al. Novel hydrolase diversity retrieved from a metagenome library of bovine rumen microflora[J]. Environmental Microbiology, 2005, 7(12): 1996-2010.
[55] BREITBART M, HEWSON I, FELTS B, et al. Metagenomic analyses of an uncultured viral community from human feces[J]. Journal of Bacteriology, 2003, 185(20): 6220-6223.
[56] SONG J S, JEON J H, LEE J H, et al. Molecular characterization of TEM-type β-lactamases identified in cold-seep sediments of Edison Seamount (south of Lihir Island, Papua New Guinea)[J]. Journal of Microbiol, 2005, 43(2): 172-178.
[57] MARTINS D S V, MULLER M, de VOS W M. Systems biology of the gut: the interplay of food, microbiota and host at the mucosal interface[J].Current Opinion in Biotechnology, 2010, 21(4): 539-550.
[58] GILL S R, POP M, DEBOY R T, et al. Metagenomic analysis of the human distal gut microbiome[J]. Science, 2006, 312: 1355-1359.
[59] QIN Junjie, LI Ruiqiang, RAES J, et al. A human gut microbial gene catalogue established by metagenomic sequencing[J]. Nature, 2010, 464:59-65.
[60] TASSE L, BERCOVICI J, PIZZUT-SERIN S, et al. Functional metagenomics to mine the human gut microbiome for dietary fiber catabolic enzymes[J]. Genome Research, 2010, 20(11): 1605-1612.
[61] PREIDIS G A, VERSALOVIC J. Targeting the human microbiome with antibiotics, probiotics, and prebiotics: gastroenterology enters the metagenomics era[J]. Gastroenterology, 2009, 136(6): 2015-2031.
[62] LAPARRA J M, SANZ Y. Interactions of gut microbiota with functional food components and nutraceuticals[J]. Pharmacological Research, 2010,61(3): 219-225.
[63] LI Fei, HULLAR M A J, SCHWARZ Y, et al. Human gut bacterial communities are altered by addition of cruciferous vegetables to a controlled fruit- and vegetable-free diet[J]. Nutrition, 2009, 139(9): 1685-1691.
[64] COETZEE B, FREEBOROUGH M J, MAREE H J, et al. Deep sequencing analysis of viruses infecting grapevines: virome of a vineyard[J]. Virology, 2010, 400(2):157-163.
[65] STREIT W R, SCHMITZ R A. Metagenomics: the key to the uncultured microbes[J]. Current Opinion in Microbiology, 2004, 7(5): 492-498.
[66] 赵越, 王晖, 鲍楠, 等. 利用宏基因组数据筛选高产核黄素的基因片段[J]. 黑龙江大学自然科学学报, 2009, 26(3): 380-384.
[67] NICOLAOU S A, GAIDA S M, PAPOUTSAKIS E T. A comparative view of metabolite and substrate stress and tolerance in microbial bioprocessing: from biofuels and chemicals, to biocatalysis and bioremediation[J]. Metabolic Engineering, 2010, 12(4): 307-331.
Research Progress and Applications of Metagenomics in Food Science
ZHANG Wei-xiao1,2,LI Jian3,QIAN Yu4,SUO Hua-yi1,2,*
(1. College of Food Science, Southwest University, Chongqing 400715, China;2. National Food Science and Engineering Experimental Teaching Center, Southwest University, Chongqing 400715, China;3. College of Life Science and Technology, Southwest University for Nationalities, Chengdu 610041, China;4. College of Biology and Chemical Engineering, Chongqing College of Education, Chongqing 400067, China)
Metagenomics is an advanced methodology by means of extracting all microbialgenomic DNAs in certain environmental habitat, constructing and screening metagenomic libraries to seek novel functional genes and biologically active compounds without cultivating. It can mine microbial resources to the greatest extent. Therefore, it is increasingly becoming one of the most important subjects in microbiological research and development. This paper reviews the recent research progress of metagenomics and its applications in the area of food science, especially in enzyme preparation development, food safety and hygiene, food fermentation, ecological evolution, food nutrition, and the discovery of new compounds, and explores its future prospects in the area of food science.
metagenomics;food science;research progress
TS201.3
A
1002-6630(2012)05-0309-06
2011-09-20
国家公益性行业(农业)科研专项资金资助项目(201203009);西南大学中央高校基本科研业务费专项资金资助项目(XDJK2009C041)
张维潇(1989—),女,本科生,研究方向为食品质量与安全。E-mail:zwxlaugh@163.com
索化夷(1978—),男,讲师,博士研究生,研究方向为食品科学。E-mail:birget@swu.edu.cn

