湖南新田大冠岭野生大豆居群遗传结构与空间分布关系
赵青松,赵云云,王 朋,邓小娟,马启彬,年 海,杨存义
(1亚热带农业生物资源保护和利用国家重点实验室/广东省植物分子育种重点实验室/国家大豆改良中心广东分中心/华南农业大学农学院,广东广州 510642;2河北农林科学院粮油作物所/国家大豆改良中心石家庄分中心,河北石家庄 050035)
中国是大豆的起源地,拥有丰富的种质资源[1].野生大豆是栽培大豆的祖先,蕴含着许多优良基因[2-5].野生大豆在中国广泛分布,从北纬53°的漠河到北纬24°的象州均有分布[6].野生大豆资源的有效保护和利用是建立在了解野生大豆遗传多样性和群体遗传结构的基础上.目前已利用形态性状、同工酶、分子标记等在不同水平上对中国野生大豆的遗传多样性进行了广泛研究.从全国范围对野生大豆遗传多样性的分析表明野生大豆拥有比栽培大豆更高的遗传多样性[7-9].在全国范围内的野生大豆地理群体中,43% ~92%的遗传变异存在于居群内,大多数居群存在遗传瓶颈效应[10-13].多个研究结果也表明西南部和南方是野生大豆的一个主要多样性中心[10-11],但也有研究结果认为中国有多个多样性中心,分别位于黄河流域、中国中部和长江流域[13].研究表明,浙江[14]、河北省[15-16]、重庆[17]、湖南和湖北[18]、江西[19]、广西[20]、西藏[21]和北京[22]等区域内的野生大豆群体间也存在丰富的遗传多样性.
小尺度空间范围对野生大豆居群的遗传多样性研究可提供居群在特定微环境中演化和分化的信息,也为自然居群的取样提供理论依据[23].从小尺度范围内已对我国辽宁新宾县野生大豆保护区野生大豆居群[24]、吉林省龙井保护区野生大豆居群[25]、山东垦利黄河口自然保护区野生大豆居群[26]、上海江湾野生大豆居群[23,27]的遗传多样性做了分析,结论不尽相同.多数分析结果认为受野生大豆种子、花粉传播和微环境的影响,居群内的遗传变异并不完全呈随机分布,而呈斑块状分布,居群的遗传多样性与地理分布存在相关.但对浙江金华的自然群体分析结果表明遗传距离与地理距离无明显相关性[13].这主要是因为小尺度范围居群的多样性主要由突变引起,随机漂变是其空间分布的主要因素,而影响大尺度居群分布的光、温等因素的作用较小.
目前已有的小尺度居群分析多集中在黄淮海流域和东北地区野生大豆群落,而多个研究都认为中国南方、西南部为中国野生大豆的多样性中心[10-11],也是野生大豆的起源中心[7,28],但对该区域内野生大豆小尺度分析及居群之间进化及其演变关系报道较少.研究认为南方野生大豆与栽培大豆的细胞器DNA最接近[8],尤其北纬25°左右野生大豆可能与栽培大豆的起源有关[29-30].上述研究结论是利用了国家资源库中的野生大豆资源得出,而利用该区域自然群体研究的报道不多.
地处北纬25°的南岭是我国重要的自然地理分界线,南岭的南北坡降水和温度差异明显.北纬25°附近也是目前已知野生大豆分布的最南端,其中湖南新田大冠岭位于南岭北,分布着丰富的野生大豆,一些野生大豆居群的遗传多样性远高于周围的自然居群[31].本研究采用SSR标记对分布在大冠岭周围的野生大豆自然居群遗传多样性、空间结构进行分析,以揭示该地区野生大豆居群间关系及其演变关系,旨在为中国南方野生大豆资源的有效利用和资源保护提供理论指导.
1 材料与方法
1.1 材料及来源
2007年在湖南省新田县大冠岭及其周围的16个野生大豆Glycine soja Sieb.et Zucc.自然居群中依据群体大小随机采集612份个体的新鲜叶子用于DNA提取和SSR分析,并用GPS定位仪对取样植株地理位置定位,记录采样点面积、生境等相关信息(表1).
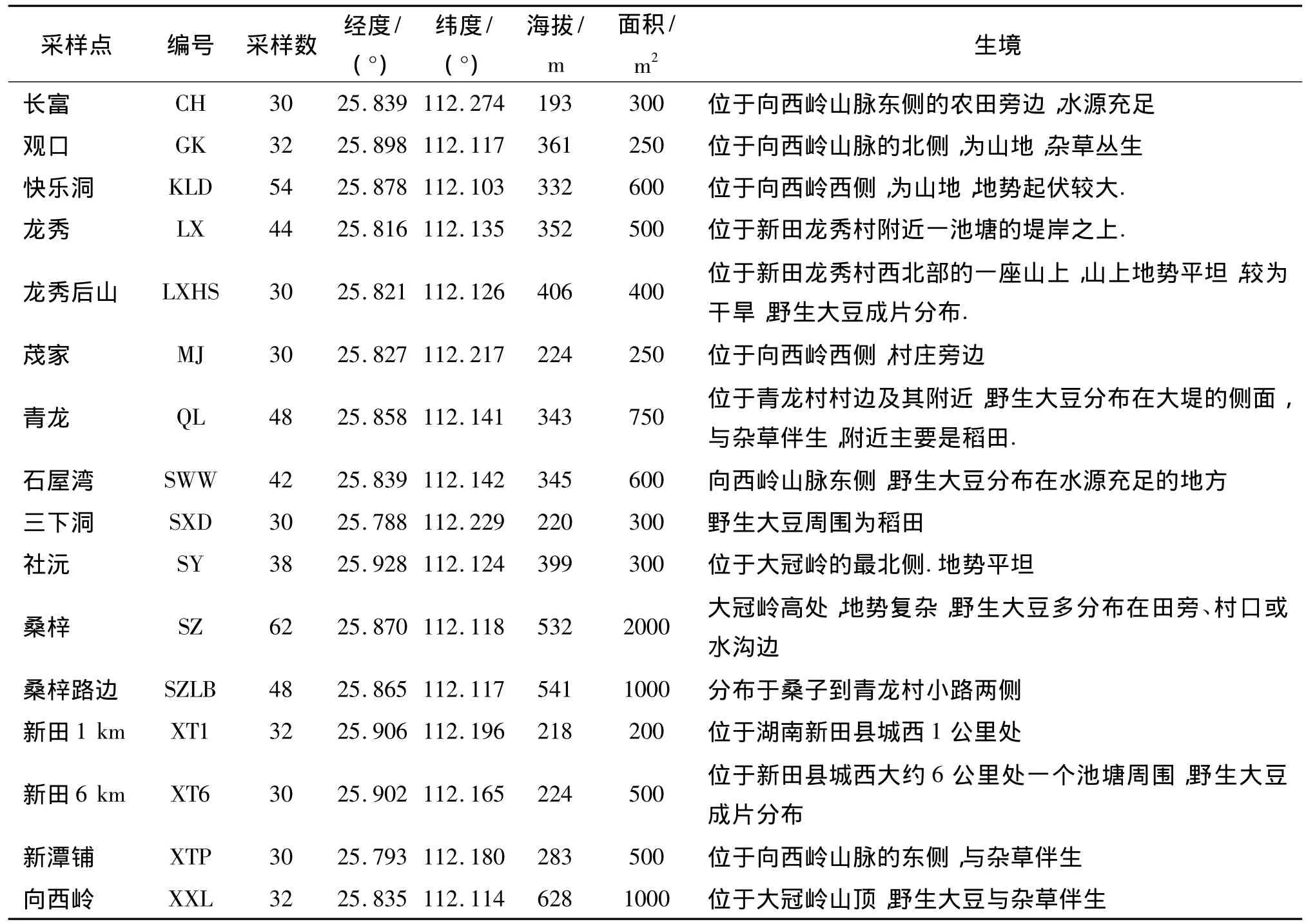
表1 野生大豆居群采集地点及相关信息Tab.1 The samp ling sites and related informations of soybean populations
1.2 基因组提取及SSR扩增
参照Cregan等[32]建立的大豆分子标记连锁图,从每个连锁群上等距离选择2~3个SSR位点,共选用41对引物(表2).基因组DNA提取和 SSR扩增与文献[31]一致.
1.3 野生大豆居群的遗传多样性分析
居群多态位点百分数(Polimophism,P):P=l/T×100%,l为居群(或个体)观测到的多态性位点数,T为总群体所观测到的多态性位点总数.位点等位基因数(Number of alleles at locus,Na):某位点在所有居群中检测到的等位变异数.群体平均等位基因数(Mean number of alleles per locus,Nap):某居群在所有位点上所检测到等位变异数的平均值.期望杂合度(Expected heterozygosity,He)和Shannon指数(I):
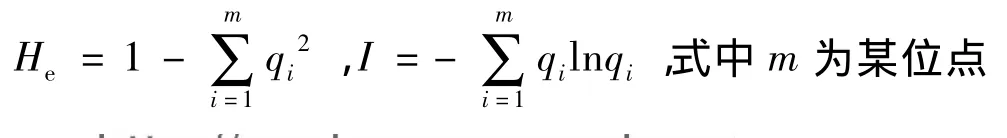

1.4 遗传结构分析及聚类分析
应用 Cregan 等[32]和 Pritchard 等[33]的基于混合模型的多位点基因型数据聚类分析方法(软件Structure 2.3)对供试野生大豆居群聚类,并确定材料的血缘组成.具体分析过程,先设定群体数目(K)为2~30,并假定位点都是独立的,将 MCMC(Markov chain Monte Carlo)开始时的不作数迭代(Length of burn-in period)设为10 000次,再将不作数迭代后的MCMC迭代设为100 000次,然后依据似然值最大的原则选取一个合适的K值并且画出基于模型的群体遗传结构图.群体的主成份分析用GenAlEx6.5软件计算[33].
1.5 遗传结构与地理分布相关评价
用Mental test矩阵相关检测遗传距离和地理距离的相关性.用空间自相关系数(r)来衡量野生大豆居群的空间遗传结构,即样本个体间空间距离对和遗传距离对之间的相关系数,以上数据采用GeneA-lEx6.5 计算[33].
2 结果与分析
2.1 大冠岭及其周围区域各居群的地理分布
调查中发现,野生大豆居群往往分布在离村庄或耕地不远的地方,如水沟边、池塘边、田埂旁、路边等地方.位于大冠岭区域的快乐洞(KLD)、桑梓村(SZ)和向西岭(XXL)一带分布较大面积的野生大豆,而远离大冠岭地区的野生大豆零星分布.在大冠岭及其周围区域共采集了16个居群612个样本(表1),其中新田县城西1 km处(XT1)和6 km处(XT6)2个较小的自然居群处于公路边,地势较平坦,长富村(CH)、茂家(MJ)和三下洞(SXD)自然居群则位于新田县城南部,其余11个居群位于大冠岭及其周边地区.大冠岭向西岭(XXL)一带海拔较高,周边海拔逐渐降低,形成复杂地形地貌.
2.2 大冠岭及其周围区域野生大豆居群遗传多样性分析
分子标记结果显示,41个位点共检测到414个等位变异,位点等位变异数目范围为4~19个,平均值为10.1个,Satt269、Satt073、Satt497等位变异最少,只有4个,而Sat_177等位变异最多,为19个.≥10个等位变异的位点有20个,<10个等位变异的位点有21个.位点的I值变化范围0.283~2.542,平均为1.751,He值变化范围0.096~0.909,平均为0.751(表2).
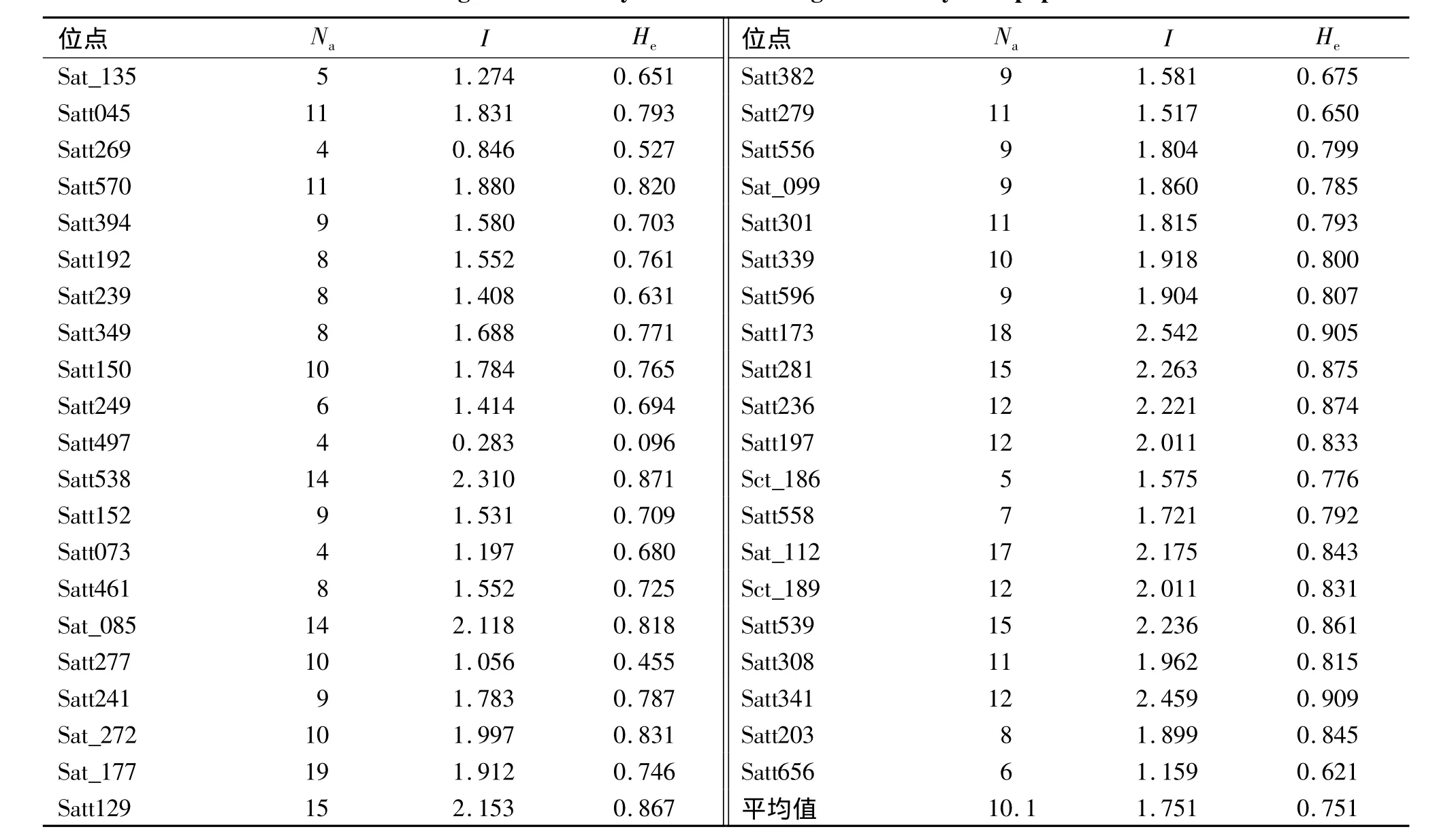
表2 41个位点在16个野生大豆居群中的遗传多样性参数1)Tab.2 The genetic diversity of 41 lociamong 16 w ild soybean populations
41对引物在各居群中均具有多态性,居群平均位点多态性为66.09%(表3).多态位点百分率大于90%的居群有 7 个,分别是 KLD、LX、LXHS、SY、SZ、SZLB、XXL,其中 XXL最高,多态位点百分数为100%.16个野生大豆居群He变化范围为0.062~0.610,信息指数变化范围为0.118~1.228.从空间分布发现大冠岭区域的向西岭(XXL)至桑梓一带野生大豆遗传多样性较高,而远离大冠岭的新潭铺(XTP)、三下洞(SXD)、茂家(MJ)、长富(CH)、新田县城西1 km(XT1)和新田县城西6 km(XT6)居群的遗传多样性较低.
在16个居群中总共检测到126个特有等位基因,不同居群内的特有等位基因数不同,XXL、SY、KLD、SZ几个居群的野生大豆的特有等位基因较多,而MJ、SZLB、XTP、QL等居群的特有等位基因较少(表3).KLD野生大豆居群位于大冠岭山上一个村庄附近,此处野生大豆居群特有等位基因最多,但遗传多样性不是最高,可能是稀有等位基因较多,桑梓路边遗传多样性较高,但特有等位基因较少,这是因为桑梓路边野生大豆居群与桑梓居群相邻,居群间有重叠,所以特有等位基因较少.
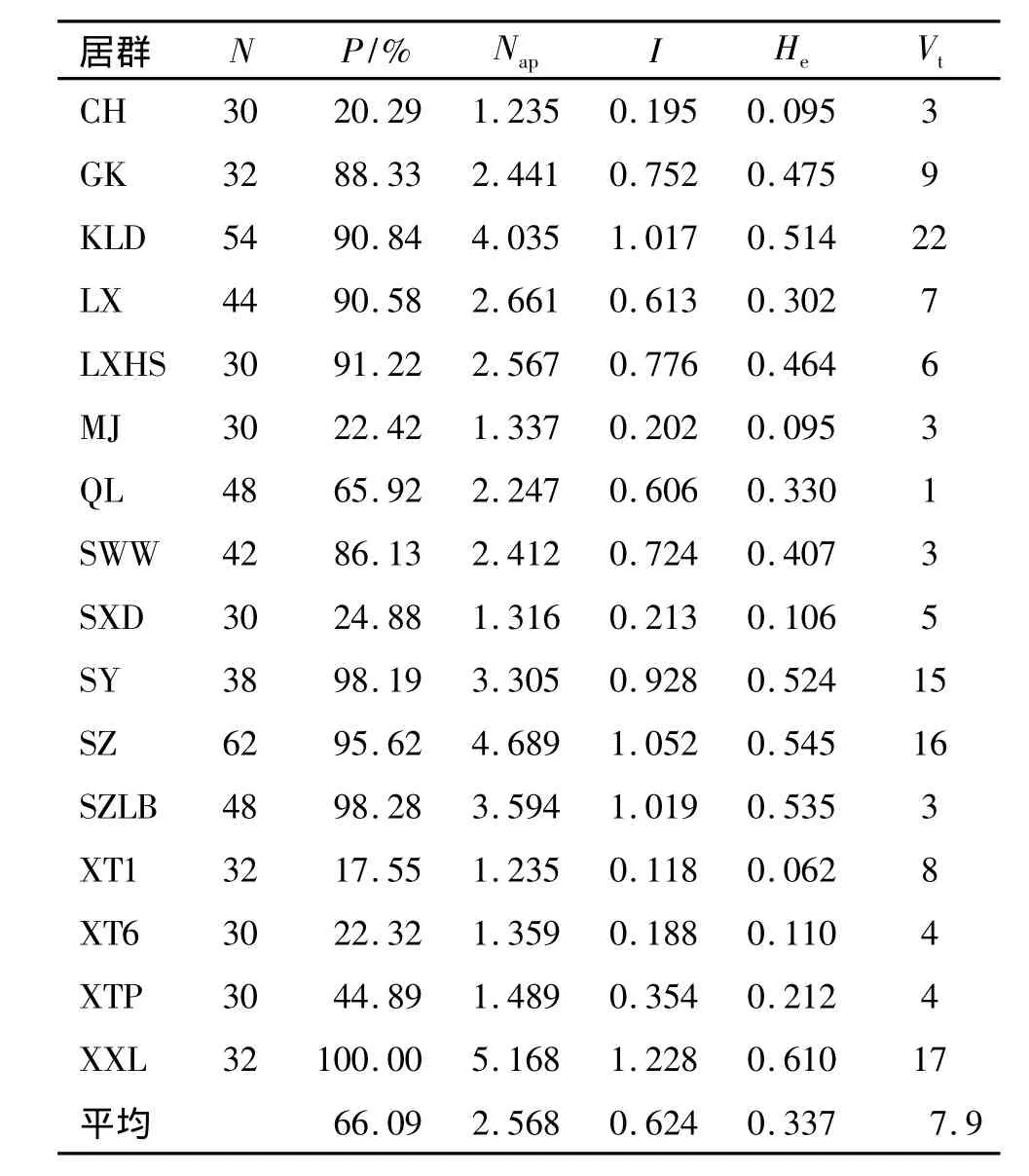
表3 16个野生大豆居群的遗传多样性参数1)Tab.3 The genetic diversity of 16 w ild soybean populations
2.3 野生大豆居群遗传分化与基因流
自然居群间分化程度可用分化系数和基因流来描述,居群间分化系数越大,基因流越小,居群差异就越大.新田大冠岭区域野生大豆自然居群间的分化系数和基因流变化范围分别为0.114~0.848和0.052~1.978.居群间基因流>1的有12对,<0.1的有10对.向西岭、桑梓、桑梓路边与周边居群分化系数较小,基因流较大(表4).推测大冠岭区域内野生大豆原来是一个大群落,可能由于人类活动、气候、地理等因素的影响而被割裂.大冠岭山上大部分居群与山脚下和远离大冠岭自然居群间分化系数和基因流处于中间,表明山下和远离大冠岭自然居群是早期由山上群体传播形成.远离大冠岭的自然居群间及与大冠岭山脚下的自然居群间分化系数大,基因流较小.新田西1 km处居群和其他居群的基因流均较小,说明新田1 km处和其他居群的来源不同(表4).主成分分析进一步表明向西岭有向外扩散趋势,由此推测大冠岭各居群可能始于向西岭居群(图1).
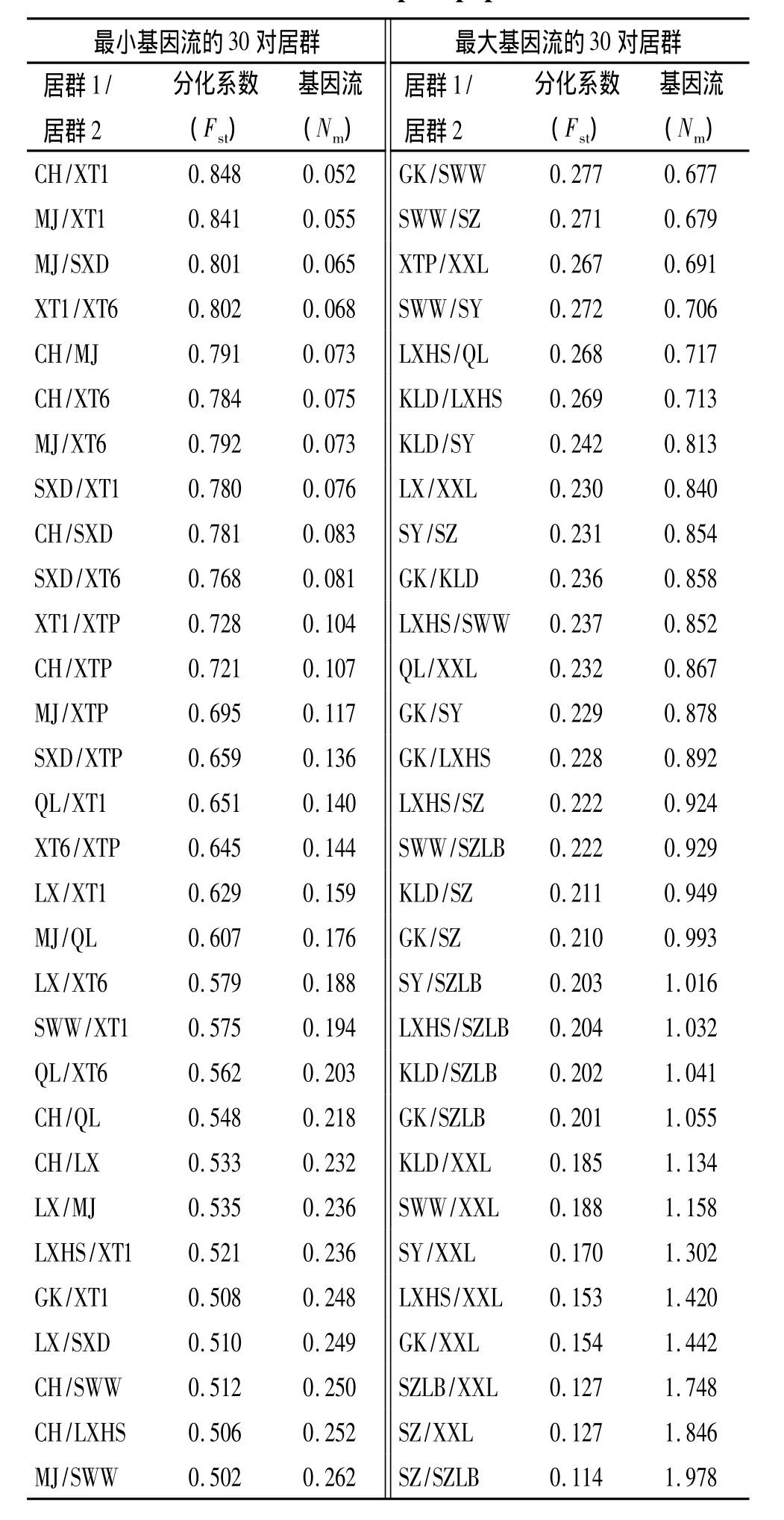
表4 30对最大和最小居群间成对基因流和分化系数Tab.4 The gene flow and differentiation coefficient of the most and the least 30 pair populations

图1 野生大豆居群主成分分析Fig.1 Principal component analysis of wild soybean populations
2.4 大冠岭及其周围区域野生大豆遗传结构分析
利用Structure 2.3软件对野生大豆居群进行遗传结构分析,在K=19时似然值最大,因此认为大冠岭野生大豆居群分为19组较为合理,其中有9个组是由1个居群的个体组成的,分别由XT1、SXD、CH、SY、MJ、XT6、LXHS、LX、GK、QL 居群组成.另外10组则包括了不同居群的个体.遗传结构图(图2a)表明,19个组界限明显,其中第8、12、15、18组成员组成较为复杂,大部分都存在居群SZ、SZBL、XXL的个体.说明这几个居群遗传多样性较高.从居群的结构图(图2b)也可以发现KLD、GK、SY等居群中个体为不同组的混合,说明这些居群个体来源于临近野生大豆居群.从空间分布看,混合组主要是由大冠岭周围的自然居群组成,远离大冠岭的自然居群只有CH和SY的部分个体进入了混合组.由此推测大冠岭山上的野生大豆自然居群曾连片分布,现有居群是近期人类活动所致.
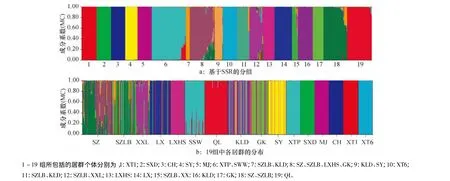
图2 大冠岭野生大豆群体的遗传结构Fig.2 The genetic structure ofwild soybean population of Daguanling
2.5 遗传结构与地理距离的关系
遗传距离矩阵和地理距离矩阵的Mental检验显示,两者之间相关系数达0.543(P<0.01),表明遗传距离和地理距离存在线性相关关系,即随着地理距离的加大,居群间的遗传距离也有增大的趋势(图3).而空间自相关分析(图4)表明,随着地理距离的增加,居群间遗传距离和地理距离的相关性呈下降趋势,在置信区间95%以下,与横坐标的截距在1 400 m以内空间自相关系数(r)为正值,在1 700 m以外r为负值.这一结果表明在1 400 m以内,野生大豆的遗传距离和地理距离呈正相关,即地理距离越近,遗传距离也越近;而在1 700 m以外,遗传距离和地理距离没有相关性.

图3 16个野生大豆居群地理距离和遗传距离相关结果Fig.3 The correlation between geographic distance and genetic distance of 16 wild soybean populations
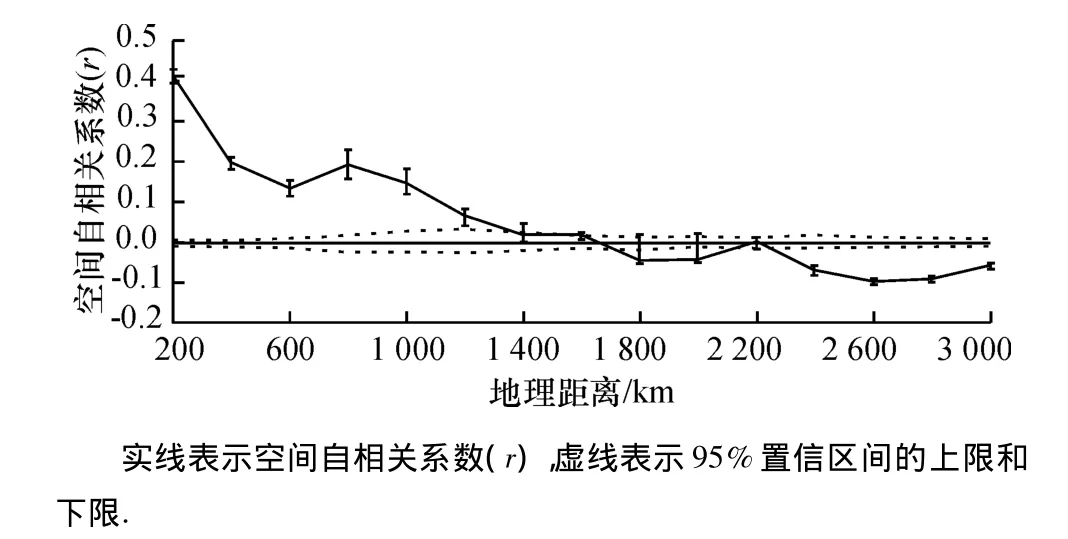
图4 基于41对引物野生大豆的空间自相关分析结果Fig.4 Spatial autocorrelation analysis of wild soybean based on 41 loci
分析海拔与遗传多样性之间的关系发现,随着海拔的升高,居群的等位变异数、期望杂合度和信息指数有逐渐升高的趋势.居群XT1海拔较低(218 m),具有最低的遗传多样性,等位变异数、信息指数和期望杂合分别为1.235、0.118和0.062;而居群XXL海拔最高(628 m),等位变异数、信息指数和期望杂合度达到最大值,分别为5.168、1.228和0.610(表1、表3).这可能是由于海拔高野生大豆居群受人类活动影响较少,保留了丰富的遗传多样性.
3 讨论
3.1 湖南新田大冠岭及其周围区域存在一个野生大豆居群的多样性中心
国内外学者近年来利用形态性状、同工酶、分子标记等从宏观尺度对中国野生大豆的遗传多样性进行了研究,发现南方野生大豆多样性高,与栽培大豆遗传亲缘近,可能是栽培大豆的起源地之一[7-8],也有学者认为有多个野生大豆的多样性中心,分别位于黄河流域、中国中部、和长江流域[11].对国内不同区域的野生大豆的群体结构分析也表明区域内野生大豆群体间也存在丰富的遗传多样性[11,16-23]等.小尺度空间范围对野生大豆居群的遗传多样性研究可提供居群在特定微环境中演化和分化的信息,也为自然居群的取样提供理论依据[24].
调查发现湖南新田县大冠岭周围广泛分布野生大豆,多样性高于远离大冠岭的自然居群,推测该区域可能存在一个野生大豆的遗传多样性中心[31].本研究在上次采样的基础之上围绕大冠岭密集调查采样,在原采样居群的基础上又增加了GK、SY、KLD、XXL、SWW等几个居群,同时在远离大冠岭地方增加了XTP、SXD、MJ、CH等几个居群,居群数目达到16个.41对SSR引物分析的结果表明,位于大冠岭山上的XXL、SZ、SZLB、KLD、SY 野生大豆居群的遗传多样性高,而远离大冠岭的 CH、MJ、SXD、XTP、XT1、XT6野生大豆居群的遗传多样性非常低.成对基因流分析结果表明XXL和其他大部分居群间的基因流均较大.由以上结果推测向西岭至桑梓一带分布的野生大豆至少是大冠岭及其周围区域野生大豆的一个多样性中心.
在小尺度范围内对我国辽宁新宾县野生大豆保护区野生大豆居群[25]、吉林省龙井保护区野生大豆居群[26]、山东垦利黄河口自然保护区野生大豆居群[27]、上海江湾野生大豆居群[24,28]的遗传多样性分析认为受野生大豆种子、花粉传播和微环境的影响,居群内的遗传变异并不完全呈随机分布,而呈斑块状分布,遗传多样性与地理分布存在相关性.湖南新田县大冠岭野生大豆多样性分布呈斑块状,但向西岭至桑梓一带分布的野生大豆应是人类活动或气候、地理因素造成的割裂,而其他居群是这一群体扩散形成的.因此湖南新田县野生大豆保护和利用应以向西岭为中心开展.
3.2 湖南新田大冠岭及其周围区域野生大豆遗传多样性地理分布及传播方式
大冠岭野生大豆居群表现出明显的特征,位于海拔较高的向西岭—桑梓一带遗传多样性较高,遗传距离与地理距离在一定范围内呈正相关.空间自相关结果显示,在1 400 m以内,遗传距离和地理距离呈正相关,而1 700 m以外遗传距离和地理距离没有相关关系,这与Wang等[34]认为野生大豆在生态系统中种子的传播半径为1.5 km相一致.
本研究认为湖南新田县大冠岭的野生大豆是以向西岭至桑梓一带为中心向四周辐射传播,主要证据可归为以下几点:1)向西岭至桑梓一带海拔较高,野生大豆居群受人类破坏少,有利于野生大豆遗传多样性保存;2)向西岭周围均有野生大豆分布;3)此处是野生大豆的一个多性性中心,多样性中心往往是起源中心;4)遗传多样性最丰富的向西岭自然居群和其他野生大豆居群均有较大的基因流,而远离大冠岭居群基因交流较小;5)此处地势较高有利于种子借重力、雨水或风力从高向低传播;6)快乐洞(KLD)与茂家(MJ)分别位于海拔最高的向西岭(XXL)两侧,地理距离较远,但KLD自然居群的部分个体与MJ自然居群的个体却聚在同一组.湖南新田县大冠岭及其周围野生大豆空间分布上的这一规律为野生大豆扩散途径研究提供了一个样本.
3.3 湖南新田大冠岭及其周围区域野生大豆群体遗传结构模式
根据居群间基因交流方式,可以把群体遗传结构分为陆岛模式、海岛模式、阶石模式、距离隔离模式和层次模式[35-38].新田大冠岭及其周围的16个野生大豆居群遗传距离和地理距离明显相关,表明居群间的遗传结构模式符合距离隔离模式,可以认为原来新田大冠岭一带是一个均匀分布的大群体,16个野生大豆居群在遗传距离约为1.0处大冠岭地区野生大豆居群聚为一类,也佐证了大冠岭一带原来可能是一个大的群体.由于野生大豆是自花授粉作物,基因交流方式之一为种子雨,野生大豆种子95%散布在3.5 m以内[37].邻近的居群间基因交流的可能性要比相距较远的居群间大得多,而且由于基因频率在居群间呈现连续变化,居群间的基因频率差异小,导致居群间在地理位置和遗传距离之间呈密切相关.
调查时发现,远离大冠岭地区的野生大豆居群面积一般较小,所以又可以把大冠岭一带野生大豆居群看成是“陆”,而把远离大冠岭的居群看成是“岛”,这样,大冠岭一带居群和远离大冠岭的其他居群的遗传结构就为陆岛模式,“陆”基因向“岛”的迁移会影响岛基因频率的变化,由于“岛”较小,易产生漂变,同时也会降低居群的多样性.所以远离大冠岭居群与在大冠岭地区居群之间以陆岛模式存在.
[1]李荣堂.大豆古今考[J].农业考古,1982(2):84-85.
[2]刘广阳,齐宁,林红,等.黑龙江省野生和栽培大豆异黄酮与其组分相关性分析[J].植物遗传资源学报,2008,9(3):378-380.
[3]樊金萍,柏锡,李勇,等.野生大豆S-腺苷甲硫氨酸合成酶基因的克隆及功能分析[J].作物学报,2008,34(9):1581-1587.
[4]王臻昱,才华,柏锡,等.野生大豆GsGST19基因的克隆及其转基因苜蓿的耐盐碱性分析[J].作物学报,2012,38(6):971-979.
[5]王昌陵,王文斌,闫春娟,等.野生大豆蛋白质优异基因源种间杂交遗传规律及其应用研究进展[J].辽宁农业科学,2010(6):41-44
[6]全国野生大豆考察组.中国野生大豆资源考察报告[J]. 中国农业科学,1983,16(6):69-75.
[7]许东河,高忠,盖钧镒,等.中国野生大豆与栽培大豆等位酶、RFLP和RAPD标记的遗传多样性与演化趋势分析[J].中国农业科学,1999,32(6):16-22.
[8]盖钧镒,许东河,高忠,等.中国栽培大豆和野生大豆不同生态类型群体间遗传演化关系的研究[J].作物学报,2000,26(5):513-520.
[9]赵青松,赵云云,马启彬,等.广东乳源县大桥镇野生大豆自然居群遗传结构分析[J].华南农业大学,2014,35(1):37-42.
[10]HE Shuilian,WANG Yunsheng,VOLISS,et al.Genetic diversity and population structure:Implications for conservation of wild soybean(Glycine soja Sieb.et Zucc.)based on nuclear and chloroplast microsatellite variation[J].Int JMol Sci,2012,13(10):12608-12628.
[11]WEN Zixiang,DING Yanlai,ZHAO Tuanjie,et al.Genetic diversity and peculiarity of annual wild soybean(G.soja Sieb.et Zucc.)from various eco-regions in China[J].Theor Appl Genet,2009,119(2):371-381.
[12]GUO Juan,LIU Yifei,WANG Yunsheng,et al.Population structure of thewild soybean(Glycine soja)in China:Implications from microsatellite analyses[J].Ann Bot,2012,110(4):777-785.
[13]LIX H,WANG K J,JIA JZ.Genetic diversity and differentiation of Chinese wild soybean germplasm(G.soja Sieb.&Zucc.)in geographical scale revealed by SSR markers[J].Plant Breeding,2009,128(6):658-664.
[14]府宇雷,钱吉,张美云,等.金华地区野生大豆小种群的分子生态学研究[J].复旦学报:自然科学版,1999,38(5):584-586.
[15]王克晶,李福山,曹永生,等.河北省野生大豆种群若干数量性状结构特征[J].植物生态学报,2001,25(3):351-358.
[16]王丹,乔亚科,韩粉霞,等.河北东部沿海地区野生大豆SSR多样性分析[J].大豆科学,2010(4):555-558.
[17]陈红,张志良,王克晶,等.重庆地区野生大豆资源区域居群表型多样性初步分析[J].植物遗传资源学报,2010,11(5):573-577.
[18]王瑞珍,赵朝森,程春明,等.我国中部湖南湖北两省野生大豆种群表型多样性分析[J].江西农业学报,2009,21(12):1-4.
[19]程春明,杨存义,马启彬,等.江西野生大豆遗传多样性分析[J].植物遗传资源学报,2011,12(6):928-933.
[20]曾维英,梁江,陈渊,等.广西新收集野生大豆资源的遗传多样性分析[J].大豆科学,2011(3):379-383.
[21]WANG K J,LIX H.Genetic diversity and geographical peculiarity of Tibetan wild soybean(Glycine soja)[J].Genet Res Crop Evolution,2012,59(4):479-490.
[22]严茂粉,李向华,王克晶.北京地区野生大豆种群SSR标记的遗传多样性评价[J].植物生态学报,2008,32(4):938-950.
[23]赵茹,程舟,陆伟峰,等.基于分子标记的野生大豆居群遗传多样性估算与取样策略[J].科学通报,2006(9):1042-1048.
[24]关荣霞,刘秀敏,常汝镇,等.辽宁新宾县原位保护区野生大豆(Glycin Soja Sieb.et Zucc.)遗传多样性分析[J].高技术通讯,2006,16(1):67-72.
[25]孙晓环,刘晓冬,赵洪锟,等.吉林省龙井原位保护区野生大豆遗传多样性分析[J].大豆科学,2012(3):358-363.
[26]朱维岳,周桃英,钟明,等.基于遗传多样性和空间遗传结构的野生大豆居群采样策略[J].复旦学报:自然科学版,2006,45(3):321-327.
[27]JIN Yan,ZHANG Wenju,FU Daxu,et al.Sampling strategy within a wild soybean population based on its genetic variation detected by ISSRmarkers[J].Acta Botanica Sinica,2003,45(8):995-1002.
[28]DONG Y S,ZHUANG B C,ZHAO L M,et al.The genetic diversity of annualwild soybeans grown in China[J].Theor Appl Genet,2001,103:98-103.
[29]庄炳昌,王玉民,徐豹,等.中国不同纬度不同进化类型大豆的RAPD分析[J].科学通报,1994,39(23):2178-2180.
[30]周晓馥,庄炳昌,王玉民,等.利用RAPD与SSR技术进行野生大豆种群内分化的研究[J].中国生态农业学报,2002,10(4):10-13.
[31]赵青松,年海,杨存义.湖南新田野生大豆自然居群遗传多样性分析[J].西北植物学报,2009,29(11):2221-2227.
[32]CREGAN P B,JARVIK T,BUSH A L,et al.An integrated genetic linkage map of the soybean genome[J].Crop Sci,1999,39(5):1464-1490.
[33]PEAKALL R,SMOUSE P E.GenAlEx 6.5:Genetic analysis in Excel:Population genetic software for teaching and research-an update[J].Bioinformatics,2012,28(19):2537-2539.
[34]WANG K J,LIX H.Genetic characterization and gene flow in differentgeographical-distance neighbouring natural populations of wild soybean(Glycine soja Sieb.&Zucc.)and implications for protection from GM soybeans[J].Euphytica,2012,186(3):817-830.
[35]PRITCHARD JK,STEPHENSM,DONNELLY P.Inference of population structure usingmultilocus genotype data[J].Genetics,2000,155(2):945-959.
[36]FALUSH D,STEPHENSM,PRITCHARD JK.Inference of population structure using multilocus genotype data:Dominantmarkers and null alleles[J].Mol Ecol Notes,2007,7(4):574-578.
[37]曲若竹,侯林,吕红丽,等.群体遗传结构中的基因流[J]. 遗传,2004,31(3):377-382.
[38]YOSHIMURA Y,MIZUGUTIA,MATSUO K.Analysis of the seed dispersal patterns ofwild soybean as a reference for vegetation management around geneticallymodified soybean fields[J].Weed Biol Manag,2011,11(4):210-216.
【责任编辑周志红】

