布氏菌分离株16SrDNA基因遗传进化分析
刘志国,崔步云,王 妙,刘日宏,夏咸柱
布氏菌分离株16SrDNA基因遗传进化分析
刘志国1,2,崔步云3,王 妙2,刘日宏2,夏咸柱1,4
目的 检测内蒙古布氏菌临床分离株的16SrDNA基因序列,构建16SrDNA遗传进化树,确定该菌株的分类地位。方法 用BCSP31-PCR对临床分离株检测,再进行16SrDNA双向测序;从GeneBank下载标准参考菌株和与布氏菌有共同抗原细菌的16SrDNA基因序列;用Mega6.0进行序列比对并构建遗传进化树。结果 BCSP31-PCR结果显示均有223 bp扩增条带,16SrDNA测序得到单一基因序列;比对后发现有4处特异片段,经Blast比对,位于844-1224包括380 bp的片段为布氏菌独有;进化树显示布氏菌临床分离株与标准菌株聚在1个末端分支上,遗传距离接近0,无法精细区分相互关系;与人苍白杆菌聚集在1个亚分支上,遗传距离0.019;与其它试验菌株如霍乱弧菌、小肠结肠炎耶尔菌遗传进化距离较远,遗传距离>0.02。结论 从遗传进化角度对布氏菌分离株与标准菌株的亲缘关系进行分析,布氏菌所有种型遗传距离接近,提示布氏菌16SrDNA为高度保守序列,不宜做遗传进化分析的靶基因;布氏菌与人苍白杆菌有较近的亲缘关系;布氏菌属16SrDNA特有的380 bp的基因片段可作为布氏菌快速鉴定的靶序列。
布氏菌;16S rDNA;遗传进化;分析
布氏菌病(brucellosis)是由布氏菌引起的人兽共患传染病,世界范围广泛流行,在我国流行的主要是羊、牛种布氏菌;羊种菌造成人间布病的暴发,而牛种、猪种菌则多引起散发[1-3]。
近年,细菌的分类学方法发展飞速,已从单一的表型分类发展为系统性分类即利用表型、遗传型和系统发育信息等多种途径来区分细菌,进而阐述细菌的遗传进化关系。特以16S rDNA基因序列测定鉴定布氏菌并阐述其进化关系[4-5]。
1 材料与方法
1.1 材料
1.1.1 仪器试剂 生物安全柜(美国Thermo),梯度PCR扩增仪、PCR管离心机、高速离心机均为德国Sigma公司;电泳仪(北京八一仪器厂);凝胶成像分析系统(上海天能公司)。DL2000 Marker(全氏金生物公司);DNA提取试剂盒(天根生化科技公司);BCSP31-PCR引物由天一辉远生物公司完成。
1.1.2 菌株来源 牛种标准菌株544A羊种标准菌株16M,大肠杆菌O∶157DNA均来自中国疾控中心传染病所布病室。布氏菌临床分离株系2012-2014年分离自来乌兰察布市地方病防治中心就诊的布病患者血液样本,共20株,患者均来源于乌兰察布市的8个旗县,患者均有养羊史或羊接触史,菌株编号为:C1~C17,C19、C21和C22。
1.1.3 细菌DNA的提取 按照天根细菌基因组提取试剂盒推荐步骤进行。
1.1.4 扩增引物 布氏菌属聚合酶链式反应(BCSP31-PCR)扩增引物参照参考文献[6]。
1.2.3 测序 将BCSP31-PCR扩增后,20株代表性阳性菌株DNA送天一辉远生物公司进行16SrDNA全序列测定。
1.2.4 序列比对 用Mega6.06软件中的ClustalW功能,对试验菌株的序列比对,并查找特异序列。
1.2.5 遗传进化树的构建 将上述比对结果导入Mega6.06软件中,采用Neighbor-Joining法,构建试验菌株16S rDNA进化树。
2 结 果
2.1 布氏菌BCSP31-PCR扩增结果 BCSP31-PCR扩增结果电泳显示所有临床分离菌株均在约223 bp处有扩增条带,与参考菌株一致。
2.2 16S rDNA测序结果 测序取得了单一的核苷酸序列,然后根据测序峰值图删除两端可信度较差的序列,所有测序菌株均得到了约为1 350 bp大小的基因序列,上述序列经Blast比对显示:Expect为0.0;Identities为100%,证实该菌株序列与布氏菌的16S rDNA基因序列100%吻合。
2.3 布氏菌临床分离株、标准菌株和与布氏菌有共同抗原细菌的16S rDNA比对结果 用ClustalW软件做上述序列比对后发现临床分离株和布氏菌标准株在16S rDNA基因上差异甚微;以B.abortus544A(GeneBankNR:042460)为基准,在位置101-142(序列:5′-GGAGCGGCAGACGGGTGAGTAACGCGTGGGAACGTACCATTT-3′)、228-260(序列:TGATCGGCCCGCGTTGGATTAGCTAG
TTGGTGG)和844-1224(序列:5′-CGGGGTGTTTACACTTCGGTGGCGCAGCTAACGCATTAA
ACATTCCGCCTGGGGAGTACGGTCGCAAGA
TTAAAACTCAAAGGAATTGACGGGGGCCCG
CACAAGCGGTGGAGCATGTGGTTTAATTCG
AAGCAACGCGCAGAACCTTACCAGCCCTTG
ACATCCCGGTCGCGGTTAGTGGAGACACT
ATCCTTCAGTTAGGCTGGACCGGAGACAG
GTGCTGCATGGCTGTCGTCAGCTCGTGTC
GTGAGATGTTGGGTTAAGTCCCGCAACG
AGCGCAACCCTCGCCCTTAGTTGCCAGCA
TTCAGTTGGGCACTCTAAGGGGACTGCCGGTGATAAGCCGAGAGGAAGGTGGGGATGAC
GTCAAGTCCTCATGGCCC-3′)有3处特异性序列;对这3段特异性序列在NCBI做BLAST,发现位于844~1224处包含380 bp的核苷酸序列为布氏菌属基因特异性片段,见图1。
2.4 遗传进化树构建结果 以16S rDNA基因构建的上述试验的遗传进化树表明,布氏菌临床分离株与标准参考菌株紧密聚集在进化树的同一末端分支上,遗传距离几乎相同,无法精细区分相互关系;与人苍白杆菌聚集形成了分支,未与布氏菌属聚为一类,遗传距离约为0.019,提示有较近的亲缘关系;其他实验菌株如霍乱弧菌、巴尔通体,包括与布氏菌交叉凝集的耶尔森菌O∶9型,均聚集在了不同的分支中,提示遗传进化距离较远,亲缘关系较远。见图2。
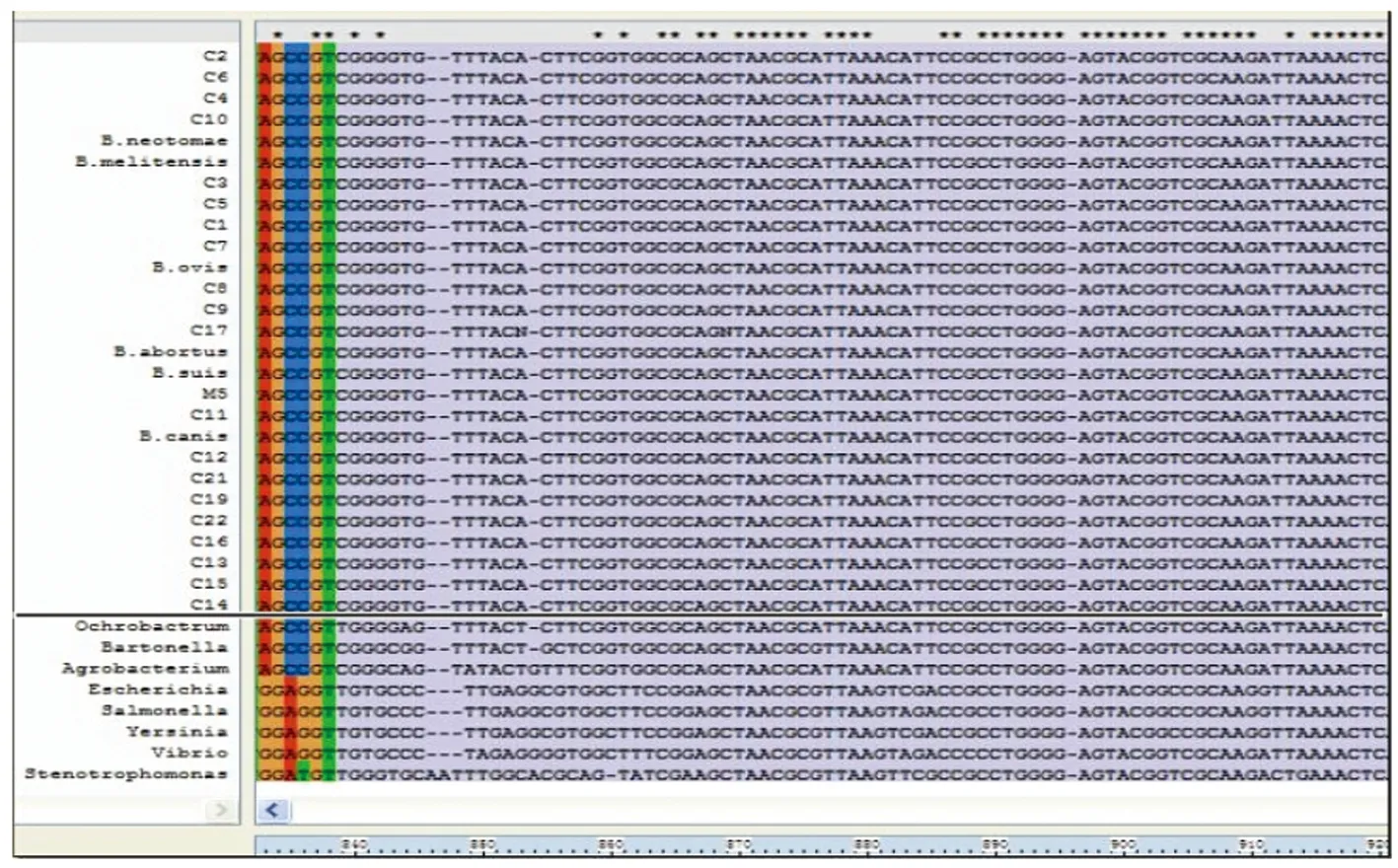
图1 布氏菌16S rDNA 844~1224处的部分特异序列
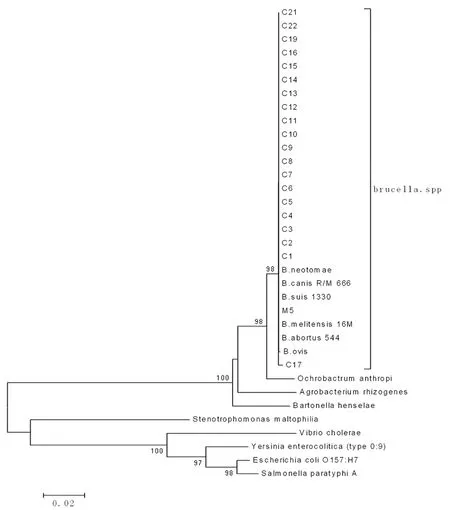
图2 布氏菌临床分离株16S rDNA遗传进化树
Fig.2 Phylogenetic tree of clinical isolates ofBrucellastrains 16S rDNA
3 讨 论
目前,布氏菌病诊断检测和基因特征分析的方法多样,血清学方法操作简单、快速适用,但由于许多菌株与布氏菌有共同抗原,容易发生交叉反应[7],导致误诊、漏诊;传统的微生物学检测方法操作繁琐、周期长、且有约10%~30%的菌株成为无法鉴定的非典型菌株[8]。所以,布氏菌的分离培养仍是布病感染的金标准,亦是进行基因研究和阐明遗传进化关系的基础。布氏菌16S rDNA基因序列分析,可以对布氏菌进行快速、准确的分类鉴定,还可用于确定菌株的分类地位、种系发生[9]。
细菌的16S rDNA 普遍存在于原核生物中,而且在生物进化的漫长历程中保持不变,可看作为生物演变的时间钟,其分子中既含有高度保守的序列区域,又有中度保守和高度变化的序列区域即含有具有交替的保守区域和异质性区域的特点[10],它适用于进化距离不同的各类生物亲缘关系的研究。本文对20株布氏菌进行16S rDNA序列测定及遗传进化分析,BCSP31-PCR的鉴定均为布菌属。BCSP31-PCR方法在属的水平上鉴定布氏菌菌株,是OIE推荐鉴定方法之一。16S rDNA基因序列测定,BLAST结果显与布氏菌属标准菌株序列相似性为100%;显示16S rDNA基因序列在布氏菌属中高度保守,与先前研究结果一致[11]。以牛种布氏菌的16S rRNA 全序列 (GenBank:NR_114469)为基准,在位置101-142 有3处为布氏菌属基因特异性片段,可作为快速分型鉴定的靶序列,可用于建立布氏菌的快速鉴定。进化树是用树状结构来表示物种或者序列之间的演化关系,揭示物种隐含的种系关系,在进行分子进化中研究重要意义。本试验基于试验菌株的16S rDNA基因,采用邻接法(NJ)构建了布氏菌及相关菌株的分子进化树。本试验构建的遗传进化树显示遗传距离为0.02,即试验菌株核苷酸序列同源性差异为2%即同源性为98%,代表试验菌株基因中每100个核苷酸/氨基酸中有2个不同;进化树的Bootstrap检验值均在97以上,表明该进化树可信度较高,一般该值大于70较为可信[12]。进化分析结果显示不同地域、年代、种型的临床分离株与标准参考菌株聚集在进化树的末端分支上,表明分离株均为布氏菌,但遗传距离很接近,无法精细的区分种型和相互关系,这与 Boye K等[13]的研究结果一致。进化树提示布氏菌16SrDNA为高度保守序列,适合种属鉴定,不宜做遗传进化分析的靶基因,应选择保守性相对较差的基因构建进化树;而布氏菌与人苍白杆菌聚集在了一个亚分支上,且16S rDNA基因同源性较高,遗传距离约为0.019,提示有较近亲缘关系,这与Velasc J等[14]的试验结果相吻合;布氏菌属与其它试验菌株如霍乱弧菌、小肠结肠炎耶尔菌等聚集在不同的进化分支上,遗传进化距离较远,提示亲缘关系相对较远。分离株C17在用常规试验方法鉴定时,鉴定为可疑布氏菌株,而16S rDNA序列测定结果显示该菌株为布氏菌属细菌,表明布氏菌16S rDNA序列测定在非典型株或可疑菌株的分类鉴定中具有较好的作用。
16SrDNA已广泛地应用于细菌遗传特征和分子差异的研究,尤其适用于临床中不能或不易培养的病原和未知病原的分类鉴定,大量已知细菌的16SrDNA数据都被测定并输入到国际基因数据库,使之成为对细菌鉴定和分类非常有用的参照系统;而且16SrDNA序列的相似性已成为划分属的标准,属内相似性不低于95%[15]。布氏菌16S rDNA序列测定不仅试验结果直观、分类鉴定准确、信息易于电子存档,便于构建数据库;而且能够进行布氏菌遗传进化、分子差异等方面的试验研究。
[1]Bureau of Disease Control and Prevention, Ministry of Health. Manual of brucellosis control[M]. Beijing: People’s Health Publish House, 2008. (in Chinese) 卫生部疾病预防控制局.布鲁氏菌病防治手册[M]. 北京:人民卫生出版社,2008:55.
[2]Liu ZG, Ren QH, Wang M, et al. Overview of specific serological detection technologies for brucellosis[J]. Chin J Zoonoses, 2013, 29(10): 1026-1031. (in Chinese) 刘志国,任清华,王妙,等.布病特异性血清学检测技术应用概述[J].中国人兽共患病学报.2013, 29(10): 1026-1031.
[3]Cui BY. Disease surveillance and control of chain brucellosis[J]. Dis Survill, 2007, 22(10): 649-651. (in Chinese) 崔步云.中国布鲁氏菌病疫情监测与控制[J].疾病监测,2007;22 (10): 649-651.
[4]Woese CR. Bacterial evolution[J]. Microbiol Rev, 1987, 51: 221-227.
[5]Jenkins C,Ling CL,Ciesielczuk HL. Detection and identification of bacteria in clinical samples by 16S rRNA gene sequencing: comparison of two different approaches in clinical practice[J]. J Med Microbiol, 2012, 61(Pt 4): 483-488. DOI:10.1099/jmm.0.030387-0
[6]Liu ZG, Cui BY, Liu RH, et al. Isolation and identification of human brucellosis pathogens in Ulanqab of Inner Mongolia[J]. Chin J Endemiology, 2014, 33(1): 49-52. (in Chinese) 刘志国,崔步云,刘日宏,等.内蒙古乌兰察布市人间布鲁杆菌病病原菌的分离鉴定[J]. 中华地方病学杂志,2014,33(1):49-52.
[7]Nielsen K. Diagnosis of brucellosis by serology[J]. Vet Microbiol, 2002, 90(1-4): 447-459.
[8]Cui BY, Yin JM, Li LY, et al. Typing ofBrucellaisolates by repetitive element sequence-base polymerase chain reaction[J]. Dis Survill, 2005, 20(8): 397-400. (in Chinese) 崔步云,尹继明,李兰玉,等.布鲁氏菌的Rep-PCR分型研究[J].疾病监测,2005,20(8): 397-400.
[9]Fox GE, Stackebrandt E, Hespell RB, et al. The phylogeny of prokaryotes[J]. Science, 1980, 209(4455): 457-463.
[10]Wolk D, Mitchell S, Patel R. Principles of molecular microbiology testing method[J]. Infect Dis Clin North Am, 2001, 15(4): 1157-1204.
[11]Tang X, Jiang H, Zhao HY, et al. Application of the 16S rDNA sequence analysis method in identification ofBrucella[J]. Microbiology, 2013, 40(7): 1290-1296. (in Chinese) 汤旭,姜海,赵鸿雁,等.16Sr DNA序列分析在鉴定布鲁氏菌中的应用[J].微生物学通报.2013, 40(7): 1290-1296.
[12]Xue ZQ. Tools for analysis of DNA and protein sequence data[M]. 3rd ed. Beijing: Science Press, 2014: 61. (in Chinese) 薛庆中,等.DNA和蛋白质序列数据分析工具[M].3版.北京:科学出版社,2014,06:61.
[13]Boye K, Hansen DS. Sequencing of 16S rDNA ofKlebsiella: taxonomic relations within the genus and to otherEnterobacteriaceae[J]. Int J Med Microbiol, 2003, 292(7-8): 495-503. DOI:10.1078/1438-4221-00228
[14]Velasco J, Romero C, Lopez-Goni I, et al. Evaluation of the relatedness ofBrucellaspp. andOchrobactrumanthropiand description ofOchrobactrumintermediumsp. nov., a new species with a closer relationship toBrucellaspp[J]. Int J Syst Bacteriol, 1998, 48 (Pt 3): 759-768. DOI:10.1099/00207713-48-3-759
[15]Woo PC, Lau SK, Teng JL, et al. Thenand now: use of 16SrDNA gene sequencing for bacterial identification and discovery of novel bacteria in clinical microbiology laboratories[J]. Clin Microbiol Infect, 2008, 14(10): 908-934. DOI:10.1111/j.1469-0691.2008.02070.x
s: Xia Xian-zhu, Email: xiaxzh@cae.cn; Cui Bu-yun, Email: cuibuyun@icdc.cn
Gene genetic evolution of 16SrDNA forBrucellastrains
LIU ZHi-guo1,2,CUI Bu-yun3,WANG Miao2,LIU Ri-hong2,XIA Xian-zhu1,4
(1.CollegeofVeterinaryMedicine,InnerMongoliaAgriculturalUniversity,Hohhot010018,China;2.UlanqabCentreforEndimicDiseaseControl,Ulanqab012000,China;3.NationalInstituteofInfectiousDiseasesControlandPrevention,ChineseCenterforDiseaseControlandPrevention,StateKeyLaboratoryforInfectiousDiseasePreventionandControl,CollaborativeInnovationCenterforDiagnosisandTreatmentofInfectiousDisease,Beijing102206,China;4.InstituteofMilitaryVeterinaryAMMS,Changchun130062,China)
We detected 16SrDNA gene sequence ofBrucellastrains of Inner Mongolia clinical isolates and constructed 16SrDNA phylogenetic tree, in order to determine the taxonomic status. Clinical isolates were identified by BCSP31-PCR, and then the 16SrDNA was bidirectionaly sequenced; 16SrDNA gene sequences ofBrucellareference strains and present common antigen bacteria withBrucellawere downloaded from the GenBank; sequence was aligned by Mega6.0 and phylogenetic tree of experimental strains was constructed. Results showed that 223 bp specific fragments were amplified from allBrucellaclinical isolates by BCSP31-PCR; single sequence was obtained by 16SrDNA sequencing. By the sequence alignment with other test strains, 4 location specific fragments were found, 380 bp gene fragment located at 844-1 224 position of 16SrDNA, which was unique ofBrucellaspp. Phylogenetic tree revealed thatBrucellaisolates and standard strains clustered in one terminal branch, and the genetic distance was approximately close to 0. It’s was unable to precise distinct between the relationship, as the evolutionary distance was much closed. TheBrucellaisolates gathered in a one sub-branch withOchrobactrumanthropi, the genetic distance was about 0.019.Brucellaspp. was closely related to theOchrobactrumanthropi. The relative distances with other strains (such asVibriocholerae,Yersiniaenterocolitis bacteria) were greater than 0.02. In conclusion, phylogenetic relationship ofBrucellastrains isolated and standard strains were analyzed from genetic evolution perspectively, the genetic distances of all specific type were close, which hinted that 16SrDNA ofBrucellawas highly conserved sequence and was not suitable as a target gene forBrucellagenetic evolution analysis.BrucellaandOchrobactrumanthropihad closer genetic relationship; 16SrDNA specific 380 bp gene fragment ofBrucellagenus could be used as target sequences for rapid identification ofBrucella.
Brucella; 16S rDNA; genetic evolution; analysis
10.3969/cjz.j.issn.1002-2694.2015.08.003
夏咸柱, Email: xiaxzh@cae.cn; 崔步云, Email: cuibuyun@icdc.cn
1.内蒙古农业大学兽医学院,呼和浩特 010018; 2.乌兰察布市地方病防治中心,乌兰察布 012000; 3.中国疾病预防中心传染病预防控制所 传染病预防控制国家重点实验室 感染性疾病诊治协同创新中心,北京 102206; 4.军事医学科学院军事兽医研究所,长春 130122
Funded by the Health Scientific Research Project of Health and Family Planing Commission of Inner Mongolia Autonomous Region (No. 201301094)
R378
A
1002-2694(2015)08-0700-04
2014-09-17;
2014-12-26

