超级杂交水稻种子内生细菌群落结构及其多样性
刘洋,赵燃,黎妮,曹艳花,张超,白飞荣,
张欣1,袁隆平1,王伟平1,3*,程池2*
1(杂交水稻国家重点实验室(湖南杂交水稻研究中心),湖南 长沙,410125) 2(中国食品发酵工业研究院 中国工业微生物菌种保藏管理中心,北京,100015) 3(南方粮油作物协同创新中心,湖南 长沙,410128)
超级杂交水稻种子内生细菌群落结构及其多样性
刘洋2,赵燃2,黎妮1,曹艳花2,张超3,白飞荣1,
张欣1,袁隆平1,王伟平1,3*,程池2*
1(杂交水稻国家重点实验室(湖南杂交水稻研究中心),湖南 长沙,410125) 2(中国食品发酵工业研究院 中国工业微生物菌种保藏管理中心,北京,100015) 3(南方粮油作物协同创新中心,湖南 长沙,410128)
通过采用构建16S rDNA克隆文库的非培养方法,对超级杂交水稻品种“深两优5814”及“Y两优900”种子内生细菌群落结构多样性进行研究。实验表明,“深两优5814”种子内生细菌含13个OUT,第一优势菌属为Pantoea,丰度为89.44%,第二优势菌和第三优势菌分别为Flavobacterium及Methylobacterium;“Y两优900”种子内生细菌含13个OUT,第一优势菌属为Pantoea,丰度为76.11%,第二优势菌和第三优势菌分别为Pseudomonas及Xanthomonas。由研究可见,泛菌属细菌为水稻种子中的优势菌群,同时,水稻种子基因型对其内生细菌的丰度及多样性具有一定的影响。
超级杂交水稻;种子内生细菌;非培养方法;群落多样性
水稻作为我国广泛种植的主要粮食作物,其年产量的保证对我国粮食安全具有重要作用。研究超级杂交水稻“深两优5814”及“Y两优900”两品种水稻种子的内生细菌群落结构多样性,可以为保障种子的健康活力,为种子生产与贮藏过程中的微生物控制与强化提供科学依据,对促进我国水稻种业发展具有重要的理论依据与实践意义。
能够定殖在健康的植物组织内,并与植物建立和谐联合关系的一类微生物称之为植物内生菌[1],它们能够通过多种方式影响植物的生长发育过程,例如通过生物防治抑制病原菌的生长,通过内生固氮等作用促进植物的生长[2]。植物种子是植物的延续器官,同时是农业生产资料中十分重要的一种[3],种子也是多种有益细菌的传递载体,许多植物病原菌也会在种子内部定殖。大量实验研究已证实,在植物种子的表面及其内部均有多种微生物存在,微生物群落十分丰富[4],在种子萌发的过程中,土壤中的微生物和存在于植物内部的细菌及真菌会起到促进植物生长的作用[5-6],其中不乏一些能够与植物协同互作的微生物,它们对提高土壤肥力和土壤酶的活性有显著作用,从而促进植物的健康生长[7]。ADAMS等[8]研究发现,在棉花种子萌发和发育成幼苗的过程中,定殖其内部的内生菌群落结构具有明显差异,这是由棉花品种不同,其在遗传亲缘关系、外部形态学和生理学特征方面的差异所造成的;NEAL等[10]研究基因型不同的小麦品种之间根际微生物群落结构的特点,发现一条染色体出现变异的小麦品种与未发生变异的小麦中根际微生物多样性不同,据此他提出了根际微生物类群的变化是由植物基因型决定的这一观点。目前,关于植物基因型与之联合的细菌的影响的相关研究尚集中于根和根际[8-13],与种子联合的微生物的相关研究还相对较少[14],且其中有关超级杂交水稻种子内生细菌的研究还鲜有报道,对于不同基因型杂交水稻种子内生细菌群落结构的研究,可为深入探讨植物基因型与其内部定殖的细菌群落之前的相关性提供理论依据和参考。因此本文以我国自主培育的超级杂交水稻品种“深两优5814”及“Y两优900”种子为研究材料,利用构建16S rDNA克隆文库的非培养方法,研究杂交水稻种子内生细菌区系和优势种群信息,并揭示其种子内生细菌群落结构与及其多样性,为指导超级杂交水稻种子生产与贮藏过程中的微生物控制与强化,提高产量以及保障种子的健康与活力提出科学依据。
1 材料与方法
1.1水稻种子的采集
试验样品为湖南杂交水稻研究中心提供的超级杂交水稻品种“深两优5814”和“Y两优900”种子,样品于2015年5月采自国家杂交水稻工程技术研究中心海南制种试验基地,样品于4 ℃保存。
1.2样品表面灭菌
先用无菌蒸馏水清洗水稻种子,将种子晾干后,依次用70%乙醇浸泡3 min,2.5%有效Cl-的次氯酸钠溶液浸泡5 min,70%乙醇浸泡30 s,接着用无菌蒸馏水淋洗种子5~7次,在无菌条件下放于滤纸上晾干[16];同时取120 μL最后一次的淋洗水涂布于LB固体平板上,28 ℃恒温培养72 h,检测水稻种子表面灭菌效果。
1.3样品总DNA提取
采用CTAB法[15]提取水稻种子样品基因组DNA,并将提取获得的DNA溶于30 μL无菌双蒸水中,-20 ℃保存。将总DNA稀释至50 ng作为PCR模板,进行下一步细菌16S rDNA 的PCR扩增。
1.4样品种子内生细菌的PCR扩增
以稀释后的水稻种子总DNA为模板,上游引物799f(5’-AACAGGATTAGATACCCTG-3’),下游引物1492r(5’-GGTACCTTGTTACGACTT-3’)[15]。PCR反应体系(50 μL)如下:50 ng模板DNA,1×Taqreaction buffer,dNTP 20 μmol,1.5单位Taq酶(Ferments),引物各20 pmol。反应程序为:94 ℃预变性5 min,94 ℃变性1 min,52 ℃复性1 min,72 ℃延伸1 min,30个循环后72 ℃延伸10 min。PCR产物约为750 bp。
1.516S rDNA克隆文库的构建
将扩增获得的PCR产物点于琼脂糖凝胶(2.0%胶浓度)电泳中,条带应位于700~800 bp之间,在紫外灯下切取条带,并用试剂盒进行胶回收,具体方法参照TIAN gel Midi Purification Kit(Tiangen,China)说明书步骤进行操作。回收后将得到的750 bp左右的DNA片段进行连接转化,具体方法参照pGEM-T Easy(Transgen,China)说明书步骤进行操作。
1.6克隆文库评价
用C值(coverage value)和aRarefactWin软件对克隆文库进行分析和评价。C值(计算公式为Cx=1-n1/N,其中N代表克隆文库的库容大小,n1代表在16S rDNA克隆文库中仅出现过一次的OTU的个数)理论上代表所构建16S rDNA克隆文库中所含有的微生物种类(OTU)占样品中全部微生物种类的比例[16-17],C值越大,克隆文库的代表性越强,从而能够反映出所构建的克隆文库内样品内生菌的代表程度。稀释曲线(rarefaction curve)是利用aRarefact Win软件对克隆文库进行分析和评价的方法,用已知的各种OTU的相对比例来推算抽取n个克隆时出现OUT数量的期望值,继而根据一组n值与其相对应的OTU数量的期望值所作出的曲线,当曲线达到平台期或趋于平缓时则可以认为库容已经足够。
1.7测序及相关分析
随机挑取160个克隆,并采用ABI 3730型DNA测序仪进行测序(ABI,USA)。将测序结果在EzTaxon server 2.1数据库中进行序列比对[18],收索与其同源性较高的近缘物种16S rDNA序列,并确定此序列与已知序列的同源关系,经比对序列相似性达到98.65% 以上的归为同一个OTU。
2 结果与分析
2.1种子内生细菌16S rDNA 的扩增及其序列测定
用CTAB法提取样品的总DNA,包括种子和内生细菌的基因组DNA,为了从DNA模板中扩增得到特异的内生细菌的16S rDNA,采用799f和1492r这一对引物[15],电泳结果显示,PCR产物为2条带,一条在1 000~1 500 bp,条带较暗,为水稻线粒体18S rDNA;在700~800 bp检测出较亮的条带,是包含有水稻种子内生细菌16S rDNA的目的条带,将该片段经回收纯化后,用于构建16S rDNA克隆文库。每个文库随机挑取200个克隆进行目的片段扩增,经PCR验证后,除去假阳性菌株并进行序列测定,之后将得到的序列信息提交到GenBank,登录号为KT012520-KT012548。
2.2克隆文库评价
“深两优5814”与“Y两优900”种子内生细菌的OUT数目均为13,这两个文库的C值分别为96.67%及98.33%。16S rDNA克隆文库的稀释曲线见图1。C值和稀释曲线均表明构建的两个水稻种子内生细菌克隆文库的库容已经足够,即该克隆文库已经可以基本代表“深两优5814”与“Y两优900”种子内生细菌群落的微生物多样性。
2.3种子内生细菌16S rDNA多样性及系统发育分析
“深两优5814”种子内生细菌群落分属Proteobacteria类群(179 clone,99.44%)及Actinobacteria(1 clone,0.56%),其中Proteobacteria包含13个OUT,Actinobacteria包含1个OUT。该群落优势属(丰度)为Pantoea(89.44%)、Flavobacterium(3.30%)及Methylobacterium(3.30%)(表1、表3)。水稻“Y两优900”种子内生细菌群落分属Proteobacteria类群(179 clone,99.44%)及Actinobacteria(1 clone,0.56%),其中Proteobacteria包含13个OUT,Actinobacteria包含1个OUT。该群落优势属(丰度)为Pantoea(76.11%)、Pseudomonas(7.22%)及Xanthomonas(6.11%)(表2、表3)。
“深两优5814”与“Y两优900”种子内生第一优势菌属相同,均为Pantoea,且丰度相当(89.44%、76.11%),优势现象十分明显;且两品种种子内生细菌所含OUT数目相同,均为14个OUT。同时,“深两优5814”与“Y两优900”种子内生细菌也存在明显的群落差异,5814种子第二优势菌和第三优势菌分别为Flavobacterium及Methylobacterium, 而900种子第二优势菌和第三优势菌分别为Pseudomonas及Xanthomonas,呈现内生细菌群落的多样性。

图1 杂交水稻种子内生细菌16S rDNA克隆文库的稀释曲线Fig.1 Rarefaction curve of the endophytic bacterial 16S rDNA clone library of rice seed
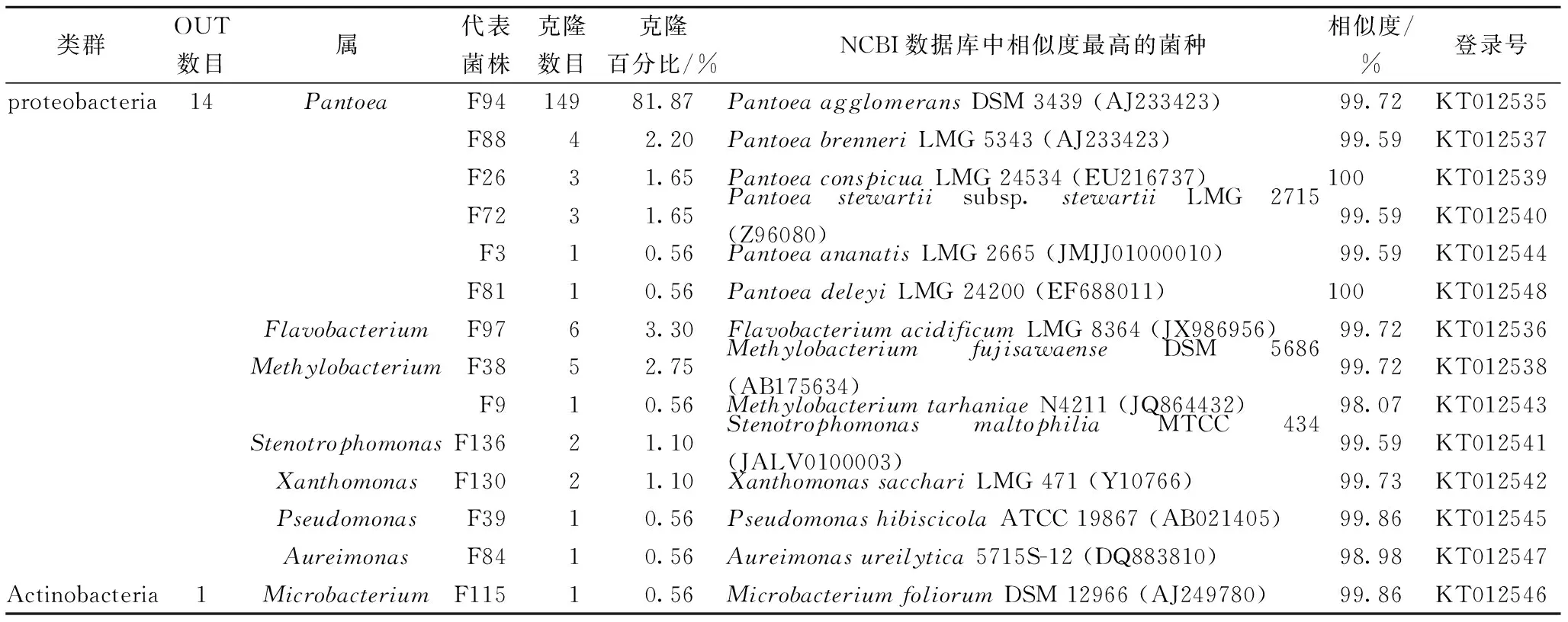
类群OUT数目属代表菌株克隆数目克隆百分比/%NCBI数据库中相似度最高的菌种相似度/%登录号proteobacteria14PantoeaF9414981.87PantoeaagglomeransDSM3439(AJ233423)99.72KT012535F8842.20PantoeabrenneriLMG5343(AJ233423)99.59KT012537F2631.65PantoeaconspicuaLMG24534(EU216737)100KT012539F7231.65Pantoeastewartiisubsp.stewartiiLMG2715(Z96080)99.59KT012540F310.56PantoeaananatisLMG2665(JMJJ01000010)99.59KT012544F8110.56PantoeadeleyiLMG24200(EF688011)100KT012548FlavobacteriumF9763.30FlavobacteriumacidificumLMG8364(JX986956)99.72KT012536MethylobacteriumF3852.75MethylobacteriumfujisawaenseDSM5686(AB175634)99.72KT012538F910.56MethylobacteriumtarhaniaeN4211(JQ864432)98.07KT012543StenotrophomonasF13621.10StenotrophomonasmaltophiliaMTCC434(JALV0100003)99.59KT012541XanthomonasF13021.10XanthomonassacchariLMG471(Y10766)99.73KT012542PseudomonasF3910.56PseudomonashibiscicolaATCC19867(AB021405)99.86KT012545AureimonasF8410.56Aureimonasureilytica5715S-12(DQ883810)98.98KT012547Actinobacteria1MicrobacteriumF11510.56MicrobacteriumfoliorumDSM12966(AJ249780)99.86KT012546
3 讨论
植物种子是植物的延续器官,同时是农业生产资料中十分重要的一种[3],种子是多种有益细菌的传递载体,许多植物病原菌也会在种子内部定殖,在种子表面及其内部均蕴藏着丰富的微生物群落资源[4]。目前对根际微生物已有较广泛及较深入的研究,但与种子相关微生物还处于初步研究阶段[14],而有关杂交水稻种子内生细菌群落结构多样性的研究还鲜有报道。本研究以我国新培育的超级杂交水稻品种“深两优5814”及“Y两优900”种子为研究对象,通过构建内生细菌16S rDNA克隆文库的非培养方法,对其内生细菌群落多样性进行研究,旨在探究具有遗传相关性的不同杂交水稻品种对其种子内生细菌群落结构的影响。

表2 “Y两优900”内生细菌多样性统计

表3 超级杂交水稻种子内生优势菌属及其丰度分析汇总
杂交水稻品种“深两优5814”及“Y两优900”在遗传上具有相关性,二者具有共同的母本和不同的父本,属同母异父。本研究发现,两品种种子内生细菌的第一优势菌种类相同,均为泛菌属细菌,它们在克隆文库中所占比例也保持一致,这与水稻种子的两个品种在遗传亲缘关系上较近有密不可分的关系,成团泛菌的优势现象十分明显说明该菌是水稻种子中的优势菌群,水稻种子中有着适宜成团泛菌生长的环境条件;同时,二者内生细菌的第二优势菌及第三优势菌均不相同,两品种杂交水稻的父本不同,在基因型上会显现出一定的差异,这直接导致了它们在内生细菌群落结构上有所不同。研究表明不同基因型的植物在内生细菌群落方面具有显著的差异性。Adams和Kloepper对不同棉花(Gossypiumspp.)栽培品种内生菌的研究发现,在棉花种子萌发和发育成幼苗的过程中,定殖其内部的内生菌群落结构具有明显差异,这是由棉花品种不同,其在遗传亲缘关系、外部形态学和生理学特征方面的差异所造成的[8]。Picard和Bosco也发现,玉米杂交子代与其亲本相比,在其能够吸引更多种类的假单胞菌微生物,这与子代玉米能够表达更丰富的多种蛋白质有关[19]。
从我们的实验结果中也可以看出,成团泛菌属在两品种水稻种子内生菌克隆文库中出现的丰度均较高,达到了80%以上,这一实验结果与前人对不同杂交组合的水稻种子内生细菌群落结构多样性的研究结果相一致[20],这也可以说明泛菌属细菌能够广泛在水稻植物中定植。泛菌属细菌属于肠杆菌科,革兰氏阴性菌,周生鞭毛,在自然界中分布广泛,从土壤、水中、植物表面、种子内部及动物和人的血液、尿液中均能分离得到,多数为与植物相联合的细菌,有的泛菌对植物能够起到促生作用,有的则对人及植物具有一定的致病性[21]。本实验数据表明,“深两优5814”及“Y两优900”两品种杂交水稻中第一优势菌均为成团泛菌(Pantoeaagglomerans),相似度达到了99.72%,且丰度很高,占整个克隆文库的80%左右。Yang等人曾在水稻“越富”的生长过程中对其内生菌多样性进行跟踪研究,发现其优势菌为成团泛菌[22],同时证明从其中分离得到的成团泛菌YS19能聚集形成称为“symplasmata”的结构[23];除此之外,也有研究表明成团泛菌可以通过其他途径对植物生长起到促进作用,例如具有固氮作用,能够分泌iP、ZR、HDZR等细胞分裂素和IAA、ABA、GA4等其它激素类物质;在水稻籽粒乳熟期前喷施YS19成团泛菌能够调控并促进水稻光合作用的产物在源、库中的分配[24]。
本研究利用非培养方法对超级杂交水稻“深两优5814”及“Y两优900”两品种种子内生细菌群落多样性进行研究,对于进一步研究植物内生细菌与其基因型相关性机理奠定了基础。同时,本研究对深入挖掘植物与其共生的内生细菌之间的互作关系提供了参考依据。从杂交水稻种子内生细菌的角度为切入点,对促进我国种业发展具有重要的理论与实践意义,但还需要不断对更多的杂交水稻品种及其亲本进行更全面的研究。
4 结论
本文对超级杂交水稻“深两优5814”及“Y两优900”两品种种子内生细菌群落多样性进行研究发现,作为同母异父的两品种种子,二者内生细菌的第一优势菌种类相同,均为泛菌属细菌,它们在克隆文库中所占比例也保持一致,这与水稻种子的两个品种在遗传亲缘关系上较近有密不可分的关系;成团泛菌的优势现象十分明显,说明该菌是水稻种子中的优势菌群,水稻种子中有着适宜成团泛菌生长的环境条件;同时,二者内生细菌的第二优势菌及第三优势菌均不相同,两品种杂交水稻在基因型上显现出的差异直接导致了它们在内生细菌群落结构上的不同。由本研究可见,泛菌属细菌为水稻种子中的优势菌群,同时,水稻种子基因型对其内生细菌的丰度及多样性具有一定的影响。
[1]KLOEPPER J W,BEAUCHAMP C J.A review of issues related to measuring colonization of plant roots by bacteria[J].Canadian Journal of Microbiology,1992,38(12):1 219-1 232.
[2]冯永君,宋未.植物内生细菌[J].自然杂志,2001,23(5):249-252.
[3]管康林.种子生理生态学[M].北京: 中国农业出版社,2009:1-7.
[4]NELSON E B.Microbial dynamics and interactions in the spermosphere[J].Annual Review of Phytopathology,2004, 42: 271-309.
[5]BACILIO-JIMÉNE M,AGUILAR-FLORES S,del Valle M V,et al.Endophytic bacteria in rice seeds inhibit early colonization of roots byAzospirillumbrasilense[J].Soil Biology and Biochemistry,2001,33(2):167-172.
[6]COTTYN B,REGALADO E,LANOOT B,et al.Bacterial populations associated with rice seed in the tropical environment[J]. Phytopathology,2001,91(3):282-292.
[7]BAREA J M, POZO M J, AZCON R,et al. Microbial co-operation in the rhizosphere[J]. Journal of Experimental Botany, 2005, 56(417): 1 761-1 778.
[8]ADAMS P D,KLOEPPER J W.Effect of host genotype on indigenous bacterial endophytes of cotton (GossypiumhirsutumL.)[J].Plant and Soil,2002,240(1):181-189.
[9]SIMON H M,SMITH K P,DODSWORTH J A,et al.Influence of tomato genotype on growth of inoculated and indigenous bacteria in the spermosphere[J].Applied and Environmental Microbiology,2001,67(2):514-520.
[10]NEAL J L,LARSON R I,ATKINSON T G.Changes in rhizosphere populations of selected physiological groups of bacteria related to substitution of specific pairs of chromosomes in spring wheat[J].Plant and Soil,1973,39(1):209-212.
[11]MICHIELS K,VANDERLEYDEN J,VAN Gool A. Azospirillum-plant root associations:a review[J]. Biology and Fertility Soils,1989,8(4):356-368.
[12]MAVINGUI P,LAGUERRE G,BERGE O,et al.Genetic and phenotypic diversity ofBacilluspolymyxain soil and in the wheat rhizosphere[J].Applied and Environmental Microbiology, 1992,58(6):1 894-1 903.
[13]SONG W,YANG H L,SUN X L,et al.The rice endophytic diazotroph and PGPR//Malik K A, Mirza M S,Ladha J K.Nitrogen Fixation with Non-Legumes[M].Kluwer Dordrecht: Academic Publishers,1998:41-48.
[14]CANKAR K, KRAIGHER H, RAVNIKAR M,et al. Bcterial endophytes from seeds of Norway spruce (PiceaabiesL. Karst) [J].FEMS Microbiology Letters, 2005, 244: 341-345.
[15]SUN L,QIU F B,Zhang X X,et al.Endophytic bacterial diversity in rice (OryzasativaL.) roots estimated by 16S rDNA sequence analysis[J].Microbiology Ecology,2008,55(3): 415-424.
[16]GOOD I J.The population frequencies of species and the estimation of population parameters[J].Biometrika,1953,40(3/4):237-264.
[17]MULLINS T D,BRITSCHGI T B,KREST R L,et al.Genetic comparisons reveal the same unknown bacterial lineages in atlantic and pacific bacterioplankton communities[J].Limnology and Oceanography,1995,40(1):148-158.
[18]CHUN J,LEE J H,JUNG Y,et al.EzTaxon: a web-based tool for the identication of prokaryotes based on 16S ribosomal RNA gene sequences[J].International Journal of Systematic and Evolutionary Microbiology,2007, 57:2 259-2 261.
[19]PICARD C,BOSCO M.Heterozygosis drives maize hybrids to select elite 2,4-diacethylphloroglucinol-producingPseudomonasstrains among resident soil populations[J].FEMS Microbiology Ecology,2006,58:193-204.
[20]邹媛媛,刘琳,刘洋,等.不同水稻品种种子固有细菌群落的多样性[J].植物生态学报,2012, 36(8):880-890.
[21]VIDEIRA S S,de ARAUJO J L S,RODRIGUES Lda S,et al.Occurrence and diversity of nitrogen- fixingSphingomonasbacteria associated with rice plants grown in Brazil[J].FEMS Microbiology Letters,2009,293:11-19.
[22]YANG H L,SUN X L,SONG W,et al.Screening, identification and distribution of endophytic associative diazotrophs isolated from rice plants[J].Acta Botanica Sinica,1999,41:927-931.
[23]FENG Y J,SHEN D L,DONG X Z,et al.Invitrosymplasmata formation in the rice diazotrophic endophytePantoeaagglomeransYS19[J]. Plant and Soil,2003,255:435-444.
[24]沈德龙,冯永君,宋未.内生成团泛菌YS19对水稻乳熟期光合产物在旗叶、穗分配中的影响[J].自然科学进展,2002,12:863-865.
Diversity of endophytic bacterial communities in seeds of super hybrid rice (OryzasativaL.)
LIU Yang2,ZHAO Ran2,LI Ni1,CAO Yan-hua2,ZHANG Chao3,BAI Fei-rong2,ZHANG Xin2,YUAN Long-ping1,WANG Wei-ping1,3*,CHENG Chi2*
1(National Key Laboratory of Hybrid Rice (Hunan Hybrid Rice Research Center),Changsha 410125,China) 2(China Center of Industrial Culture Collection, China National Research Institute of Food and Fermentation Industries,Beijing 100015,China) 3(Southern Regional Collaborative Innovation Center for Grain and Oil Crops in China, Changsha 410128,China)
The community diversities of endophytic bacteria in seeds of two types of super hybrid rice (“Shenliangyou 5814” and “Y Liangyou 900”) were studied through culture-independent method of the 16S rDNA clone library technique. Experimental result showed that the endophytic bacterial communities in “5814” included 13 OTUs, and the abundance of the first dominant bacteriumPantoeawas 89.44%, the second and third dominant bacterium wasFlavobacteriuandMethylobacterium. The endophytic bacterial communities in “900” included 13 OTUs, and the abundance of the first dominant bacteriumPantoeawas 76.11%, the second and third dominant bacterium wasPseudomonasandXanthomonas. From this study, we found thatPantoeawas the dominant genera in hybrid rice seeds, and the endophytic bacterial community structure had certain relevance to the seeds of hybrid rice which had been genetically related.
super hybrid rice; seed endophytic bacteria; bacterial diversity; culture-independent method
10.13995/j.cnki.11-1802/ts.201601006
博士,高级工程师(王伟平,程池教授级高级工程师为通讯作者,E-mail: wangweiping01@126.com;cheng100027@163.com)。
杂交水稻国家重点实验室开放课题基金(2014KF06);国家高技术研究发展计划(863计划)-强优势水稻杂交种的创制与应用(2011AA10A101);北京市科技新星计划项目(Z141105001814095)
2015-09-11,改回日期:2015-10-08

