基于CRISPR/Cas9系统的OsbHLH116基因编辑及其脱靶效应分析
杜彦修 季新 陈会杰 彭廷 张静 李俊周 孙红正 赵全志
(河南农业大学 农学院/河南粮食作物协同创新中心/河南省水稻生物学重点实验室, 郑州 450002; *通讯联系人, E-mail: qzzhaoh@126.com)
基于CRISPR/Cas9系统的OsbHLH116基因编辑及其脱靶效应分析
杜彦修 季新 陈会杰 彭廷 张静 李俊周 孙红正 赵全志*
(河南农业大学 农学院/河南粮食作物协同创新中心/河南省水稻生物学重点实验室, 郑州 450002;*通讯联系人, E-mail: qzzhaoh@126.com)
DU Yanxiu, JI Xin, CHEN Huijie, et al. CRISPR/Cas9 system-based editing ofOsbHLH116 gene and its off-target effect analysis. Chin J Rice Sci, 2016, 30(6): 577-586.
以水稻OsbHLH116 基因为编辑对象,根据基因编码区序列(CDS)在第一外显子区域设计长度为19 bp的sgRNA,化学合成sgRNA的寡核苷酸序列,然后与CRISPR/Cas9系统表达载体pBUN411连接,再用农杆菌介导法获得水稻转基因株系,最后利用酶切和测序相结合的方法对OsbHLH116 突变体进行了筛选鉴定和脱靶效应分析。结果表明,所构建的pBUN411-gRNA载体成功实现了对基因OsbHLH116 的定向编辑。酶切分析表明在选取的10株T0代转基因苗中得到了6个OsbHLH116 突变单株。对6个突变单株进行了TA克隆测序分析,发现了纯合突变、双等位突变和杂合突变3种类型。酶切分析表明2个潜在脱靶位点均未发生脱靶效应。
水稻; 基因编辑; CRISPR/Cas9; 脱靶效应;OsbHLH116
精确而简单的基因组编辑方法是研究基因功能的重要手段之一。最新发现的CRISPR/Cas (clustered regularly interspaced short palindromic repeats/CRISPR-associated protein) 系统因其简单和高效被广泛应用于包括植物在内的多种生物的基因组编辑中。其原理是Cas9蛋白与sgRNA形成复合体,切割与sgRNA上的间隔序列(spacers)互补的基因组 DNA,造成双链DNA损伤,随后通过体内的NHEJ (non-homologous end joining) 修复机制引入基因突变[1]。2013年,Cong等[2]利用靶点特异性的RNA (sgRNA) 将Cas9核酸酶带到基因组上的具体靶点,首次实现了在人类和小鼠细胞中内源基因的定点敲除。目前,CRISPR/Cas系统已经成功在8种植物中实现了基因组的定点编辑,且在水稻中获得了稳定的突变体植株[3, 4],其强大的基因组编辑功能得到充分体现。
碱性-螺旋-环-螺旋(basic-helix-loop-helix, bHLH)转录因子家族在动植物中广泛存在,其结构域约由60个氨基酸组成,包括碱性(Basic)区域和螺旋环螺旋(HLH)区域,碱性区域位于bHLH结构域N端,主要与DNA结合有关;HLH区域分布在bHLH的C端,主要由疏水氨基酸残基构成,利于HLH之间相互作用形成二聚体[5, 6]。bHLH转录因子碱性区域的某些氨基酸能够与基因启动子区域的E-box (5′-CANNTG-3′)结合,从而调控靶基因的表达[7, 8]。研究表明,水稻中至少有173个bHLH转录因子[9],参与众多代谢过程的调控,在水稻生长发育、形态建成、生物合成、信号传导和抗逆性方面起着重要作用[10-12]。
作者在前期研究中发现,红光处理后水稻胚乳中OsbHLH116 表达量极显著降低。本研究利用CRISPR/Cas9系统对水稻OsbHLH116 基因进行编辑,通过转化水稻,获得了OsbHLH116 突变体,并对其突变位点进行鉴定和脱靶效应分析,以期为深入研究其功能提供实验材料,同时也为其他研究者利用CRISPR/Cas9系统构建突变体提供技术参考。
1 材料与方法
1.1 试验材料
转基因受体材料为日本晴(Oryzasativassp.japonicacv. Nipponbare),CRISPR/Cas9载体pBUN411由中国农业大学陈其军教授课题组惠赠[13]。大肠杆菌感受态细胞DH5α、T4 DNA 连接酶、PCR产物回收试剂盒和质粒小提试剂盒购于北京天根生化科技有限公司。TaqMix和TA克隆试剂盒购于北京康为世纪生物科技有限公司。硫酸卡那霉素和氨苄青霉素钠购于上海生工生物技术有限公司。BsaⅠ和EarⅠ限制性内切酶购于NEB (New England Biolabs)有限公司。引物由苏州金唯智生物科技有限公司合成(表1)。
1.2 gRNA靶点设计及寡核苷酸链合成
根据水稻生物学网站(http://rice.plantbiology.msu.edu/)提供的OsbHLH116 (LOC_Os12g40730)基因序列设计gRNA靶点序列。设计原则如下:1)选择PAM (protospacer-adjacent motif) 序列 (NGG) 5′ 端的一段碱基序列作为原间隔序列,即敲除的靶位点;2)靶标DNA必须在外显子中,且尽量靠近所编码蛋白质的N′端;3) PAM 5′ 端尽量存在酶切位点,以方便后期突变体的检测;4)在水稻全基因组中比对候选序列,尽量避免同源序列出现,防止脱靶效应产生。
表1 本研究所用引物序列
Table 1. Primer sequence used in the study.

引物名称Primername序列Sequence用途UsagesgRNA-F5'-GGCGCCTCCATCGGAGGAAGAGA-3'靶序列合成ConstructionoftargetsitesgRNA-R5'-AAACTCTCTTCCTCCGATGGAGG-3'pBUN411-VF5'-CCATGAAGCCTTTCAGGACATGTA-3'载体构建验证VerificationofvectorconstructionpBUN411-VR5'-ACGCTGCAAACATGAGACGGAGAA-3'Basta-F5'-AAGCACGGTCAACTTCCGTA-3'除草剂基因验证VerificationofBtBasta-R5'-GAAGTCCAGCTGCCAGAAAC-3'OsbHLH116-F5'-GTTGATGTGGCAAGGAGGAG-3'靶点两侧序列扩增AmplificationoftargetregionOsbHLH116-R5'-TACGCACCAGACAGTTCACC-3'LOC_Os01g01380-F5'-ACAAGCAATGCAAATGTTGG-3'脱靶位点扩增Off-targetamplificationLOC_Os01g01380-R5'-CTCTTCGCCCACACCATC-3'Chr4:+30193341-F5'-AATAGATCACGCCGTCAACC-3'脱靶位点扩增Off-targetamplificationChr4:+30193341-R5'-CGAGACGAATCTTTTGAGCA-3'
靶点序列正义链和反义链的互补单链DNA设计,是在靶序列的5′ 端添加ggcg,靶序列互补链的5′ 端添加aaac,使其与质粒经BsaI 酶切后形成黏性互补末端。2条引物65℃下退火5 min形成互补双链DNA,直接用于后续的载体构建。
1.3 pBUN411-gRNA载体构建
对pBUN411质粒进行BsaI酶切,酶切体系包括pBUN411质粒2 μg,10×NEB缓冲液 5 μL,BsaI 10 U,加ddH2O至50 μL。酶切后加入如下连接体系:T4连接酶1 μL,10×缓冲液 1 μL,靶序列双链DNA 2 μL,线性化质粒pBUN411 6 μL。用热击法将重组质粒转入大肠杆菌DH5α,培养过夜后挑取单克隆,根据载体序列分别设计载体正反向引物pBUN411-VF和pBUN411-VR,对含有重组质粒的大肠杆菌进行菌落 PCR 验证,选取验证片段大小正确的抽提质粒并测序。
1.4 转基因水稻阳性鉴定和突变位点分析
表达载体转化农杆菌后侵染水稻愈伤组织,使用Nishimura等[14]报道方法进行转基因。转基因再生苗总DNA采用SDS法提取,利用抗除草剂(Basta)基因引物进行转基因阳性鉴定。以水稻阳性转基因苗DNA为模板,OsbHLH116-F和 OsbHLH116-R为引物, PCR扩增sgRNA靶向位点两侧序列,然后使用EarI进行酶切,酶切体系包括PCR产物5 μg,10×NEB缓冲液5 μL,EarI 20 U,加ddH2O至50 μL。37℃下酶切过夜,使用2%琼脂糖凝胶电泳检测突变单株。将酶切检测为突变单株的PCR产物直接测序初步验证,随后对测序出现套峰单株PCR产物连接pUC-T载体,进行TA克隆,随机选取5~9个单克隆进行测序。测序结果与基因组DNA进行序列比对,检测是否成功靶向水稻的OsbHLH116 基因。根据测序得到的各突变单株的DNA序列,使用BioEdit软件[15](http://www.mbio.ncsu.edu/BioEdit/bioedit.html)将编码区核苷酸序列转换成氨基酸序列并进行比对;使用GeneDoc软件[16](http://www.psc.edu/index.php/user-resources/software/genedoc)对比对结果进行编辑和描影,分析各突变单株氨基酸序列是否发生改变。
1.5 脱靶效应分析
用gRNA靶点序列在NCBI (http://www.ncbi.nlm.nih.gov/) 中进行比对,根据序列比对结果,挑选3′端含有PAM序列(NGG)且PAM序列前碱基与靶点序列同源性较高的2个位点作为潜在出现脱靶效应的位点,通过PCR扩增这2个位点,然后使用EarI进行酶切,酶切后使用2%琼脂糖凝胶电泳检测。
2 结果与分析
2.1 OsbHLH116靶位点的选择和pBUN411-gRNA表达载体构建
根据CRISPR/Cas9靶位点设计原则,在OsbHLH116 基因第一外显子区域设计19 bp的gRNA靶点序列CCTCCATCGGAGGAAGAGA,其中包含限制性内切酶EarI识别位点5′…▲(N)4GAAGAG…3′,在靶序列3′端为PAM序列TGG (图1-B)。退火形成的双链互补靶序列与载体pBUN411连接形成重组载体pBUN411-gRNA (图1-A)。pBUN411空载体扩增片段长度为1538 bp,经BsaI酶切后连接,靶序列插入扩增片段大小应为360 bp,菌落PCR鉴定1、3和4号菌落片段大小与预期结果一致(图2),证明pBUN411-gRNA表达载体构建成功。
2.2 OsbHLH116突变体的酶切鉴定
将pBUN411-gRNA表达载体利用农杆菌导入粳稻品种日本晴,以抗除草剂(Basta)基因为筛选标记鉴定筛选转基因阳性植株,选择其中10株阳性植株,在sgRNA靶向位点两侧设计引物,PCR扩增片段大小为613 bp。然后使用EarI进行酶切,如果靶位点序列未发生突变,EarI酶可以将PCR产物全部切开,得到一条430 bp和一条183 bp的条带(图3中泳道1、2、7和8),表明转基因植株1、2、7和8没有发生突变;如果PCR产物不能被EarI切开,琼脂糖凝胶电泳检测为613 bp的单一条带(图3中泳道4、6、9和10),表明两条同源染色体靶标区的EarI酶切位点识别序列均被破坏,EarI酶无法切割PCR产物,则转基因单株为纯合突变或双等位突变;如果PCR产物被EarI酶切后得到3条条带,一条为613 bp且不能被EarI酶切的突变DNA扩增条带,另外两条为未突变DNA扩增的PCR产物EarI酶切后产生430 bp和183 bp的条带(图3中泳道3和5),表明转基因植株3和5为OsbHLH116杂合突变。
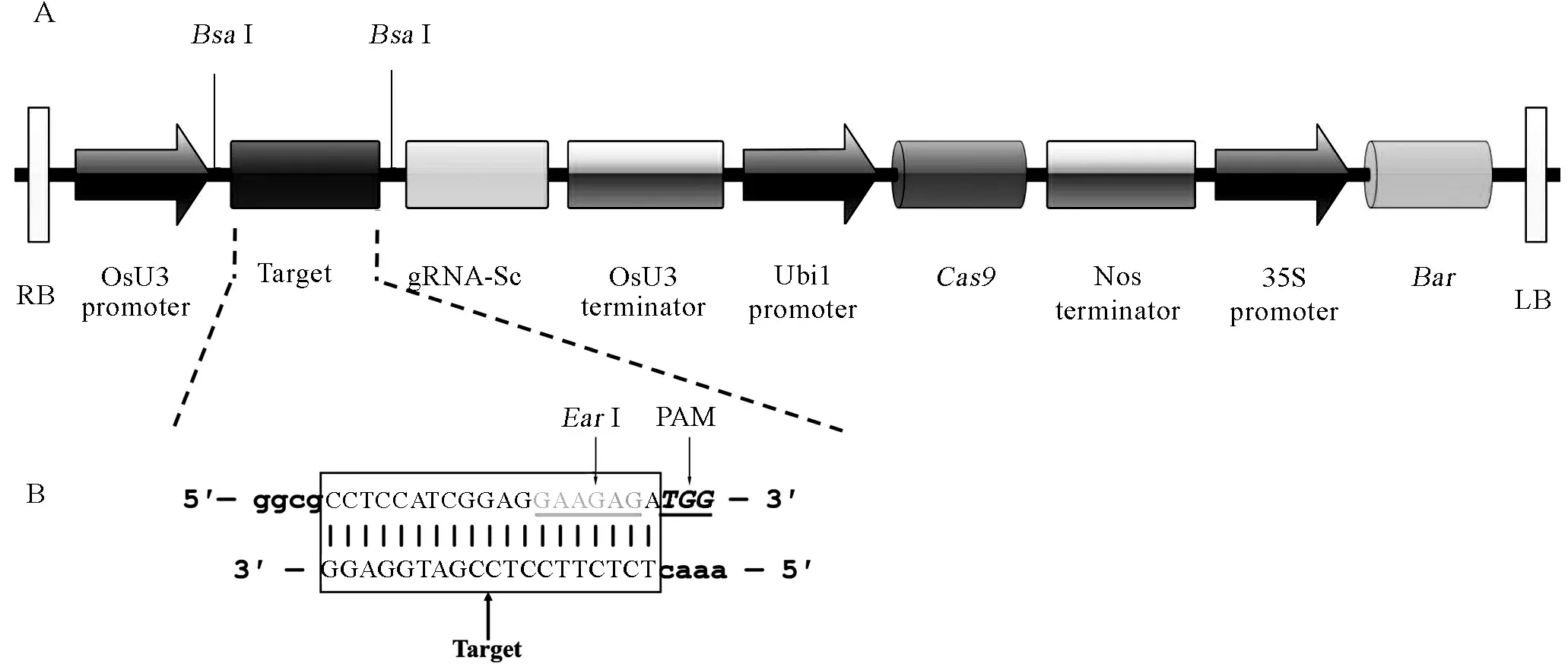
A-表达载体pBUN411-gRNA的LB和RB之间线性结构。 竖线-酶切位点BsaⅠ; RB-T-DNA右边界; OsU3 promoter-水稻U3启动子;gRNA-Sc-向导RNA支架; OsU3 terminator-水稻U3终止子; Ubi1 promoter-玉米泛素基因1(Ubi1)启动子;Cas9-Cas9基因; Nos terminator-农杆菌胭脂碱合成酶基因(Nos)终止子; 35S Promoter-花椰菜花叶病毒(CaMV)35S启动子;Bar-抗除草剂基因; LB-T-DNA左边界。B-gRNA靶序列互补双链DNA。方框内为gRNA靶点序列; 灰字体为EarⅠ酶切位点; 斜体为PAM序列(不在所构建载体上);小写字体为BsaⅠ酶切后黏性互补末端。
A, Physical map between RB and LB of pBUN411-gRNA expression vectors. Vertical lines stand forBsaⅠ restriction sites; RB, Right border of T-DNA; OsU3 promoter, Rice U3 promoters; gRNA-Sc, Guide RNA scaffold; OsU3 terminator, Rice U3 terminators; Ubi1 promoter, Maize ubiquitin gene promoter;Cas9,Cas9 gene; Nos Terminator,Agrobacteriumtumefaciensnopaline synthase gene (Nos) terminator; 35S Promoter,Cauliflowermosaicvirus(CaMV) 35S promoter;Bar, Herbicide resistanceBargene; LB, Left border of T-DNA. B, The target sites of gRNA complementary with double-stranded DNA. The target sequences are in the box; Gray letters stand forEarⅠ restriction sites; Letters with italics stand for PAM (not in the expression vectors); The lowercase stand for sticky ends ofBsaⅠ.
图1 pBUN411-gRNA表达载体LB和RB之间线性结构及gRNA靶序列合成
Fig. 1. Linear structure of pBUN411-gRNA expression vectors between LB and RB and the construction of target site.

1~4-表达载体的4个菌落PCR; 5-pBUN411空载体(对照); M-DM2000 标记。
1-4, Four independent colonies to detect expression vectors; 5, pBUN411 empty vectors (negative contrast); M, DM2000 marker.
图2 pBUN411-gRNA表达载体菌落PCR
Fig. 2. Verification of pBUN411-gRNA expression vectors via colony PCR.
2.3 OsbHLH116突变位点测序分析
为进一步验证OsbHLH116 突变单株,对酶切鉴定出的突变单株进行测序分析。图4-B中10号单株的PCR产物测序峰图,起始峰图正常,在靶序列突变位点附近开始出现套峰,表明该突变体为双等位突变体,而未突变的7号单株(图4-A)和4号纯合突变单株(图4-C)PCR产物测序峰图正常。对杂合突变(3号和5号)和测序出现套峰的双等位突变(6号和10号),进一步采用TA克隆的方法,进行了测序再验证(图5)。
综合分析测序结果,酶切鉴定的6个植株OsbHLH116 确实发生了突变(图5)。4号、6号、9号和10号植株为两条同源染色体靶标区均发生了突变,测序结果表明4号和9号为纯合突变,4号植株从PAM序列前第7个碱基开始向PAM方向缺失了6个碱基“GAAGAG”,9号植株从PAM序列前第7个碱基开始向PAM方向缺失了4个碱基“GAAG”。6号和10号植株虽然两条同源染色体靶标区均发生了突变,但突变不一致, 6号单株一条同源染色体靶标区的PAM前第5个碱基“A”缺失,另一条同源染色体靶标区PAM前2个碱基“GA”缺失;10号单株一条同源染色体靶标区在PAM前第3和第4碱基间插入1个碱基“G”,另一条同源染色体的PAM前2个碱基“GA”缺失。3号和5号为杂合突变,3号单株一条同源染色体靶标区的PAM前第7个碱基开始向PAM方向缺失了“GAAG”,5号单株一条同源染色体靶标区的PAM前第7个碱基开始向PAM方向缺失了4个碱基“GAAG”。 利用CRISPR/Cas9系统对OsbHLH116 基因进行编辑,发生了碱基缺失和插入2种类型,并且碱基缺失发生的概率较大;从突变位点看,突变主要发生在PAM前8个碱基内;从同源染色体突变情况看,发生了同源染色体突变一致的纯合突变(4号和9号)、同源染色体均突变但突变不一致的双等位突变(6号和10号)和只有一条同源染色体突变的杂合突变(3号和5号)3种情况。

1-10—10株转基因植株; WT-野生型; M-DM1000 标记;-RE-未进行酶切;+RE-使用EarI酶切; 箭头示酶切后PCR片段。
1-10, 10 transgenic lines; WT, Wild type; M, DM1000 marker; -RE, PCR product without restriction enzyme digestion; +RE,EarⅠ digested PCR product; Arrowheads, PCR fragments afterEarI digested.
图3 10株T0代转基因植株PCR产物EarⅠ酶切分析结果
Fig. 3. Mutation analysis of 10 T0transgenic lines by EarⅠ digestion of PCR product.
2.4 OsbHLH116突变位点氨基酸序列分析
根据测序获得的各突变单株OsbHLH116 CDS序列,使用BioEdit软件翻译成氨基酸并与野生型进行比对,结果如图6所示。4号突变单株由于缺失“GAAGAG”6个碱基,发生了整码突变,在62和63位置缺失了两个谷氨酸,其余氨基酸均无改变,bHLH结构域氨基酸序列亦无改变,因此4号植株OsbHLH116 发生的突变是否会对表型产生影响有待进一步的表型观察;6号、9号和10号突变单株缺失或插入的碱基造成了移码突变,使翻译均被提前终止。参考Li等[17]对OsbHLH116 基因bHLH结构域的预测结果,发现6号、9号和10号3个单株OsbHLH116 的 bHLH结构域已受到破坏;由于3号和5号植株为杂合突变,其中一条同源染色体能够正常翻译蛋白,需在后继世代突变单株中分析蛋白序列,因此本研究没有对3号和5号植株进行蛋白序列分析。本研究获得的OsbHLH116 突变体虽然蛋白序列发生了改变,但是否功能丧失,仍需进一步观察表型进行验证。
2.5 脱靶效应位点分析
由于PAM序列5′ 端碱基序列决定了gRNA的特异性,因此将gRNA靶序列在NCBI数据库中进行比对,获得了与gRNA同源性较高的2个可能出现脱靶效应的位点,分别位于基因LOC_Os01g01380内含子区域和第4染色体基因间区+30 193 341(Chr4:30193363..30193341)。这两个位点在紧邻PAM序列的5′ 端均有11~12个碱基的同源序列(图7)。通过PCR扩增脱靶位点序列,使用EarI限制性内切酶进行酶切分析脱靶情况(图8)。图8-A示基因LOC_Os01g01380扩增脱靶位点扩增片段大小为597 bp,10株转基因苗的PCR产物被全部酶切形成371 bp 和226 bp大小的片段,表明未发生脱靶;图8-B表明位点第4染色体+30 193 341靶序列扩增出557 bp的PCR产物均被酶切为大小为295 bp和 262 bp的小片段,表明该位置也未发生脱靶。

下划线-PAM序列TGG; 箭头-DNA预期被切割处; 方框—套峰。A-7号未突变单株PCR产物测序峰; B-10号双等位突变单株PCR产物测序峰; C-4号纯合突变单株PCR产物测序峰。
Underlines stand for PAM (TGG); The arrows stand for the intended cleavage site; The double peak phenomenon is in the box. A, Sanger sequencing results for PCR products of non-mutation number 7 ; B, Sanger sequencing results for PCR products of biallelic mutation number 10; C, Sanger sequencing results for PCR products of homozygous mutation number 4.
图4 4号、7号和10号单株PCR产物测序峰图
Fig.4. Sanger sequencing results for PCR products of number 4, 7 and 10.
3 讨论
基因定点编辑是研究基因功能的重要手段之一,锌指核酸酶(ZFNs)和类转录激活因子效应物核酸酶(TALENs)能够使DNA的靶位点产生DNA双链断裂进而实现基因组定点编辑[18, 19]。但ZFN制备复杂,成本昂贵,而TALENs对每一个基因位点的编辑都需要重新设计、合成和组装2个核酸酶,载体构建复杂,技术难度大,这也制约了其应用。2013年,一种全新的基因编辑技术CRISPR/Cas9出现,只需合成一个sgRNA就能实现对基因的特异性修饰,操作简单,成本低,作用高效[20]。目前在水稻中,利用CRISPR/Cas9技术已对OsBADH2、OsPDS[4]、OsLAZY1[3]和OsMYB1[21]等多个基因实现了定点编辑且得到了稳定遗传的突变植株。本研究基于CRISPR/Cas9系统,从水稻OsbHLH116 基因靶位点的选择,CRISPR/Cas9表达载体的构建、突变位点和脱靶位点的分析,系统阐述了利用CRISPR/Cas9技术构建OsbHLH116 基因突变体和验证方法,为研究OsbHLH116 基因功能奠定了材料基础,同时也为其他研究者利用CRISPR/Cas9构建突变体材料提供了技术参考。
蓝色字体-gRNA靶序列; 黄色高亮-PAM序列; 红色删除线-缺失碱基; 红色小写字体-插入碱基; -表示缺失;+表示插入;(0/0)-PCR产物测序。
The gRNA target site is shown in blue; The yellow highlighting denote PAM; Red dashes deleted bases; Insertion nucleotides are shown in red lowercases; -, Deletion; +, Insertion; (0/0)-Sequencing by using PCR product.
图5 野生型与6株突变体突变位点序列比对
Fig. 5. DNA sequence alignment of wild type with six mutant versions.
蓝色直线和方框-碱性-螺旋-环-螺旋结构域; 红色高亮-氨基酸缺失; 黄色高亮-翻译终止。
Blue lines and boxes denote basic-helix-loop-helix domain; The red highlighting denote amino acid deletion; The yellow highlighting denote translation termination.
图6 野生型与株突变体氨基酸序列比对
Fig.6. Comparison of the amino acid sequences between wild type and OsbHLH116 mutants.

方框内为PAM (NGG) 序列,红色字体为与靶序列比对不匹配碱基。
The PAM motif (NGG) is marked by black box; Mismatching bases are shown in red color.
图7 推定的脱靶位点序列与靶位点序列比对分析
Fig. 7. Sequences comparative analysis between the putative off-target sites and target sites.

1~10-10株转基因植株; WT-野生型; M-DM1000 标记; -RE-未进行酶切; +RE-使用EarI酶切; 箭头-酶切后PCR片段。A-基因LOC_Os01g01380脱靶位点酶切鉴定; B-Chr4:+30193341脱靶位点酶切鉴定。
1-10, 10 transgenic lines; WT, Wild type; M, DM1000 Marker; -RE, PCR product without restriction enzyme digestion; +RE,EarI digested PCR product; Arrowheads, PCR fragments afterEarI digestion. A, PCR/RE assay to detect off-target of LOC_Os01g01380; B, PCR/RE assay to detect off-target of Chr4: +30 193 341.
图8 10株T0代转基因植株脱靶位点酶切验证
Fig. 8. PCR/ restriction enzyme (PCR/RE) assay to detect off-target of 10 plants of T0transgenic lines.
本研究采用限制性核酸内切酶和测序相结合的方法来筛选鉴定突变体。Jinek等[22]证明CRISPR系统主要使靶DNA的PAM序列上游3~8 bp处形成双链断裂,当靶位点被切割后,相应的限制性内切酶识别序列会被破坏,以此来检测DNA靶标是否被切割。由于酶切位点序列多为4~6 bp,应用此方法可筛选大部分突变单株,但如果突变位点发生在酶切位点之外,就有可能发生漏判。限制性内切酶酶切PCR产物时,如果PCR产物浓度过高,上样量过大或者酶切时间较短而导致酶切不完全,可能会造成部分PCR产物未被切割而形成杂合子突变的假象造成误判。因此也可采用Surveyor酶切法[23]或T7E1酶切法[24]来检测CRISPR/Cas9系统编辑效果,这两种酶均可识别双链DNA中的错配碱基。但对于纯合突变,二者可能无法有效识别,另外T7E1酶除了能识别错配碱基外,还能识别十字型结构DNA、交叉DNA和异源双链DNA等,可能导致假阳性的出现。因此,本研究中将酶切法与测序相结合,高效、准确地对突变体进行了筛选鉴定,避免了以上问题的出现。
sgRNA的前导序列与基因组上的靶标序列直接结合来确定Cas9切割的部位,因而PAM序列前14个碱基主要负责CRISPR/Cas9系统的特异性[1],因此在基因编辑过程中可能存在一定程度的脱靶效应。研究表明,CRISPR/Cas9系统在植物中脱靶效应并不严重。张锋课题组通过全基因组测序和对可能脱靶位点的测序分析在拟南芥中未检测到任何的脱靶效应[25]。Zhang等[26]在水稻中对3个靶基因的11个潜在脱靶效应位点进行了检测,仅有1个靶基因的1个脱靶位点检测到了不到1/10的突变。鉴于存在潜在的脱靶效应,本研究选择了与PAM序列5′端11~12个碱基同源性较高的2个位点进行了酶切验证,在这两个位点均未检测到脱靶效应的产生,表明本研究中CRISPR/Cas9系统介导的靶点序列特异性较高。虽未检测出脱靶效应,但后续仍需对构建的独立基因敲除个体进行表型等综合分析和评判,以避免其他可能出现的脱靶效应产生干扰。如需要更全面准确地寻找脱靶位点,可通过全基因组测序的方法进行检测。对于脱靶产生的突变位点可通过与亲本多次回交的方法进行去除,其分离产生的含有脱靶位点突变的基因敲除个体还可作为其他突变材料加以研究和利用。
OsbHLH116 基因属于bHLH家族的转录因子,作者前期研究中发现,红光处理后胚乳中OsbHLH116 表达量极显著降低(数据未发表)。Li等[27]通过聚类分析推测OsbHLH116 功能和光敏色素互作因子OsPIL12 (OsbHLH103) 功能相近。Gu等[28]将强种壳休眠的杂草稻SS18-2中的主效休眠QTLqSD12定位在标记间距小于75 kb的区段内,预测OsbHLH116 等9个基因为候选基因。本研究基于CRISPR/Cas9系统对水稻OsbHLH116 基因进行了定向编辑并得到了相应突变体,这为深入研究OsbHLH116 在水稻生长发育中的功能提供材料。
[1] Wiedenheft B, Sternberg S H, Doudna J A. RNA-guided genetic silencing systems in bacteria and archaea.Nature, 2012, 482(7385): 331-338.
[2] Cong L, Ran F A, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems.Science, 2013, 339(6121): 819-823.
[3] Miao J, Guo D, Zhang J, et al. Targeted mutagenesis in rice using CRISPR-Cas system.CellRes, 2013, 23(10): 1233.
[4] Shan Q, Wang Y, Li J, et al. Targeted genome modification of crop plants using a CRISPR-Cas system.NatBiotechnol, 2013, 31(8): 686-688.
[5] Atchley W R, Terhalle W, Dress A. Positional dependence, cliques, and predictive motifs in the bHLH protein domain.JMolEvol, 1999, 48(5): 501-516.
[6] Nesi N, Debeaujon I, Jond C, et al. TheTT8 gene encodes a basic helix-loop-helix domain protein required for expression ofDFRandBANgenes inArabidopsissiliques.PlantCell, 2000, 12(10): 1863-1878.
[8] Massari M E, Murre C. Helix-loop-helix proteins: Regulators of transcription in eucaryotic organisms.MolCellBiol, 2000, 20(2): 429-440.
[9] Pires N, Dolan L. Origin and diversification of basic-helix-loop-helix proteins in plants.MolBiolEvol, 2010, 27(4):862-874.
[10]Komatsu K, Maekawa M, Ujiie S, et al. LAX and SPA: Major regulators of shoot branching in rice.PNAS, 2003, 100(20): 11765-11770.
[11]Sakamoto W, Ohmori T, Kageyama K, et al. The Purple leaf (Pl) locus of rice: The Plw allele has a complex organization and includes two genes encoding basic helix-loop-helix proteins involved in anthocyanin biosynthesis.PlantCellPhysiol, 2001, 42(9): 982-991.
[12]Seo J S, Joo J, Kim M J, et al. OsbHLH148, a basic helix-loop-helix protein, interacts with OsJAZ proteins in a jasmonate signaling pathway leading to drought tolerance in rice.PlantJ, 2011, 65(6): 907-921.
[13]Xing H, Dong L, Wang Z, et al. A CRISPR/Cas9 toolkit for multiplex genome editing in plants.BMCPlantBiol, 2014, 14(1): 327.
[14]Nishimura A, Aichi I, Matsuoka M. A protocol forAgrobacterium-mediated transformation in rice.NatProtoc, 2006, 1(6): 2796-2802.
[15]Hall T A. BioEdit: A user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT.NuclAcidsSymposSer, 1999, 41: 95-98.
[16]Nicholas K B, Nicholas H, Deerfield D W. GeneDoc: Analysis and visualization of genetic variation.EmbnewNews, 1997, 4: 1-4.
[17]Li X, Duan X, Jiang H, et al. Genome-wide analysis of basic/helix-loop-helix transcription factor family in rice andArabidopsis.PlantPhysiol, 2006, 141(4): 1167-1184.
[18]Carroll D, Morton J J, Beumer K J, et al. Design, construction and in vitro testing of zinc finger nucleases.NatProtoc, 2006, 1(3): 1329-1341.
[19]Li T, Liu B, Spalding M H, et al. High-efficiency TALEN-based gene editing produces disease-resistant rice.NatBiotechnol, 2012, 30(5): 390-392.
[20]Mussolino C, Cathomen T. RNA guides genome engineering.NatBiotechnol, 2013, 31(3): 208-209.
[21]Mao Y, Zhang H, Xu N, et al. Application of the CRISPR-Cas system for efficient genome engineering in plants.MolPlant, 2013, 6(6): 2008.
[22]Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity.Science, 2012, 337(6096): 816-821.
[23]Ran F A, Hsu P D, Lin C, et al. Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity.Cell, 2013, 154(6): 1380-1389.
[24]Zhou H, Liu B, Weeks D P, et al. Large chromosomal deletions and heritable small genetic changes induced by CRISPR/Cas9 in rice.NuclAcidsRes, 2014, 42(17): 10903-10914.
[25]Feng Z, Mao Y, Xu N, et al. Multigeneration analysis reveals the inheritance, specificity, and patterns of CRISPR/Cas-induced gene modifications inArabidopsis.ProcNatlAcadSci, 2014, 111(12): 4632-4637.
[26]Zhang H, Zhang J, Wei P, et al. The CRISPR/Cas9 system produces specific and homozygous targeted gene editing in rice in one generation.PlantBiotechnolJ, 2014, 12(6): 797-807.
[27]Li X, Duan X, Jiang H, et al. Genome-wide analysis of basic/helix-loop-helix transcription factor family in rice andArabidopsis.PlantPhysiol, 2006, 141(4): 1167-1184.
[28]Gu X, Liu T, Feng J, et al. The qSD12 underlying gene promotes abscisic acid accumulation in early developing seeds to induce primary dormancy in rice.PlantMolBiol, 2010, 73(1-2): 97-104.
CRISPR/Cas9 System-based Editing ofOsbHLH116 Gene and Its Off-target Effect Analysis
DU Yan-xiu, JI Xin, CHEN Hui-jie, PENG Ting, ZHANG Jing, LI Jun-zhou, SUN Hong-zheng, ZHAO Quan-zhi*
(AgronomyCollege,HenanAgriculturalUniversity/CollaborativeInnovationCenterofHenanGrainCrops/HenanKeyLaboratoryofRiceBiology,Zhengzhou450002,China;*Correspondingauthor,E-mail:qzzhaoh@126.com)
WithOsbHLH116 as the editable object, the 19-bp sgRNA was designed in the exon 1 site according to the coding sequence (CDS) and the oligonucleotides of sgRNA were chemically synthesized and inserted into linearized plasmid pBUN411. The transgenic lines were obtained byAgrobacterium-mediated transformation method. Identification of mutants and off-target effects analysis ofOsbHLH116 were conducted by combing restriction enzyme digestion with sequencing. The results showed that the recombinant vector of pBUN411-gRNA succeeded in oriented editing ofOsbHLH116. The restriction enzyme analysis results indicated that we got 6 mutants from 10 randomly selected transgenic lines. Sequence analysis of the six mutants indicated that homozygous mutation, biallelic mutation and heterozygous mutation occured. After searching the rice genome using the target sequence with PAM, two highly identical sites were found. However, we did not observe any mutation at these sites by restriction enzyme.
rice; gene editing; CRISPR/Cas9; off-target effects;OsbHLH116
2016-02-19; 修改稿收到日期: 2016-06-03。
河南省现代农业技术体系专项经费资助项目(S2012-04-02)。
Q755; S511.01
A
1001-7216(2016)06-0577-10
杜彦修,季新,陈会杰,等. 基于CRISPR/Cas9系统的OsbHLH116基因编辑及其脱靶效应分析. 中国水稻科学, 2016, 30(6): 577-586.

