紫花苜蓿MsSOS1基因的克隆与表达分析
方志红, 崔苗苗, 张 燕, 任彬琳, 石永红, 刘建宁,王运琦,董宽虎, 高洪文, 吴欣明*
(1. 山西省农业科学院畜牧兽医研究所, 山西 太原 030032; 2. 中国农业科学院北京畜牧兽医研究所, 北京 100193;3. 山西农业大学动物科技学院, 山西 太谷 030801)
盐胁迫是影响植物生长发育和产量的重要因素之一[1],全世界约有8亿公顷的土地被盐化,其中约有五分之一的可耕地受到盐胁迫的危害[2-3],土壤中的Na+是影响植物生长的主要因子,高浓度的Na+会对植物体造成生理性干旱和离子毒害[4],从而抑制植物的生长甚至会引起植物死亡[5-6],为了生存,植物会利用各种各样的机制来抵御环境中的有害因子[7-8]。SOS(salt overly sensitive)信号通路是植物抵御盐胁迫的一个至关重要的途径,它主要揭示了在盐胁迫下植物体内关于离子稳态调节和耐钠性的机理[9],SOS1、SOS2和SOS3在一条共同的SOS信号转导途径中起作用,通过对该通路中SOS1、SOS2和SOS3突变体的研究发现,SOS1(Salt Overly Sensitive 1)是植物在抵御盐胁迫的过程中必需的一个重要的、与植物耐盐性关系最直接的一个基因[4],它编码的SOS1蛋白可以将植物体内的多余的Na+进行区隔化或外排来降低细胞中的Na+浓度来适应高浓度的盐环境[3,10-12],从而降低盐胁迫的伤害[13-15]。
已经有很多研究表明,SOS1基因在植物的耐盐方面起着重要作用,它主要在介导Na+外排、参与Na+的长距离运输、介导K+的吸收或转运和调节细胞质pH几个方面起着重要的作用[16-18]。近些年来,质膜Na+/H+逆向转运蛋白(SOS1)在植物耐盐性研究中的作用受到广泛重视,SOS1基因最初是在拟南芥中被发现的,拟南芥SOS1突变体对盐超敏感,对其进行过表达发现可以提高转基因拟南芥的耐盐性,表明它在植物的耐盐机制中起着非常重要的作用[19-20]。其后许多植物的SOS1基因已经被克隆鉴定和进行了功能分析,如十字花科植物拟南芥(Arabidopsisthaliana)[21]、盐芥(Thellungiellasalsuginea)[22]、芥菜(Brassicajuncea)[23],杨柳科植物胡杨(Populuseuphratica)[24],藜科植物盐地碱蓬(Suaedasalsa)[25],番杏科植物海马齿(Sesuviumportulacastrum)[26],蒺藜科植物霸王(Zygophyllumxanthoxylum)[27],禾本科植物小麦(Triticumaestivum)[28]、水稻(Oryzasativa)[29]、大麦(Hordeumvulgare)[30]、獐茅(Aeluropuslittoralis)[31],茄科植物番茄(Lycopersiconesculentum)[32],豆科植物大豆(GlycinemaxandGlycinesoja)[33]和草木樨(Melilotusofficinalis)[34]。这些研究都可以表明,在盐胁迫下SOS1蛋白可以通过减少根部Na+的累积来提高植物的耐盐性。很多实验研究也都可以表明SOS1基因可以恢复并使其转化体的耐盐能力增强[26,28,33,35-41]。
虽然SOS1基因在很多植物中被克隆鉴定,但是在豆科植物中研究的比较少,只是在大豆和草木樨中被报道,紫花苜蓿SOS1基因的研究还未见报道。紫花苜蓿易于栽培、产量高,是可以为家畜提供优良蛋白饲料的重要豆科牧草[42-43],但是在盐碱地上不能大面积的种植[44],为了解决这一问题,利用生物技术手段培育抗逆性强的品种则成为一条切实可行的有效途径[45],本研究利用同源克隆的方法获得了紫花苜蓿MsSOS1基因的全长cDNA序列并对其进行了分析和不同逆境胁迫下的表达分析,以期为探讨SOS1基因所在的SOS途径在紫花苜蓿中的作用机制和合理开发利用紫花苜蓿培育抗逆新品种提供理论依据。
1 材料与方法
1.1 试验材料
以紫花苜蓿(Medicagosativa)种子(中苜1号,北京畜牧兽医研究所提供)为实验材料。实验所需的试剂为ExTaq(Takara)、DNA Maker(Takara)、pMD®18-Tvector(Takara),Total RNA(Invitrogen),RevertAidTMFirstStrand cDNA Synthesis Kit(Fermentas公司),大肠杆菌菌株E.coliDH5α(北京天根生化科技有限公司),凝胶DNA回收试剂盒(Axygen),SMARTTM RACE cDNA Amplification Kit(Clontech),In-Fusion®HD Cloning Kit(Clontech),其他常用试剂为国产分析纯。
1.2 试验方法
1.2.1材料培养与处理 挑选籽粒饱满且整齐度一致的紫花苜蓿种子,消毒后用灭菌蒸馏水冲洗2~3遍,播种于铺有滤纸的培养皿中,置于光照培养箱中培养,待长出两片子叶后,移至1/2Hoagland营养液中于培养箱中进行培养;培养箱温度为(28±2)℃/(23±2)℃,光照时间16 h·d-1,光照强度约600 μmol·m-2·s-1,相对湿度60%~80%。培养4周后选取长势一致的紫花苜蓿幼苗进行胁迫处理,每个处理3次重复。干旱和ABA处理分别用25% PEG6000和100 μmmol·L-1ABA处理0,12,24,48,72 h,冷胁迫(4℃)处理0,4,8,12,24 h后,取其叶片;组织表达特异性分析是分别采取同一株紫花苜蓿上的根、茎、叶和花等组织,-80℃保存备用。
1.2.2总RNA的提取及cDNA第一链的合成 参照Trizol总RNA抽提试剂盒说明书(Invitrogen公司)提取1.2.1中样品的总RNA,经1%琼脂糖凝胶电泳检测RNA质量后进行cDNA的合成(cDNA Synthesis Kit,Fermentas公司),产物于-20℃保存备用。
1.2.3引物的设计及合成 根据NCBI上查找到的其它物种的SOS1基因序列进行同源性比对,排除相似性不高的序列,找到高度保守的区域,利用Primer 5.0软件设计特异性引物MsSOS1-F和MsSOS1-R(表1),送英俊公司合成。
1.2.4紫花苜蓿MsSOS1基因保守区片段的克隆 PCR扩增体系为(10 μl体系):10×Ex Taq Buffer 1 μl,dNTP Mixture 1 μl,cDNA(<500 ng) 0.5 μl,TaKaRa Ex Taq(5 U/μl)0.1 μl,引物MsSOS1-F(10 μm)1 μl,MsSOS1-R(10 μm)1μl,ddH2O 5.4 μl。扩增条件为:94℃ 5 min,94℃ 30 s,56℃ 60 s,30 cycles;72℃ 10 min。扩增产物回收后连接到载体pMD®18-T上,转化DH5α后挑取阳性克隆测序。
1.2.5紫花苜蓿MsSOS1基因全长克隆 根据克隆得到的紫花苜蓿MsSOS1基因中间片段设计3′-RACE引物MsSOS1-F1和5′-RACE引物MsSOS1-F2(表1),进行全长PCR的扩增,获得的PCR产物连接载体进行转化,并挑取阳性克隆送公司测序。

表1 实验中的引物Table 1 Primers used in the experiment
1.2.6紫花苜蓿MsSOS1基因的生物信息学分析 利用软件DNAStar进行紫花苜蓿MsSOS1基因ORF的查找和氨基酸翻译;利用ExPASy工具对MsSOS1蛋白进行理化性质、高级结构和生物学功能的预测分析、信号肽预测、亚细胞定位情况进行分析;利用DNAMAN6.0进行序列的同源性比对分析,MEGA 5.10软件构建系统进化树(NJ法)。
1.2.8Real-time PCR检测紫花苜蓿MsSOS1基因的组织表达特异性 根据紫花苜蓿MsSOS1基因的全长序列设计Real-time PCR引物qRT-MsSOS1-F、qRT-MsSOS1-R(表1),以Actin基因为内参设计特异性引物Actin-F、Actin-R(表1)。使用Bio-Rad CFX96进行Real-time PCR扩增,扩增程序为:94℃ 2min,94℃ 5sec,58℃ 15sec,72℃ 10sec,45个循环,每个样品3次重复。
利用2-△△Ct方法[46]中的△△Ct=(CtTarget-CtActin)处理-(CtTarget-CtActin)对照公式计算紫花苜蓿根、茎、叶、花中MsSOS1基因的表达量并绘出柱形图。
2 实验结果与分析
2.1 紫花苜蓿MsSOS1基因中间片段的获得
以反转录得到的cDNA为模版,PCR扩增得到约784 bp的目的片段(如图1所示),将PCR结果进行回收、连接、转化并测序,测序结果进行Blast比对分析,结果显示获得的目的片段序列与鹰嘴豆(Cicerarietinum)、大豆(Glycinemax)、花生(Arachisipaensis)、绿豆(Vignaradiata)等基因的同源性达到80%以上,据此推断该序列是紫花苜蓿MsSOS1基因的一部分,可以进行后续实验。
2.2 紫花苜蓿MsSOS1基因5′端序列和3′端序列的获得
根据获得的目的片段设计RACE引物进行PCR扩增,获得了5′端片段序列(约1 785 bp)和3′端片段序列(约2 712 bp)(如图1所示),利用软件将获得的5′端序列、3′端序列和中间序列进行拼接,最终获得紫花苜蓿MsSOS1基因的全长cDNA序列(约3 757 bp)。设计ORF(开放阅读框)引物进行PCR扩增,获得约3 000 bp的特异性目的条带,测序后确定该序列为紫花苜蓿MsSOS1基因ORF序列(约2 580 bp)。
2.3 紫花苜蓿MsSOS1蛋白的生物信息学分析
2.3.1紫花苜蓿MsSOS1基因全长序列及蛋白的理化性质分析 紫花苜蓿MsSOS1基因全长cDNA序列长3 757 bp,包含922 bp的5′非编码区,2 580 bp的开放阅读框(ORF)和255 bp的3′非编码区(图2),编码一个分子式为C4337H6816N1156O1258S31、相对分子量为96.3 kDa、理论等电点为6.51的氨基酸序列(859 aa)(表3)。含有碱性氨基酸(K,R)88个;酸性氨基酸(D,E)94个;疏水氨基酸(A,I,L,F,W,V)320个;极性氨基酸(N,C,Q,S,T,Y)218个。该蛋白中Leu(97个,11.3%),Ser(86个,10.0%),Val(59个,6.9%),Glu(58个,6.8%)和Ala(58个,6.8%)相对含量比较多;Asp+Glu为94,Arg+Lys为88;亲水性平均数为-0.075,脂肪指数(AI)为95.23,蛋白质三级结构不稳定系数(II)为47.13,表现为不稳定状态,由此我们可以推测其可能为不稳定的酸性蛋白质。

图1 紫花苜蓿MsSOS1基因cDNA克隆过程的PCR产物电泳图谱Fig.1 Electrophoresis in the process of MsSOS1 cDNA cloning注:M为DL5000 DNA Marker;1为RT-PCR产物;2为5′-RACE PCR产物;3为3′-RACE PCR产物;4为MsSOS1基因全长PCR产物Note:M:DL5000 DNA Marker;1:RT-PCR product of MsSOS1 gene;2:5′-RACE PCR product of MsSOS1 gene;3:3′-RACE PCR product of MsSOS1 gene;4:The full-length PCR product of MsSOS1 gene

图2 紫花苜蓿MsSOS1基因cDNA及其推导氨基酸序列Fig.2 Nucleotide sequence and predicted amino acid sequence of MsSOS1注:左侧和右侧的数字分别代表核苷酸和氨基酸位点,绿色区域为起始密码子(ATG)和终止密码子(TGA),黄色区域代表3′及5′非编码区,下划线为预测的跨膜区域Note:The nucleotide sequences and amino acid residues are indicated by numbers of the left and right. The start codon(ATG) and the stop codon(TGA) were green shaded. The red shaded was 3′-UTR and 5′-UTR;The underline was predictive transmembrane region
2.3.2紫花苜蓿MsSOS1蛋白的同源性分析 与其他植物SOS1蛋白的同源性比较结果显示,紫花苜蓿MsSOS1蛋白与鹰嘴豆(Cicerarietinum)、大豆(Glycinemax)、羽扇豆(Lupinusangustifolius)、花生(Arachisipaensis)、葡萄(Vitisvinifera)等的同源性相对较高(表2),由此可以确定该序列确实是紫花苜蓿SOS1基因,命名为MsSOS1。

表2 紫花苜蓿SOS1蛋白与其它植物SOS1蛋白的同源性分析Table 2 Homology analysis of the MsSOS1 withSOS1 from some higher plants
2.3.3紫花苜蓿MsSOS1蛋白二级结构预测 对紫花苜蓿MsSOS1蛋白二级结构进行预测发现其主要由α-螺旋、β-转角、无规则卷曲和β-折叠组成(图3),其中以α-螺旋为主,占46.22%;其次是无规则卷曲,占37.25%;β-转角含量最少,仅占5.01%。
2.3.4紫花苜蓿MsSOS1蛋白的保守结构域分析 运用生物信息学软件对MsSOS1蛋白的保守结构域进行分析,结果发现紫花苜蓿MsSOS1蛋白的N-端含有NhaP、Na+/H+Exchanger结合位点和b-cpal、Na+/H+Exchanger superfamily等保守结构域,C-端含有cNMP_binding结合位点和Crp、CAP_ED、CAP_ED superfamily、Crp superfamily等保守结构域,表明该基因应该是Na+/H+转运蛋白家族中的一员(图4)。
2.3.5紫花苜蓿MsSOS1蛋白的跨膜结构分析 对紫花苜蓿MsSOS1蛋白的跨膜结构进行分析发现,MsSOS1蛋白的N端和C端均位于细胞外,含有4个跨膜区域,其跨膜区氨基酸序列位置分别为29-51,63-85,100-118,138-160(图5);对信号肽位点进行分析发现,紫花苜蓿MsSOS1蛋白无信号肽位点存在(图6)。

图3 MsSOS1蛋白的二级结构Fig.3 Predicted secondary structure for MsSOS1 protein

图4 MsSOS1蛋白的功能结构域Fig.4 The conserved domains of the MsSOS1 protein

图5 MsSOS1蛋白的跨膜区分析Fig.5 Analysis of transmembrane helicesof MsSOS1 protein

图6 MsSOS1蛋白信号肽预测分析NN-法Fig.6 Analysis and prediction of signal peptideof MsSOS1 based on NN-method
2.3.6紫花苜蓿MsSOS1蛋白的疏水性分析 运用ProtScale程序对紫花苜蓿MsSOS1蛋白的疏水性进行,发现该蛋白中含有疏水区域,N-末端具有4个跨膜区,C-末端有一个很长的亲水性尾部,面向细胞膜内腔(图7),这与先前报道过的拟南芥(Arabidopsisthaliana)、水稻(Oryzasativa)、獐茅(Aeluropuslittoralis)的SOS1蛋白的研究结果相似[26-27,30]。

图7 MsSOS1蛋白疏水性分析Fig.7 Analysis of hydrophobility of MsSOS1 protein
2.3.7紫花苜蓿MsSOS1蛋白亚细胞定位预测 对紫花苜蓿MsSOS1蛋白进行亚细胞定位结果显示:denoplasmic reticulum(47.8%) > plasma membrane(26.1%) > nuclear(4.3%) > vesicles of secretorysystem(4.3%) > vacuolar(4.3%) > mitochondrial(4.3%) > cytoplasmic(4.3%) > Golgi(4.3%),由此可以预测紫花苜蓿MsSOS1蛋白可能主要定位于内质网中。
2.3.8紫花苜蓿MsSOS1蛋白的氨基酸序列多重比对及系统进化树分析 将紫花苜蓿MsSOS1蛋白序列同Genbank上查找的其他已知物种的SOS1蛋白序列进行多重比对发现,MsSOS1蛋白与豆科植物鹰嘴豆(Cicerarietinum)CaSOS1(XM_004504555.2)、大豆(Glycinemax)GmSOS1(NM_001258010.1)和绿豆(Vignaradiata)VrSOS1(KC855193.1)的同源性非常近,在85%以上,分别为91%、87%、85%(图8)。
利用MEGA5.0软件对紫花苜蓿MsSOS1蛋白同其他植物的Na+/H+逆向转运蛋白进行系统进化树分析发现,紫花苜蓿MsSOS1蛋白与已经报道的具有Na+/H+逆向转运蛋白运输功能的几种植物的同源性很近,而与定位在内膜系统上的NHX类的Na+/H+逆向转运蛋白的同源性较低(图9),这些都可以说明MsSOS1可能是质膜型Na+/H+逆向转运蛋白(SOS1),可能与紫花苜蓿的耐盐性有着密切的关系。

图8 紫花苜蓿MsSOS1与其它植物SOS1蛋白的多重比对分析Fig.8 Multiple alignment analysis of MsSOS1 with other plant SOS1 proteins注:多重比对采用DNAMAN软件进行。Ms:紫花苜蓿Medicago sativa;Ca:鹰嘴豆Cicer arietinum;Ai:花生Arachis ipaensis;Gm:大豆Glycine max;Vr:绿豆Vigna radiata。横线表示跨膜区Note:The sequence were aligned with DNAMAN software;The transverse line was transmembrane helices
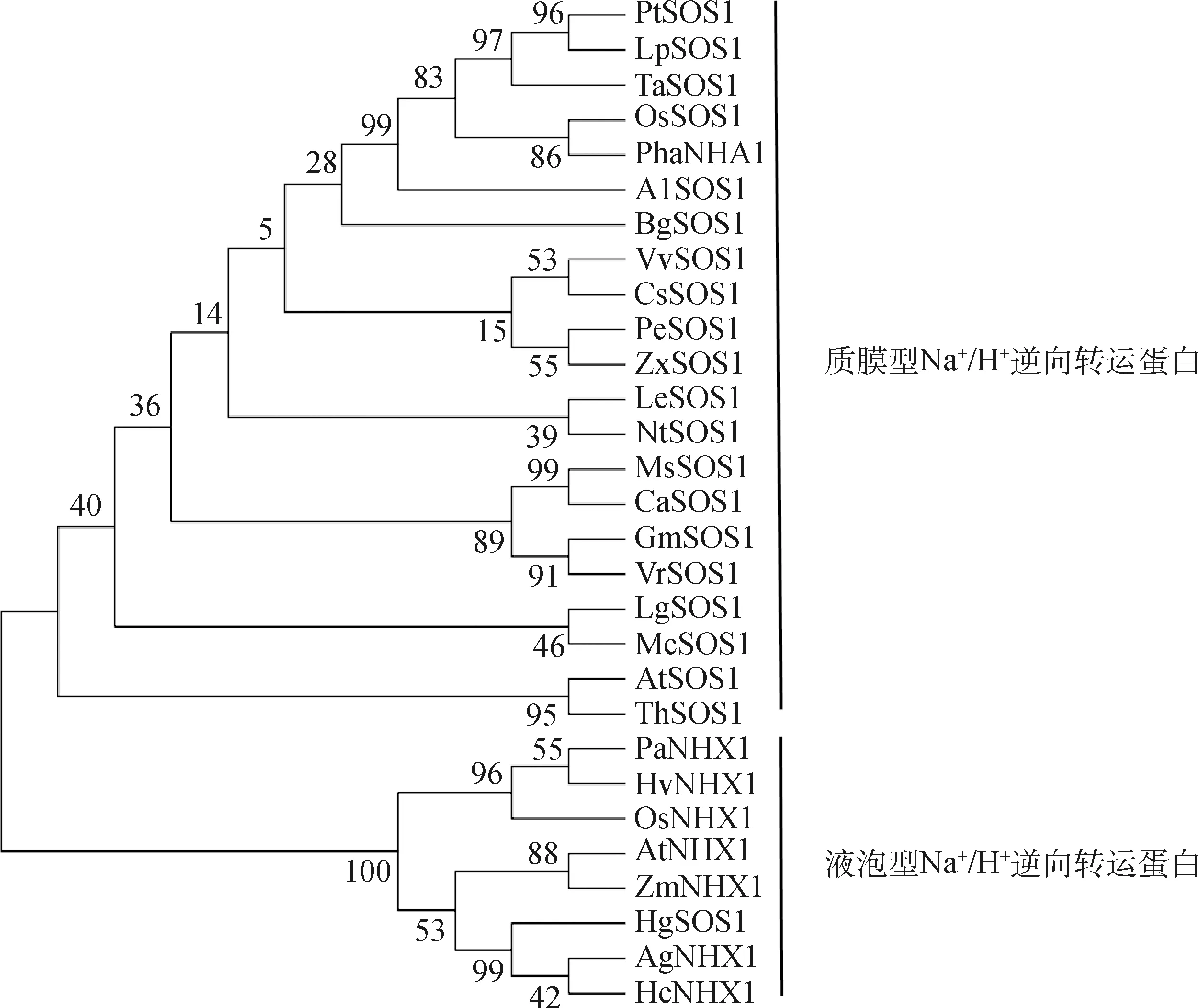
图9 紫花苜蓿MsSOS1与其他植物Na+/H+逆向转运蛋白的系统进化树分析Fig.9 Phylogenetic analysis of MsSOS1 in Medicago sativa with closely related Na+/H+antiporters from different plant species
2.4 紫花苜蓿MsSOS1基因的组织特异性表达分析
对MsSOS1基因在同一株紫花苜蓿不同组织中的相对表达量进行分析并作图(图10),以花中MsSOS1基因的表达量为对照发现,在正常生长条件下,MsSOS1基因在紫花苜蓿各组织中均有表达,在根中的表达量最高,且明显高于其它组织,是对照的11.9倍,叶中的表达量次之,是对照的6.7倍,茎中的表达量不是很明显,但也达到了对照的3.4倍(图10)。由此结果可以看出,MsSOS1基因在紫花苜蓿的各个器官中均有表达,只是表达峰度不同。

图10 MsSOS1基因的组织表达特异性Fig.10 Organ-specific expression pattern of MsSOS1
2.5 不同非生物胁迫下紫花苜蓿MsSOS1基因的表达分析
为了研究紫花苜蓿MsSOS1基因是否受非生物胁迫的诱导,分别对紫花苜蓿幼苗进行了不同的非生物胁迫处理,分析不同组织中MsSOS1基因的表达情况。
2.5.14℃胁迫下MsSOS1基因在紫花苜蓿中的表达量分析 4℃胁迫条件下,以0 h为对照分析MsSOS1基因在不同时间段的表达差异。结果发现,该基因受4℃胁迫诱导比较明显。叶片中MsSOS1基因的表达量随着胁迫时间的增加呈现出先升高后降低的趋势,且于8 h达到最大值;茎和根中的表达量随着处理时间的增加均呈现先降低后增高的趋势,但均低于对照(图11)。
2.5.2PEG6000胁迫下MsSOS1基因在紫花苜蓿中的表达量分析 25% PEG6000模拟干旱条件下,以0 h为对照分析MsSOS1基因在不同时间段的表达差异。结果发现,该基因受PEG6000诱导表达比较明显,在根、茎和叶中均有不同程度的表达(图12)。在叶中,随着PEG诱导时间的变化,MsSOS1基因的表达呈逐渐上升的趋势,于72 h表达量显著增加且达到最大表达量,是对照的7.5倍;随着PEG胁迫时间的延长,MsSOS1基因在根和茎中的表达量基本上都是出现先升高后降低的趋势,根中的表达量于12 h达到最大值,是对照的3.67倍;茎中的表达量于24 h达到最大值,是对照的8.23倍。

图11 MsSOS1基因在4℃胁迫下的表达模式分析Fig.11 Expression pattern of MsSOS1 in response to 4℃ stress

图12 MsSOS1基因在PEG胁迫下的表达模式Fig.12 Expression pattern of MsSOS1 in response to PEG stress
2.5.3脱落酸(ABA)胁迫下MsSOS1基因在紫花苜蓿中的表达量分析 以0 h为对照分析MsSOS1基因在ABA胁迫下的不同时间段的表达差异。结果发现,在ABA诱导下,随着时间的延长,MsSOS1基因主要在茎和叶中表达(图13),在叶中,MsSOS1基因的表达量在12 h达到最低值,24 h达到最大值,是对照的3.14倍;在茎中,随着处理时间的延长,MsSOS1基因的表达量呈现先升高后降低的趋势,且于24 h达到最大值,是对照的9倍;ABA处理对MsSOS1基因在根部的表达影响不显著,随着胁迫时间的延长,表达量几乎不变。
3 讨论
目前,很多研究已经确定了从蓝细菌到真菌、从藻类到高等植物中耐盐性的决定因素。在正常条件下,植物体细胞质中维持比较高的K+/Na+,而当它受到盐胁迫时,植物就会启动体内多种适应机制如传感、信号转导、基因表达和代谢途径等来适应这些不利环境。为了了解这些机制中有哪些部分可以提高植物对盐胁迫的适应性,我们重点研究了紫花苜蓿中与盐度耐受性相关的主要基因的分离、鉴定和表达模式分析。在本研究中,我们克隆得到了紫花苜蓿MsSOS1基因的全长序列并对其进行了序列和表达分析。紫花苜蓿MsSOS1蛋白N-端具有4个高度同源的、具有水溶性特征的、主要负责Na+转运的功能结构域,这与前人所报道的一般具有10~12个跨膜结构域有所不同,这可能是由于物种之间的差异所造成的;C-端是可以确定其确实是属于参与细胞内Na+调控转运的Na+/H+逆向转运蛋白家族成员的高度亲水的调控结构域,末端还有一个很长的、面向细胞质内腔的亲水性尾部,推测它可能是通过与逆境相关的调控因子发生互作来提高植物的抗性;另外,C-末端还存在1个SOS1蛋白的活性调节区(即磷酸化调节位点),这与拟南芥(Arabidopsisthaliana)、水稻(Oryzasativa)和獐茅(Aeluropuslittoralis)等的SOS1蛋白结构相似[26-27,31],只是不同的物种之间存在一些差异。

图13 MsSOS1基因在ABA处理下的表达模式Fig.13 Expression pattern of MsSOS1 in response to ABA treatment
对编码后的MsSOS1蛋白序列进行分析发现,该蛋白与已经报道的高等植物的SOS1基因蛋白序列相似度比较高;通过比对分析发现该蛋白与豆科植物鹰嘴豆(Cicerarietinum)、大豆(Glycinemax)等豆科植物的SOS1蛋白的同源性比较高,与其它科属植物的同源性比较低,说明SOS1蛋白在不同的物种之间虽然具有较高的保守性,但也存在一定的差异;但是,将其与模式植物拟南芥相比,同源性相对较低,说明这两种蛋白之间的差异较大,由此可以推测它们在结构和功能上也可能会存在不同的差异[32],这也就需要我们通过进一步的功能表达分析研究该基因在耐盐性状方面的特点来进行鉴别。另外,已有研究报道SOS1基因在植物的耐盐方面起着至关重要的作用,同时研究发现SOS1基因超表达可以使植物的耐盐性增强[47],这也预示着紫花苜蓿的MsSOS1基因在植物的耐盐方面可能也起着重要的作用。
采用实时荧光定量的方法对紫花苜蓿MsSOS1基因进行组织特异性表达分析,结果发现该基因在紫花苜蓿生育期的根、茎、叶、花中均有表达,这与拟南芥AtSOS1[21]和菊苣CiSOS1[48]在根和地上部分均有表达的结果相一致。从图11中也可以看到,紫花苜蓿MsSOS1基因在生育期的表达峰值为:根>叶>茎>花,具有根部表达的特性,它主要表现为根部组织通过对紫花苜蓿MsSOS1基因的表达进行上调的方法,将根系中多余的Na+排除体外,降低根中Na+浓度来抵御Na+对植物体本身的毒害作用。对紫花苜蓿MsSOS1基因在不同非生物胁迫下的表达模式研究发现,不同的非生物胁迫表达模式有所区别,Munns等[49]研究认为植物激素ABA是植物适应盐胁迫过程最初的信号物质,在植物应答盐胁迫的过程中具有重要的作用。已有文献报道,ABA处理植株能增强其在盐胁迫条件下的适应能力[50],Shi等研究发现拟南芥AtSOS1基因在ABA处理下表达量没有影响,而在本研究中,MsSOS1基因在根中的表达影响不明显,主要在茎和叶中表达,茎中的表达相对比较明显,推测该基因可能具有在茎维管束薄壁细胞表达的特异性[51];本研究中,MsSOS1基因还受到PEG胁迫的强烈诱导,无论是在根还是在茎和叶中均有不同程度的表达,与Song等对大岛野路菊(Chrysanthemumcrassum)CcSOS1基因的研究相一致[52],说明该基因也可能参与了紫花苜蓿的抗旱过程。
4 结论
本研究从豆科植物中苜1号紫花苜蓿中克隆得到一个MsSOS1基因,对其进行序列分析发现,该基因包含一个2 580 bp的开放阅读框,编码859个氨基酸;蛋白序列与豆科植物鹰嘴豆的氨基酸序列的同源性最高,为91%,进化树分析发现该基因属于质膜型Na+/H+逆向转运蛋白。另外,利用qRT-PCR技术对其进行表达分析发现,该基因的表达具有组织表达特异性,且在根中的表达量最大,其次是茎和叶;4℃、PEG和ABA等非生物胁迫都会诱导MsSOS1基因的表达,只是表达峰度不同,推测该基因可能在紫花苜蓿的抗逆分子机制中发挥一定的作用。这对于了解豆科植物Na+/H+逆向转运蛋白基因参与植物抗逆性的分子机制有了一定的了解,并为深入解析紫花苜蓿MsSOS1基因的功能和培育抗逆品种提供了良好的理论基础。

