两种梧桐叶绿体基因组密码子使用偏性分析
陆奇丰 骆文华 黄至欢
摘要: 为了分析美丽梧桐、云南梧桐叶绿体基因组密码子的使用偏性,该研究通过筛选美丽梧桐、云南梧桐叶绿体基因组中各52条蛋白编码序列,并利用CodonW、CUSP和SPSS软件对其密码子使用模式及偏性进行了分析。结果表明:(1)美丽梧桐、云南梧桐的GC含量分别为38.12%、38.05%,表明叶绿体基因组内富含A/T碱基。(2)有效密码子数(ENC)范围为36.91~56.46、36.55~58.04,表明多数密码子偏性较弱。(3)相对同义密码子(RSCU)分析显示,RSCU>1的密码子各有29个,其中28个以A、U结尾。(4)中性绘图显示,GC3与GC12的相关性不显著,回归曲线斜率分别为0.195和0.304,说明密码子偏好性主要受到自然选择的影响。(5)ENC-plot分析中大部分基因分布于曲线的周围和下方,ENC比值多分布于-0.04~0.10之间,表明突变会影响密码子偏性的形成。此外,17、18个密码子分别被鉴定为美丽梧桐、云南梧桐的最优密码子。以上结果说明美丽梧桐、云南梧桐叶绿体基因组的密码子使用偏性可能受选择和突变共同作用,且使用模式较为相似,但具有一定的差异,可能与适应环境的进化机制有关。
关键词: 美丽梧桐, 云南梧桐, 叶绿体基因组, 密码子偏性, 最优密码子
中图分类号: Q943.2文献标识码: A
文章编号: 1000-3142(2020)02-0173-11
Abstract: In order to analyze the codon usage patterns of the chloroplast genome of Firmiana pulcherrima and F. major. Fifty-two genes selected from chloroplast genome (Cp genome) of these two species were analyzed using CodonW, CUSP and SPSS software. The results showed that: (1) GC content in Firmiana pulcherrima was 38.12%, and that in F. major was 38.05%, suggesting that A/T base was rich in chloroplast genome. (2) The effective number of codons in F. pulcherrima ranged from 36.91 to 56.46, and that in F. major ranged from 36.55 to 58.04, meaning the codon bias was weak in F. pulcherrima and F. major. (3) Both F. pulcherrima and F. major had 29 codons with relative synonymous codon usage (RSCU) greater than 1 and 28 codons ending with A and U base. (4) Neutrality plot showed that there was not significant correlation between GC3 and GC12, and the regression slope was 0.195 in F. pulcherrima, and 0.304 in F. major, suggesting that codon bias was mainly affected by natural selection. (5) ENC-plot analysis showed that most of genes fell below and followed the standard curve, and ENC ratio of most genes ranged from -0.04 to 0.10, suggesting that the mutation also affected the formation of codon bias in F. pulcherrima and F. major. In addition, 17 codons were identified as optimal codons in F. pulcherrima, and 18 codons in F. major. We suggest that the codon usage bias of F. pulcherrima and F. major are formed under effect of mutation and selection. Although the codon usage pattern of F. pulcherrima and F. major are very similar, there are some differences, which may be related to the evolutionary me-chanism of adaptive environment.
Key words: Firmiana pulcherrima, F. major, chloroplast genome, codon usage bias, optimal codons
自然界中,基因序列根據三联密码子法则翻译成氨基酸序列,并进一步组装修饰形成具有特定空间结构和功能活性的蛋白质。密码子由3个相连的核苷酸碱基所组成,一共存在64个密码子,除去3个终止密码子 (UAA、UAG、UGA),61个密码子对应特定的氨基酸。天然氨基酸有20种,除了甲硫氨酸(AUG)、色氨酸(UGG)对应单一密码子编码以外,其余18种氨基酸还均对应2~6个编码密码子。编码相同氨基酸的不同密码子又称为同义密码子,同义密码子使用频率在基因内不是随机的,而是优先使用其中一些密码子,这一现象被称为密码子使用偏性(Gustafsson et al., 2004;Li et al., 2017)。影响密码子偏性的因素较多,如自然选择下DNA的变异(Ashesh, 2002;Suzuki, 2011)、tRNA丰度(Buchan et al., 2006)、基因序列长度(Gabriel et al., 2001;Stoletzki et al., 2011;Ribeiro et al., 2012)、蛋白质翻译效率(Haupt et al., 2009;Li et al., 2017)等。研究密码子偏性不仅有助于解释物种间进化规律,而且还对基因表达、载体构建、未知功能基因的分析具有重要意义。
叶绿体属于半自主性细胞器,是进行光合作用和固定CO2的主要场所,参与植物生长环境胁迫(干旱、盐、光等)相关的重要代谢过程(Liu et al., 2018)。此外,叶绿体具有母系遗传、结构稳定和基因组较小等特性,从而广泛运用于植物多样性、系统发育、DNA 条码开发、植物适应性和遗传工程研究中(Sajjad et al., 2017;Liu et al., 2018)。不同物种叶绿体基因组密码子使用偏性存在一定差异(Liu & Xue, 2005;郑薇玮等,2016)。密码子使用模式对基因功能和表达有一定的影响作用,基因的密码子偏性越强,其表达水平越高(Zhou et al.,2016)。因此,为提高叶绿体遗传转化效率及目的基因表达水平,很有必要开展叶绿体全基因组密码子使用模式的研究。
美丽梧桐(Firmiana pulcherrima)、云南梧桐(F. major)是梧桐属内的重要物种,具有较高的研究价值和经济价值。但是,由于生态环境的破坏,美丽梧桐、云南梧桐野外資源日益减少,已被列为濒危树种(中国科学院,1984;余志祥等,2013;周亚东等,2013)。目前,对美丽梧桐、云南梧桐的研究主要集中于资源调查(黎国运等,2011;Yang et al., 2018)、种苗繁育(黎国运等,2011;余志祥等,2013;周亚东等,2013)等方面,两种梧桐叶绿体全基因组虽已完成测序 (Wang et al., 2018;Ya et al., 2018),但关于两者的叶绿体基因组编码基因的密码子使用模式比较的研究却尚未见有报道。因此,本研究在前人研究的基础上,对美丽梧桐和云南梧桐的叶绿体基因组中52个基因的密码子碱基组成特点及使用偏性进行比较分析,揭示其密码子偏性的影响因素,并分析最优密码子,旨在为美丽梧桐、云南梧桐制定全面的保育策略以及为梧桐科叶绿体基因组学研究提供参考。
1材料与方法
1.1 叶绿体基因序列数据
从NCBI 分别检索下载美丽梧桐、云南梧桐叶绿体全基因组序列信息,登录号分别为NC_036395.1(F. pulcherrima 美丽梧桐)、NC_037242.1(F. major 云南梧桐),分别获得82条和85条编码基因。对所获得的编码基因按以下条件进行筛选:基因长度大于300 bp,以ATG为起始密码子,以TAA、TAG、TGA为终止密码子,并剔除重复的编码基因,最终获得各52条编码基因进行后续分析。
1.2 方法
1.2.1 密码子相关参数的计算分别利用软件CodonW(http://codonw.sourceforge.net/)和CUSP(http://imed.med.ucm.es/EMBOSS/)分析CDS的有效密码子数(ENC)、密码子第3位碱基组成特性,以CT3、CC3、CA3、CG3表示,统计密码子第1位、第2位、第3位碱基的GC含量及52个基因总GC含量,分别用CGC1、CGC2、CGC3、GC表示。
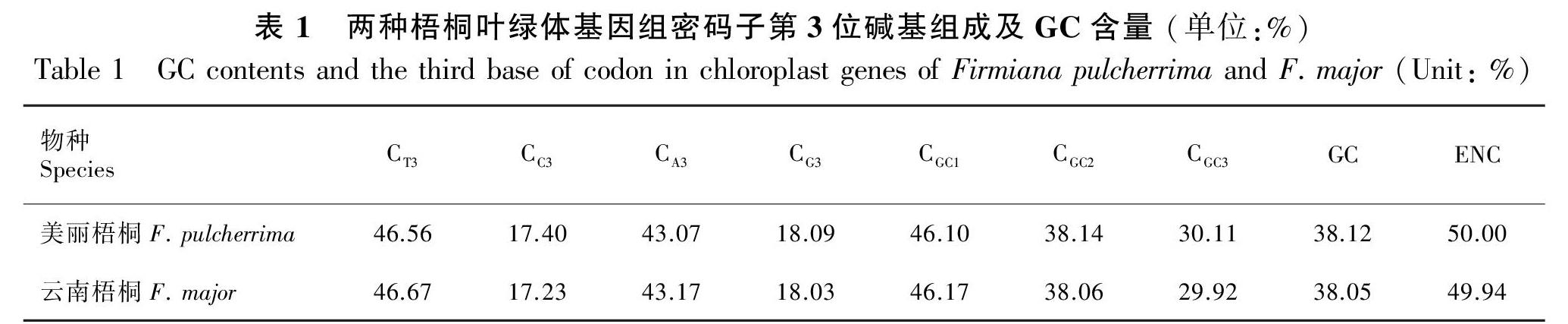
1.2.2 中性绘图分析计算各CDS的CGC1、CGC2的平均值,记为CGC12,以此为纵坐标,以CGC3为横坐标绘制散点图,分析CGC3与CGC12的相关性,并推测影响密码子偏性的因素。若CGC3与CGC12呈显著相关,表明密码子对碱基使用无差异,突变是密码子偏性的主要影响因素;若CGC3与CGC12相关性不显著,且斜率接近0,表明密码子第1位、第2位碱基与第3位碱基使用存在差异,密码子偏性与第3位碱基显著相关,选择压力对密码子偏性的影响较强(尚明照等,2011; 秦政等,2018)。
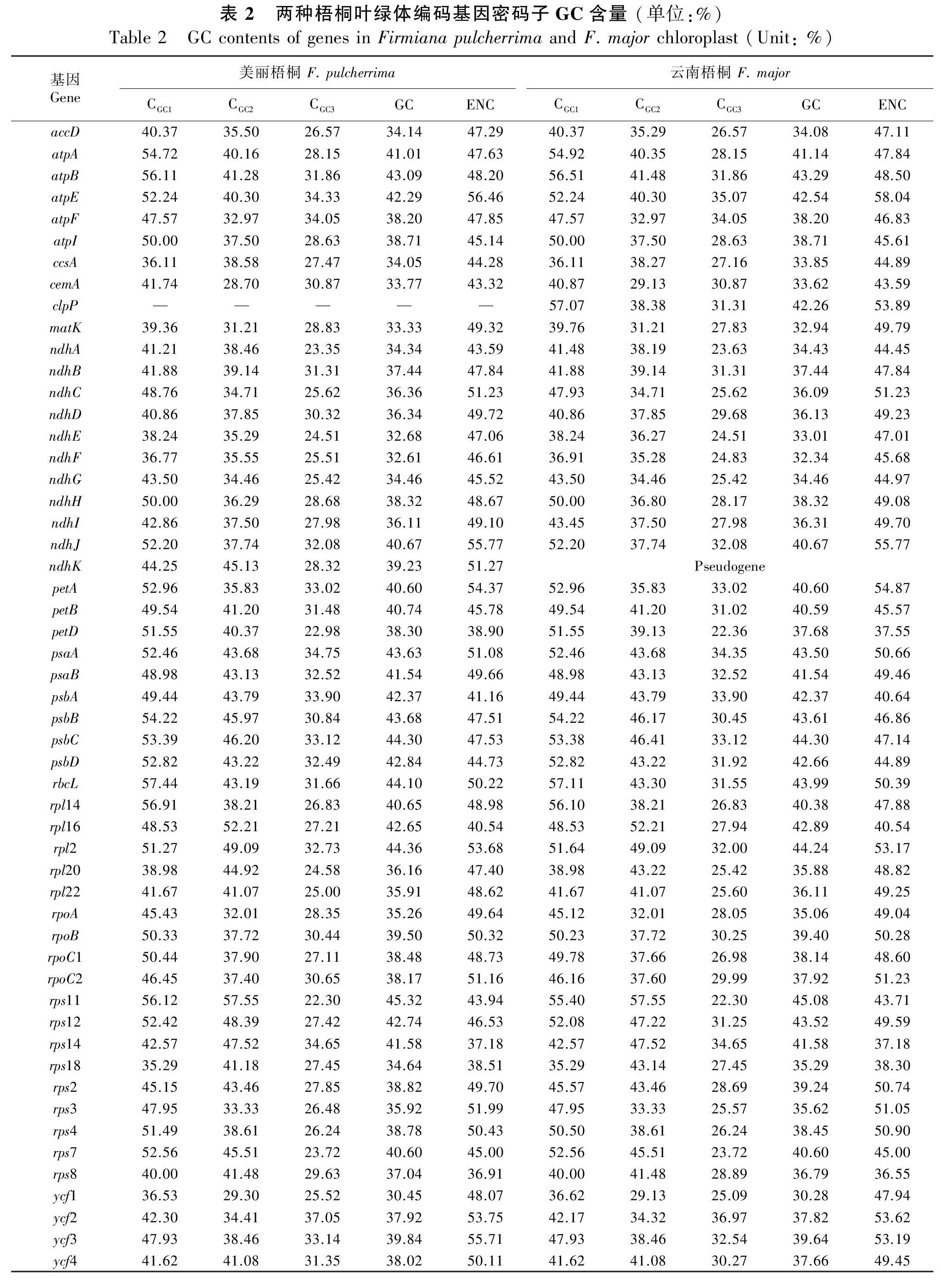
1.2.3 PR2-plot绘图分析PR2-plot分析是考察突变和自然选择对密码子偏性影响的方法之一(尚明照等,2011;刘慧等,2017)。为避免密码子第3位碱基上的A/T和G/C突变的不平衡,通过统计两种梧桐叶绿体52条基因中编码丙氨酸、甘氨酸、亮氨酸(CTT、CTC、CTA、CTG)、脯氨酸、精氨酸(CGT、CGC、CGA、CGG)、丝氨酸(TCT、TCC、TCA、TCG)、苏氨酸、缬氨酸的密码子第3位碱基的T3、C3、A3、G3的组成情况,并以A3/(A3+T3)为纵坐标,以G3/(G3+C3)为横坐标绘制散点图。
1.2.4 ENC-plot绘图分析为进一步了解两种梧桐碱基组成对密码子偏性的影响,以两种梧桐叶绿体中各基因的ENC、GC3值为纵、横坐标做散点图,并与ENC期望值(ENC =2+GC3+29/([GC32+ (1-GC3) 2]) 进行比较,期望值代表密码子偏性仅受碱基组成的影响。若基因分布于期望值曲线的上方,其密码子偏性仅受突变影响;若基因分布于曲线下方较远位置,表明选择是影响密码子偏性的主要因素(牛元等,2018)。
1.2.5 相对同义密码子使用度分析为考察同义密码子偏好性情况,以相对同义密码子使用度(RSCU)表示特定密码子在编码对应氨基酸的同义密码子间的相对使用概率(杨国锋等,2015)。当RSCU>1时,表示该密码子使用频率较高,偏好性较强;当RSCU<1时,表示该密码子使用频率较低,偏好性较弱;当RSCU=1时,该密码子无偏好性。
1.2.6 最优密码子分析对CDS的ENC值进行排序,从两极各选出5个基因作为高、低表达组,分别计算对应的密码子RSCU值,筛选高低表达组内对应密码子△RSCU之差大于0.08作为优越高表达密码子,并与整体密码子RSCU进行结合比较,将△RSCU≥0.08,且整体RSCU>1的密码子定义为最优密码子(秦政等,2018;石元豹等,2018 )。
2结果与分析
2.1 密码子组成分析
通过CodonW和在线CUSP对美丽梧桐、云南梧桐叶绿体编码基因组的52条CDS进行分析,结果如表1所示,美丽梧桐、云南梧桐叶绿体编码基因密码子CT3和CA3的含量分别为46.56%、46.67%与43.04%、43.17%,表明两种梧桐叶绿体编码基因第3位碱基以A/T结尾为主。密码子上不同位置碱基GC含量有所差异,第1位碱基的GC含量最高,分别为46.10%、46.17%;其次为第2位,分别为38.14%、38.06%;最低为第3位碱基,分别为30.11%、29.92%,且两种梧桐叶绿体基因组GC含量分别为38.12%、38.05%,表明叶绿体编码基因富含A/T碱基。ENC值可反映密码子偏性程度,其取值范围为20~61,ENC值越小表明偏性越强,ENC值越大则密码子偏性越弱,一般以ENC的值为35时区分密码子偏性强弱(Jiang et al.,2008)。本研究中,两种梧桐叶绿体基因组的ENC值为50左右,大于35表明其密码子偏性较弱。
利用在线CUSP对各基因密码子不同位置碱基的GC含量进行分析,结果如表2所示,不同基因的GC含量存在一定的差异,CGC1、CGC2与CCG3差异较大,两种梧桐叶绿体编码基因ENC范围分别为36.91~56.46、36.55~58.04,且大部分基因的ENC大于44,表明叶绿体编码基因密码子偏性较弱。美丽梧桐、云南梧桐中atpE基因的ENC值最大,分别为56.46和58.04,说明美丽梧桐中编码atpE基因密码子偏性强于云南梧桐,同时两种梧桐叶绿体编码基因GC含量、ENC值之间也存在差异,但差异不大。此外,本研究还发现,美丽梧桐中不存在clpP基因及云南梧桐中的ndhK基因为假基因,表明两种梧桐叶绿体基因组具有较高的保守性,但种间却存在一定的差异。
CGC1、CGC2、CCG3、GC、ENC及序列长度(sequence length, SL)相关性分析结果如表3所示,CGC1与CGC2极显著相关,CGC1、CGC2均与CCG3相关性不显著,表明两种梧桐叶绿体CDS密码子的第1位、第2位碱基组成相似,且与第3位碱基组成存在较大差异,GC与CGC1、CGC2、CCG3均呈极显著相关,SL与CGC2呈负相关。此外,美丽梧桐的ENC与CCG3呈显著相关,与CGC1、CGC2、GC、SL均未达到显著水平,表明美丽梧桐叶绿体编码基因密码子第3位碱基组成对密码子偏性影响较大。云南梧桐中ENC与CCG1、CGC3显著相关,相关值分别为0.29、0.32,与CGC2、GC、SL相关性较低,表明CCG3对云南梧桐叶绿体密码子偏性的影响强于CGC1。两种梧桐密码子参数的相关性分析结果存在差异,其中 CGC1与云南梧桐叶绿体基因组密码子偏性显著相关,而美丽梧桐叶绿体基因组密码子偏性与CGC1相关不显著,进一步说明两种植物叶绿体基因组密码子偏性的影响因素有所差异。
2.2 中性绘图分析
如图1所示,美丽梧桐的叶绿体CDS的CGC3与CGC12含量分布范围分别为0.223 0~0.370 5、0.329 2~0.568 4,云南梧桐为0.223 0~0.369 7、0.328 8~0.564 8,两者的分布范围几乎重合,说明两种梧桐叶绿体基因组较为保守。两种梧桐的CGC3与CGC12的Pearson相关系数分别为0.121和0.206,进一步说明CGC1和CGC2均与CCG3相关性不显著,密码子的第1位、第2位碱基与第3位碱基组成存在显著差异,密码子偏性与第3位碱基显著相关。同时,回归曲线斜率分别为0.195和0.304,表明选择压力对两种梧桐属叶绿体基因组密码子偏性的影响较强。此外,突变对密码子偏性也有一定的影响,且云南梧桐叶绿体密码子偏性受到突变的影响高于美丽梧桐。
2.3 PR2-plot绘图分析
PR2-plot绘图分析是指通过对密码子第3位碱基组成进行分析,探讨突变和自然选择对密码子使用偏性的影响,当密码子第3位碱基表现为A=T和C=G时,表明DNA两条互补链上不存在选择效应。突变是影响密码子偏性的唯一因素(Sueoka, 2001;Zhang et al., 2018)。本研究中,两种梧桐大部分基因分布在左下方区域(图2),T、C碱基频率高于A、G碱基,说明密码子第3位碱基偏爱使用嘧啶碱基,表明两种梧桐叶绿体编码基因密码子使用偏性不仅受到突变的影响,而且还受到其他因素的影响,如自然选择压力等。
2.4 ENC-plot绘图分析
从图3可以看出,两种梧桐的大部分基因较集中分布于标准曲线的两侧,说明其ENC值与ENC期望值较为接近,而rps8、rps14、rps18、petD、rpl16、psbA基因偏离标准曲线较远,且ENC值较小,密码子偏性较强。根据公式(ENC期望-ENC实际)/ENC期望统计基因ENC的分布频数(表4),结果显示大部分基因的ENC频数集中于-0.04~0.10之间,进一步表明大部分基因的ENC值与ENC期望值差异较小,表明这部分基因密码子偏性主要受到突变的影响。rps8、rps14、rps18、petD、rpl16、psbA分布于0.18~0.33之间,与0点相差较远,表明这部分基因密码子偏性主要受到选择压力的影响。
2.5 RSCU分析
利用CondonW程序对两种梧桐叶绿体基因组52个编码基因进行RSCU分析,结果如表5所示,两种梧桐的高频密码子(RSCU>1)各有29个,除了UUG以外,其余密码子均以A/U结尾,表明两种梧桐密码子偏好使用以A/U结尾的密码子,且两种梧桐植物对应密码子RSCU值极为接近,进一步说明其叶绿体基因组密码子使用偏性较为接近,叶绿体基因组较为保守,种间差异较小。
2.6 最优密码子分析
对两种梧桐植物叶绿体基因组基因的ENC值进行排序,选出两极的10%基因作为高、低表达基因库,其中两种梧桐属叶绿体基因组高表达基因为rps8、rps14、petD、rps18、rpl16,平均ENC值分别为38.41(F. pulcherrima)、38.02(F. major);F. pulcherrima低表达基因为ycf2、petA、 ycf3、ndhJ、atpE,图 3ENC-plot绘图分析
Fig. 3Analysis of ENC-plot (ENC value against CGC3)而F. major为ycf2、clpP、petA、ycf3、ndhJ、atpE,平均ENC值分别为55.21(F. pulcherrima)、55.24(F. major)。分别计算高、低表达基因库的RSCU值,并计算对应密码子的△RSCU值(高表达库RSCU值-低表达库RSCU值),同时与整体密码子的RSCU值结合比较,筛选出△RSCU值≥0.08,且整体RSCU>1的密码子,定义为优越密码子。最终分别确定了17、18个表达优越密码子(表4),分别为GCA、UGU、GAA、UUU、GGA、GGU、CAU、AUU、AAA、UUA、CAA、CGA、CGU、AGU、ACA、ACU(为F. major优越密码子)、GUA、UAU,这些优越密码子均以A/U结尾。此外, 我們观察到两种梧桐植物叶绿体基因组使用的优越密码子基本相同,但其△RSCU值存在一定的差异,F. pulcherrima 中的GAA、 CGA密码子△RSCU>0.08且<0.30, 而F.
3讨论与结论
密码子是遗传信息的基本单位,是核酸与蛋白质的联系纽带,研究其使用情况对了解物种基因组进化具有重要意义(李平等,2011)。密码子使用偏性是物种不断进化适应的结果,其形成机制受多种因素的影响,其中突变作用和自然选择压力是其重要影响因素(王鹏良等, 2018)。不同物种、基因家族中密码子使用偏性存在一定的差异,因此密码子使用偏性是研究物种基因组进化关系及影响的重要指标之一。
碱基组成差异对密码子的使用有重要的影响。密码子在第1位、第2位碱基上的突变通常会导致编码氨基酸的改变,第3位碱基存在简并性,其突变对编码氨基酸的改变通常无影响(尚明照等,2011)。因此,密码子第3位碱基的使用模式与密码子偏性密切相关。有研究表明,植物叶绿体基因组中密码子第3位碱基偏好使用A/T(Zhou et al., 2008;秦政等,2018 )。本研究中,两种梧桐植物CT3和CA3高于CG3、CC3,且CGC1、CGC2均与CCG3相关性不显著,CCG3比例最低,说明梧桐属植物叶绿体基因组密码子偏好使用A/T碱基结尾,与大花香水月季(Rosa odorata var. gigantea)、表 5两种梧桐密码子RSCU及最优密码子樟树(Cinnamomum camphora)、甜荞(Fagopyrum esculentum) (罗洪等,2015;牛元等,2018;秦政等,2018)等植物的研究结果一致。
自然選择和突变是影响密码子使用偏性的重要因素(Sharp et al.,2010;Rao et al.,2011)。本研究通过对两种梧桐叶绿体基因组52个编码基因CGC3与CGC12相关分析表明,密码子第3位碱基GC含量与第1位、第2位碱基相关性弱,且线性回归斜率较小,表明两种梧桐密码子的使用受到选择压力因素较强。此外,PR2-plot分析发现,密码子第3位碱基上A/T碱基和C/G碱基使用频率存在差异,更偏好使用T、C碱基,进一步表明两种梧桐叶绿体基因组密码子使用不仅与碱基突变的有关,而且还更多受到选择的影响。ENC-plot分析显示:大部分基因的ENC值与期望ENC值较为接近,其分布频数集中于-0.04~0.10之间,表明这部分基因受密码子偏性与第3位碱基的GC含量相关,突变是主要影响因素;而rps8、rps14、rps18、petD、rpl16、psbA分布于标准曲线的下方较远位置,自然选择影响其密码子偏性的主要因素。综合中性绘图分析、ENC-plot、PR2-plot等分析发现,突变和自然选择等多重因素共同影响着两种梧桐叶绿体密码子使用偏性。这与柿属植物、普通油茶、苦荞、甜荞等物种的叶绿体基因组密码子偏性研究结果相似(罗洪等,2015;胡莎莎等,2016;傅建敏等,2017;王鹏良等,2018)。
本研究发现,两种梧桐中29个高频密码子(RSCU>1)第3位碱基主要以A/U结尾,与前人在其他植物中的研究结果相似(尚明照等,2011;秦政等,2018)。与高表达密码子进行综合分析,最终确定美丽梧桐的最优密码子为17个,云南梧桐18个,分别为GCA、UGU、GAA、UUU、GGA、GGU、CAU、AUU、AAA、UUA、CAA、CGA、CGU、AGU、ACA、ACU(为F. major优越密码子)、GUA、UAU,这些最优密码子以A/U结尾,与大多植物叶绿体基因组最优密码子相似,表明梧桐与其他双子叶植物叶绿体基因组密码子第3位碱基偏爱使用A/U结尾(Kawabe & Miyashita, 2003)。有研究表明,物种间的亲缘关系越近,其密码子使用模式越相似(张太奎等,2017;张孟伟等,2018)。本研究中,两种梧桐叶绿体基因组密码子使用偏性极为相似,但存在一定的差异。此外,本研究还发现,美丽梧桐中叶绿体clpP编码基因丢失,云南梧桐的ndhK为假基因,推测两种梧桐适应环境改变的进化趋势有所不同,有待深入研究。
本研究分析了美丽梧桐、云南梧桐叶绿体基因组编码基因的密码子偏性形成的影响因素和特点,以及两种梧桐叶绿体基因组的最优密码子,为外源基因密码子改造及梧桐属叶绿体基因组进化机制研究提供一定的理论基础。
参考文献:
ASHESH N, 2002. Investigations on evolutionary changes in base distributions in gene sequences [J]. Internet Electr J Mol Design, 1: 545-558.
BUCHAN JR, AUCOTT LS, STANSFIELD I, 2006. tRNA properties help shape codon pair preferences in open reading frames [J]. Nucl Acids Res, 34(3): 1015-1027.
Chinese Academy of Sciences, 1984. Flora Reipublicae Popularis Sinicae [M]. http: //frps.iplant.cn/, 49(2): 136-137. [中国科学院, 1984. 中国植物志 [M]. http: //frps.iplant.cn/, 49(2): 136-137.]
FU JM, SUO YJ, LIU HM, et al., 2017. Analysis on codon usage in the chloroplast protein coding genes of Diospyros spp. [J]. Nonw For Res, 35(2):38-44. [傅建敏, 索玉静, 刘慧敏, 等, 2017. 柿属植物叶绿体蛋白质编码基因密码子用法 [J]. 经济林研究, 35(2): 38-44.]
GABRIEL M, LAURENT D,2001. Synonymous codon usage, accuracy of translation, and gene length in Caenorhabditis elegans [J]. J Mol Evol, 52: 275-280.
GUSTAFSSON C, GOVINDARAJAN S, MINSHULL J,2004. Codon bias and heterologous protein expression [J]. Trends Biotechnol, 22(7): 346-253.
HAUPT S, ZIEGLER A, COWAN G,et al., 2009. Studies of the role and function of barley stripe mosaic virus encoded proteins in replication and movement using GFP fusions [M]. Methods Mol Biol, 515: 287-297.
HU SS, LUO H, WU Q, et al., 2016. Analysis of codon usage bias of chloroplast genome of tartary buckwheat [J]. Mol Plant Breed, 14(2): 309-317. [胡莎莎, 羅洪, 吴琦, 等, 2016. 苦荞叶绿体基因组密码子偏爱性分析 [J]. 分子植物育种, 14(2): 309-317.]
JIANG Y, DENG F, WANG HL, et al., 2008. An extensive analysis on the global codon usage pattern of baculoviruses [J]. Arch Virol, 153(12): 2273-2282.
KAWABE A, MIYASHITA NT, 2003. Patterns of codon usage bias in three dicot and four moncot plant species [J]. Genes Genet Syst, 78(5): 343-352.
LI GY, SHENG XH, XU PL, 2011. A preliminary report on wild resource survey of Firmiana pulcherrima [J]. Torp For, 39(1): 50-52. [黎国运, 盛小彬, 徐佩玲, 2011. 美丽梧桐野生资源调查初报 [J]. 热带林业, 39(1): 50-52.]
LI GY, GUAN XJ, SHENG XB, et al., 2011. Research on shoot cutting propagation of Firmiana pulcherrima Hsue [J]. Torp For, 39(4): 36-38. [黎国运, 关秀娟, 盛小彬, 等, 2011. 美丽梧桐扦插育苗技术研究 [J]. 热带林业, 39(4): 36-38.]
LI J, LI HY, ZHI JK, et al., 2017. Codon usage of expansin genes in Populus trichocarpa [J]. Current Bioinform, 12: 452-461.
LI P, BAI YF, FENG RY, et al., 2011. Analysis of codon bias of NAD-ME gene in Amaranthus hypochondriacus [J]. Chin J Appl Environ Biol, 1: 12-17. [李平, 白云凤, 冯瑞云, 等, 2011. 籽粒苋苹果酸酶(NAD-ME)基因密码子偏好性分析 [J]. 应用与环境生物学报, 1: 12-17.]
LIU QP, XUE QZ, 2005. Comparative studies on codon usage pattern of chloroplasts and their host nuclear genes in four plant species [J]. J Genet, 84(1): 55-62.
LIU X, ZHOU BY, YANG HY, et al., 2018. Sequencing and analysis of Chrysanthemum carinatum Schousb and Kalimeris indica. The complete chloroplast genomes reveal two inversions and rbcL as barcoding of the vegetable [J]. Molecules, 23(6): 1358.
LUO H, HU SS, WU Q, et al., 2015. Analysis of buckwheat chloroplast genome bias [J]. Genom Appl Biol, 34(11): 2457-2464. [罗洪, 胡莎莎, 吴琦, 等, 2015. 甜荞叶绿体基因密码子偏爱性分析 [J]. 基因组学与应用生物学, 34(11): 2457-2464.]
NIU Y, XU Q, WANG YD, et al., 2018. An analysis on codon usage bias of chloroplast genmoe of Rosa odorata var. gigantea [J]. J NW For Univ, 33(3): 123-130. [牛元, 徐琼, 王嵛德, 等, 2018. 大花香水月季叶绿体基因组密码子使用偏性分析 [J]. 西北林学院学报, 33(3): 123-130.]
QIN Z, ZHENG YJ, GUI LJ, et al., 2018. Codon usage bias analysis of the chloroplast genome of camphor tree (Cinnamomum camphora) [J]. Guihaia, 38(10): 1346-1355. [秦政, 郑永杰, 桂丽静, 等, 2018. 樟树叶绿体基因组密码子偏好性分析 [J]. 广西植物, 38(10): 1346-1355.]
RAO Y, WU G, WANG Z, et al., 2011. Mutation bias is the driving force of codon usage in the gallus gallus genome [J]. DNA Res, 18(6): 499-512
RIBEIRO AS, HKKINEN A, LLOYD-PRICE J, 2012. Effects of gene length on the dynamics of gene expression [J]. Comput Biol Chem, 41(4): 1-9.
SAJJAD A, MUHAMMAD W, ABDUL LK, et al., 2017. The complete chloroplast genome of wild rice (Oryza minuta) and its comparison to related species [J]. Front Plant Sci, 8: 304.
SHANG MZ, LIU F, HUA JP, et al., 2011. Analysis on codon usage of chloroplast genome of Gossypium hirsutum [J]. Sci Agric Sin, 44(2):245-253. [尚明照, 刘方, 华金平, 等, 2011. 陆地棉叶绿体基因组密码子使用偏性的分析 [J]. 中国农业科学, 44(2):245-253.]
SHARP PM EMERY LR, ZENG K, 2010. Forces that influence the evolution of codon bias [J]. Phil Trans R Soc B, 365(1544): 1203-1212.
SHI YB, WANG GB, YANG XM, et al., 2018. Analysis of codon usage of WRKY transcription factors in Ginkgo biloba [J]. Mol Plant Breed, http:kns.cnki.net/kcms/detail/46.1068.S.20180809.1423.008.html. [石元豹, 汪貴斌, 杨晓明, 等, 2018. 银杏WRKY家族基因密码子使用偏向性分析 [J]. 分子植物育种, http://kns.cnki.net/kcms/detail/46.1068.S.20180809.1423.008.html.]
STOLETZKI N, 2011. The surprising negative correlation of gene length and optimal codon use-disentangling translational selection from GC-biased gene conversion in yeast [J]. BMC Evol Biol, 11(1): 1179-1187.
SUEOKA N, 2001. Near homogeneity of PR2-bias fingerprints in the human genome and their implications in phyloge-netic analyses [J]. J Mol Evol, 53: 469 –476.
SUZUKI Y, 2011. Statistical methods for detecting natural selection from genomic data [J]. Genes Genet Syst, 85(6): 359-376.
WANG JH, CAI YC, ZHAO KK, et al., 2018. Characterization of the complete chloroplast genome sequence of Firmiana pulcherrima (Malvaceae) [J]. Conserv Genet Resour, 10(3): 445-448.
WANG PL, YANG LP, WU HY, et al., 2018. Codon preference of chloroplast genome in Camellia oleifera [J]. Guihaia, 38(2): 135-144. [王鹏良, 杨利平, 吴红英, 等, 2018. 普通油茶叶绿体基因组密码子偏好性分析 [J]. 广西植物, 38(2): 135-144.]
YA JD, YU ZX, YANG YQ, et al., 2018. Complete chloroplast genome of Firmiana major (Malvaceae), a critically endangered species endemic to Southwest China [J]. Conserv Genet Resour, 10(4): 713-715.
YANG GF, SU KL, ZHAO YR, et al., 2015. Analysis of condon usage in the chloroplast genome of Medicago truncatula [J]. Atca Pratac Sin, 24(12): 171-179. [杨国锋, 苏昆龙, 赵怡然, 等, 2015. 蒺藜苜蓿叶绿体密码子偏好性分析 [J]. 草业学报, 24(12): 171-179.]
YANG J, CHEN G, SUN WB, 2018. Conserving Firmiana major, a tree species endemic to China [J]. Oryx, 52(2): 211-213.
YU ZX, YANG YQ, MO X, et al., 2013. A shudy of artificial breeding of an endangered panlt (Firmiana major) [J]. J Sichuan For Sci Technol, 34(2): 62-65. [余志祥, 杨永琼, 莫旭, 等, 2013. 濒危植物云南梧桐(Firmiana major)人工繁育研究 [J]. 四川林业科技, 34(2): 62-65.]
ZHANG H, LI J, WANG RX, et al., 2018. Comparative analysis of expansin gene codonusage patterns among eight plant species [J]. J Biomol Struct Dyn, 7: 1-8.
ZHANG MW, ZHANG TK, LIU CY, et al., 2018. Biosysthsis pathway evolution and codon usage bias analysis of lignin in Punica granatunm [J]. Mol Plant Breed, 16(7): 2131-2138. [张孟伟, 张太奎, 刘翠玉, 等, 2018. 石榴木质素合成途径进化与密码子使用偏好性分析 [J]. 分子植物育种, 16(7): 2131-2138.]
ZHANG TK, QI GH, YE HL, et al., 2017. Codon usage bias in pomegranate transcriptome [J]. Acta Hortic Sin, 44 (4): 675-690. [张太奎, 起国海, 叶红莲, 等, 2017. 石榴转录组密码子使用偏向性 [J]. 园艺学报, 44 (4): 675-690.]
ZHENG WW, CHEN JH, HAO ZD, et al., 2016. Analysis on codon usage bias of chloroplast genes from five conifers including Cunninghamia lanceolata [J]. Mol Plant Breed, 14(5): 1091-1097. [鄭薇玮, 陈金慧, 郝兆东, 等, 2016. 杉木等5种针叶树叶绿体密码子偏好性分析 [J]. 分子植物育种, 14(5): 1091-1097.]
ZHOU YD, ZHOU YB, YAN SL,et al., 2013. Effects of different substrates on seed germination and seeding growth of Firmiana pulcherrima Hsue [J]. J Trop Biol, 4(4): 322-326. [周亚东, 周有标, 颜速亮, 等, 2013. 不同基质对美丽梧桐种子育苗的影响 [J]. 热带生物学报, 4(4): 322-326.]
ZHOU ZP, DANG YK, ZHOU M, et al., 2016. Codon usage is an important determinant of gene expression levels largely through its effects on transcription [J]. Proc Natl Acad Sci USA, 26: e6117-e6125.
(责任编辑 蒋巧媛)

