粒子群优化RBF神经网络的DNA序列分类
孙倩 赵昕


摘 要: 为提高径向基神经网络算法在DNA序列分类问题应用上的准确率和收敛速度,首先,以20种氨基酸的含量作为特征向量;其次,针对由于随機选取径向基神经网络初始参数引起的收敛速度慢的问题,选择粒子群算法优化神经网络参数。应用真实的DNA序列对优化前后的两种神经网络算法进行比较,结果显示,优化后算法在迭代12次左右误差即稳定,分类结果正确率达到93.75%,远高于未经优化的神经网络70%的正确率。由实验结果可知,在DNA序列分类问题中,粒子群优化径向基神经网络对提高收敛速度和正确率方面是有效的。
关键词: DNA序列分类; PSO?RBF神经网络; 特征提取; 分类模型建立; 参数优化; 分类效果对比
中图分类号: TN911.1?34; TP183 文献标识码: A 文章编号: 1004?373X(2020)09?0087?05
DNA sequence classification based on PSO?RBF neural network
SUN Qian1, ZHAO Xin1, 2
(1. College of Information and Technology, Jilin Agricultural University, Changchun 130118, China;
2. School of Mathematics and Big Data, Huizhou University, Huizhou 516007, China)
Abstract: In order to improve the accuracy and convergence speed of RBF (radial basis function) neural network algorithm in the application of DNA sequence classification, the contents of 20 kinds of amino acids are taken as the feature vectors of the DNA sequence; in view of the slow convergence speed caused by randomly?selected initial parameters of RBF neural network, the PSO (particle swarm optimization) is selected to optimize the RBF neural network parameters. The optimized neural network algorithm and the unoptimized neural network algorithm are contrasted by means of the real DNA sequences. The results show that the error of the optimized algorithm keeps stable after 12 iterations, and the classification accuracy reaches 93.75%, which is much higher than that (70%) of the unoptimized neural network algorithm. It is concluded from the experimental results that the PSO?RBF (particle swarm optimization radial basis function) neural network is effective in improving the convergence speed and accuracy of DNA sequence classification.
Keywords: DNA sequence classification; PSO?RBF neural network; feature extraction; classification model establishment; parameter optimization; classification effect contrast
0 引 言
DNA分类的主要思想是通过研究待分类DNA序列在碱基组成以及排列顺序等方面与已知类别DNA序列的相似度来判断该序列属于哪一类。该研究的目的是预测未知DNA序列的类别,从而了解其特性,对判断其是否属于隐存物种、外来物种或者濒危物种有重要意义。尤其是对细菌和病毒等种群,DNA序列分类技术得到了广泛的应用[1]。目前,DNA序列分类的数学方法有很多,具有代表性的主要有隐马尔可夫模型[2]、支持向量机[3]、神经网络[4]等。文献[5]首次将RBF神经网络应用于DNA序列分类问题中,但其在特征提取方面采用了单碱基、双碱基以及4D表示法相融合的方法,该方法运算复杂且只考虑了DNA序列的排列信息,忽视了其生物学意义。
人工神经网络模型[6]在1943年被首次提出,之后迅速被应用于各个领域[7?8]。作为一种前馈型网络,径向基函数(RBF)神经网络因其只有一个隐含层的简单构造,较快的学习速度,不容易陷入局部最小化,具有良好的泛化能力等优点,在近些年来受到越来越多的关注[9?11]。而在处理实际问题时,RBF神经网络各参数初始值的选取对性能有着较大影响,若初始值选取不当,可能使该网络收敛速度较慢。粒子群优化(PSO)算法有着较强的全局收敛能力以及鲁棒性,模型比较简单,不涉及到复杂的遗传问题。作为一种神经网络训练算法,PSO也体现出了很好的应用效果[12?13]。
针对DNA序列的RBF神经网络分类问题,本文以氨基酸含量作为特征向量,并使用PSO优化RBF神经网络。最后用GenBank中真实的DNA序列數据对优化前后两种算法进行实验,实验结果表明,与优化前的RBF算法相比较,PSO?RBF算法在分类准确率和收敛速度两个方面均有非常明显的改善。
1 DNA序列分类模型建立
1.1 RBF神经网络分类模型原理
RBF神经网络是一个三层结构的网络,如图1所示。第一层为输入层,顾名思义,该层用来接收数据的特征向量,每个单元代表一个特征,输入数据的特征向量的维数即为该层单元个数。其次是隐含层,RBF神经网络中第一层与第二层之间并不通过权值连接,而是直接到隐含层。在隐含层中,数据通过基函数将线性数据映射到非线性空间中。最后为输出层,得到最终结果。第二层与第三层之间由权值连接,即这次映射是线性的。输出层单元个数由具体问题分析,本文中最后输出的分类结果是一个实数,因此,输出层只有一个单元。由上述分析可知,若要构造一个RBF神经网络,除了要确定网络中各层节点数和权值,还要考虑隐含层中基函数的选取。本文选择形式简单,由具有良好解析性能的高斯函数作为径向基函数,其基本形式为:
[φx-cj=exp-x-cjTx-cjδ2j] (1)
式中:[cj]为数据中心;[δj]为参数宽度。从式(1)可得,RBF神经网络的训练参数有三个,分别为:数据中心[cj]、数据宽度[δj]以及权值[ωj]。
一般情况下选取梯度下降法训练RBF神经网络以得到各参数最优值。具体算法步骤如下:
1) 设置神经网络隐含层节点数[M],初始化数据中心[cj]、数据宽度[δj]以及权值[ωj]。误差[E=12i=1pe2i]作为目标函数,则第[i]个样本的误差表示为:
[ei=di-FXi=di-j=1MωjGXi-cj] (2)
2) 用下列公式计算以及调整网络中各隐含层单元到输出层之间的权值:
[Δωj=-η?E?ωj=-ηj=1PeiGXi-cjωj+1=ωj-η?E?ωj] (3)
式中[P]为训练样本数。
3) 隐含层数据中心为:
[Δcj=-η?E?cj=ηωjδ2jj=1PeiGXi-cjXi-cjΔcj+1=Δcj-η?E?cj] (4)
4) 隐含层数据中心的宽度为:
[Δδj=-η?E?δj=ηωjδ3jj=1PeiGXi-cjXi-cj2Δδj+1=Δδj-η?E?δj] (5)
5) 返回步骤2),当目标函数值达到误差要求时停止。迭代结束后得到的初始化数据中心[cj]、数据宽度[δj]以及权值[ωj]即为该神经网络的参数最优值。
1.2 粒子群优化算法原理
粒子群优化算法[14]将群体中单个个体看作是空间中以一定速度运动着的粒子,通过不停地更新每个粒子的最佳位置以及种群里最优的粒子位置,对参数进行优化。实现步骤如下[15]:
1) 数据初始化,其中,包括种群规模以及粒子初始速度和位置。
2) 根据模型评估每个粒子的位置。
3) 对比每个粒子当前位置与之前最优位置[Pbest]。若当前位置优,则记录下新位置,同时更新[Pbest]。
4) 将种群内最优的粒子位置记为[Gbest]。
5) 根据公式:
[vi+1=ωvi+c1r1Pid-xi+c2r2Pgd-xi] (6)
更新每个粒子的速度。[c1]和[c2]表示学习因子;[r1]和[r2]是[0,1]范围内的两个随机数。
6) 根据公式:
[xi+1=xi+vi+1] (7)
更新每个粒子的位置。
7) 重复步骤2)~步骤6)进行迭代,直到满足条件为止。
1.3 PSO优化RBF神经网络分类算法实现
1.3.1 数据样本选取
本文实验样本来源于GenBank,是由美国国家生物技术信息中心(NCBI)管理的DNA序列数据库,其数据主要来源于专业工作人员的真实测序结果。共选取了两类,在GenBank中的编号分别为ANFO01000063.1和NZ_COGN01000034.1,将这两组数据分别标记为A类和B类。分别对A类和B类数据进行截取,每300个碱基长度为一组,共分为298组。将这298组DNA序列分成训练样本和测试样本两部分,具体分类情况如表1所示。
1.3.2 DNA序列特征提取
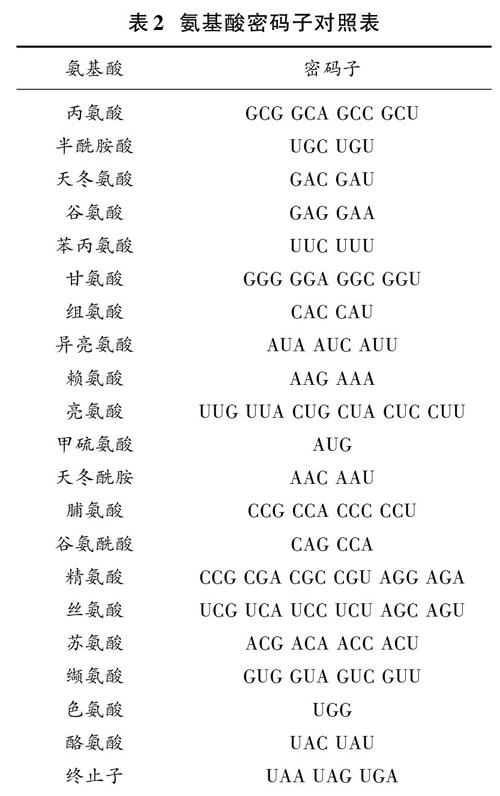
特征提取指的是把待识别对象通过一定的方法映射到新的特征空间里面,是特征的形成过程。DNA序列特征提取即将原始由A,C,G,T四个碱基构成的数据用数值化表示。一般情况下,DNA序列特征提取都是基于碱基含量和排列顺序这两个方面。根据生物学知识可知,在遗传的过程中,DNA序列首先要转录成信使RNA,信使RNA上相邻三个碱基的组成叫作密码子。所以说,当DNA序列的碱基排列顺序确定时,该序列所决定的密码子的个数以及种类也都是确定的。DNA序列中三个碱基的排列组合共有64种,这64种密码子决定了三个终止密码子以及20种氨基酸。对照情况如表2所示。
本文将DNA序列中氨基酸含量作为DNA特征。首先,氨基酸由三个不同的碱基组成,其含量不仅可以体现碱基含量也可体现排列序列。其次,对于不同的生物来说,氨基酸的种类和含量都有明显的不同,具有生物学意义。最后,DNA特征向量即为神经网络的输入,由于一共只有20种氨基酸,以其含量作为特征向量可以有效降低数据维度,简化网络机构。表3为部分数据示例(因为两类DNA序列都仅含有以下4种氨基酸,因此,其他氨基酸含量不予列出)。
1.3.3 PSO优化RBF神经网络过程
用PSO算法来优化RBF神经网络的过程为:
1) 初始化粒子群和RBF神经网络参数,确定粒子规模,将每个粒子的维度对应到神经网络的每一个参数上,即数据中心、宽度系数和权值。通过“试选法”确定RBF神经网络隐含层数目为3,设定群体规模为20,迭代次数为300。
2) 将RBF神经网络目标函数,即误差均方差设定为PSO适应度函数。
3) 将RBF连接权值的参数设置为向量,表示种群中的个体,并进行迭代,找到最优解。最后将算法结束后产生的最优解还原成网络参数。
4) 对测试样本进行分类。
PSO?RBF算法流程如图2所示。
2 结果分析
DNA序列分类问题中,将分类结果分别用0,1来表示,即神经网络输出期望为0或者1,实际输出结果为一个实数,若输出结果更接近0,则认为该DNA序列属于A类别;反之,则认为该DNA序列属于B类。输出结果与期望输出越接近,表明分类误差越小,RBF神经网络性能越好。
粒子群算法相关参数的设置也影响着其性能以及收敛速度,当参数设置过小时,网络收敛速度慢,参数设置过大时,算法过于复杂且运算时间过长。本文通过多次实验对比设置相关参数如下:总体规模数[m=20],总迭代次数[G=300],学习因子[c1=c2=2],权重[ω=0.1]。
图3为PSO优化RBF神经网络适应度曲线,从中可得,随着迭代次数的增加,曲线开始变得越来越平稳,即收敛速度开始变慢。当迭代300次之后,适应度数值达到7.65。
为证明PSO?RBF算法在DNA序列分类应用中的优越性,本文将RBF神经网络算法与PSO?RBF神经网络算法在网络训练阶段和测试样本分类结果两方面进行比较。
1) 在神经网络训练阶段的对比
图4表示RBF神经网络和PSO?RBF神经网络的训练均方差曲线图,从中可得,RBF神经网络在迭代209次之后均方差稳定在[0.08,0.09]区间,而PSO?RBF神经网络在迭代12次之后,均方差稳定在[0.07,0.08]区间。实验结果说明,对比未经优化的RBF神经网络算法,PSO?RBF算法收敛速度明显更快。
图5表示两种算法分别对训练样本的输出结果。从中可以看出,前130个训练样本期望输出为0,而PSO?RBF算法实际输出大部分都更加接近0,这种现象在后88个训练样本的实际输出中体现的更加明显。综合两类训练样本的实际输出可知,与RBF神经网络相比,PSO?RBF神经网络实际输出结果训练误差更小。
2) 对测试样本分类结果的对比
为验证两种算法的分类效果,分别用训练好的两个网络对剩余80个测试样本进行分类。表4为RBF神经网络算法和PSO?RBF算法对DNA序列测试样本分类结果。从表4中可以看出,對同一批测试样本分类,RBF神经网络算法分类错误数目为24个,正确率仅为70%,PSO?RBF神经网络算法分类错误数目为5个,正确率达到93.75%。实验结果表明,在DNA序列分类问题上,PSO?RBF算法正确率明显高于未经优化的RBF神经网络。
3 结 语
针对径向基神经网络在DNA分类方法应用中出现的分类精度低、性能不稳定的问题,本文在以下两方面进行了改进:在DNA特征提取方面,以氨基酸含量代替单双碱基含量作为DNA序列特征;在提高RBF神经网络收敛速度方面,选择PSO算法对RBF神经网络进行优化。为改进实验效果,选择了GenBank DNA序列库中的真实数据作为实验样本对改进前后两种算法进行对比。结果表明,与传统RBF神经网络相比,本文方法收敛速度更快、分类正确率更高。证明PSO算法优化RBF神经网络对其性能的提高是有效的。当然,本文算法也有一定的局限性,当数据量过大时,PSO算法耗时较长,如何平衡运行时间与正确率之间的关系也是待研究的问题。
注:本文通讯作者为赵昕。
参考文献
[1] 宋会银,张琪,胡愈炘,等.球状绿藻的隐性生物多样性及其分类学进展[J].生物多样性,2015,23(3):383?397.
[2] 郭彦明,陈黎飞,郭躬德.DNA序列的二阶隐马尔科夫模型分类[J].计算机系统应用,2015,24(9):22?28.
[3] 冼广铭,曾碧卿,冼广淋.最小二乘小波支持向量机的DNA序列分类方法[J].计算机工程与应用,2009,45(12):222?225.
[4] 敖丽敏,罗存金.基于神经网络集成的DNA序列分类方法研究[J].计算机仿真,2012,29(6):171?175.
[5] 孙晓楠.基于RBF神经网络的DNA序列分类方法[D].长春:吉林大学,2009.
[6] MCCULLOCH W S, PITTS W. A logical calculus of the ideas immanent in nervous activity [J]. The bulletin of mathematical biophysics, 1943, 5(4): 115?133.
[7] 于明,安梦涛,刘依.基于多特征与卷积神经网络的人脸表情识别[J].科学技术与工程,2018,18(13):104?110.
[8] 夏思为,段书凯,王丽丹,等.基于忆阻神经网络PID控制器设计[J].计算机学报,2013,36(12):2577?2586.
[9] 朱国俊,冯建军,郭鹏程,等.基于径向基神经网络?遗传算法的海流能水轮机叶片翼型优化[J].农业工程学报,2014,30(8):65?73.
[10] LUO S, LI S, FARID T. Chaos and nonlinear feedback control of the arch micro?electro?mechanical system [J]. Journal of systems science & complexity, 2018, 31(6): 1510?1524.
[11] ABDELATTI Marwan, YUAN Chengzhi, ZENG Wei, et al. Cooperative deterministic learning control for a group of homogeneous nonlinear uncertain robot manipulators [J]. Science China (Information sciences), 2018, 61(11): 63?81.
[12] 南敬昌,陆亚男,高明明.基于改进混合算法优化RBF网络的滤波器建模[J].计算机工程与科学,2018,40(7):1329?1336.
[13] 袁艳,叶俊浩,苏丽娟.基于改进的粒子群径向基神经网络的目标识别[J].计算机应用,2018,38(z1):6?8.
[14] 林玉娇.基于改进粒子群的PID参数整定应用研究[D].南宁:广西大学,2015.
[15] KEERTHI S S, LIN C J. Asymptotic behaviors of support vector machines with Gaussian kernel [J]. Neural computation, 2003, 15(7): 1667?1689.

