基于羧基功能化碳球的溶菌酶固定化和表面印迹研究
刘文倩,许 亮,钱立伟,徐甜甜,陈昊楠,张素风
(陕西科技大学 轻化工程国家级实验教学示范中心 陕西省造纸技术及特种纸品开发重点实验室 教育部轻化工助剂化学与技术重点实验室,陕西 西安 710021)
0 引言
近十几年来,在生物技术和生命科学领域,大量的研究致力于蛋白质药物的研发、医学诊断以及基于蛋白质组学的重大疾病发病机制的探究,在这些研究中,蛋白质的纯化、分离与检测至关重要[1].为了实现目标蛋白质的分离或分析,基于抗体-抗原特异性识别作用的方法是被认为最有效的方法,如免疫亲和色谱和酶偶联免疫吸附剂分析.然而,抗体的筛选与生产十分复杂,其成本较高,稳定性差,不易贮存,所以价格十分昂贵[2].因此,研究廉价、稳定、可反复使用的识别分离材料来取代生物抗体非常重要.
其中,以蛋白质为模板的分子印迹技术被认为是最有希望的一种方法[3].分子印迹技术是创造一种对模板分子在形状、大小以及官能团具有相互匹配功能的识别性材料的过程,合成的分子印迹聚合物(Molecularly Imprinted Polymers,MIPs)对模板分子具有较高的亲和力和选择性[4-6].MIPs是通过聚合物中构筑与模板分子形状、大小以及官能团具有相互匹配功能的印迹孔穴,来实现对特定蛋白的识别.在分子印迹领域中,以小分子为模板的分子印迹技术发展较为成熟[7-9],与之相比,以生物大分子为模板,特别是蛋白质分子,由于其庞大的分子体积使得高识别性蛋白印迹材料的制备依然面临许多挑战.
近些年来,将模板固定化法与表面印迹技术结合的蛋白MIPs制备策略成为热点[10].这是因为:(1)通过蛋白的固定化,使其位于基质材料表面相似位置与高度,因而有利于实现印迹层厚度与识别性、模板洗脱能力之间的规律调控;(2)通过调节基质表面官能团种类和数目,可有效增强蛋白的固载量,从而提高蛋白MIPs中印迹孔穴数目与识别性.我们研究的意义是寻求一种简单高效的、适用于蛋白质等大分子的印迹策略,为蛋白质等大分子的纯化、分离与检测提供新的思路和方法.
分子印迹技术被认为是在模板存在下,功能单体与交联剂共聚制备人工受体的最有利技术之一,然而,相较于印迹效果良好的小分子模板,生物大分子蛋白由于具有结构易变性、分子尺寸大和复杂的表面结构等特性,其成功的印迹仍面临许多挑战.针对这些问题,设计了多种策略来解决这些局限性,如原位印迹,微接触印迹和表面印迹.在这些方法中,表面印迹技术近年来受到了越来越多的关注.
本文采用模板固定化策略和表面印迹技术相结合的方法,选用蛋白负载量高的碳纳米微球用于蛋白固定化作为分子印迹聚合物的载体,选择聚合可控、能发生自聚合作用的多巴胺为功能单体来印迹蛋白质,固定化策略可以使目标蛋白定向富集在材料表面,得到的印迹孔穴较为规整,表面印迹技术使其印迹空穴位于表面,利于模板蛋白的传质过程,最终得到对目标蛋白具有高识别性能、良好吸附性量的印迹识别材料.
1 实验部分
1.1 实验原料及仪器
(1)主要原料
实验所有原料及试剂见表1所示.
表1 材料、试剂及其规格
(2)主要仪器
傅里叶红外光谱仪,德国布鲁克Bruker;扫描电子显微镜,捷克TESCAN;X射线光电子能谱分析仪,英国岛津;紫外可见分光光度计,上海佑科仪器仪表有限公司;Zeta电位测定仪,德国Mutek;SDS-PAGE凝胶电泳仪,北京六一仪器厂.
1.2 实验方法
1.2.1 羧基功能化碳纳米微球的制备(CFCs)
先将3 g葡萄糖溶解在30 mL去离子水中,然后加入不同浓度(相对于葡萄糖剂量为0 wt%、1 wt%、2 wt%、5 wt%、10 wt%和20 wt%)的丙烯酸.然后将反应混合物在190 ℃的条件下加水热处理5 h,得到的物料用水和乙醇洗两次,在60 ℃的条件下于真空条件下烘干,获得的样品[11].
1.2.2 蛋白印迹聚合物的制备(CFC@MIPs)
为制备溶菌酶表面印迹微球,首先将0.04 g CFCs在溶菌酶浓度为1.2 mg/mL的10 mL Tris-HCl缓冲液(0.01 M,pH 8.5)中孵育,室温下搅拌30 min,得到预反应溶液.接下来,将0.2 g多巴胺添加到溶液中在25 ℃下反应40 min.所得产品被混合溶液的2%(v/v)醋酸和2%(w/v)SDS去除模板,直到紫外线分光光度计在280 nm没有探测到吸附的蛋白质信号.所得到的印迹聚合物(CFC@MIPs)用水和乙醇洗涤数次,最后在60 ℃真空干燥备用.作为参考,非印迹聚合物(CFC@NIPs)以与CFC@MIPs相同的方法制备,但不添加模板蛋白.
1.2.3 溶菌酶浓度-吸光度标准曲线的绘制
以水作溶剂,配制0.1~1.2 mg/L不同浓度的溶菌酶水溶液,以水作参比,测定各浓度溶液在最大吸收波长(280 nm)下的紫外吸收强度,并绘制溶菌酶浓度-吸光度(Abs)标准曲线[12].
1.2.4 吸附实验表征
(1)吸附量计算
将测定的上清液的紫外吸光度在浓度-紫外吸收标准曲线上标定,计算上清液蛋白质浓度,用式(1)计算溶菌酶吸附量[13,14].
(1)
式(1)中:q-吸附量,mg/g;C0-初始浓度,mg/L;Ct-吸附后的浓度mg/L;V-体积,L;m-吸附剂质量,g.
(2)吸附动力学
为了研究颗粒的吸附动力学,将10 mg的CFC@MIPs(NIPs)悬浮于含10 mL 1.1 mg/mL的溶菌酶磷酸盐缓冲液(0.01 M,pH7.0)中.按规定时间间隔,在25 ℃下孵育10~110 min,从溶液中离心样品.用紫外可见分光光度计在280 nm检测波长下测定了残液中溶菌酶的浓度,并用准一级和准二级模型对数据进行拟合,以得到最合适的描述.
(3)吸附等温线
在吸附等温实验中,使用不同初始浓度的溶菌酶进行了测试,从0.1~1.2 mg/mL在25 ℃下孵化110 min后,离心分离后,用紫外可见分光光度计在280 nm检测波长下测定了上清液中溶菌酶的浓度,并用Langmiur和Freundlich模型对数据进行拟合,以得到最合适的描述.
(4)选择性吸附
在选择性吸附实验中,将25 mg CFC@MIP(NIPs)添加到离心管中,该离心管中含有25 mL 浓度为1.1 mg/mL的蛋白质溶液.在温育110 min后,将溶液通过离心分离,并使用紫外可见分光光度计测量上清液中蛋白质的浓度.
(5)竞争吸附
在竞争性吸附实验中,有10 mg颗粒放入10 mL磷酸盐缓冲液(0.01 M,pH 7.0)中,每种浓度的BSA和溶菌酶的蛋白质混合物1.0 mg/mL,在25 ℃下.孵育120 min后,将颗粒用2%(v/v)乙酸和2%(w/v)SDS的混合溶液洗涤去除模板蛋白直到紫外可见分光光度计在280 nm处检测不到蛋白吸附信号.合并蛋白洗脱液,并使用带有分子截止值为500 Da透析,然后冷冻干燥.将获得的产物溶于3 mL磷酸盐缓冲液(0.01 M,pH 7.0),并使用12.5%聚丙烯酰胺分离凝胶和5%聚丙烯酰胺堆积凝胶进行SDS-PAGE测量.
2 结果与讨论
本研究以葡萄糖为原料,以水热碳化制备的碳纳米微球为蛋白印迹的载体,以多巴胺作为功能单体和交联剂,将模板蛋白固定化策略与表面印迹技术相结合,制备了溶菌酶印迹核壳碳微球.在MIPs的制备过程中,通过调节丙烯酸的用量和多巴胺的聚合时间,可以方便地控制碳微球基质上羧基的含量和印迹聚合物的厚度.因此,制备的溶菌酶印迹微球在理论上可以表现出优异的识别能力和良好的吸附性能.
2.1 以碳纳米微球为载体的蛋白印迹聚合物的表征
2.1.1 丙烯酸添加量对蛋白固定量的影响
对于羧基功能化碳微球基质,丙烯酸用量是控制其表面电荷、形貌和蛋白固定化能力的重要参数.如图1所示,随着丙烯酸用量从0~2 wt%的增加,CFCs的负表面电荷显著增加.但碳球上溶菌酶的固定化量呈现先增加后减少的趋势,在2 wt%的丙烯酸用量下,最大固定化量约为59 mg/g.这可能是由于丙烯酸的过量,会导致羧基功能化碳纳米微球之间的交联和粘附,从而减少了基体中结合位点的数量.
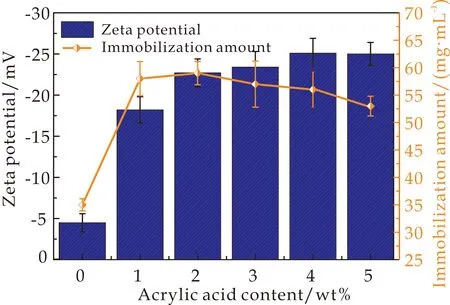
图1 丙烯酸添加量对碳球表面电荷和蛋白固定化影响
2.1.2 形貌表征
图2所示的SEM图像显示了CFCs、CFC@MIPs和CFC@NIPs的形态学特征.如图2(a)~(c)所示,CFCs、CFC@MIPs和CFC@NIPs的SEM证实了单分散纳米颗粒具有明确的球形形状和300~500 nm范围内的平均粒径.此外,图2(b)、(c)的SEM图像显示,与CFCs相比,CFC@MIPs和CFC@NIPs均表现出团聚现象,这可能是由于碳球表面包覆了一层聚多巴所导致的.此外,由于碳球基体的电子密度与印迹层相近,而印纹层的厚度又很薄,因此很难对印迹层厚度的准确厚度进行评估.

(a)CFCs
2.1.3 结构表征
由图3(a)全反射红外光谱图可以看出,与CFCs相比CFC@MIPs和CFC@NIPs在3 400 cm-1左右的峰变宽,这是由于PDA中羟基吸附水和氨基重叠造成的;此外,在1 506 cm-1和1 645 cm-1分别对应于CFC@MIPs和CFC@NIPs中PDA苯基C=C拉伸和N-H弯曲的典型特征峰[15],证明多巴胺成功包覆于碳纳米微球上,成功制备了蛋白印迹材料和非印迹材料.采用XPS法对印迹聚合物的表面化学成分进行了表征,通过XPS分析确定了0~1 200 eV范围内的碳(C)、氧(O)和氮(N)元素.
如图3(b)所示,CFCs中没有氮的信号,但是在DA自聚合后,N峰值出现在CFC@MIPs和CFC@NIPs中的400.84 eV处,这与PDA中氮元素所对应,进而说明了CFC@MIPs和CFC@NIPs的成功合成.
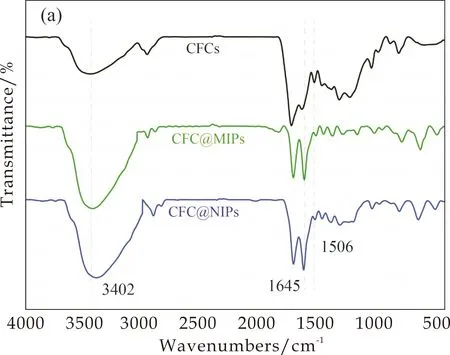
(a)FT-IR图
2.2 溶菌酶印迹聚合物吸附性能表征
2.2.1 溶菌酶浓度-吸光度(Abs)标准曲线
通过紫外分光光度法测得温度为25 ℃,pH值为7时,不同浓度下溶菌酶水溶液的吸光度,从而制得溶菌酶溶液-吸光度(Abs)标准曲线,对曲线进行线性拟合,如图4所示,标准曲线拟合后方程表示为y=2.293 6x+0.073 2,R2=0.998 4.
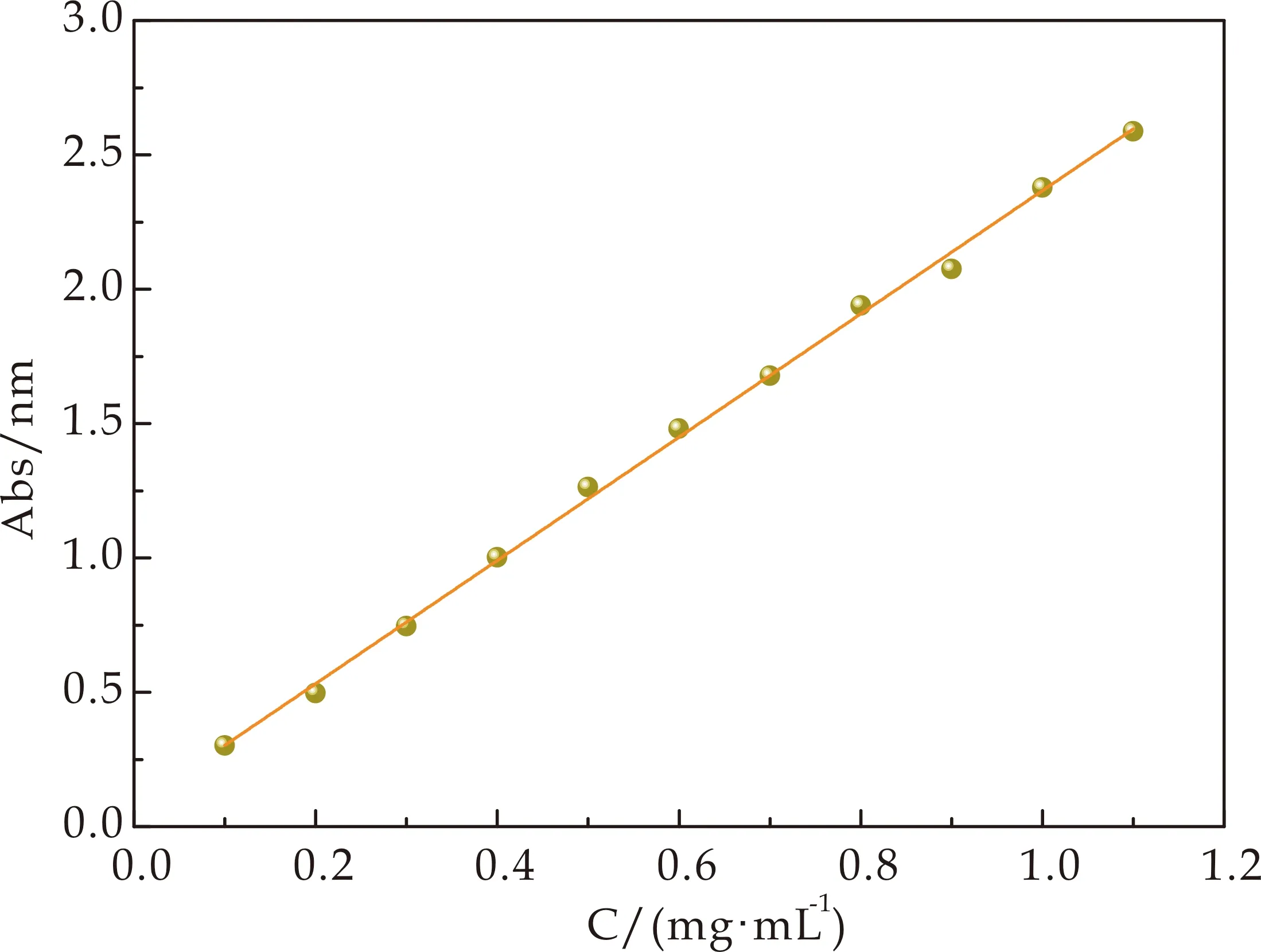
图4 溶菌酶浓度-吸光度(Abs)标准曲线
2.2.2 多巴胺聚合时间对印迹效果的影响
在表面印迹技术中,印迹层的厚度是影响MIPs识别能力的重要因素,因此本文研究了多巴胺聚合时间对CFC@MIPs识别的影响[16-18].如图5所示,聚合时间不到40 min的时候,识别能力CFC@MIPs随着反应时间的增加而显著增加,表明聚多巴胺层的增厚的初始阶段聚合有利于建设更完整的印迹腔,从而达到更好的识别能力.但随着聚合时间的延长,CFC@MIPs的识别能力下降.这可能是由于印迹层太厚,这很容易导致模板分子包埋难以洗脱,减少了印迹腔的数量.因此,为了获得最佳的识别能力,选择40 min的聚合时间作为制备CFC@MIPs的最佳反应条件.

图5 多巴胺聚合时间对印迹效果的影响
2.2.3 吸附动力学研究
为了确定接触时间对吸附容量的影响,分别对CFC@MIPs和CFC@NIPs微球的吸附动力学进行了评价.如图6所示,在1~100 min内,印迹微球的吸附速度比非印迹微球更快.这表明,CFC@MIPs和溶菌酶之间的相互作用明显强于CFC@NIPs,这很可能是印迹微球内存在溶菌酶形状的印迹孔穴.吸附100 min后,CFC@MIPs和CFC@NIPs的吸附速率均逐渐降低,说明微球内外部环境的驱动力降低.此外,值得注意的是,印迹和非印迹微球均能在110 min内达到平衡,这可能与表面印迹技术制备的吸附剂传质阻力低有关.因此,在接下来的吸附研究中,CFC@MIPs和CFC@NIPs接触时间均选择了110 min.
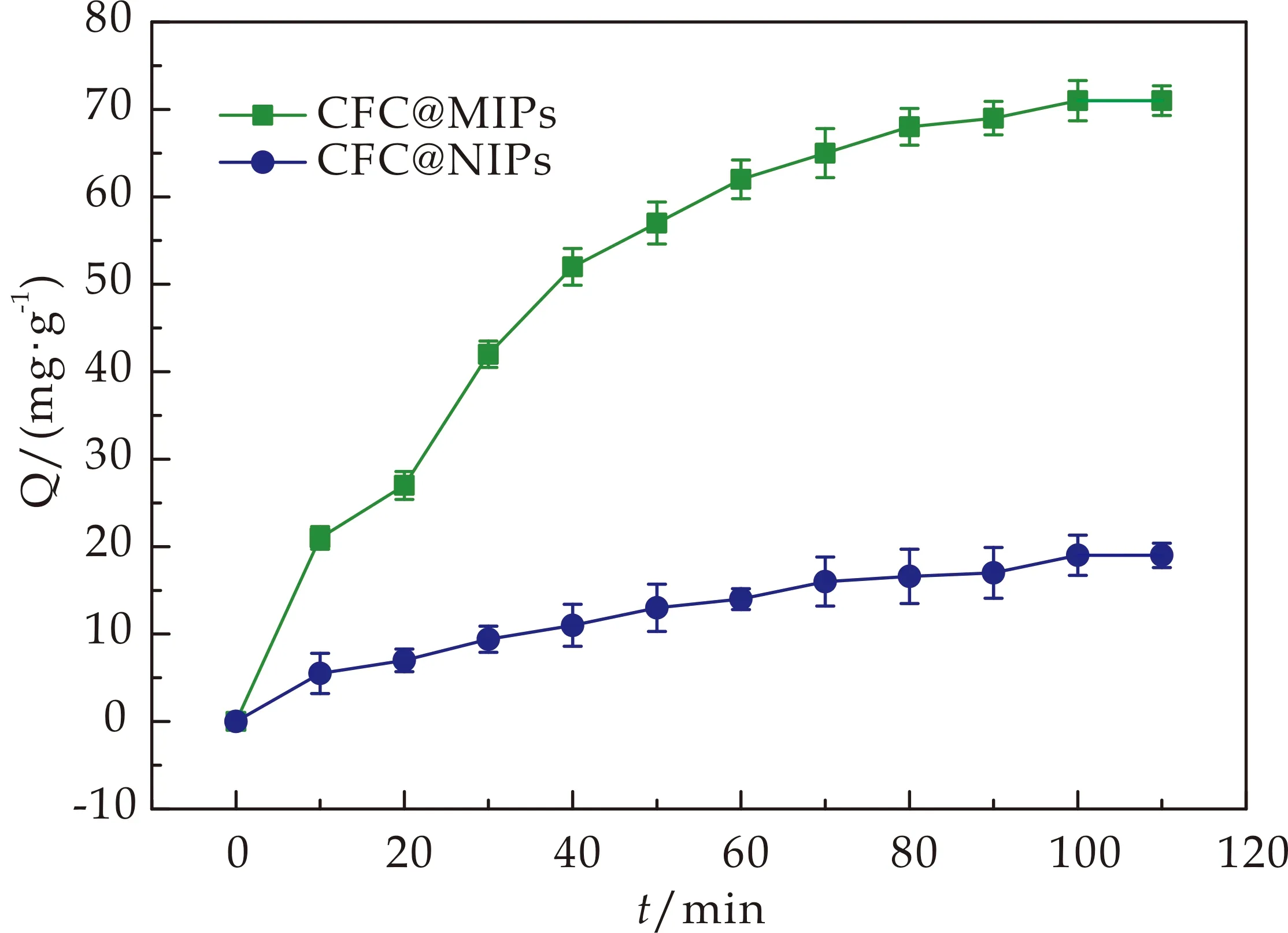
图6 不同接触时间内CFC@MIPs和CFC@NIPs吸附性能对比
为了进一步研究CFC@MIPs的吸附和识别行为,建立了准一级动力学模型和准一级动力学模型对数据进行拟合,以期得到最适合的描述.准一级模型与准二级模型公式如式(2)、(3)所示[19,20]:
ln(Qe-Qt)=lnQe-k1t
(2)
(3)
式(2)、(3)中:Qe(mg/g)和Qt(mg/g)分别为平衡时刻和不同时刻对应的吸附容量.k1(min-1)和k2(mg·g-1min-1)分别是准一级和准二级吸附速率常数.准一阶模型描述了吸附位点的占用率与未占据位点的数量成正比,而准二阶模型则是吸附质与吸附质的化学吸附量的总和.
CFC@MIPs在温度为25 ℃,pH值为7时,对浓度为1.1 mg/g的溶菌酶吸附动力学准一级和准二级动力学拟合结果如图7和图8所示,拟合结果总结如表1所示.
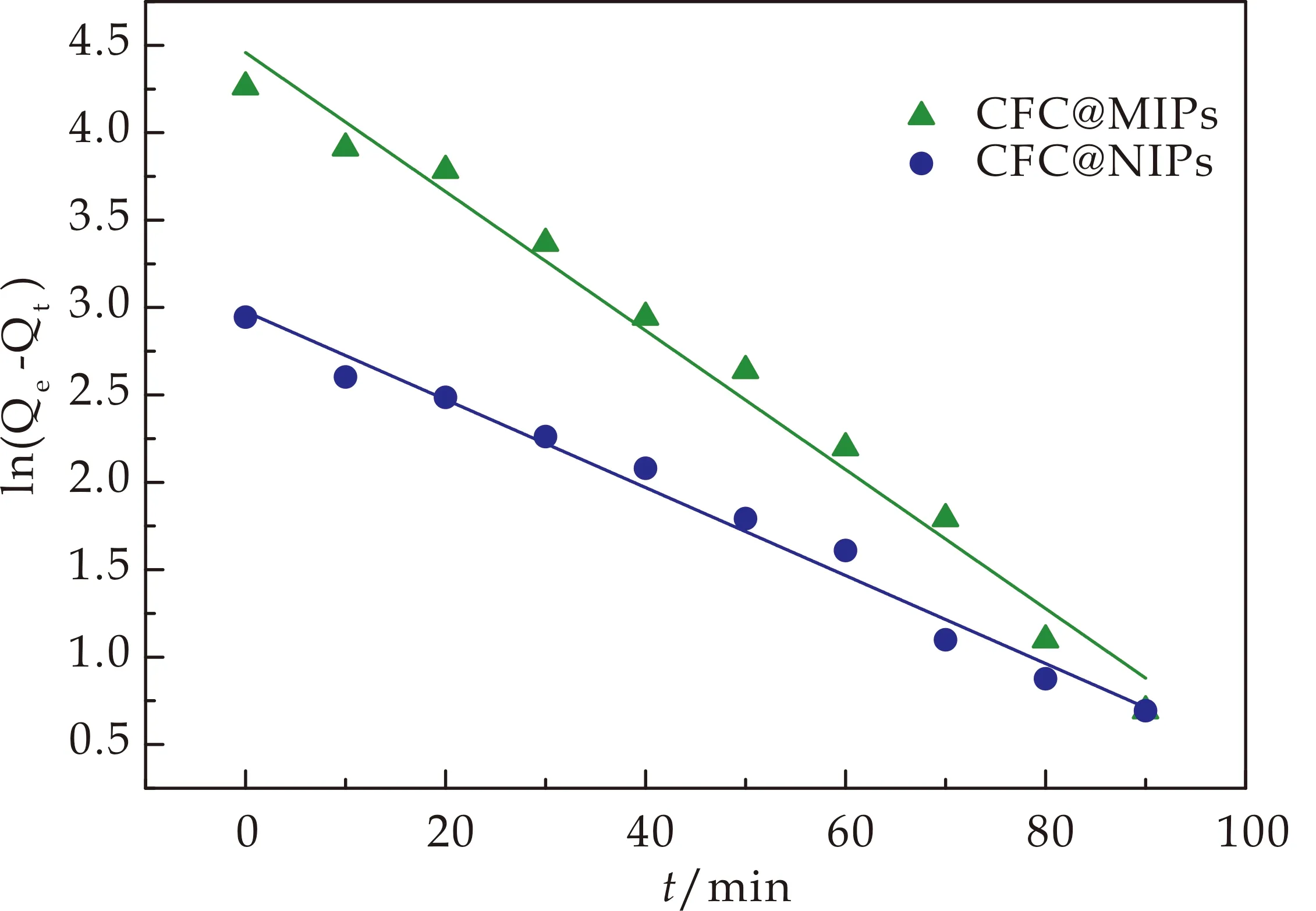
图7 CFC@MIPs和CFC@NIPs的准一级吸附动力学模型拟合

图8 CFC@MIPs和CFC@NIPs的准二级吸附动力学模型拟合

表1 吸附动力学拟合参数
如表1所示,CFC@MIPs和CFC@NIPs的伪二阶模型与实验数据拟合较好,R2值比伪一阶模型高,表明吸附可能是由于化学吸附.此外,CFC@MIPs的Qe值明显大于CFC@NIPs,说明它们对溶菌酶有良好的印迹作用.
2.2.4 等温吸附研究
为了研究初始浓度对吸附容量和识别能力的影响,对MIPs和NIPs样品进行吸附等温线实验.从图9可以看出,随着溶菌酶浓度从0.1增加到1.0 mg/mL,CFC@MIPs的吸附量迅速增加,在1.1 mg/mL以上达到了71 mg/g的饱和吸附能力.另外,CFC@MIPs吸附的溶菌酶量明显高于CFC@NIPs,1.1 mg/mL溶菌酶溶液的最大IF值也达到3.3.在随后的吸附实验中,选择溶菌酶浓度为1.1 mg/mL,识别效果最佳.
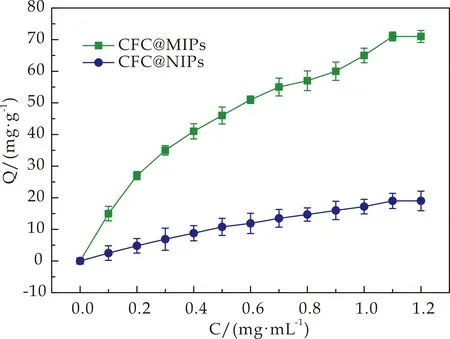
图9 溶菌酶不同初始浓度下CFC@MIPs(NIPs)吸附性能对比
为了进一步研究CFC@MIPs和CFC@NIPs对溶菌酶的吸附和识别机制,分别采用Langmuir模型和Freundlich模型对吸附等温曲线进行分析.Langmuir等温线模型假设吸附发生在特定的均相位点和单层吸附,每个位点只能吸附一个分子,而Freundlich等温线模型适用于非均相表面的多层吸附.这些模型表示如式(4)、(5)所示[21,22]
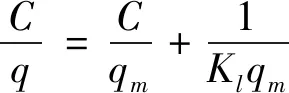
(4)
(5)
式(4)、(5)中:qm(mg/g)和Kl(mL/mg)分别为理论最大吸附容量和结合极限的Langmuir常数.Kf(mg/g)是与吸附能力有关的Freundlich常数.
CFC@MIPs和CFC@NIPs在温度为25 ℃,pH值为7时,对不同浓度的溶菌酶吸附Langmuir模型结果如图10所示.它们在温度为25 ℃,pH值为7时,对不同浓度的溶菌酶吸附Freundlich模型结果如图11所示,拟合结果如表2所示.
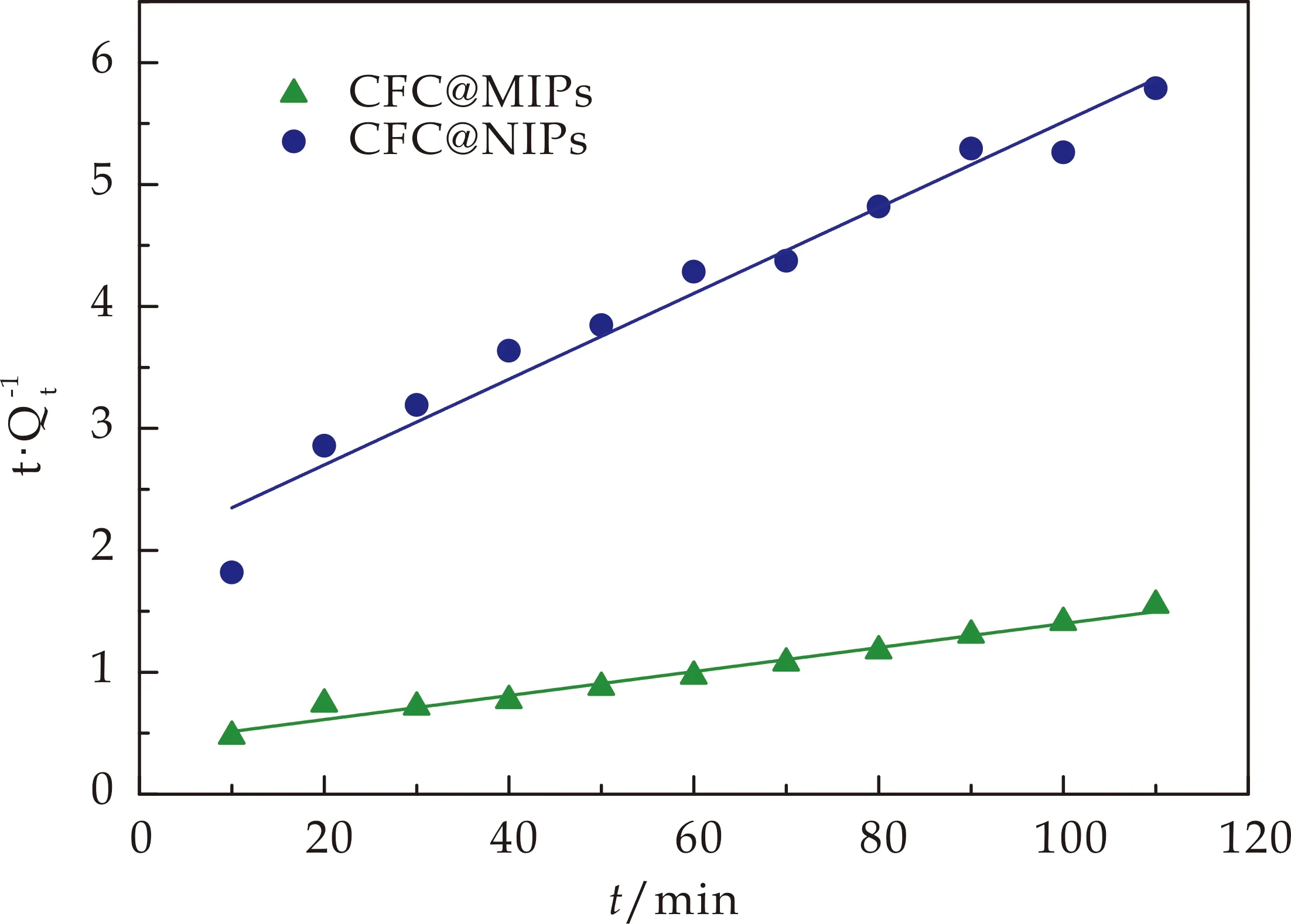
图10 CFC@MIPs和CFC@NIPs的Langmuir吸附等温模型拟合
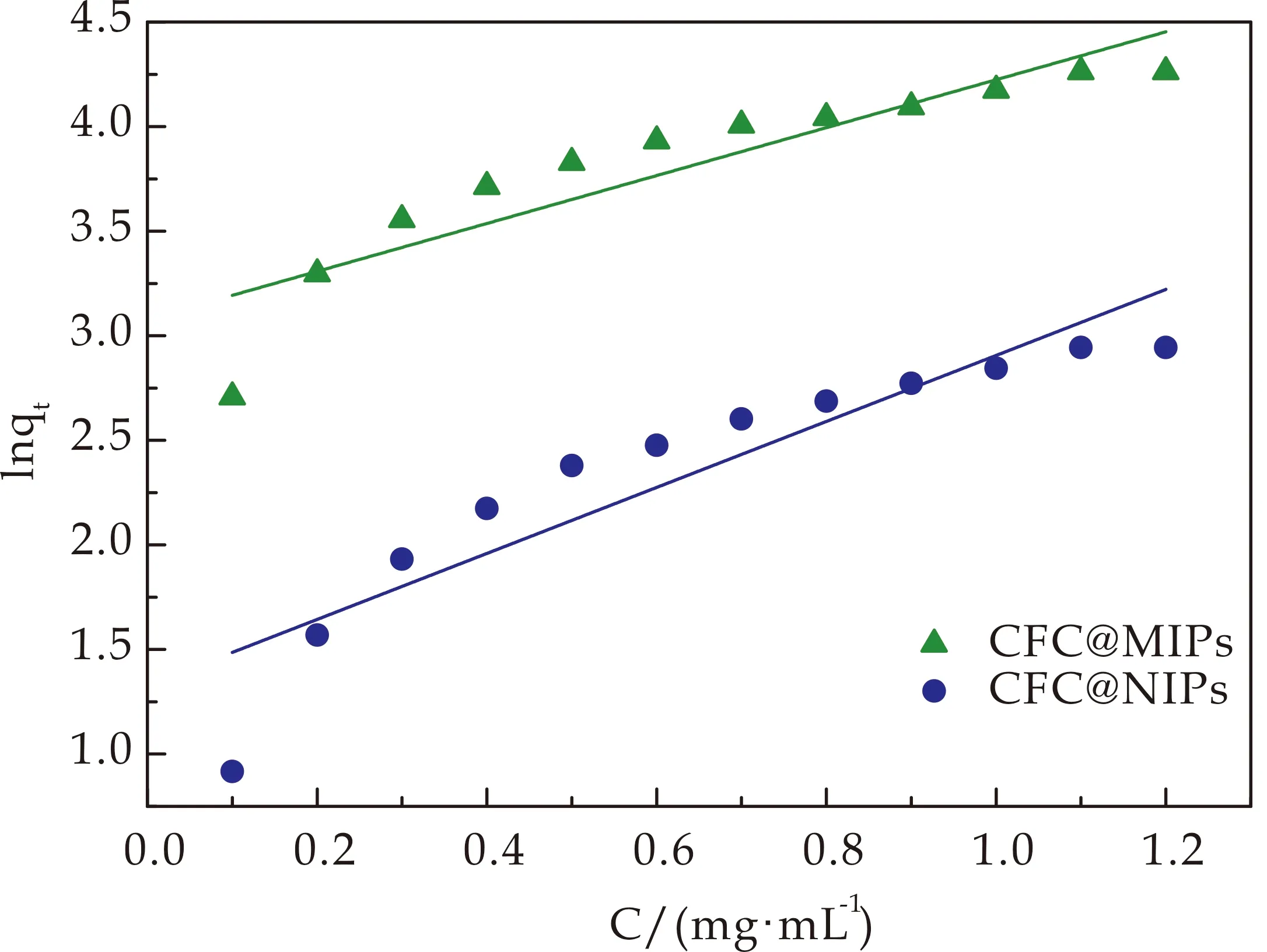
图11 CFC@MIPs和CFC@NIPs的Freundlich吸附等温模型拟合
如表2所示,与Freundlich模型拟合结果相比,CFC@MIPs和CFC@NIPs的实验数据与Langmuir模型拟合较好,R2都大于0.98,说明两种材料对溶菌酶的吸附过程类似于单分子层吸附,并且材料中的吸附位点分布较为均匀,进一步证实了CFC@MIPs中存在溶菌酶大小的印迹空腔.

表2 吸附等温线拟合参数
2.2.5 选择性吸附实验
选择几种蛋白包括卵清白蛋白(Mw=43 kDa,pI=4.5),牛血红蛋白(Mw=65 kDa,pI=6.9),牛血清白蛋白(Mw=66.4 kDa,pI=4.8)和细胞色素C(Mw=13 kDa,pI=10.8)作为参比蛋白,以研究CFC@MIPs和CFC@NIPs对溶菌酶的识别能力.如图12所示,CFC@MIPs可以选择性地吸附溶菌酶,印迹因子可达3.3.但是,仍然可以观察到有一些牛血红蛋白和细胞色素C与MIPs结合,这可能是由于π-π堆积,氢键和蛋白质和聚多巴胺层之间的静电相互作用.此外,与牛血红蛋白和细胞色素C相比,CFC@MIPs吸附牛血清白蛋白和卵清白蛋白的容量更少,说明两者之间存在较强的静电排斥作用[23].
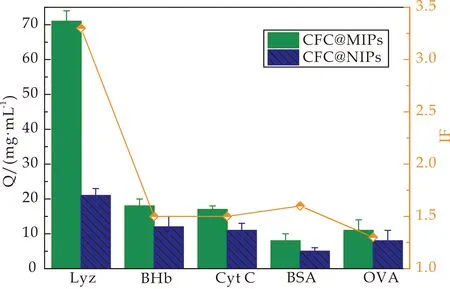
图12 选择性吸附实验
2.2.6 竞争吸附
通过竞争吸附实验进一步研究了MIPs在蛋白混合溶液中对溶菌酶的识别能力.从图13可以看出,在SDS-PAGE分析中,泳道4的两条明显的条带显示了NIPs可以通过非特异性吸附提取牛血清白蛋白(Mw=66.4 kDa,pI=4.8)和溶菌酶(Mw=14.4 kDa,pI=11.2).然而,与NIPs相比,只在泳道3中发现了一条条带,且颜色更深,而在14.4 kDa处代表溶菌酶,进一步说明MIPs表现出了良好的特异性识别能力.从SDS-PAGE分析结果可以推断,CFC@MIPs纳米颗粒具有大量与模板蛋白溶菌酶互补的印迹空腔.
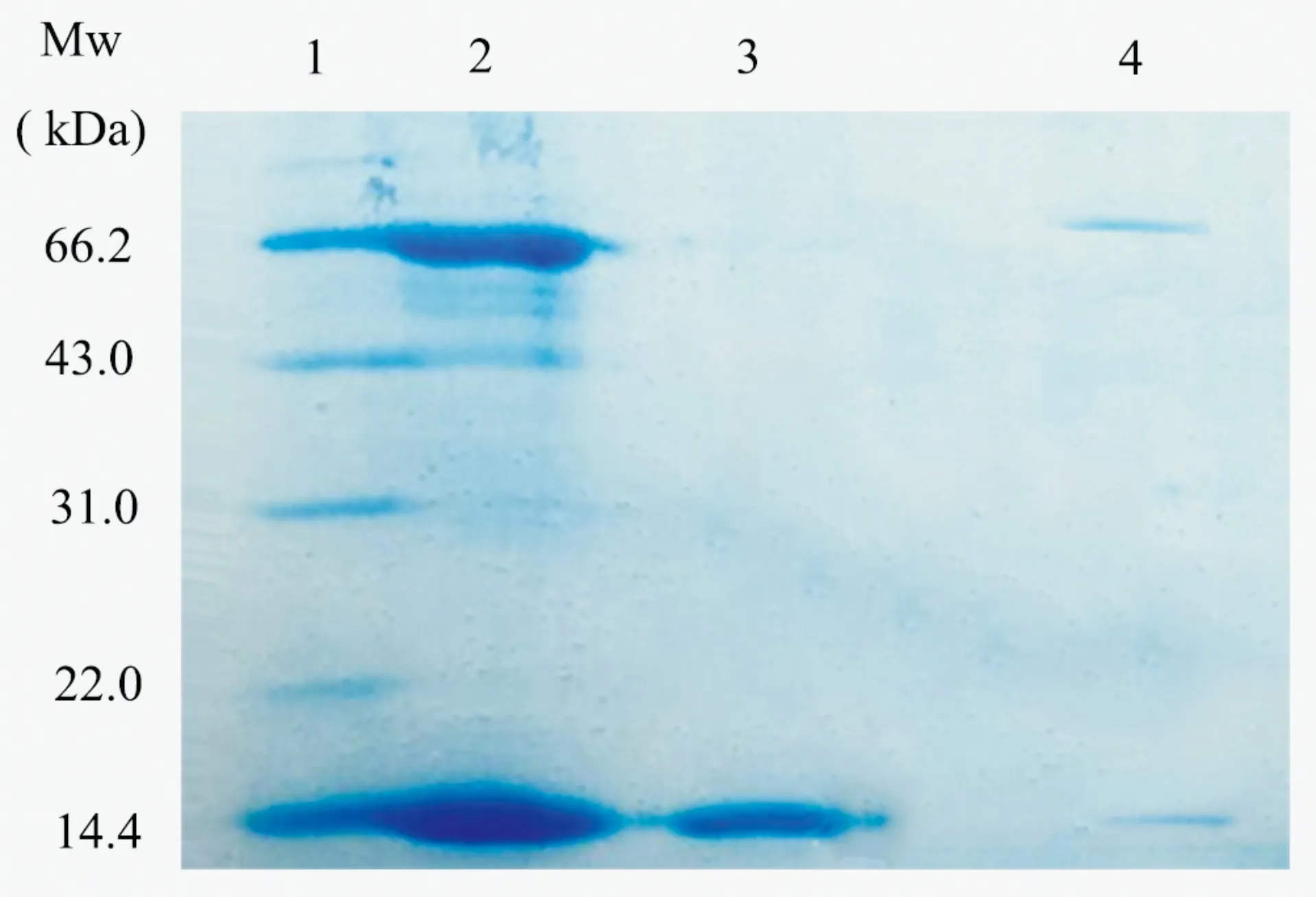
条带1为蛋白质Marker;条带2为溶菌酶和牛血清白蛋白的混合蛋白溶液:条带3和4分别为从CFC@MIPs和CFC@NIPs中得到的洗脱蛋白图13 SDS-PAFE凝胶电泳分析
3 结论
对基于羧基功能化的碳纳米微球为载体的蛋白印迹聚合物通过扫描电子显微镜(SEM)、X射线光电子能谱分析仪(XPS)和傅里叶红外光谱仪(FT-IR)进行了物理和化学性能检测及分析,可以证实多巴胺成功包覆于羧基功能化碳纳米微球表面,成功制备了溶菌酶蛋白印迹聚合物.
吸附实验结果证明,在温度为25 ℃、pH为7的环境下,该印迹纳米材料对目标蛋白有较高的吸附能力,吸附量为71 mg/g,快速平衡时间为110 min,溶菌酶印迹聚合物的吸附动力学数据符合准二级动力学模型,等温吸附数据符合Langmuir模型,吸附属于化学单层吸附,且对目标蛋白有较好的选择性,印迹因子可达3.3.

