基于转录组测序分析自然感染的绵羊肺腺瘤肿瘤组织差异表达基因富集的生物学功能模块
杨 惠,刘淑英*
(1.内蒙古农业大学兽医学院,内蒙古 呼和浩特 010018;2.农业部动物临床诊疗技术重点实验室,内蒙古 呼和浩特 010018;3.内蒙古基础兽医学重点实验室,内蒙古 呼和浩特 010018)
绵羊肺腺瘤(Ovine pulmonary denocarcinoma,OPA)是由绵羊肺腺瘤病毒(Jaagsiekte sheep retrovirus,JS⁃RV)引起的绵羊可传播性的肺脏肿瘤[1]。OPA 临床表现为呼吸困难、咳嗽、流鼻液、精神萎靡,最终导致患羊肺泡功能丧失,呼吸困难而死亡。该病目前已在世界范围内广泛传播,并严重影响我国绵羊产业的发展[2]。尽管国内外关于OPA 肿瘤机制的研究已经持续多年,然而关于OPA 疾病分子机制的了解仍不全面。
大量研究显示,转录组测序(Transcriptome se⁃quencing,RNA-Seq)可在RNA 水平上研究基因表达差异,目前,RNA-Seq 技术已应用于肝炎、艾滋病、布鲁氏菌病等疾病的分子致病机制研究[3-4]。此外,该技术还大量应用于动物健康管理[5]、营养代谢[6]、育种[7]等畜牧生产领域。2019 年Karagianni 等人对短时期人工感染的JSRV 羔羊进行了RNA-Seq研究,寻找到1 971 个差异表达转录本[8],但Karagi⁃anni 等人所提供的RNA-Seq 信息对临床症状明显的OPA 自然感染病例并不适用。一方面,OPA 疾病主要发生在成年羊个体当中,偶见羔羊感染[2];另一方面,JSRV 感染的潜伏期较长,短时期人工感染JSRV 无法模拟长期JSRV 感染造成的遗传信息积累[2]。目前尚无利用RNA-Seq 技术系统揭示自然感染OPA 肺肿瘤组织转录本变化的相关研究。因此,需要针对临床症状明显的自然感染的OPA 病例进行RNA-Seq 研究,以进一步完善OPA 分子致病机制的具体信息。
本研究收集了自然感染的OPA 肺肿瘤组织及绵羊健康肺组织,利用RNA-seq 技术进行测序分析,鉴定差异表达基因(Differentially expression genes,EGs),通过GO 功能富集分析,分别寻找表达上调基因及表达下调基因富集的相关生物学功能。为进一步研究OPA 致癌机制提供基础的参考资料。
1 材料与方法
1.1 实验动物及主要试剂3 例临床症状明显的自然感染OPA 病羊及3 例健康羊来源于内蒙古自治区四子王旗某羊场(内蒙古,乌兰察布市)。TRIzol、Prime ScriptTMRT reagent Kit with g DNA Eraser 和SYBR GreenⅠ购自TaKaRa 公司;六碱基随机引物购自生工生物工程(上海)有限公司;poly-T 寡核苷酸磁珠、AMPure XP beads、RNase-Free DNase Set、T4 DNA 聚 合 酶、Klenow DNA 聚合酶均购自Invitrogen 公司;RNAClean XP Kit 购自Beckman 公司;Agilent Bioan-alyzer 2100 购 自Agilent 公 司;Fragmentation Buffer 购自Epicentre 公 司。
1.2 样品采集及总RNA 提取采集健康羊肺组织及自然感染OPA 的羊肺肿瘤组织样品,利用TRIzol试剂分别提取3 例绵羊肺腺瘤肺肿瘤组织及3 例健康羊肺脏组织的总RNA,利用Nano Drop 核酸蛋白仪测定总RNA 的浓度和纯度,利用Agilent Bioana⁃lyzer 2100 系统分析所提取RNA 的RIN(RNA Integri⁃ty Number,RIN)值,并检测RNA 的完整性。
1.3 cDNA 文库的制备及质量控制总RNA 经质检合格后,使用连接有Oligo(dT)的磁珠富集样本mRNA。抽提的mRNA 经片段化试剂(Fragmentation Buffer)随机打断,以mRNA 为模板,用六碱基随机引物合成单链cDNA,随后加入dNTPs、RNaseH 和DNA Polymerase I 进行双链cDNA 的合成。采用AM⁃Pure XP beads 纯化双链产物,利用T4 DNA 聚合酶和Klenow DNA 聚合酶将DNA 的粘性末端修复为平末端,3'末端加碱基A 并加接头,利用AMPureXP beads进行片段选择并扩增获得最终测序文库。文库质检合格后由联川生物技术有限公司,利用Illumina Hiseq 4000测序,测序读长为双端2×150 bp。
1.4 数据处理通过Seqtk(2.0)程序将测序结果中总体质量较低、含有测序引物、末端质量偏低等不合格的Reads 进行过滤,得到可用于数据分析的clean Reads;然后应用bowtie 2 采用局部比对将reads 进行基因匹配,以UCSC(http://genome.ucsc.edu/)数据库中的绵羊基因组(Ovis_aries_3.1.88)作为参照进行拼接;应用HTSeq(2.0)对TopHat(2.0)比对后的reads进行计数,基因表达水平以基因表达丰度值(Frag⁃ments Per Kilobase of exon model per Million mapped reads,FPKM)来表示。
1.5 生物信息学分析使用DESeq R(1.18.0)对患OPA的绵羊肿瘤组织及绵羊健康肺组织的表达基因进行统计,将同时满足Fold-change 2 与p 0.05 条件的基因,进一步使用Benjamini&Hochberg方法调整所得p 值。最终满足adjust p<0.05 的基因被认定为DEGs。将DEGs 中表达上调和表达下调的基因分别映射 至Gene Ontology(GO)(http://www.geneontology.org/)数据库中,将DEGs 分别富集于生物过程(Biological process)、细胞成分(Cellular component)和分子功能(Molecular function)层面,不同DEGs 富集到同一GO功能条目下即被划分为一个生物学模块,模块化富集网络由Cytoscape 中的Clue GO 插件生成。
1.6 转录组结果的RT-qPCR 验证随机选取7 个DEGs,根据GenBank 中的基因序列,采用Primer 6.0 软件设计引物(表1),以GAPDH、18S rRNA、ACTB 作为内参基因[9],以1.3 中制备的cDNA 为模板,采用SYBR GreenⅠ方法经RT-qPCR 检测,以验证转录组测序结果的可靠性。每个样品设3 个重复,反应程序为50 ℃2 min;95 ℃10 min、95 ℃15 s、60 ℃1 min, 40 个循环,相对定量的结果采用2-ΔΔCt法计算[10]。

表1 7 个DEGs 及内参基因的引物信息Table 1 Primer sequencesof 7 DEGs and reference genes
2 结 果
2.1 转录组测序数据分析利用Nano Drop 核酸蛋白仪测定OPA 患羊肺肿瘤组织和健康羊肺组织提取的RNA 质量合格, 6 个样本的RNA 平均浓度为399.30±12.64 ng/μL。通过Illumina Hiseq 4000 分析,样本平均包含7.33±1.14 G 的原始读数。质量控制后测序质量值 20(Q20)的碱基所占平均比例为99.63%,测序质量值 30(Q30)的碱基所占平均比例为95.63%,GC 平均含量值为52.50%。超过85.77%的纯净读数被映射到绵羊参考基因组,大约90% 的有效读数被映射到绵羊参考基因组的外显子区域。综合以上数据表明,转录组测序结果质量较高,可用于后续生物信息学分析。
2.2 自然感染OPA 羊肺肿瘤组织与健康羊肺组织DEGs 相关分析通过从RNA-Seq 产生的高质量序列鉴定DEGs,结果显示,对比绵羊参考基因组,6个检测样本拥有共同注释基因15 304 个。在OPA 患羊肺肿瘤组织中发现了366 个DEGs,其中154 个表达上调基因,212 个表达下调基因(图1A),366 个DEGs 对应2 143 个差异转录本,其中1 360 个表达上调转录本,783 个表达下调转录本(图1B)。以上数据表明,自然感染OPA 羊肺肿瘤组织与健康羊肺组织之间存在大量DEGs,可作进一步功能富集分析。
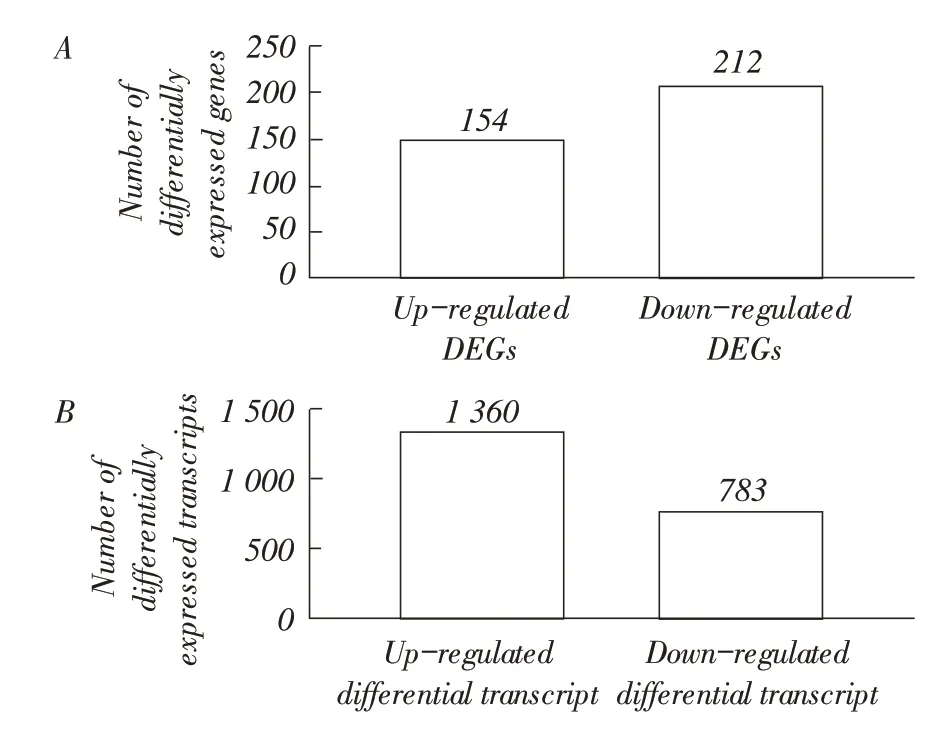
图1 自然感染OPA 羊肺肿瘤组织与健康羊肺组织DEGs 的分析Fig.1 Differential gene expression between OPA tumor tissue and normal lung tissue
2.3 自然感染OPA 羊肺肿瘤组织与健康羊肺组织DEGs 的GO 功能分类为进一步了解自然感染OPA 羊肺肿瘤组织相关生物学功能的变化,本研究将表达上调的DEGs 和表达下调的DEGs 分别进行GO富集,结果显示,自然感染OPA 羊肺肿瘤组织DEGs在分子功能网络中存在4 个上调功能模块(L-亮氨酸跨膜转运蛋白活性、铜还原酶活性、NADH 脱氢酶活性、NAD+二磷酸酶活性)和2 个下调功能模块(III 型转化生长因子β 受体结合、DNA 导向聚合酶活性);在生物学过程中存在1 个上调模块(磷酸蛋白磷酸酶活性的负调控)和2 个下调模块(胃气化趋同延伸、面部发育);在细胞成分网络中存在1 个上调模块(Nem1-Spo 7 磷酸酶复合物)。综合以上数据,自然感染OPA羊肺肿瘤组织DEGs 共富集上调功能模块6 个(表2),下调功能模块4 个(表3)。表明自然感染OPA 羊肺肿瘤组织DEGs 富集的生物学模块主要与细胞增殖调节相关,即在OPA 肿瘤形成过程中,细胞增殖相关的生物学功能被大量激活并伴随大量的能量代谢紊乱。
2.4 转录组结果的RT-qPCR 验证在筛选到的DEGs 中随机挑选7 个基因进行RT-qPCR 验证,结果显示,7 个DEGs 在RT-qPCR 的检测结果与RNAseq 中的检测结果基本一致(图2)。表明转录组筛选出的DEGs 可信程度较高。
3 讨 论
近年来通过转录组测序了解疾病分子机制的研究已经十分普遍,大量的RNA-seq 相关研究重点多集中于寻找药物治疗靶点及疾病生物学标志物等[9-11]。本研究采集自然感染OPA 羊肺肿瘤组织和绵羊健康肺组织,利用高通量测序RNA-seq 技术筛选自然感染OPA 羊肺肿瘤组织中的DEGs,通过GO 功能注释划分DEGs 富集的相关生物学功能模块,为深入研究自然感染OPA 的分子致病机制提供了基础的基因表达信息。本实验利用的研究方法均符合转录组数据分析处理标准[9-11]。
与其它肿瘤RNA-seq 分析报告相比,本研究关于DEGs 的筛选数量相对较少,这样的分析结果主要由本研究的实验材料所决定。一方面,自然感染OPA 羊肺肿瘤组织中除肿瘤细胞外还掺杂单核巨噬细胞、血细胞等,与单一细胞类型的体外研究相比,完整组织的基因表达丰度本身极不均一,容易造成测序结果的统计误差[11]。另一方面,大多数肿瘤研究都是根据肿瘤病理学变化将肿瘤疾病进程分期,综合同一肿瘤不同发病时期的DEGs,可以得到更多、更具体的差异表达基因信息[12]。在本研究中,仅针对OPA 临床特征明显的自然感染晚期的病例进行研究,缺失OPA 肿瘤形成早期JSRV 诱导的DEGs。由于JSRV 感染早期介导的部分DEGs 在肿瘤发展进程中,通过细胞自我平衡(自噬、凋亡等)以及机体稳态平衡(信号通路代偿、肿瘤微环境调节等)在肿瘤形成中后期可以恢复正常表达水平,导致本实验筛选到的DEGs 与其它肿瘤分期试验相比相对较少[13]。

表2 DEGs 富集的上调生物学功能模块Table 2 Up-regulated biological function modules for differentially expressed gene enrichment

表3 DEGs 富集的下调生物学功能模块Table 3 Down-regulated biological function modules for differentially expressed gene enrichment

图2 DEGs 的RT-qPCR 验证Fig.2 Differential expression genes confirmed by RT-qPCR assay
本研究中DEGs 集中富集在以下生物学过程:L-亮氨酸跨膜转运蛋白活性、铜还原酶活性、NADH 脱氢酶活性、NAD+二磷酸酶活性、磷酸蛋白磷酸酶活性的负调控、Nem1-Spo7 磷酸酶复合物、III 型转化生长因子β 受体结合、 DNA 导向的DNA聚合酶活性、胃气化趋同延伸、面部发育。更具体地说,对于生物学过程而言,DEG 主要与细胞复制后修复和核苷代谢过程(POLH、UBE2N、EN⁃SOARG00000014411、UPRT)直接相关,从而影响了基因组的稳定性[14]、肿瘤发生[15]和致癌信号转导[16]。对于细胞成分而言,主要表现为多个DEGs(TGFβ 3、FGF11、FYVE、RhoGEF、FGD1)集 中 富 集 于Nem1-Spo7 磷酸酶复合物这一生物学功能模块下,该功能主要参与癌细胞侵袭和转移[17],肿瘤发生(TGFβ3、FGF11)[18]和 癌 症 易 感 性(FGF11、FYVE、FGD1)[19]。对于分子功能而言,DEGs 主要为FGF11、FGF18、INHA、PGF、TGFβ3、ARHGEF37、DNMBP、FGD1、RGL2。以上DEGs 涉及NADH 脱氢酶活性、NAD+二磷酸酶活性、DNA 导向的DNA 聚合酶活性。这些酶直接参与细胞增殖、存活、分化等生理过程[20]。此外,NADH 脱氢酶还参与诱导肿瘤的发生和迁移[21-22]并促进癌细胞生长[22]。由此推断,自然感染的OPA 肺脏肿瘤组织DEGs 主要富集于肿瘤发生、癌细胞侵袭和转移等生物学过程中[17-22]。
目前,尚无利用RNA-seq 分析自然感染的OPA肺组织致病分子机制的研究报道。本研究首次利用生物信息学分析手段筛选自然感染OPA 肿瘤组织的DEGs,针对DEGs 进行GO 功能注释分析富集相关生物学功能模块。本实验鉴定出2 143 个差异转录本与Karagianni 等针对人工感染JSRV 66 d~85 d 的羔羊经转录组分析鉴定出1 971 个差异转录本[8]基本一致,均为参与基因组稳定性(BRCA1、ARID1A、SET⁃BP1 等)、肿瘤发生(ERBB2、ASXL1、TAX1BP1 等)和致癌信号转导(FGD1、TGFβ3、FGF11 等)的基因[14-15]。本研究鉴定的差异转录本数量大于Karagianni 的试验,多出的转录本主要与肿瘤生成(SLC38A7、NDUFS1、NUDT12 等)及能量代谢(POLH、UBE2N、UPRT 等)相关[15-19]。进一步说明临床症状明显的自然感染OPA 病例肺组织肿瘤化程度相较于短时期人工感染的羔羊更为严重,表现出更多转录层面的差异表达。此外在以往的研究报道中,关于绵羊全基因组分析的研究有很多[9-11],但针对绵羊单一组织的转录组报道大多集中在卵巢[9],骨骼肌[10],脂肪[11]等组织,并且以往针对肿瘤的RNA-seq 研究对象主要集中在人[18],狗[23]和小鼠[13]。本研究首次报道了患OPA 绵羊肺脏组织转录组的基础数据,为今后绵羊肺脏转录组研究提供基本参考。
综上所述,本实验采用转录组测序技术对OPA患羊肺脏肿瘤组织的基因变化进行了研究,揭示了自然感染OPA 羊肺肿瘤组织中的基因表达变化,能够引起一系列复杂生物学过程的改变,这些数据为进一步研究OPA 致癌机理提供了基础试验数据。

