陆地棉REM 基因家族全基因组鉴定及表达分析
石荣康,张冬梅,孙正文,刘正文,解美霞,张艳,马峙英,王省芬
(华北作物改良与调控国家重点实验室/ 河北省棉花产业协同创新中心/ 河北省作物种质资源重点实验室/河北农业大学农学院,河北 保定071001)
棉花是重要的纤维作物和油料作物,在全世界广泛种植。 陆地棉是我国种植最广泛、产量最高的栽培种,其基因组数据也在不断完善[1]。 B3超家族是植物中特有的一个基因家族,在植物的生长发育以及应对环境胁迫中发挥着重要的调控作用,该家族编码一类含有B3 功能域(与DNA 结合的高度保守结构域)的转录因子[2]。 B3功能域的最初命名源于玉米中发现的VIVIPAROUS1(VP1)蛋白的第三个基本结构域,包含90~120 个氨基酸[3]。 按照基因结构和功能特征,B3 超家族进一步分为4 个亚家族,包括LAV(LEAFY COTYLEDON2[LEC2]-ABSCISIC ACID INSENSITIVE3[ABI3]-VAL)、ARF(AUXIN RESPONSE FACTOR)、RAV (RELATED TO ABI3 AND VP1)和REM(REPRODUCTIVE MERISTEM)[4-6]。
目前, 植物中关于LAV、ARF 2 个亚家族的研究报道较多, 拟南芥LAV 亚家族分为LEC2-ABI3 和VAL 两 组,LEC2 和FUSCA3(FUS3)促进胚胎发育[7-8],ABI3 在种子发育过程中调控脱落酸响应基因[9],这3 种基因都在种子中大量表达, 在种子发育过程中发挥着重要作用。 拟南芥的ARF 亚家族含有生长素响应元件,参与生长素介导的各种生理过程,AtARF1 和AtARF2 突变导致开花延迟[10],AtARF6 和AtARF8的双缺失突变体会导致花器官成熟滞后以及繁殖力降低[11],AtARF5 和AtARF7 促进极性生长以及细胞扩增[12-13]。 而有关RAV、REM 2 个亚家族的功能研究报道较少, 拟南芥中RAV 亚家族成员有13 个, 过量表达AtRAV1 的拟南芥表现为侧根少、莲座叶少以及开花延迟[14],在烟草中表达大豆的RAV1 基因也产生了相似的表型[15],表明AtRAV1 在植物生长和开花中作为负调控因子起作用。拟南芥中REM 亚家族成员只含有B3 功能域, 但是每个成员在结构域的个数上存在差异,REM 亚家族中最先确定功能的基因是AtVRN1,该基因以非特异性序列的方式结合到DNA 上,并对维持拟南芥春化反应发挥作用[16]。 对于其他AtREM 基因, 目前只有一小部分有关基因表达特性的研究报道, 比如AtREM22 在雄蕊发育中发挥作用[17],AtREM01 和AtREM23 在花芽的分化期发挥作用[18-19],REM 家族的AtVDD 可以与MADS-box 转录因子结合,在植物胚珠发育中发挥作用[20-21]。 椰菜BoREM1 在生殖分生组织表达,在花芽分化期发挥作用[22]。 同时对番茄、毛竹和玉米等作物REM 基因家族进行了生物信息学分析, 为研究REM 家族在作物中的功能提供了理论基础[23-25]。
目前, 有关棉花REM 家族的研究还未见报道。 为深入了解REM 家族基因在棉花生长发育和逆境中的作用, 本研究基于陆地棉TM-1 基因组测序数据,运用生物信息学手段结合公共数据库中的转录组数据[26-27],对陆地棉基因组中的REM 家族成员进行全面系统的鉴定, 并对REM家族基因在基因组中的分布、REM 蛋白的理化性质、亚细胞定位、进化以及表达特性进行分析,利用qRT-PCR (Quantitative real-time ploymerase chain reaction, 实时荧光定量聚合酶链式反应)对部分基因进行表达验证, 为后续研究REM 家族基因的功能提供理论依据。
1 材料与方法
1.1 陆地棉REM 基因家族成员鉴定及理化性质分析
以基于三代测序组装的陆地棉TM-1 基因组数据作为参考[28],从CottonFGD (http://www.cottonfgd.org/)下载基因序列和氨基酸序列,利用REM家族特有的B3 结构域(Pfam accession 02362)搜索陆地棉REM 氨基酸序列, 将得到的候选基因在NCBI-CDD(https://www.ncbi.nlm.nih.gov/Structure/bwrpsb/bwrpsb.cgi)数据库中进一步确认。 通过 ExPASy-ProtParam tool (https://web.expasy.org/protparam/)在线软件,分析REM 家族成员编码蛋白的氨基酸数量、相对分子质量、理论等电点及不稳定指数等[29]。
1.2 陆地棉REM 基因家族的染色体定位和亚细胞定位
从CottonFGD 获取陆地棉REM 基因家族成员的染色体位置信息[30],利用Mapchart 2.32 绘制REM 家族基因的染色体定位图。使用CELLO v.2.5 (http://cello.life.nctu.edu.tw/) 在 线 网 站 对REM 编码蛋白进行亚细胞定位预测[32]。
1.3 陆地棉REM 基因家族的保守基序分析和基因结构分析
利用The MEME Suite(http://meme-suite.org/)分析REM 家族氨基酸序列的保守基序, 基序数量设置为10, 其他参数为默认值。 通过软件TBtoolsv0.66831 绘制REM 家族的基因结构图[31]。
1.4 REM 基因家族的系统进化分析
从TAIR(https://www.arabidopsis.org/)网 站下载拟南芥REM 家族基因序列。 利用软件MEGA 7.0 的Muscle 功能对陆地棉与拟南芥REM 家族基因编码的氨基酸序列进行比对,采用相邻连接法(Neighbor-joining,NJ)构建系统进化树,其中Bootstrap 值设定为1 000[33]。
1.5 陆地棉REM 基因的上游顺式作用元件分析
从CottonFGD 提取陆地棉REM 家族基因上游2 000 bp DNA 序列,用PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)在线网站预测可能存在的顺式作用元件。
1.6 陆地棉REM 基因家族的共线性分析
使用MCScanX[34]软件检测陆地棉基因复制事件,Tbtools 软件提取并展示REM 家族成员复制事件。
1.7 REM 基因家族在棉花不同组织及逆境胁迫下的表达分析
从NCBI sequence read archive(SRA)数据库下载陆地棉REM 基因家族成员的根、茎、叶、雌蕊、雄蕊、花萼、花瓣和花托等8 个组织和胚珠、纤维不同发育时期的转录组数据(PRJNA 248163),以及4 种逆境胁迫(冷、热、PEG 和盐胁迫)的转录组数据。 棉花材料处理和数据获取过程详见Zhang 等[35]的研究报道。 以FPKM(Fragments per kilobase of transcript per million fragments mapped)>1 作为基因表达的筛选标准,对转录组数据进行log2(1+FPKM)标准化处理。 利用TBtools v0.66831 软件绘制REM 家族成员的表达热图[31]。
1.8 部分REM 家族基因在不同发育时期纤维中的qRT-PCR 分析
按照RNAprep Pure Plant Plus kit 试剂盒说明书提取陆地棉品种农大棉13 号开花后5、10、15、20 天(Days post anthesis,DPA)的纤维RNA。经1.5%琼脂糖凝胶电泳和Nano Drop 2000 分光光度计检测RNA 样品的质量和浓度后, 使用TransScriptOne-Step gDNA Removal and cDNA Synthesis SuperMix 反转录试剂盒合成cDNA。采用TransStartTop Green qPCR SuperMix (+Dye II)定量试剂盒进行qRT-PCR 分析,GhHis3 作为内参[36]。反应体系共20 μL,包括2×SuperMix 10 μL,ddH2O 7 μL,cDNA 1 μL, 正向引物1 μL 以及反向引物1 μL。反应程序为:50 ℃2 min,95 ℃预变性10 min,95 ℃变性15 s,60 ℃退火1 min,40 次循环。 引物信息详见表1。 每个样品进行3次重复试验, 采用2-ΔΔCt法计算基因的相对表达量[37]。 使用GraphPad Prism7 软 件 对 数 据 进 行Tukey 多重比较和绘图。

表1 本研究所用引物Table 1 Primers used in this study
2 结果与分析
2.1 陆地棉REM 基因家族成员全基因组鉴定
基于陆地棉TM-1 基因组测序数据, 从陆地棉基因组中共鉴定出79 个GhREM 基因, 至少都含有一个B3 功能域。 根据其在染色体上的位置信息,依次命名为GhREM01~GhREM79(图1)。 从基因在染色体上的分布来看,除了A 亚组的第9 染色体没有REM 家族成员外, 其他每条染色体上至少含有一个GhREM, 其中A 亚组第2 号和第3 号染色体上最多, 每条染色体均包含7 个GhREM。 此外,我们在陆地棉REM 家族成员中发现了9 对串联重复基因, 分布在A、D 亚组,推测这些基因可能在棉花发生多倍化事件时产生。

图1 陆地棉REM 基因的染色体定位Fig. 1 Chromosome location of Gossypium hirsutum REM genes
2.2 陆地棉REM 家族成员编码蛋白的理化性质和亚细胞定位分析
对REM 家族成员编码的蛋白质进行理化性质分析(表2),其编码的肽链由119~749 个氨基酸组成,相对分子质量介于13.81~83.90 kDa,平均41.04 kDa。理论等电点介于5.54~10.10,平均值为8.50, 多数偏碱性。 蛋白不稳定指数介于22.10~70.10, 平均值为42.28, 其中有30 个GhREM 成员为稳定蛋白(蛋白不稳定指数<40);其余的49 个基因为不稳定蛋白 (蛋白不稳定指数>40)。 亚细胞定位预测显示,REM 家族基因主要定位于细胞核、细胞质以及线粒体中,以细胞核中分布最广泛, 其中有58 个基因定位在细胞核中,25 个定位在细胞质中,18 个定位在线粒体,4 个定位在胞外,2 个定位在叶绿体。

表2 陆地棉REM 基因信息Table 2 The information of Gossypium hirsutum REM gene

表2 (续)Table 2 (Continued)

表2 (续)Table 2 (Continued)
2.3 陆地棉REM 家族成员的保守基序和基因结构分析
利用79 条陆地棉REM 家族基因的氨基酸序列构建系统进化树,并对家族成员的基因结构和保守基序进行可视化分析。 依据Motif 的种类与数量(图2A),内含子与外显子的数量和位置(图2B), 将陆地棉REM 家族分为6 个亚组,即Ⅰ、Ⅱ、Ⅲ、Ⅳ、Ⅴ和Ⅵ。 一些成员含有相同的基序(Motif 1、Motif 2、Motif 3、Motif 4、Motif 5、Motif 8和Motif 9), 所有GhREM 基因的外显子数量在1~10。 Ⅰ亚组中含有6 个外显子的基因占多数,Motif 的个数在8~13, 是6 个亚组中最多的;Ⅱ亚组含有4 个外显子的基因居多,Motif 8 只存在于第Ⅱ亚组;Ⅲ亚组外显子数目介于1~7,含有2~3 个Motifs;Ⅳ亚组的基因大多含有4 个外显子,Motif 9 只存在于第Ⅳ亚组; Ⅴ亚组含有1 个外显子的基因居多, 并且只含有1~2 个Motif;Ⅵ亚组包含4~6 个外显子,Motif 的个数在2~8。 所有REM 基因包含1~4 个B3 功能域,其中46 个成员包含1 个B3 功能域,17 个成员和12个成员分别包含2 个和3 个B3 功能域,4 个成员包含4 个B3 功能域。大多数B3 功能域位于肽链的N 端,C 端次之,中部最少。
2.4 陆地棉与拟南芥REM 基因家族的系统进化分析

图2 陆地棉REM 家族成员的保守基序和基因结构分析Fig. 2 Conserved motif and gene structure analyses of REM family members in G. hirsutum

图3 陆地棉REM 基因家族保守基序Fig. 3 Conserved motifs of REM gene family in G. hirsutum

图4 陆地棉与拟南芥REM 基因家族的系统进化分析Fig. 4 Phylogenetic analysis of REM genes from G. hirsutum and Arabidopsis thaliana
REM 基因家族是功能研究较少的基因家族,拟南芥中含有45 个REM 基因,按功能分为5 个亚组[5]。 将79 个GhREMs 与45 个AtREMs编码的氨基酸序列构建系统进化树(图4),所有的陆地棉REM 基因与拟南芥基因并没有因为物种差异而单独分成2 类, 陆地棉REM 基因也分为5 个亚组, 即A~E 亚组。 A 亚组包含13 个GhREMs 以及15 个AtREMs, 其中At4G31610、At4G31615 和At4G31620 在花分生组织和花发育的早期阶段中表达并发挥作用[18];B 亚组包含12 个GhREMs 和3 个AtREMs;C 亚组包含5 个GhREMs 和3 个AtREMs,其中At3G19184 响应花、叶分生组织的发育[5];D 亚组包含41 个陆地棉和 6 个拟南芥成员, 其中 At3G18990(AtVRN1)是拟南芥中发现最早、功能最先确定的AtREM 基因[16],维持拟南芥对春化作用的响应;E 亚组包含8 个GhREMs 和18 个AtREMs,其中At3G17010 为雄蕊发育的标志基因[17],At2G16210、At2G35310 和At5G09780 在 花 芽 的分化期表达并发挥作用[18]。 推测与拟南芥REM家族成员亲缘关系较近的陆地棉REMs, 可能具有相似的生物学功能。
2.5 陆地棉REM 基因家族共线性分析
为了进一步探索陆地棉REM 家族基因的进化过程, 本研究分析了REM 家族不同基因间的共线性关系,共发现44 对REM 基因存在共线性关系(图5),除了A 亚组和D 亚组对应染色体上的REM 基因之间存在共线性关系外,A 亚组和D 亚组内分别有11 对和9 对REM 基因也存在共线性关系, 而且REM 基因家族存在单个基因对应多个基因的情况,说明该家族在进化过程中发生了染色体片段复制事件。
2.6 陆地棉REM 家族启动子顺式作用元件分析

图5 陆地棉REM 基因共线性分析Fig. 5 Collinearity analysis of REM genes in upland cotton
通过分析陆地棉REM 基因家族上游2 000 bp的序列,发现数量不等的顺式作用元件(图6),包括光信号响应元件、激素响应元件、环境胁迫响应元件等。首先,GhREM 家族每个成员都含有大量 的 光 信 号 响 应 元 件(G-box、GT1-motif、TCT-motif 等), 说明REM 家族成员的启动子具有光诱导的特性, 并参与到光周期的调控途径。其次,大多数成员都含有各种激素响应元件以及环境胁迫响应元件, 例如,ABA 响应元件(ABRE)、GA 响 应 元 件(GARE-motif、P-box、TATC-box)、IAA 响应元件(AuxRR-core、TGAelement)、MeJA 响 应 元 件 (CGTCA-motif、TGACG-motif)、ET 响应元件(ERE)、SA 响应元件(TCA-element)、干旱响应元件(MBS)、低温响应元件(LTR)、防御与胁迫响应元件(Defense and stress)及抗氧化响应元件(ARE)等,推测陆地棉REM 家族基因可能参与响应激素调控及抵御干旱、低温等胁迫过程。 另外,还发现部分基因含有许多响应调控昼夜节律(Circadian)、分生组织表达(CAT-box)、胚乳表达(GCN4-motif)、种 子萌发(RY-element)等生长发育相关元件。 在6 个响应激素的调控元件中, 每个GhREM 至少含有1个激素调控元件,其中52 个基因含有ABA 调控元件,67 个基因含有ET 调控元件,40 个含有MeJA 调控元件,如GhREM08 含有11 个响应乙烯调控元件,GhREM10 含有12 个响应MeJA 调控元件,GhREM50 含有6 个ABA、6 个MeJA 调控元件;此外,63 个基因还含有响应干旱、低温等逆境胁迫的调控元件。 以上结果表明, 陆地棉REM 家族成员通过不同的信号通路参与棉花生长发育以及环境胁迫。

图6 GhREM 顺式作用元件分析Fig. 6 Cis-acting element analysis of GhREM genes
2.7 陆地棉REM 家族的组织表达特性分析
根据陆地棉TM-1 的转录组数据, 分析了GhREM 家族成员在棉花根、茎、叶、雌蕊、雄蕊、花萼、花瓣和花托等8 个组织以及胚珠和纤维不同发育时期中的表达模式。 以FPKM>1 作为基因表达的筛选标准, 发现其中25 个基因在所有组织中的FPKM 数值均小于1,可以认为它们在组织中不表达或者表达量极少; 剩下的54 个基因具有组织表达特异性,并可分为5 种表达模式(图7)。 模式Ⅰ包括12 个GhREM 成员,它们在各种组织中均表达, 且胚珠及纤维中表达量高,GhREM02、GhREM24、GhREM41、GhREM60 和GhREM76 在5 DPA 胚珠中的表达量较其他组织更高,GhREM13、GhREM17 和GhREM37 在5 DPA 和10 DPA 纤维中表达量更高。 模式Ⅱ包括16 个成员, 这些基因在不同发育时期的胚珠和纤维中的表达量较模式Ⅰ低,部分成员在一些组织中不表达。 模式Ⅲ仅包括3 个成员:GhREM74、GhREM06 和GhREM35,主要在花瓣等花器官中表达量较高,在其他组织中表达量较低或不表达。 模式Ⅳ包括12 个成员,在各组织中的表达量都较低。 模式Ⅴ包括11 个成员,在大部分组织中的表达量都较低,但在1 DPA 或5 DPA的胚珠中均有较高表达, 且GhREM08、GhREM20 和GhREM57 在雌蕊中表达量较高,GhREM27、GhREM62、GhREM63 在5 DPA 或10 DPA 的纤维中表达量较高。
2.8 陆地棉REM 家族响应逆境胁迫的表达分析
进一步分析所有GhREMs 在冷、热、盐和干旱4 种逆境胁迫下的表达规律, 发现50 个基因在所有逆境胁迫下均不表达, 其余29 个基因在不同逆境条件下表现出不同的表达规律。
冷胁迫处理后(图8A),与相应时间点的对照比,28 个GhREMs 的表达发生了变化。 处理后1 h,13 个GhREMs(GhREM06、GhREM21 等)表达 下 调,GhREM43、GhREM46 和GhREM74 表达上调;处理后3 h,GhREM13 和GhREM41 表达上 调;GhREM18、GhREM21 等8 个 基 因 在 处 理后6 h 表达上调。 处理后4 个时间点表达量均下调的有10 个基因,包括GhREM02、GhREM24 等。
热胁迫处理后(图8B),28 个GhREMs 的表达发生了变化。 处理后1 h,GhREM18、GhREM19、GhREM41 和GhREM43 上 调 表 达,13 个 基 因下 调 表 达;GhREM06、GhREM18、GhREM24、GhREM31、GhREM43 和GhREM60 在处理后6 h和12 h 表达量明显上调;GhREM18、GhREM24、GhREM41 和GhREM43 在4 个时间点均上调表达。
盐胁迫处理后(图8C),27 个基因的表达发生 了 改 变, 其 中 GhREM32、GhREM41 和GhREM46 在盐处理后1 h 表达上调,8 个基因表达 下 调;GhREM31、GhREM43 和GhREM58 在处理后6 h 表达上调,GhREM24 和GhREM50在处理后12 h 表达上调;GhREM41、GhREM60和GhREM76 在4 个时间点表达量均上调。
聚乙二醇(Polyethylene glycol,PEG)模拟干旱胁迫处理后(图8D),有27 个基因的表达发生变化。处理后1 h,7 个GhREMs 的表达上调,包括GhREM02、GhREM17 等,GhREM21、GhREM36、GhREM68 和GhREM77 下调表达; 处理后6 h,GhREM24、GhREM31、GhREM43、GhREM44 和GhREM58 上调表达量;GhREM17 在4 个时间点的表达均表现为上调。
以上分析结果显示,REM 家族成员在棉花应对逆境胁迫时可能发挥着重要作用。
2.9 部分REM 家族基因在不同发育时期纤维中的qRT-PCR 分析
从陆地棉REM 家族中选择有代表性的6 个基因, 利用不同发育时期的纤维RNA 进行qRT-PCR 验证,结果显示,GhREM03、GhREM36和GhREM78 在10 DPA 的棉纤维中显著高表达, 之后表达量明显下降;GhREM13 和GhREM27 在5 DPA 和10 DPA 的纤维中表达量最高,之后表达量显著下降;GhREM50 在纤维发育到20 DPA 时表达量最高(图9),这与转录组数据反映的基因表达模式一致,也预示着这些基因在棉纤维发育过程中可能具有重要作用。
3 讨论
本研究运用生物信息学的手段首次从陆地棉TM-1 基因组中鉴定到79 个GhREM 成员,不均匀地分布在除了A 亚组第9 号染色体外的其他染色体上。 REM 家族成员含有的氨基酸残基的数量介于119~749, 氨基酸数量的较大差别,与其他作物如毛竹、 番茄和拟南芥报道的REM家族成员一致[6,23-24]。 B3 超家族中的LAV、ARF和RAV 亚家族除了含有特有的B3 功能域外,还包括AP2 结构域、 生长素响应结构域以及CW锌指蛋白结构域。然而REM 亚家族只包含B3 功能域[2,5],通过对陆地棉REM 家族基因进行分析,成员都含有特有的B3 功能域, 与已报道的描述一致。 此外,部分成员含有一些未知功能域(Domains of unknown function,DUF),这些结构域可能在棉花生长发育中发挥特定的作用。 对该家族成员进行亚细胞定位预测,58 个基因定位在细胞核中,部分基因定位在其他部位,与其转录因子家族的特征相符[2]。 根据79 个GhREM 成员基因结构及保守基序的相似性,将所有基因分成6 个亚组,部分成员有相同的结构,但整体差异较大,某些保守基序只存在于其中某一亚组。 例如,Motif 8 只存在于第Ⅱ亚组,Motif 9 只存在于第Ⅳ亚组。 基因片段复制往往促进了基因家族的扩大[38],通过对陆地棉REM 家族基因进行共线性分析,发现该家族也同样发生了一些染色体片段复制事件,一共包含44 对共线性关系,存在着单个基因对应多个基因的情况,说明该家族在进化中发生了扩张。
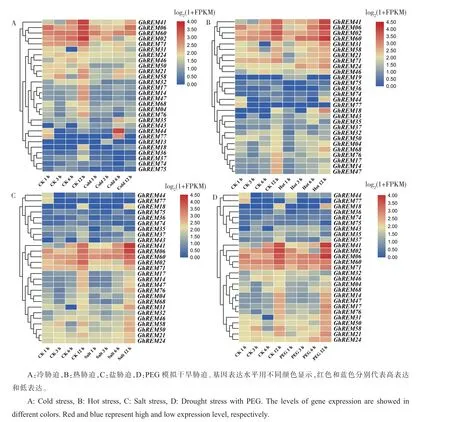
图8 陆地棉REMs 在4 个逆境胁迫下的表达Fig. 8 Profile of GhREMs expression under four stresses

图9 陆地棉REM 基因纤维发育时期的表达分析Fig. 9 The expression analysis of REM genes during fiber development in G. hirsutum
通过对陆地棉与拟南芥REM 家族基因进行系统进化分析,按照拟南芥REM 基因的功能,将棉花REM 家族基因分成A~E 5 个小家族,D 亚组GhREM 成员最多,其中AtVRN1 在春化反应中发挥作用,过量表达该基因可以引起休眠的抑制,甚至致死[16]。 A 和E 亚组中的AtREM23 和AtREM01 在花芽分化中发挥作用[18-19],AtREM22是雄蕊发育的标志基因[17],和它们属于同一组的GhREMs 可能发挥着同样的作用。 本研究发现,在79 个REM 基因中, 有25 个基因在不同组织中低表达或不表达,54 个基因在不同组织中的表达呈现出差异,大部分成员在胚珠和纤维中表达量较高, 而GhREM02、GhREM60、GhREM76 等在所有组织中均表达,说明这些基因可能在棉花整个生长发育中都发挥作用。 植物在生长发育过程中需要各种激素的调控,也常常会遇到逆境胁迫的不利因素, 本研究通过分析REM 家族基因上游2 000 bp 的序列, 发现了很多响应激素、逆境胁迫的顺式调控元件, 说明REM 家族成员可能通过参与不同的信号通路在植物生长发育和应对逆境胁迫中发挥作用。 本研究发现GhREM36、GhREM50 和GhREM78 上游含有响应IAA、ET 和GA 等参与棉花纤维发育的激素调控元件, 而转录组和qRT-PCR 的分析显示它们在纤维发育不同时期的表达量不同,预示着这些基因可能在棉纤维发育中行使着重要的功能。此外, 对REM 家族在逆境胁迫下的表达分析发现, 部分基因如GhREM41 等在响应棉花热、盐胁迫,GhREM43 等在响应干旱胁迫时表达上调,说明这些基因能快速地参与抗逆反应。 关于上述基因在棉花中的功能和作用机制还有待今后进一步研究。
4 结论
本研究从陆地棉基因组中鉴定出79 个REM 基因家族成员, 明确了基因在染色体上的分布特征、基因结构、理化性质以及系统进化特征,在转录水平上初步揭示了其在生长发育和抗逆中的功能, 为后续REM 家族基因的功能研究提供了理论依据。

