基于小麦SNP芯片对簇毛麦6V#2和6V#4染色体及其与小麦6A、6D染色体的多态性分析
许志英,王佰翠,马晓兰,贾子苗,叶兴国,林志珊,胡汉桥
基于小麦SNP芯片对簇毛麦6V#2和6V#4染色体及其与小麦6A、6D染色体的多态性分析
许志英1,2,王佰翠2,马晓兰2,贾子苗2,叶兴国2,林志珊2,胡汉桥1
1广东海洋大学滨海农业学院,广东湛江 524088;2中国农业科学院作物科学研究所,北京 100081
【】利用大数据比较2条不同的簇毛麦6V(#2和#4)染色体及其与小麦6A、6D染色体间DNA水平上的差异,为小麦-簇毛麦靶向易位的精准设计育种提供依据。以6V#4(6D)异代换系RW15为父本和6V#2(6A)异代换系南87-88为母本进行杂交,获得F2分离群体,利用6V#4S/6V#2S/6AS/6DS/6VL特异分子标记检测F2植株,筛选新类型的代换系,并用分子标记结合基因组原位杂交(genomichybridization,GISH)对新类型代换系进行确认,再利用小麦55K芯片中的6A、6D探针,对新代换系及其双亲南87-88和RW15进行分析;结合660K芯片6A、6D探针对2份簇毛麦的SNP分析结果,筛选6V特异SNP。GISH分析表明,19EL124和19EL134的体细胞染色体数2n=42,分别携带2条完整的外源染色体;分子标记鉴定结果表明,19EL124含有6V#4S/6DS特异标记带,缺失了6V#2S/6AS特异带,而19EL134含有6V#2S/6AS特异标记带,缺失了6V#4S/6DS特异带;19EL124和19EL134都含有6VL的特异带,证明19EL124为6V#4(6A)异代换系,19EL134为6V#2(6D)异代换系。55K芯片检测结果表明,异代换系中关键染色体探针的检测效率显著低于其他染色体,且对同类型异代换不同系的检测效率也有所不同。1 177个6A探针中,63.21%不能对6A代换系南87-88分型,68.90%不能对6A异代换系19EL124分型,22.51%检测到6V#2和6V#4间的多态性,其中88个只能检测到6V#4染色体,而155个只能检测到6V#2染色体;479个6D探针中,49.48%不能对6D异代换系RW15分型,53.44%不能对6D代换系19EL134分型,16.70%检测到6V#2和6V#4间的多态性,其中23个只能检测到6V#2染色体,42个只能检测到6V#4染色体。整合55K和660K芯片的共有探针,分别从395个6A、231个6D探针筛选获得簇毛麦6V特异的SNP标记22个和15个,其中3个可在6V#2和6V#4染色体间显示多态性。小麦染色体的缺失与外源染色体的替换,使相应染色体探针的检测效率大幅降低,NA分型比例极大增加,且多数NA分型在2条不同的外源染色体间显示多态;相同探针对2条外源染色体的检测效率不同,小麦6A探针可以更好地检测6V#2,而小麦6D探针可更好检测6V#4;在簇毛麦与异代换系6V染色体的一致性分型中,筛选获得簇毛麦6V特异的SNP标记37个。
小麦;簇毛麦;异代换系;小麦SNP芯片;多态性
0 引言
【研究意义】簇毛麦(,2n= 14,VV)是小麦的二倍体近缘种属,耐寒、耐旱,抗全蚀病、眼斑病、黄花叶病、白粉病等多种小麦病害,且籽粒蛋白质含量高,是小麦品种改良的重要种质资源[1-3]。由于簇毛麦与小麦染色体之间不能自由交换,给育种带来了极大的困难。基于大数据,从DNA水平上分析不同簇毛麦的同源染色体及其与小麦部分同源染色体间的多态性,为靶向易位设计育种提供理论依据。【前人研究进展】据报道,已收集超过300份的簇毛麦种质资源,一些不同来源的簇毛麦染色体导入小麦基因组中已育成各种异附加系、代换系和易位系[4-6],其中,簇毛麦6V#2和6V#4染色体对白粉菌表现明显的抗性,具有良好的应用潜力,因此得到了较为深入的研究。在过去的研究中,基于表型和部分基因序列的比较均表明6V#2和6V#4染色体存在差异。如携带不同来源6V染色体的代换系和易位系对小麦瘿螨及其传播的病毒的抗性表现不同[7];基于表达序列的引物设计与PCR扩增片段的比较,表明6V#2和6V#4上某些基因序列存在差异,并开发了可用于区分6V#2和6V#4的特异PCR标记,其中,刘畅等[8]开发的P461-5a在6V#2和6V#4间存在扩增片段长度的多态性,而P259-1只能检测6V#4;Li等[9]基于转录组测序序列开发了25个6V特异性标记,3个在6V#2和6V#4间存在多态性;BIE等[10]开发了可同时鉴定6AS/6DS/6V#2S/6V#4S的多态性标记MBH1;陈竟男等[11]利用238对EST-SSR引物对小麦中国春与簇毛麦Dv#4和簇毛麦Dv#2的基因组DNA进行扩增,结果显示,47对在簇毛麦#2和簇毛麦#4之间存在多态性。最近,MA等[12]观察到在非易位的完整染色体状态下,小麦背景中的簇毛麦6V#2和6V#4染色体中期I不配对的频率达20.24%,且在配对的二价体中,形成环状二价体的频率低于棒状二价体,说明染色体6V#2和6V#4在染色体水平上也存在较大差异。但在整条染色体的DNA水平上的差异目前尚缺乏相关的信息,难以确定簇毛麦6V#2和6V#4染色体之间及其与小麦部分同源染色体之间的多态性。【本研究切入点】随着单核苷酸多态性(single nucleotide polymorphism,SNP)分析检测技术的迅猛发展,基于Illumina技术平台的9K和90K高通量SNP芯片以及最新开发的660K、55K和820K芯片已被应用于目标基因定位、高密度遗传图谱构建和群体结构分类等小麦诸多研究领域[13-15]。Raz等[16]利用90K iSelect SNP对硬粒小麦和野生二粒小麦杂交构建的140个重组自交系(recombinant inbred lines,RILs)进行全基因组扫描,构建了全长为2 110 cM,包含14 088个SNP位点的遗传连锁图谱,其中,相邻标记之间的平均距离为0.92 cM。Stuart等[17]对384HT系列硬粒小麦群体进行研究,构建了包含1 345个SNP标记的复合遗传图谱。Zhou等[18]利用660K芯片对2个异花授粉的二倍体冰草Z1842和Z2098种间杂交产生的F1代以及35个小麦-冰草附加系/代换系进行基因分型,利用分布在跨839.7 cM 7个连锁群中的913个SNP标记构建遗传连锁图谱,确定了P基因组和小麦基因组之间的同源关系。曹延杰等[19]利用Illumina 90k iSelect SNP标记技术对96个小麦品种进行全基因组扫描,表明不同品种间亲缘关系较近,在育种中迫切需要引入新的种质资源以拓宽遗传背景。目前,利用高通量SNP芯片在DNA水平上对不同来源簇毛麦6V染色体间及其与小麦6A、6D染色体间的多态性分析鲜见报道。【拟解决的关键问题】不同来源的簇毛麦同源染色体需要在相同代换类型的条件下,才可能利用共同的小麦探针对其进行相互比较,因此,本研究通过分子标记和基因组原位杂交技术,对2个不同的异代换系南87-88和RW15杂交的F2群体筛选出的6V#4(6A)异代换系19EL124和6V#2(6D)异代换系19EL134在F3代进行确认,进一步利用小麦55K芯片中的6A、6D探针对异代换系与相同代换类型的双亲分别进行多态性分析,同时整合660K芯片上第6同源群探针对2份簇毛麦的分析结果,筛选6V染色体特异的标记,拟为靶向易位的精准设计育种提供理论依据。
1 材料与方法
1.1 试验材料
源于英国剑桥的簇毛麦Dv#2、6V#2(6A)异代换系南87-88(2n=42=21II)由南京农业大学陈佩度教授惠赠。引自俄罗斯的簇毛麦No.1026、6V#4(6D)异代换系RW15(2n=42=21II)、6V#4S·6DL易位系Pm97033和6V#2S·6AL易位系10SR3109以及普通小麦品种宛7107均由中国农业科学院作物科学研究所陈孝研究员提供。19EL124和19EL134来自杂交组合南87-88×RW15 F3的2个株系,系谱号分别为18GL101-2-5和18GL101-2-29。
1.2 样本DNA的提取
用植物材料新鲜叶片提取gDNA,方法参照DNA提取试剂盒(北京康为世纪生物科技有限公司)提供的步骤进行,并最终稀释成100 ng·μL-1,-20℃保存备用。
1.3 PCR扩增反应
PCR反应体系见表1,反应条件为95℃ 5 min;95℃ 30 s,60℃ 30 s,72℃ 1 min,35个循环;72℃ 8 min,4℃保存。扩增产物经1.5%琼脂糖凝胶电泳,在紫外成像系统下扫描拍照。所用分子标记相关信息列于表2。
1.4 原位杂交
GISH参照WEI等[21]描述的方法,以簇毛麦gDNA作为探针,高温煮沸打断的中国春gDNA作为封阻,用地高辛试剂盒标记探针(Roche,德国),荧光素异硫氰酸酯(fluorescein isothiocyanate,FITC)地高辛试剂盒(Roche,德国)检测杂交信号,在荧光显微镜40×下(BX51,OLYMPUS,日本)观察拍照。
1.5 芯片分析
取4份异代换系(RW15、南87-88、19EL124、19EL134)和2份簇毛麦(CMM和No.1026)的叶片提取DNA,委托中玉金标记(北京)生物技术股份有限公司分别进行55K和660K芯片分析:设阈值DQC≥0.82,异代换系CR≥94,簇毛麦CR≥60,用Affymetrix(Thermo Fisher)AxiomAnalysisSuite软件的apt- genotype-axiom、ps-metrics和ps-classification模块进行基因分型分析,对标记类型为otv的标记进行otv- caller分析后,得到样品原始的数字型基因型;利用软件的apt-format-result模块得到原始的基因型数据。本次分析所用的参考基因组为:IWGSC_RefSeq_v1.0(https://wheat-urgi.versailles.inra.fr/Seq-Repository/Assemblies)。

表1 PCR 扩增反应体系
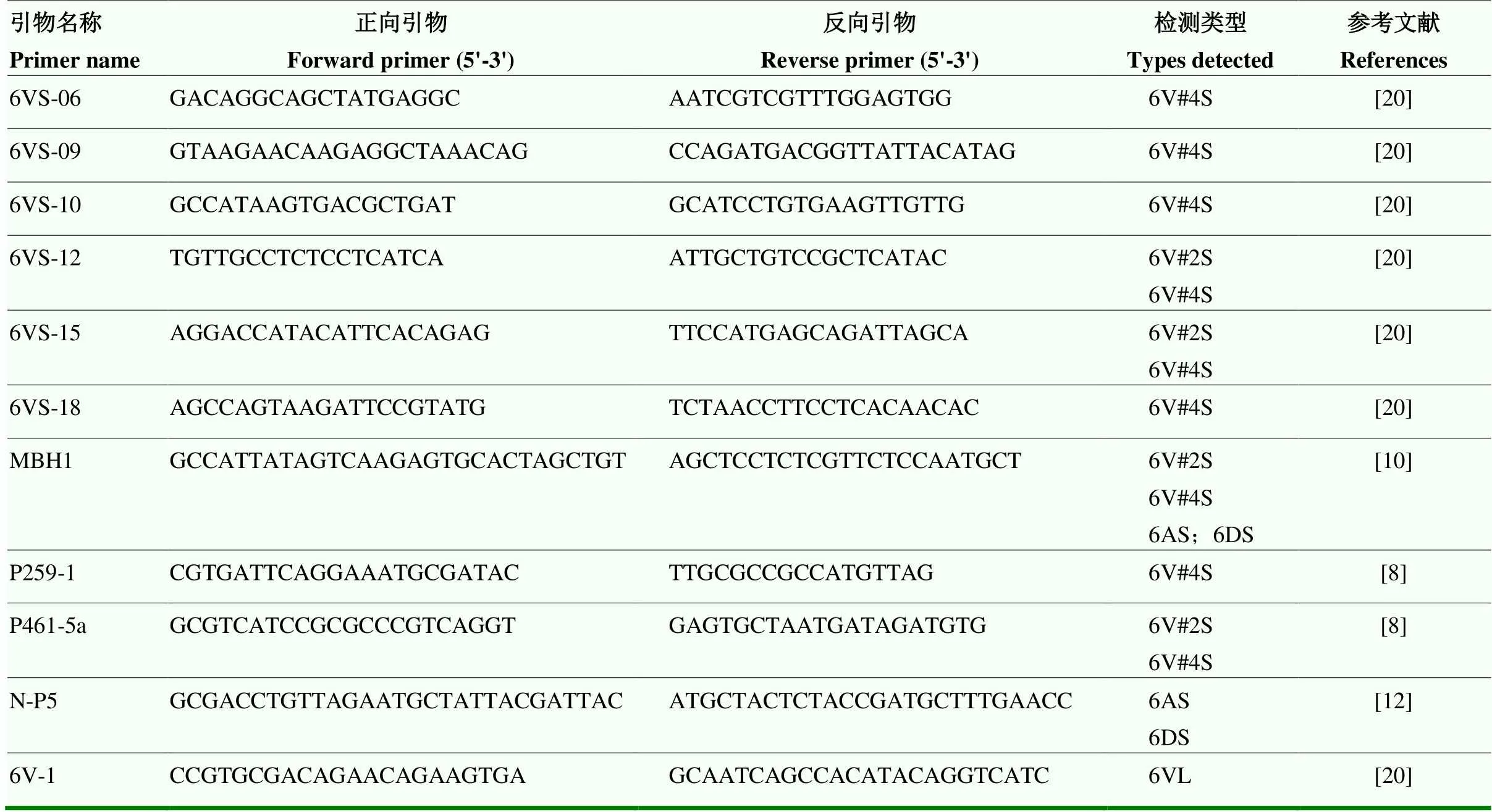
表2 PCR分子标记引物序列信息
2 结果
2.1 19EL124和19EL134的细胞学鉴定
以簇毛麦gDNA为探针,对2个亲本及衍生的19EL124、19EL134的根尖体细胞进行GISH鉴定(图1),发现探针的杂交信号显示清晰,亲本RW15、南87-88及19EL124、19EL134均为带有2条外源染色体,体细胞的染色体数目2n=42的正常整倍体。
2.2 19EL124和19EL134的分子标记鉴定
以2个亲本和宛7107、Pm97033、10SR3109为对照,用11个特异分子标记,包括5个外源染色体短臂6V#4S特异标记6VS-06、6VS-09、6VS-10、6VS-18和P259-1,3个6V#2S/6#4S共显性标记6VS-12、6VS-15和P461-5a,1个6V#2S/6#4S/6AS/6DS共显性标记MBH1,1个外源染色体长臂标记6V-1以及1个小麦6AS/6DS共显性标记N-P5对新筛选的材料19EL124和19EL134进行PCR检测(图2),19EL124与RW15有6V#4染色体特异带而缺失6V#2染色体特异带,19EL134和南87-88有6V#2染色体特异带而缺失6V#4染色体特异带;19EL124和南87-88有6D染色体特异带而缺失6A染色体特异带;19EL134和RW15有6A染色体特异带而缺失6D染色体特异带。综上所述,RW15为6V#4(6D)异代换系;南87-88为6V#2(6A)异代换系;19EL124为6V#4(6A)代换系;19EL134为6V#2(6D)代换系。
2.3 6A、6D探针对双亲南87-88和RW15基因分型的差异
1 177个高质量的6A探针用于检测分析。如图3A-a所示,探针的物理位置为1.74—617.99 Mb,涵盖小麦6A染色体约616.25 Mb的物理区域,探针平均相距约523.58 kb;1 010个探针在南87-88和RW15间显示不同的分型结果,占6A总探针85.81%。其中包括碱基差异(图3A-b)及位点缺失差异(图3A-c和图3A-d),位点缺失差异是指特定探针对一方不能进行基因分型(not available,NA),而对另一方可以分型(如6V#4NA6V#2+表示探针不能对6V#4分型而可对6V#2分型)。从图3A-c和图3A-d可知,较多的6A探针可在含有6A染色体的6V#4(6D)异代换系RW15中正常分型,而相应的探针大多对不含6A染色体的6V#2(6A)异代换系南87-88不能分型。
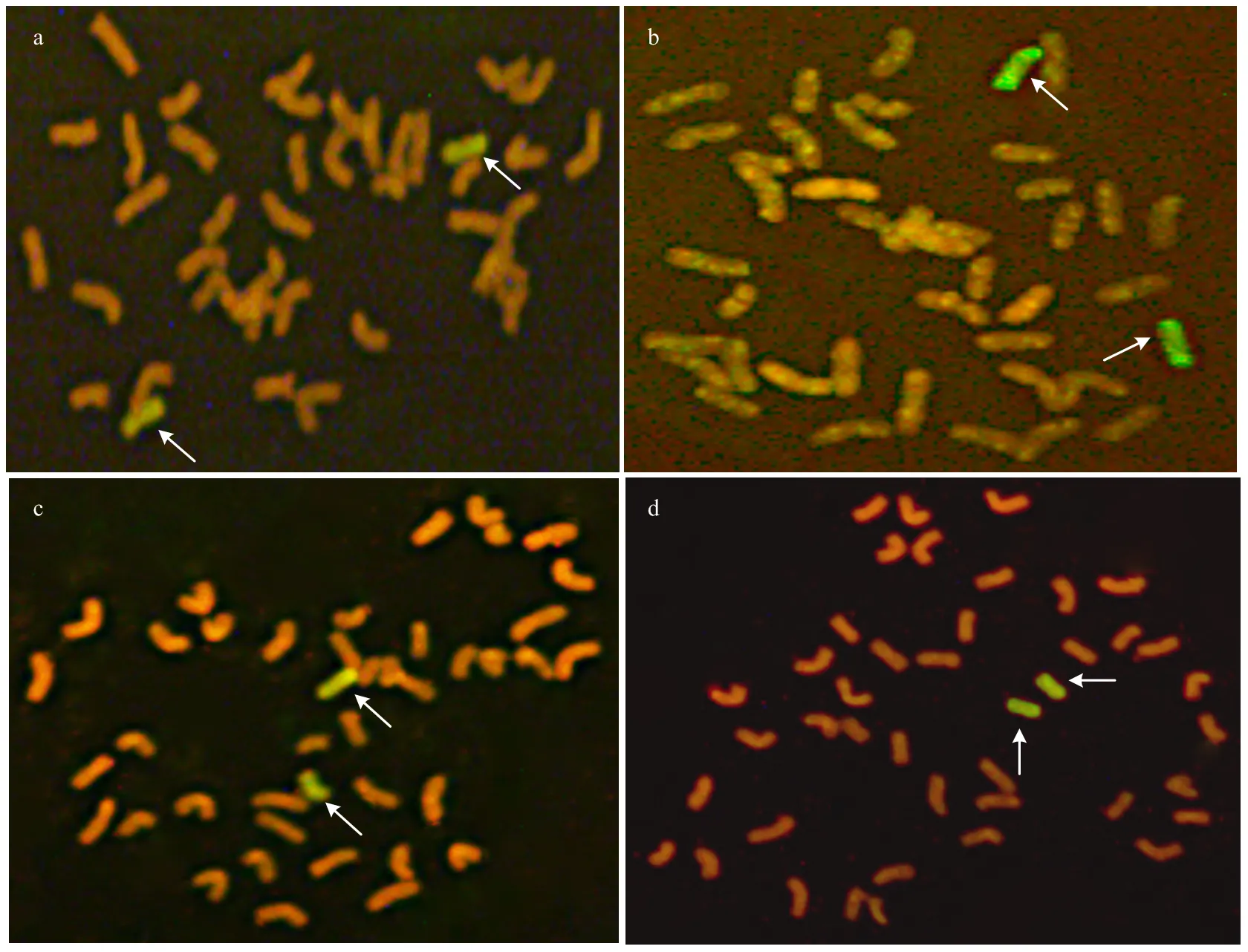
红色表示用碘化丙啶(PI)复染的小麦染色体,箭头所示黄绿色表示外源染色体。a—d:RW15、南87-88、19EL124和19EL134,2n=42
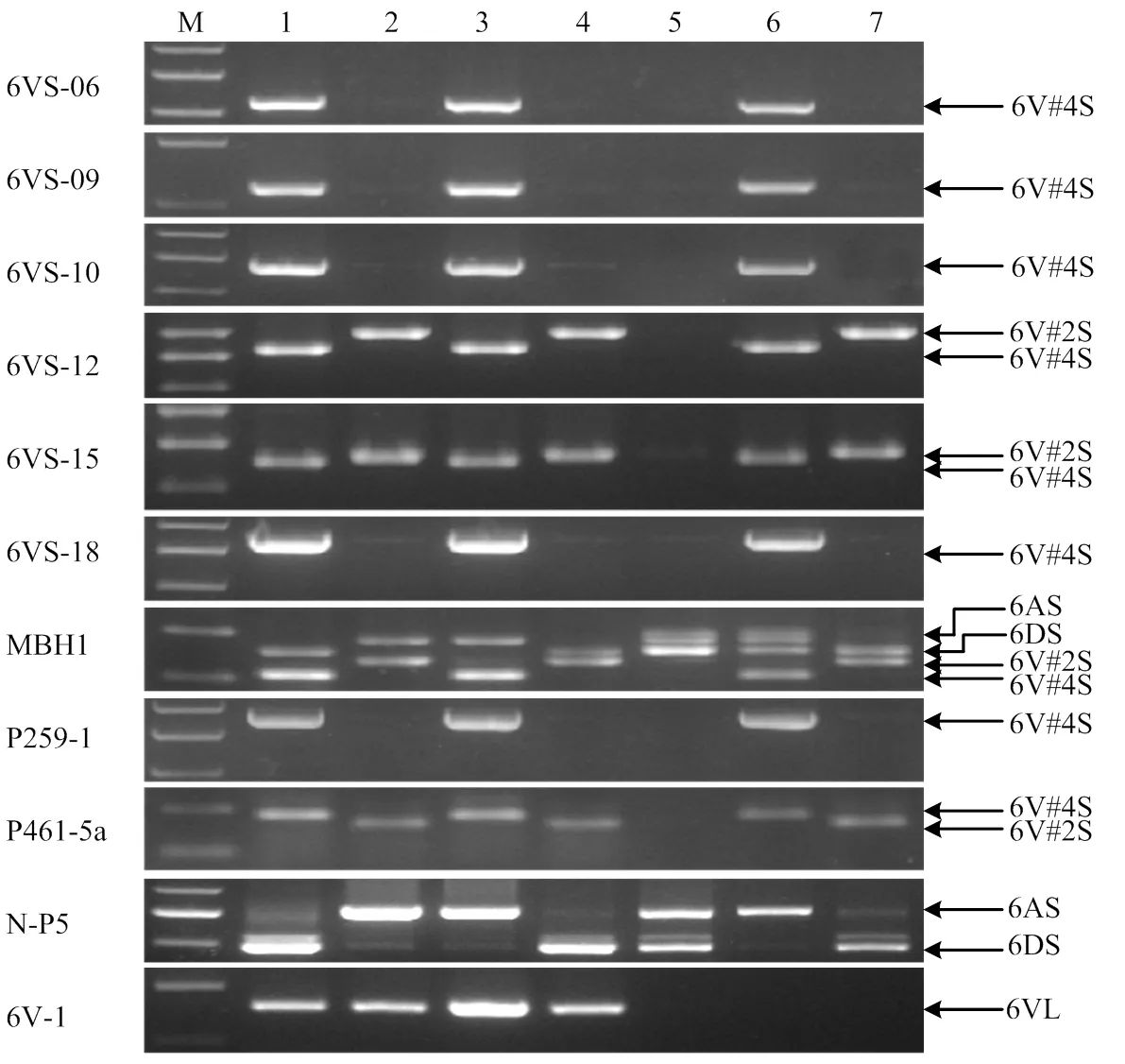
M:DL2000 marker;1—7:19EL124、19EL134、RW15、南87-88、宛7107、Pm97033和10SR3109
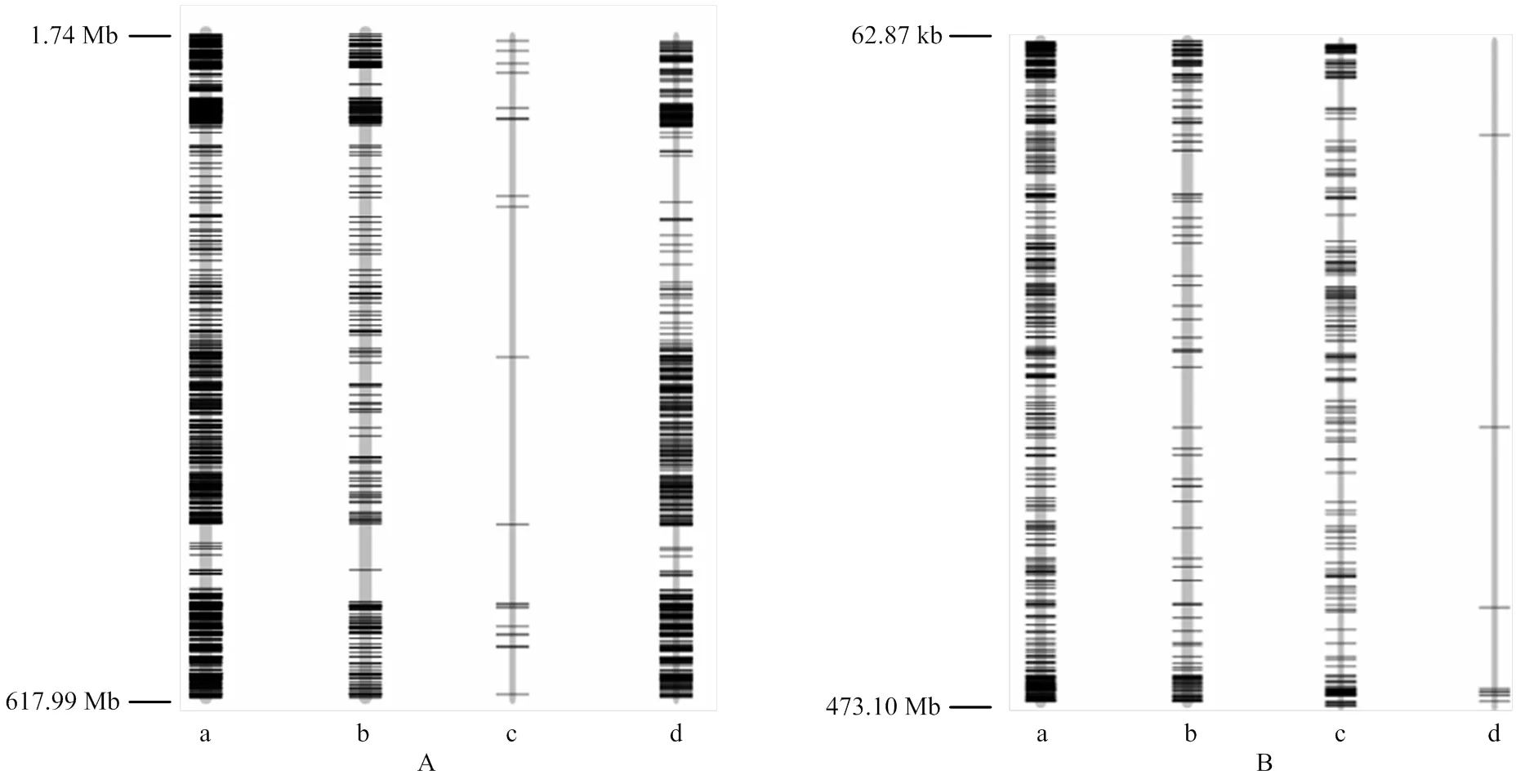
A:6A探针;B:6D探针。a:总探针数及其在染色体上的分布;b:碱基替换型SNP;c:缺失型SNP(RW15-);d:缺失型SNP(南87-88-)
479个高质量的6D探针用于检测分析。如图3B-a所示,探针物理位置从62.87 kb—473.10 Mb,涵盖小麦6D染色体约473.03 Mb的物理区域,探针平均相距约每987.55 kb;其中372个探针(77.66%)在南87-88和RW15的分型不同。二者的分型差异主要分为碱基替换的差异(图3B-b)以及NA的差异(图3B-c和图3B-d),从图3B-b和图3B-c可知,在南87-88中较少出现分型失败,而6V#4(6D)异代换系RW15,缺失6D染色体,大量6D探针不能在RW15分型。
上述结果表明,NA可能是替换小麦6A或6D的外源染色体分型的特征,为了验证这一点,调查了55K芯片所有探针中NA分型的探针在基因组各染色体的占比(表3和图4)。结果表明,在异代换系南87-88和19EL124中,6A的NA分型探针的占比最高,分别为63.21%和68.90%;在异代换系RW15和19EL134中,6D的NA分型探针的占比最高,分别为49.48%和53.44%。与6A、6D探针相比较,其余染色体探针在南87-88、RW15、19EL124和19EL134检测到的NA数量较少,占比极低,平均为0.94%。这些结果与验证的南87-88和19EL124为6A代换系、RW15和19EL134为6D代换系的结果一致。值得注意的是,尽管19EL124为6A代换系,含有6D染色体,但6D探针检测到其上的NA占比达6.89%,高于其他小麦染色体的平均值。
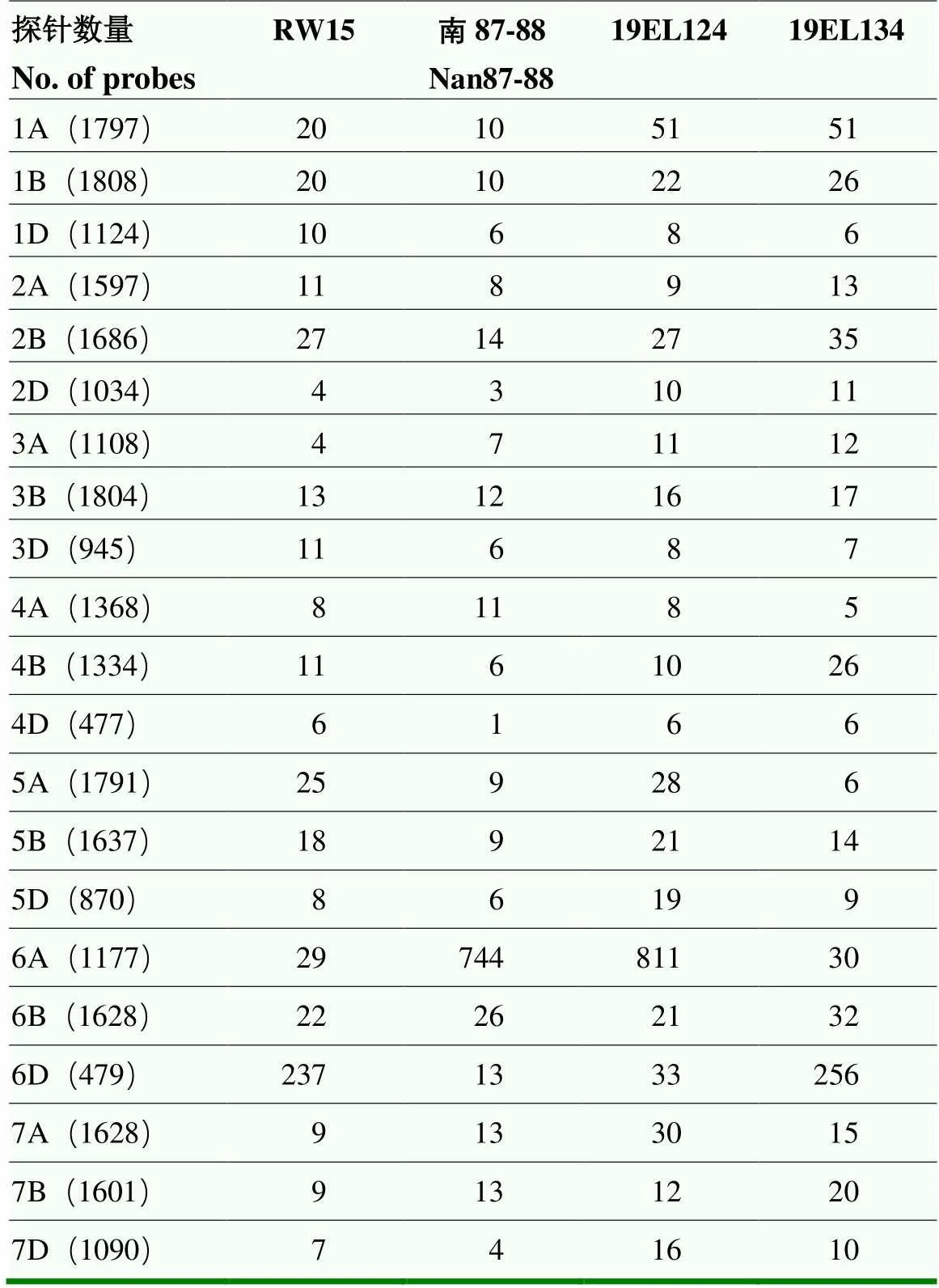
表3 探针在异代换系间检测到的NA数量
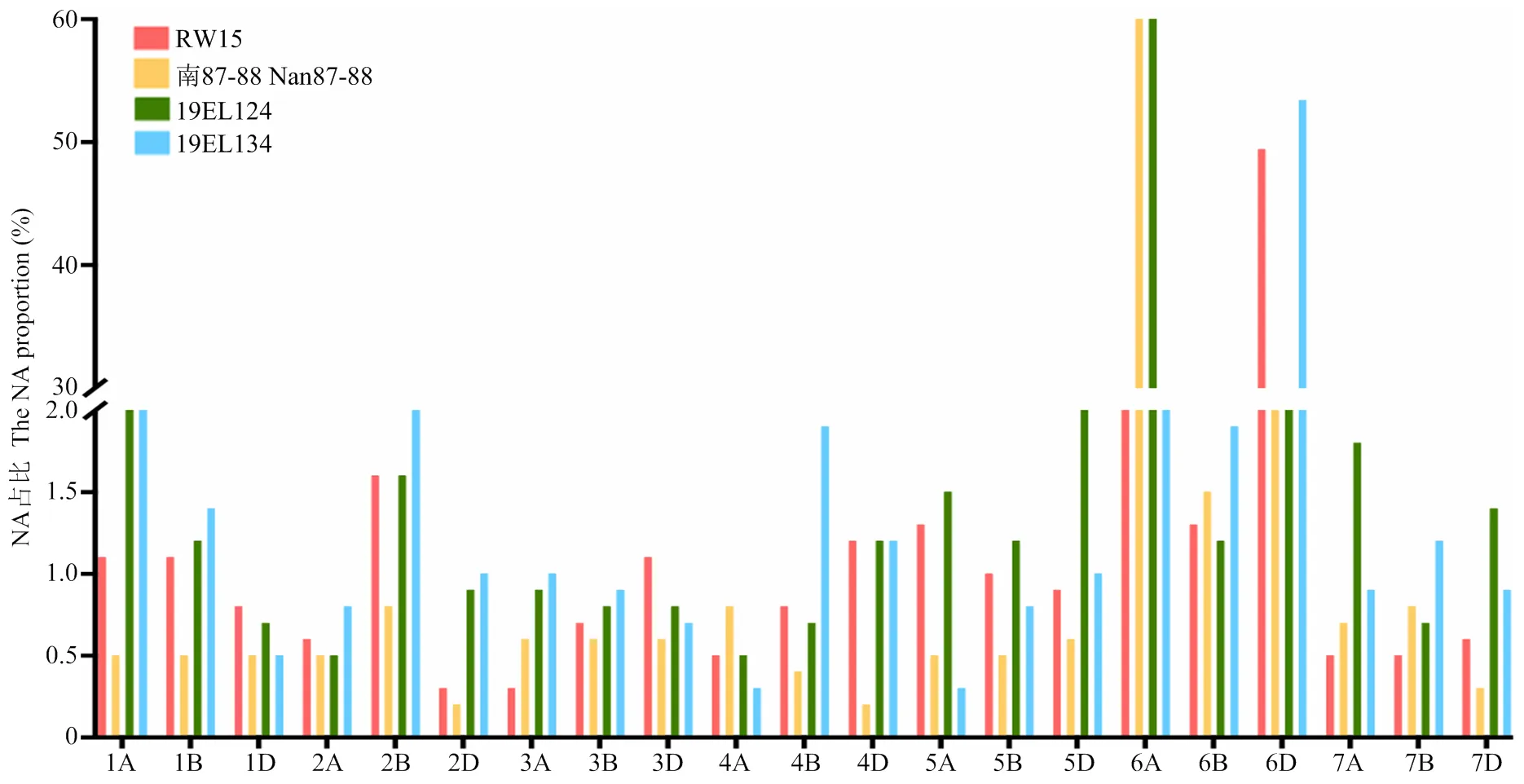
图4 28 020个物理定位的小麦55K探针检测到的NA在4个异代换系染色体的占比
2.4 6A、6D探针对同类型异代换系分型的比较
将6A、6D探针对19EL124和19EL134与相同代换类型的双亲分型进行比较。19EL124与母本南87-88同属于6A异代换类型,1 177个6A探针中,有265个显示2个系间分型不同,占6A总探针22.51%。同时,6A异代换系都含有6D染色体,479个6D探针中,有72个在2个系间表现分型差异,占6D总探针15.03%,表明二者间的6D染色体具有一定的差异。19EL134与父本RW15同属于6D异代换类型,479个6D探针中,共80个探针显示2个系间分型不同,占6D总探针16.70%。同时,6D异代换系都含有6A染色体,1 177个6A探针检测到二者间SNP 54个,占6A总探针的4.56%,说明二者间的6A染色体高度相似。
2.5 6A和6D探针对6V#2和6V#4的SNP分析
在1 177个6A探针中,有22.51%显示6V#2(南87-88)和6V#4(19EL124)间分型的差异(图5A-a)。进一步分析发现,其中,仅有22个(8.3%)碱基替换型差异(图5A-b),其余是NA的差异,如19EL124与南87-88比较,显示6V#4NA6V#2+的分型差异有155个(图5A-c),而6V#4+6V#2NA的分型差异有88个(图5A-d)。这表明了2条外源染色体相比,6A探针可以更多地检测6V#2。在479个6D探针中,有80个(15.03%)探针显示19EL134和RW15间分型的不同(图5B-a),进一步分析发现,15个(18.75%)为碱基替换型的差异(图5B-b),其余为缺失型差异(图5B-c和图5B-d),其中6V#2NA6V#4+和6V#2+6V#4NA的多态性探针各有42和23个。与6A探针相比,6D探针可以更多地检测6V#4。
2.6 660K芯片小麦第6同源群染色体探针对2份簇毛麦的SNP分析及6V特异标记的筛选
用小麦660K芯片第6群染色体的探针分析2份簇毛麦CMM(含有6V#2染色体)和No.1026(含有6V#4染色体)间的SNP(表4),结果表明,4 796个6A探针中,1 710个显示CMM和No.1026间的分型差异,占6A总探针的35.65%,其中NA分型多态性探针在6A差异分型中占48.07%,CMM和No.1026各占24.21%和23.86%。14 352个6B探针中,5 626个显示二者间的分型差异,占6B总探针的39.20%,其中NA分型多态性探针占6B差异分型探针的46.02%,CMM和No.1026各占24.46%和21.56%。3 051个6D探针中,1 206个显示二者间的分型差异,占6D总探针的39.53%,其中NA多态性探针占6D差异分型探针的39.88%,CMM和No.1026各占20.48%和19.40%。可见,6A、6D的NA分型差异探针的比例在2份簇毛麦间是比较接近的。
将用于2份簇毛麦和4份异代换系分析的6A、6D探针进行整合,2份芯片中共享的6A探针有395个,其中65个探针在CMM和南87-88中显示一致的分型,61个在No.1026和19EL124中显示一致分型,二者共享的探针25个,占6A总共享探针的6.33%。这些探针在2条外源染色体间有3个探针显示SNP,均为NA多态性,其中CMM有1个,No.1026有2个。与2份6D异代换系的6A染色体分型相比,这25个探针有22个显示SNP,占25个总共享探针的88%。2份芯片中共享的6D探针有231个,其中CMM和19EL134分型相同的探针有40个,No.1026和RW15间相同分型的探针数41个,二者共享的探针15个,占6D 231个总共享探针的6.49%。这些探针在2条外源染色体的分型完全一致,没有多态性。但与2份6A异代换系相比,所有分型都不相同,显示出100%的差异。

A:6A探针检测6V(6A)异代换系19EL124与南87-88的分型差异,a:总差异探针的分布;b:碱基替换型差异探针的分布;c:缺失型(19EL124-)差异探针的分布;d:缺失型(南87-88-)差异探针的分布。B:6D探针检测6D异代换系19EL134与RW15,a:总差异探针的分布;b:碱基替换型差异探针的分布;c:缺失型(19EL134-)差异探针的分布;d:缺失型(RW15-)差异探针的分布
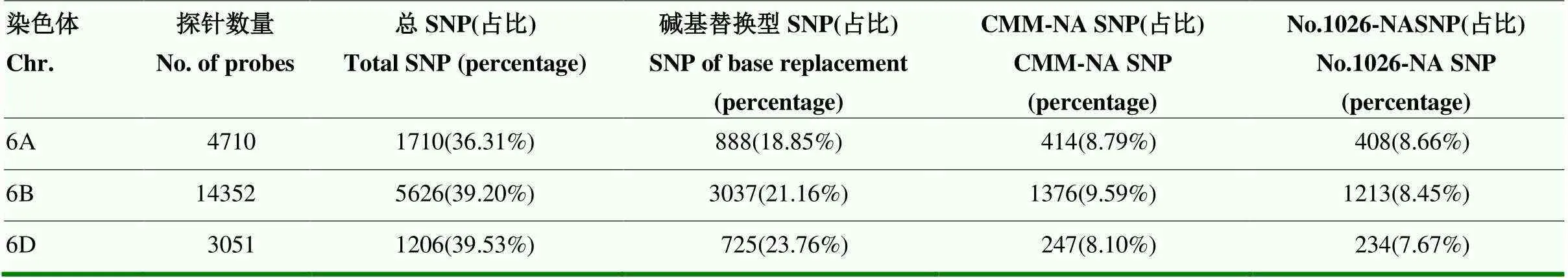
表4 660K芯片6A、6D探针对簇毛麦CMM和No.1026的检测
2.7 6A和6D探针对双亲与衍生系中6A与6D染色体的SNP分析
RW15和19EL134都含有6A染色体,6A的探针检测到二者碱基替换型SNP有17个,显示NA多态性的探针各有18和19个,各占总探针的1.44%、1.53%和1.61%。南87-88和19EL124中都含有6D染色体,6D探针检测到二者碱基替换型SNP有38个,显示NA多态性的探针各有7和27个(图5A-d),各占总探针的7.93%、1.46%和5.64%。可见,6D染色体在子代与亲代间的差异比较明显。
3 讨论
3.1 GISH及染色体特异标记鉴定的必要性
GISH是以外源物种的基因组DNA为探针的DNA分子杂交技术,是鉴定外源染色体最为直观有效的方法,可鉴定各种附加系、代换系及易位系等材料中的外源染色体状况。GISH已成功应用于鉴定小麦遗传背景中的簇毛麦、大麦、新麦草、大赖草、黑麦、拟斯卑尔山羊草等外源染色体[22-25]。在2份异代换系的杂种后代分离群体中,笔者曾筛选到缺失6VS标记、仅含6VL的材料,说明在小麦-外源异染色体系的杂交过程中,一些外源染色体可能通过着丝粒的错分裂与小麦染色体产生臂间的易位或端体。由于GISH只能检测到外源染色体,但不能确定具体的外源染色体的身份,在筛选异代换系早期,主要利用了6VS特异的分子标记跟踪6V染色体,本研究利用6V长短臂的分子标记及原位杂交相结合的方法追踪外源染色体,同时通过细胞学观察体细胞染色体数目,并利用相应的6AS、6DS特异分子标记确认缺失的小麦染色体以鉴定筛选目标材料,确保用于芯片分析的材料是准确的染色体异代换系。
3.2 大量6A和6D探针对外源染色体分型的失败,反映了小麦与近缘种部分同源染色体在DNA水平上存在较大差异
本研究利用小麦55K芯片6A和6D上的探针进行分析,发现小麦与外源部分同源染色体间极高的差异。6A探针检测出双亲间的差异实际上是南87-88中6V#2染色体与RW15中的6A染色体间的多态性。6D探针检测出的差异实际上是南87-88中的6D染色体与RW15中的6V#4染色体间的多态性,而无论6A或6D探针,绝大多数的差异属于小麦染色体可明确分型而外源染色体无法分型的缺失型多态性。此外,从与660K芯片整合的数据中,筛选到40个簇毛麦与代换系中6V染色体分型一致的6A和6D探针,其中有37个(92.5%)显示6V与小麦6A或6D间的SNP,这些结果表明了小麦染色体和外源部分同源染色体间在DNA水平上存在较大差异,反映了二者具有较远的亲缘关系。NA多态性探针在整条染色体上较为均匀地分布(图5)也与整条染色体替换的事实相符合。这些结果与Bai等[26]利用660K芯片对小麦华山新麦草2Ns/2D异代换系H139检测表明其2D上染色体丢失标记率显著高于对照7182的结果极为相似。最近LAUREN等[27]基于35K SNP芯片对17个中国春-长穗偃麦草附加系进行了分析,发现1 594个标记在小麦和长穗偃麦草之间显示出多态性。通过Flapjack™分析删除了497个错误标记,并移除834个单分离或分离不一致的标记后仅筛选到263个用于构建遗传连锁图谱的SNP标记。
3.3 同类异代换系间外源同源染色体的NA多态性,反映了2条不同来源的6V染色体在DNA水平上存在差异
Qi等[28]和Liu等[29]曾报道,利用6V#3异附加系与小麦6D单体杂交,希望通过双单体诱导6V#3与6D染色体间发生罗伯逊易位,但在其后代中选择到的易位全部发生在6V#3与6A之间,尚未发现发生在预期的靶标染色体之间的例子,作者推测可能与所涉及易位的染色体着丝粒间的亲缘关系或与遗传背景相关。在迄今报道所获得的易位系中,也只存在6V#2S·6AL和6V#4S·6DL 2种类型,虽然最近通过2对不同易位系杂交已在其后代中获得2个外源染色体臂的重组子[30]。本研究中,2对不同的外源同源染色体分别取代了相同的小麦染色体,因此,应用小麦芯片靶标染色体上的探针尝试对不同来源的外源染色体进行多态性分析。我们注意到,缺失小麦6A或6D染色体的异代换系,不能被相应的小麦SNP芯片上的6A或6D染色体的大多数探针所分型,NA比例显著增加是4份异代换系的共同特征(表3和图3),2份6A异代换系用6A探针检测时有22.51%的探针显示分型的多态性,其中91.70%的多态性分型由NA的差异构成;2份6D异代换系用6D探针检测时有16.70%的探针显示分型的多态性,其中81.25%的多态性分型由NA的差异构成,NA的多态性表明不同外源染色体DNA水平上存在较大的差异。对于6A异代换系,与6V#4染色体相比,6A染色体探针可以更多地检测6V#2染色体;反之,对于6D异代换系,与6V#2染色体相比,6D染色体探针则可以更多地检测6V#4染色体。相比之下,660K芯片6A和6D探针对2份不同簇毛麦的基因分型结果,NA多态性的比例较低,占6A和6D染色体总多态性探针的48.07%和39.88%,说明不同遗传背景对基因分型存在影响。
异源六倍体小麦基因组含有3个基因序列和排序十分近似的亚基因组,存在大量高度相似的同源基因或序列,虽然SNP芯片的探针在设计上是染色体特异的和唯一的,但染色体的缺失和异源染色体的替换可能增加了基因分型的复杂性。本研究由于2组芯片共有且在异代换系和相应簇毛麦间分型一致的6A、6D探针数只有40个,仅获得37个6V染色体特异标记,有3个标记可区分2条外源同源染色体。
3.4 19EL124/19EL134与双亲6A/6D染色体的SNP
6D异代换系19EL134与RW15中,6A染色体间SNP占总探针的4.59%,而6D探针检测2个6A异代换系19EL124和南87-88的SNP所占比例达15.03%。因为父本RW15缺失6D染色体,19EL124的6D染色体理论上来源于母本南87-88,但检测结果显示,南87-88和19EL124中的6D间存在较大的差异,以此联想到,在南87-88和RW15的杂种F1减数分裂中期I,没有观察到6A和6D 2个预期的单价体,而是观察到一个单价体和1个小麦染色体构成的三价体,鉴于其F2代6D的出现频率高于6A,因而推测6D染色体参与了三价体的形成[12]。本研究芯片分析的结果似乎进一步支持了这一推断,由于杂种中6D参与了三价体的形成,意味着6D染色体发生了交换和重组,因此,后代的6D染色体与亲本的6D染色体间存在明显的差异。
4 结论
小麦芯片上的探针对近缘种簇毛麦部分同源染色体的检测效率比小麦显著降低,揭示出2个属间具有较远的亲缘关系;相同染色体探针对不同的簇毛麦同源染色体的检测效率不同,6A探针可以更好地检测6V#2,而6D探针可更好地检测6V#4;通过簇毛麦与异代换系6V染色体分型的关联分析,筛选获得6V染色体特异的SNP标记37个。
[1] GREADZIELEWSKA A. The genuspart 2.-a wild species used in wheat improvement. Euphytica, 2006, 152(3): 441-454.
[2] MINELLI S, CECCARELLI M, MARIANI M, DE P C, CIONINI P G. Cytogenetics of×hybrids and derived lines. Cytogenetic and Genome Research, 2005, 109(1/3): 385-392.
[3] ZHANG Q P, LI Q, WANG X Y, WANG H Y, LANG S P, WANG Y N, WANG S L, CHEN P D, LIU D J. Development and characterization of atranslocation line T4VS·4DL conferring resistance to wheat spindle streak mosaic virus. Euphytica, 2005, 145(3): 317-320.
[4] ZHANG R Q, FAN Y L, KONG L N, WANG Z J, WU J Z, XING L P, CAO A Z, FENG Y G., an adult-plant powdery mildew resistance gene introgressed fromchromosome arm 2VL into wheat. Theoretical and Applied Genetics, 2018, 131(12): 2613-1620.
[5] DE P C, VACCINO P, CIONINI P G, PASQQUINI M, BIZZARRI M, QUALSET C O. Wild crop relatives: genomic and breeding resources: cereals. Heidelberg: Springer, 2011: 185-292.
[6] Zhang R Q, Zhang M Y, Wang X E, Chen P D. Introduction of chromosome segment carrying the seed storage protein genes from chromosome 1V ofshowed positive effect on bread-making quality of common wheat. Theoretical and Applied Genetics, 2014, 127(3): 523-533.
[7] LI H J, CONNER R L, CHEN Q, JIA X, LI H, GRAF R J, LAROCHE A, KUZYK A D. Different reactions to the wheat curl mite and Wheat streak mosaic virus in various wheat6V and 6VS lines. Plant Disease, 2002, 86(4): 423-428.
[8] 刘畅, 李仕金, 王珂, 叶兴国, 林志珊. 簇毛麦6VS特异转录序列P21461及P33259的获得及其分子标记在鉴定小麦-簇毛麦抗白粉病育种材料中的应用. 作物学报, 2017, 43(7): 983-992.
LIU C, LI S J, WANG K, YE X G, LIN Z S. Developing of specific transcription sequences P21461 and P33259 on6VS and application of molecular markers in identifying wheat-breeding materials with powdery mildew resistance. Acta Agronomica Sinica, 2017, 43(7): 983-992. (in Chinese)
[9] LI S J, WANG J, WANG K Y, CHEN J N, WANG K, DU L P, NI Z F, LIN Z S, YE X G. Development of PCR markers specific togenome based on transcriptome data and their application in breeding#4 alien chromosome lines. BMC Genomics, 2019, 20: 289.
[10] BIE T D, ZHAO R H, ZHU S Y, CHEN S L, CEN B Q, GAO D R, JIANG Z N, CHEN T T, WANG L, WU R L, HE H G. Development and characterization of marker MBH1 simultaneously tagging genesandconferring resistance to powdery mildew in wheat. Molecular Breeding, 2015, 35(10): 189.
[11] 陈竟男, 马晓兰, 王振, 李仕金, 谢皓, 叶兴国, 林志珊. 基于簇毛麦No.1026转录组的SSR序列分析及其PCR标记开发. 中国农业科学, 2019, 52(1): 6-15.
CHEN J N, MA X L, WANG Z, LI S J, XIE H, YE X G, LIN Z S. ssr sequences and development of pcr markers based on transcriptome ofNo.1026. Scientia Agricultura Sinica, 2019, 52(1): 6-15. (in Chinese)
[12] MA X L, XU Z Y, WANG J, CHEN H Q, YE X G, LIN Z S. Pairing and exchanging betweenchromosomes 6V#2 and 6V#4 in the hybrids of two different wheat alien substitution lines. International Journal of Molecular Sciences, 2019, 20(23): 6063.
[13] COLIN R, CAVANAGH, CHAO S M, WANG S C, HUANG B E. Genome-wide comparative diversity uncovers multiple targets of selection for improvement in hexaploid wheat landraces and cultivars.Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(20): 8057-8062.
[14] WANG S C, WONG D B, KERRIE F. Characterization of polyploid wheat genomic diversity using the high-density 90,000 SNP array. Plant Biotechnology Journal, 2014, 12(6): 1-10.
[15] SRBHI G, STELLA H E, YANG C Y, DUNCAN S, STEPHEN A, AMANDA B, PAUL A W, KING L P, KING J L. Detection ofintrogressions in wheat and development of a panel of interspecific introgression lines. Frontiers in Plant Science, 2018, 9: 1565.
[16] RAZ A, MORAN N, TAMAR E, HANAN S. Ultra-dense genetic map of durum wheat×wild emmer wheat developed using the 90K iSelect SNP genotyping assay. Molecular Breeding, 2014, 34(4): 1549-1562.
[17] STUART J, LUCAS, AYTEN S, SELAMI Y. High-throughput SNP genotyping of modern and wild emmer wheat for yield and root morphology using a combined association and linkage analysis. Functional and Integrative Genomics, 2017, 17(6): 667-685.
[18] ZHOU S G, ZHANG J P, CHE Y G, LIU W H, LU Y Q, YSNG X M, LI X Q, JIA J Z, LIU X, LI L H. Construction ofgenetic linkage maps using a wheat 660K SNP array reveals a homoeologous relationship with the wheat genome. Plant Biotechnology Journal, 2018, 16(3): 818-827.
[19] 曹廷杰, 谢菁忠, 吴秋红, 陈永兴, 王振忠, 赵虹, 王西成, 詹克慧, 徐如强, 王际睿, 罗明成, 刘志勇. 河南省近年审定小麦品种基于系谱和SNP标记的遗传多样性分析. 作物学报, 2015, 41(2): 197-206.
CAO T J, XIE J Z, WU Q H, CHEN Y X, WANG Z Z, ZHAO H, WANG X C, ZHAN K H, XU R Q, WANG J R, LUO M C, LIU Z Y. Genetic diversity of registered wheat varieties in henan province based on pedigree and single-nucleotide polymorphism. Acta Agronomica Sinica, 2015, 41(2): 197-206. (in Chinese)
[20] Li S J, Lin Z S, Liu C, Wang K, Du L P, Ye X G. Development and comparative genomic mapping of6V#4S- specific PCR markers using transcriptome data., 2017, 130(10): 2057-2068.
[21] WEI W H, QIN R, SONG Y C, NING S B, GUO L Q, GU M G. Genomic in situ hybridization analysis for identification of introgressed segments in alloplasmic lines from×.Hereditas, 2003, 138(1): 21-26.
[22] 陈佩度, 周波, 齐莉莉, 刘大钧. 用分子原位杂交(GISH)鉴定小麦-簇毛麦双倍体、附加系、代换系和易位系. 遗传学报, 1995, 22(5): 380-386.
CHEN P D, ZHOU B, QI L L, LIU D J. Identification of wheat-amphiploid, addition, substitution and translocation lines by in situ hybridization using biotin-labelled genomic DNA as a probe. Acta Genetica Sinica, 1995, 22(5): 380-386. (in Chinese)
[23] MUKAI Y, GILL B S. Detection of barley chromatin added to wheat by genomic in situ hybridization. Genome, 1991, 34(3): 448-452.
[24] FRIEBE B, JIANG J, GILL B S, DYCK P L. Radiation-induced nonhomoeologous wheat-intermedium chromosomal translocations conferring resistance to leaf rust. Theoretical and Applied Genetics, 1993, 86(2/3): 141-149.
[25] 赵万春. 普通小麦-簇毛麦整臂互补易位系T1DS·1AL和T1DL·1VS的创制、鉴定和性状评估[D]. 杨凌: 西北农林科技大学, 2010.
ZHAO W C. Development, identification and characterization of two newconpensation robersonian translocation lines T1DS·1AL and T1DL·1VS[D]. Yangling: Northwest A&F University, 2010. (in Chinese)
[26] BAI S S, YUAN F P, ZHANG H B, ZHANG Z Y, ZHAO J X, YANG Q H, WU J, CHEN X H. Characterization of theKeng 2Ns/2D substitution line H139: A novel germplasm with enhanced resistance to wheat take-all. Frontiers in Plant Science, 2020, 11: 233.
[27] LAUREN B, SURBHI G, YANG C Y, STELLA H E, DUNCAN S, STEPHEN A, AMANDA J, ALEXANDRA M, PRZEWIESLIK A, PAUL A, KING L P, KING J L. Exploiting the genome ofto expand the gene pool of hexaploid wheat. Theoretical and Applied Genetics, 2020, 133(7): 2213-2226.
[28] Qi L L, PUMPHRAY M O, FRIEBE B, ZHANG P, QIAN C, BOWDEN R L, ROUSE M N, JIN Y, GILL B S. A novel Robertsonian translocation event leads to transfer of a stem rust resistance gene () effective against race Ug99 frominto bread wheat. Theoretical and Applied Genetics, 2011, 123(1): 159-167.
[29] LIU C, QI L L, LIU W X, ZHAO W C, JAMIE W, BERND F, BIKRAM S G. Development of a set of compensating-robertsonian translocation lines. Genome, 2011, 54(10): 836-844.
[30] ZHANG R H, LIU B L, JIANG Z N, CHEN T T, WANG L, JI Y Y, HU Z B, HE H G, BIE T D. Comparative analysis of genetic effects of wheat-translocations T6V#2S·6AL and T6V#4S·6DL. Frontiers in Plant Science, 2019, 138: 503-512.
Polymorphism Analysis Among Chromosomes of6V#2 and 6V#4 and Wheat 6A and 6D based on wheat SNP Chip
XU ZhiYing1,2, WANG BaiCui2, MA XiaoLan2, JIA ZiMiao2, YE XingGuo2, LIN ZhiShan2, HU HanQiao1
1College of Coastal Agricultural Sciences, Guangdong Ocean University, Zhanjiang 524088, Guangdong;2Institute of Crop Science, Chinese Academy of Agricultural Sciences, Beijing 100081
【】 The polymorphism among the chromosomes of6V#2 and 6V#4 and their wheat homeologous 6A and 6D in the DNA level was compared in this study based on big data to provide a theoretical basis for precision designing breeding of wheat-targeted chromosome translocation.【】A 6V#4(6D) alien disomic substitution line RW15 was crossed with a 6V#2(6A) alien disomic substitution line Nan 87-88, and their F2plants were detected using 6V#4S/6V#2S/6AS/6DS/6VL specific molecular markers, and the new types of substitution lines were confirmed in F3employing the above-mentioned markers again as well as genomichybridization (GISH) technique. Subsequently, polymorphism among the targeted chromosomes in the new types of the substitution lines and their parents Nan 87-88 and RW15 were detected by the probes of 6A and 6D-specific in the wheat 55K chip, and combined with the results of SNP analysis of two accessions ofwith 6A and 6D probes in 660k chip to screen 6V specific SNPs. 【】 GISH analysis showed that 19EL124 and 19EL134 had 42 chromosomes including two complete foreign chromosomes in the somatic cells of root tips. Identification using molecular markers showed that 19EL124 had 6V#4S/6DS and missed 6V#2S/6AS specifically amplified bands, while 19EL134 displayed 6V#2S/6AS distinctive bands and lacked 6V#4S-specific bands; both 19EL124 and 19EL134 contained 6VL specific amplified bands, which confirmed that 19EL124 is a 6V#4(6A) disomic substitution line, and 19EL134 is a 6V#2(6D) disomic substitution line. The results of 55k chip showed that the detected efficiency of the key chromosome probes in the alien substitution lines was significantly lower than that of other chromosome probes, and the detected efficiency of different lines in the same type of substitution was also different. Among the 1 177 probes of 6A, the 6A substitution line Nan87-88 and 19EL124 could not be genotyped by 63.21% and 68.90% of the probes, respectively. And the polymorphism between 6V#2 and 6V#4 was detected by 22.51% of the probes. Among the 479 probes of 6D, 49.48% of the probes could not identify the 6D substitution line RW15, and 53.44% could not genotype the 6D substitution line 19EL134. the polymorphism between 6V#2 and 6V#4 was indicated by 16.70% of the 6D probes, there were 23 and 42 probes that could only detect 6V#2 and 6V#4 chromosome, respectively. Twenty-two and fifteen 6V specific SNPs were screened from 395 6A and 231 6D probes through integrating the same genotypes detected by probes which were shared by 55k and 660k chips in this study, and three of them showed polymorphism between 6V#2 and 6V#4 chromosomes. 【】 The detection efficiency of the wheat chromosome probes was greatly reduced when the target wheat chromosome was replaced by the alien chromosome, and the proportion of NA genotyping was greatly increased, and most of the NA genotypes showed polymorphism between the two different alien chromosomes in the same type of substitution. the detection efficiency of probe for two alien chromosomes in the same type of alien substitution lines was different, wheat 6A probe could detect 6V#2 better, and wheat 6D probe could detect 6V#4 better. thirty-seven SNP markers of 6V specific were obtained base on the association analysis of 6V genotyping betweenand the alien substitution lines.
wheat;; alien disomic substitution; wheat SNP chip; polymorphism

10.3864/j.issn.0578-1752.2021.08.001
2020-08-24;
2020-10-28
国家重点研发计划(2016YFD0102002)
许志英,E-mail:13414916566@163.com。通信作者胡汉桥,E-mail:huhanqiao@sina.com。通信作者林志珊,E-mail:linzhishan@caas.cn
(责任编辑 李莉)

