基于影像组学与集成学习的脑胶质瘤分级预测
戴 宏, 符冉迪, 金 炜
基于影像组学与集成学习的脑胶质瘤分级预测
戴 宏, 符冉迪*, 金 炜
(宁波大学 信息科学与工程学院, 浙江 宁波 315211)
脑胶质瘤的术前分级对治疗决策和预后评估至关重要. 为了提高分级精度, 提出了一种基于影像组学和集成学习的无创胶质瘤术前分级方法. 首先, 从不同序列的感兴趣区域提取428个影像组学特征, 采用递归特征消除算法进行特征选择, 采用6种不同的机器学习算法对脑胶质瘤进行分级, 并对各自的性能进行评估; 然后, 根据评估结果, 选取逻辑回归、决策树和多层感知机3种分类器作为脑胶质瘤分级预测的机器学习算法; 最后, 将3种分类器的输出采用投票方式进行集成, 并评估硬投票机制与软投票机制的性能. 实验结果表明, 对于数据集BraTS2019, 基于硬投票机制的集成学习算法的性能较好, 受试者工作特性曲线下面积为0.933±0.031, 准确度为0.886±0.048, 敏感度为0.872±0.077, 特异度为0.905±0.105. 该方法不仅能增加胶质瘤分级模型的可解释性, 而且可以提高分级精度.
脑胶质瘤分级; 影像组学; 递归特征消除; 集成学习
脑胶质瘤是人体脑部最为常见的一种恶性肿瘤, 约占中枢神经系统原发性肿瘤及其他肿瘤的24.7%, 占恶性肿瘤的74.6%[1]. 世界卫生组织将脑胶质瘤分为WHO-Ⅰ级至WHO-Ⅳ级, 其中WHO-Ⅰ级、WHO-Ⅱ级被称为低级别胶质瘤(Low Grade Glioma, LGG), WHO-Ⅲ级、WHO-Ⅳ级被称为高级别胶质瘤(High Grade Glioma, HGG)[2]. LGG生长速度较慢, 在进行手术切除后通常可以治愈; HGG生长迅速、侵袭性高、预后差, 患者在进行手术切除后仍有很高的病死率, 术后需要进行辅助化疗和放疗. 因此, 准确分级预测脑胶质瘤, 将有助于提供精确的治疗方案, 优化患者的预后[3].
在脑胶质瘤分级预测研究中, 通常采用影像组学与机器学习方法[4]. 文献[5]采用不同磁共振图像(Magnetic Resonance Imaging, MRI)、不同机器学习模型来建立脑胶质瘤术前分级的影像组学模型, 比较分析了2种序列的MRI图像在3种机器学习方法下的分级性能. 文献[6]通过提取小波特征作为影像组学特征, 利用偏最小二乘法(Partial Least Squares, PLS)进行降维, 有效提高了脑胶质瘤的分级性能. 但这些研究中分级预测模型均采用单一序列的MRI图像及单一的机器学习方法, 泛化性能不高, 导致胶质瘤的分级性能难以进一步提高.
本研究将集成学习思想引入脑胶质瘤术前的分级预测中, 通过提取多个序列下脑胶质瘤MRI图像的影像组学特征, 采用递归特征消除(Recursive Feature Elimination, RFE)算法对多序列影像组学特征进行筛选, 选择6种机器学习算法进行训练和评估, 最后构建逻辑回归(Logistic Regression, LR)、多层感知机(Multilayer Perceptron, MLP)和决策树(Decision Trees, DT)3种分级预测算法的集成学习模型对脑胶质瘤术前进行分级预测, 以期提高脑胶质瘤的分级精度.
1 材料与方法
胶质瘤的分级主要包含: 图像预处理、特征提取、特征选择、模型训练和测试4个步骤(图1).

GLDM为灰度相关矩阵; GLCM为灰度共生矩阵; GLSZM为灰度区域大小矩阵; GLRLM为灰度游程矩阵; RFE为递归特征消除算法; LR为逻辑回归; MLP为多层感知机; DT为决策树; SVM为支持向量机; NB为朴素贝叶斯; KNN为K近邻; AUC为受试者工作特性曲线下面积; ROC为受试者工作特性.
1.1 数据集
本研究的影像数据均来自MICCAI网站(http:// www.braintumorsegmentation.org)上BraTS2019数据集[7], 该数据集包含有334例病例, 其中高级别脑胶质瘤259例, 低级别脑胶质瘤75例. 每位患者均接受了4种MRI序列扫描, 即T1(T1 Weighted)、T1ce (T1 Weighted with Contrast Enhanced)、T2(T2 Weighted)和Flair(Fluid-Attenuated Inversion Recovery)序列, 4个序列的原始图像和感兴趣区域(Region of Interest, ROI)的图像显示如图2所示.
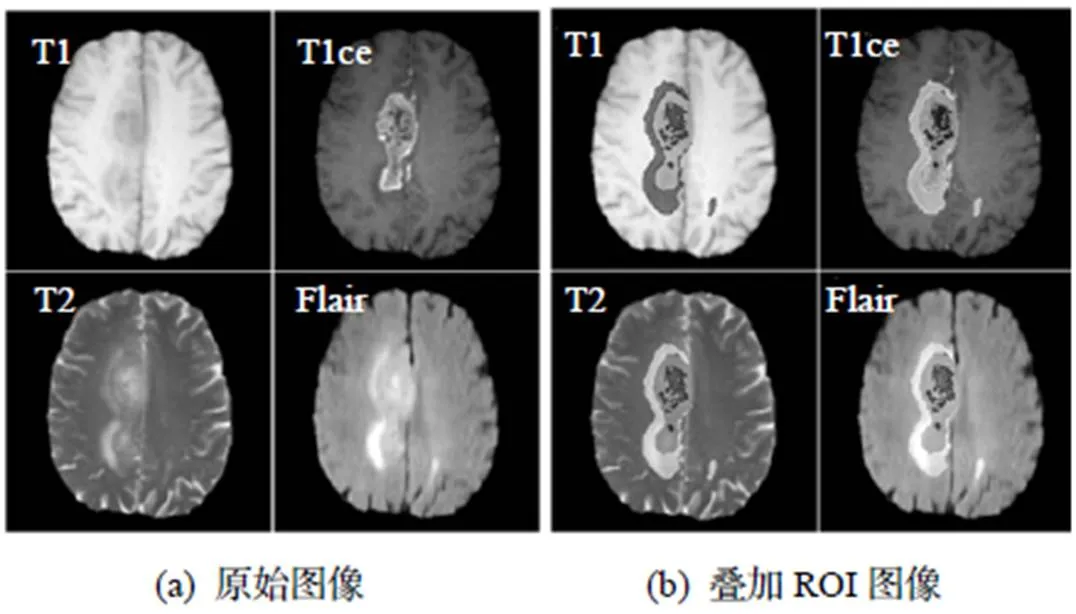
图2 4个序列的原始图像及叠加ROI图像
将所有数据集内的胶质瘤图像去除颅骨, 然后进行图像配准, 并以1mm3的分辨率进行插值, 以确保4个序列下的图像能严格匹配[8-9]. ROI由经验丰富的神经影像科医生手工分割而成, 包括坏死和非增强肿瘤核心(Necrosis and Non-enhancing Tumor, NCR/NET)、瘤周水肿(Peritumoral Edema, ED)、Gd增强的肿瘤部分(Gadolinium-enhancing Tumor, ET). 不同MRI序列对脑胶质瘤的表达能力有所区别. 在T2和Flair序列上, 坏死区和非增强肿瘤核心区域有高信号; 在T1ce序列, 利用一个阈值可以分割得到Gd增强的肿瘤部分; 瘤周水肿区域在T2序列能得到很好的检测, 并在Flair序列下将其与血液部位进行区分.
1.2 影像组学特征提取
在不同MRI序列下, 高级别脑胶质瘤与低级别脑胶质瘤的影像学表现存在差异. 高级别脑胶质瘤的MRI信号明显不均匀, 呈混杂T1/T2信号影, 周边可见明显的指状水肿影, 增强扫描呈花环状及结节样异常强化影; 低级别脑胶质瘤的MRI信号相对均匀, 表现为长T1、长T2和Flair高信号, 一般无强化[10]. 通过多个MRI序列的影像组学特征进行脑胶质瘤分级的准确度高于单序列[11], 因此本研究提取了T1、T1ce、T2和Flair这4个序列下ROI的影像组学特征. 所有影像组学特征的提取均采用Python 3.7平台下的Pyradiomics插件进行, 每个序列分别提取107个影像组学特征, 其中形状特征14个、一阶特征18个、灰度共生矩阵特征24个、灰度相关矩阵特征14个、灰度游程矩阵特征16个、灰度区域大小矩阵特征16个、相邻灰度差分矩阵特征5个, 4个序列共提取了428个影像组学特征.
1.3 特征选择
为了消除特征间数值差异过大所造成的影响, 首先对数据集进行标准化处理. 计算公式为:
由于训练的样本有限, 过多冗余和无关的特征会导致过拟合, 同时也降低了模型的可解释性, 增加模型的训练时间. 为了降低过拟合, 增加模型的可解释性, 本研究利用RFE算法进行特征选择. RFE是一种寻找最优特征集的贪心算法, 主要通过多轮训练来搭建模型, 每轮训练结束后, 消除对模型训练结果影响小的若干特征, 再基于新的特征集进行筛选, 其代价函数为:


1.4 分类模型
对逻辑回归、支持向量机(Support Vector Machines, SVM)、多层感知机网络、随机决策树、近邻((-nearest Neighbors, KNN)分类器和朴素贝叶斯(Naive Bayes, NB)分类器这6种常用的机器学习方法在胶质瘤分级中的性能进行比较. 将筛选后的数据集输入不同的分类器中, 根据评估标准选出性能最好的3种分类器. 不同分类器对同一特征的敏感性不同, 充分考虑3种分类器的分类结果能够更客观、更稳定地反映特征性能.
基于选出的3种分类器, 提出一种基于投票机制的集成方法, 流程如图3所示. 同时对2种不同的投票机制进行评价: 硬投票(基于多数票的投票)和软投票(基于平均概率的投票). 将3种最佳分类器的分类结果提供给集成分类方法, 进而得到最终的分类结果.
1.4.1 机器学习方法
逻辑回归[12]是常用的二元分类模型, 该模型通过假设输出的因变量服从伯努利分布, 输入自变量与输出因变量之间存在线性关系. 利用Sigmoid函数处理非线性数据, 从而解决二分类问题.
支持向量机在医学分类上应用广泛. 该模型通过求解一个二次优化问题来建立数据集之间的最优分类边界, 因此也被称为大间距分类器[13]. 当使用不同的核函数时, 支持向量机会表现出不同的分类性能.
多层感知机是一种包含多个感知器的人工神经网络[14], 该模型主要分为3层(输入层、隐藏层和输出层), 其中隐藏层的数目决定了网络的复杂程度. 多层感知机通常应用于有监督学习算法中, 根据输入输出变量之间的相关性来学习模型.
随机决策树是一种流行的有监督算法[15], 该算法根据最大化数据分离的标准反复分割数据集, 从而形成树状结构. 最常用的标准是信息增益, 通过识别并选择有助于分类的重要属性, 最终达到分类的目的.
近邻不同于其他分类算法, 该算法直接使用数据进行分类.近邻中唯一可调整的参数是, 在训练集中找到一组最接近测试样本的个对象, 并基于该领域中特定类的流行程度来分配标签. 为了获得更好的性能, 需要选择合适的值来训练模型.

图3 基于多数投票机制的集成方法在脑胶质瘤分级中应用流程
朴素贝叶斯是基于贝叶斯定理和最大后验假设的分类模型. 该模型假设一个属性对一个给定类的影响独立于其他属性, 被称为“条件独立”. 通过将最大概率分配给正确的类别, 从而达到分类的效果, 同时不需要准确的概率估计.
1.4.2 投票机制




1.5 模型评估
采用五折交叉验证的方法进行模型训练和测试. 根据真阳性(True Positive, TP)、真阴性(True Negative, TN)、假阳性(False Positive, FP)、假阴性(False Negative, FN)计算敏感度、特异度以及准 确度, 绘制受试者工作特性(Receiver Operating Characteristic, ROC)曲线, 并计算受试者工作特性曲线下面积(Area Under the Receiver Operating Characteristic Curve, AUC), 对不同分类器模型进行评估, 其计算公式分别为:



2 实验结果
本研究所有方法都在Python3.7(https://www. python.org)、R3.6.0(http://www.Rproject.org)上实现. 采用Python软件提取和筛选影像组学特征, 并建立分类模型; 采用R软件进行统计分析. 所有实验都在Windows10操作系统下进行(CPU为Intel Core i7-8700@3.20GHz, GPU为NVIDIA GeForce RTX 2080Ti, 内存为16GB).
2.1 特征选择
利用递归特征消除算法, 从428个影像组学特征中提取15个特征, 其中包含一阶特征6个、灰度相关矩阵1个、灰度区域大小矩阵6个、灰度共生矩阵2个, 具体提取特征见表1.

表1 RFE算法所选择的特征
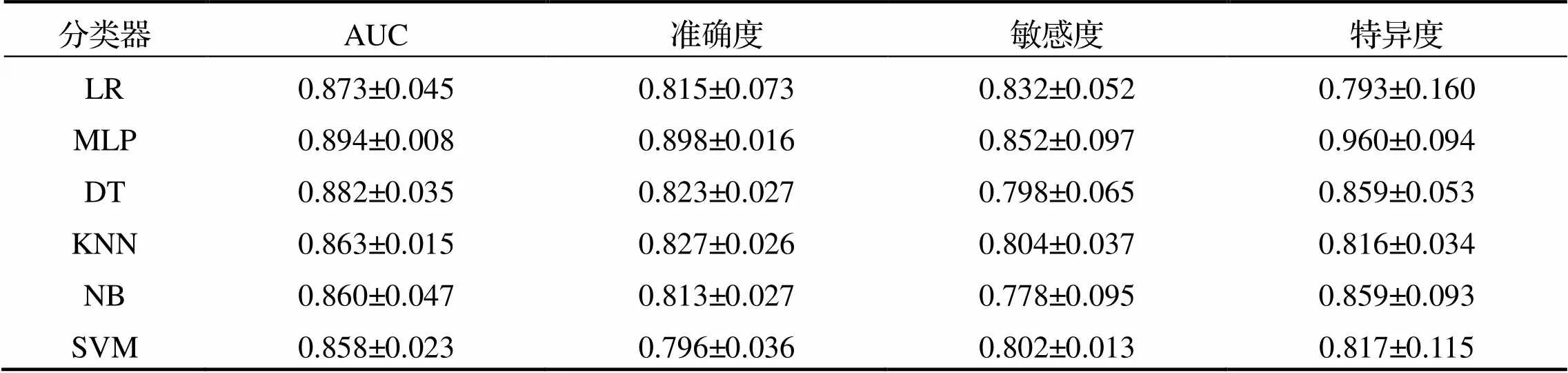
表2 各种机器学习算法的分级性能
2.2 性能比较
将筛选后的最优数据集输入各种分类器中, 并利用五折交叉验证的方法进行模型训练和评估. 6种分类器的平均分级性能见表2.
从表2可以看出, MLP有着最高的分级性能, 其AUC为0.894±0.008, 精确度为0.898±0.016, 敏感度为0.852±0.097, 特异度为0.960±0.094. DT和LR的分级性能也较好, 其中DT的AUC为0.882± 0.035; 而LR的AUC为0.873±0.045. 相比之下, KNN、NB以及SVM这3种分类器的分级性能相对较低, AUC分别为0.863±0.015、0.860±0.047和0.858±0.023.
对各种分类器结果进行分析, 根据AUC选出3种最佳分类器, 即逻辑回归、多层感知机以及决策树, 将这3种分类器的输出结果传递给不同的投票机制来预测最终的输出(图4). 图4分别给出了逻辑回归、多层感知机和决策树在五折交叉验证下的ROC曲线.
表3为集成学习中基于2种投票机制的结果. 从表3可知, 通过对3种分类器进行集成学习, 胶质瘤的分级性能得到了明显提高.其中, 硬投票中基于多数的投票机制相比于其他投票机制有更好的分级性能.

图中灰度区域为与平均ROC距离一个标准差的范围.
表3中的性能评估都是五折交叉验证后的结果, 基于硬投票的集成学习方法有着最高的分级性能: AUC为0.933±0.031, 精确度为0.886±0.048,敏感度为0.872±0.077, 特异度为0.905±0.105. ROC曲线如图5所示.

表3 2种投票机制的分级性能

图中灰度区域为与平均ROC距离一个标准差的范围.
将本文研究方法与其他优秀预测方法进行对比, 文献[16]建立了4个放射学特征数据集, 并采用最大熵判别决策树模型来完成对脑胶质瘤的分级. 该方法在BraTS2017数据集上进行了验证, 其AUC为0.818, 敏感度为0.878, 特异度为0.814. 文献[17]提出了一个带有门控、多模态单元融合的三维卷积神经网络(3-Dimensional Convolutional Neural Network, 3DCNN)模型, 以融合多模态信息和决策信息, 并在BraTS2015数据集上进行了模型的验证.本文提出的方法与文献[16-17]提出的方法的分类性能比较结果见表4.
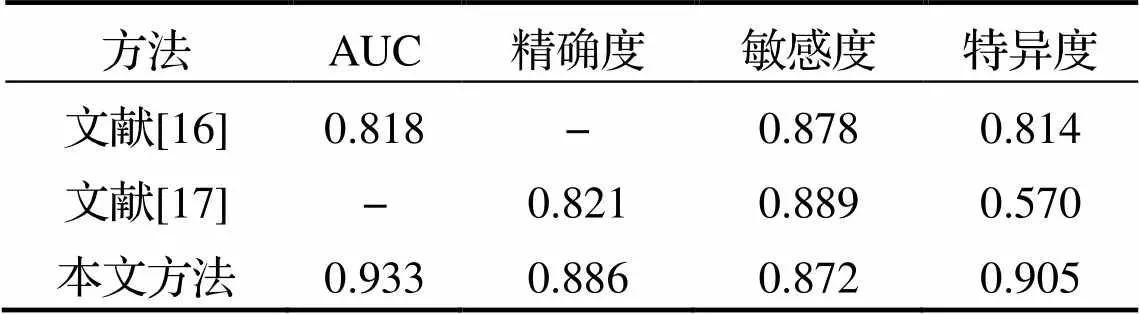
表4 本文方法与其他文献方法性能比较
注: “-”表示文献中未提及.
从表4可知, 本文所提出的脑胶质瘤分级预测方法的性能优于文献[16]和文献[17]中提出的分级方法.
3 结语
本文提出了一种新的基于影像组学与集成学习的脑胶质瘤分级方法, 通过提取4个不同序列的胶质瘤MRI图像的影像组学特征, 利用RFE进行特征选择, 并利用逻辑回归、支持向量机、多层感知机、决策树、朴素贝叶斯、近邻分类器进行胶质瘤分级. 评估这6种分类器的不同性能指标, 即AUC、准确度、敏感度和特异度. 然后选出AUC评分最高的3种分类器, 提出一种基于投票机制的集成方法, 并评估了硬投票机制与软投票机制的性能. 研究结果表明, 本文提出的基于多数投票机制的集成学习方法有着较高的分级性能, 其结果优于其他投票机制的集成方法, 并远远高于单种分类器的分级性能.
[1] Ostrom Q, Cioffi G, Gittleman H, et al. CBTRUS statistical report: Primary brain and other central nervous system tumors diagnosed in the United States in 2012-2016[J]. Neuro-Oncology, 2019, 21(S5):v1-v100.
[2] Wesseling P, Capper D. WHO 2016 classification of gliomas[J]. Neuropathology and Applied Neurobiology, 2018, 44(2):139-150.
[3] Zacharaki E I, Wang S M, Chawla S, et al. Classification of brain tumor type and grade using MRI texture and shape in a machine learning scheme[J]. Magnetic Resonance in Medicine, 2009, 62(6):1609-1618.
[4] 韩文静, 胡贵祥, 胡玲静. 影像组学在脑胶质瘤中的研究进展[J]. 国际医学放射学杂志, 2020, 43(2):183-187.
[5] 穆建华, 张雁伟, 吴志钢. 基于常规MRI图像的不同影像组学模型在脑胶质瘤术前分级中的应用[J]. 磁共振成像, 2020, 11(1):55-59.
[6] Chen Q J, Wang L H, Wang L, et al. Glioma grade prediction using wavelet scattering-based radiomics[J]. IEEE Access, 2020, 8:106564-106575.
[7] Menze B H, Jakab A, Bauer S, et al. The multimodal brain tumor image segmentation benchmark (BRATS)[J]. IEEE Transactions on Medical Imaging, 2015, 34(10): 1993-2024.
[8] Bakas S, Akbari H, Sotiras A, et al. Advancing the cancer genome atlas glioma MRI collections with expert segmentation labels and radiomic features[J]. Scientific Data, 2017, 4:170117.
[9] Clark K, Vendt B, Smith K, et al. The cancer imaging archive (TCIA): Maintaining and operating a public information repository[J]. Journal of Digital Imaging, 2013, 26(6):1045-1057.
[10] 国家卫生健康委员会医政医管局. 脑胶质瘤诊疗规范(2018年版)[J]. 中华神经外科杂志, 2019, 35(3):217- 239.
[11] Qin J B, Liu Z Y, Zhang H, et al. Grading of gliomas by using radiomic features on multiple magnetic resonance imaging (MRI) sequences[J]. Medical Science Monitor, 2017, 23:2168-2178.
[12] Landwehr N, Hall M, Frank E. Logistic model trees[J]. Machine Learning, 2005, 59(1/2):161-205.
[13] Nello C. An Introduction to Support Vector Machines and Other Kernel-based Learning Methods[M]. Cambridge: Cambridge University Press, 2000.
[14] Pal S K, Mitra S. Multilayer perceptron, fuzzy sets, and classification[J]. IEEE Transactions on Neural Networks, 1992, 3(5):683-697.
[15] Leo B. Classification and Regression Trees[M]. Belmont, CA: Wadsworth Publishing Company, 1984.
[16] Wu Y P, Hao H H, Li J, et al. Four-sequence maximum entropy discrimination algorithm for glioma grading[J]. IEEE Access, 2019, 7:52246-52256.
[17] Ye F Y, Pu J, Wang J, et al. Glioma grading based on 3D multimodal convolutional neural network and privileged learning[C]//2017 IEEE International Conference on Bioinformatics and Biomedicine (BIBM), Kansas City, MO, USA, 2017:759-763.
Glioma grading prediction based on radiomics and ensemble learning
DAI Hong, FU Randi*, JIN Wei
( Faculty of Electrical Engineering and Computer Science, Ningbo University, Ningbo 315211, China )
Glioma grading before surgery is very critical for the treatment planning and prognosis. In order to improve the grading accuracy, a non-invasive method for predicting the glioma grades based on radiomics and ensemble learning is proposed. First, 428 radiomics features are obtained from the region of interest (ROI) with different sequences. Feature selection is executed using Recursive Feature Elimination (RFE) algorithm, and 6 different machine learning algorithms are used to predict the glioma grade. Then, according to the evaluation results, three best classifiers, that is, Logistic Regression (LR), Decision Tree (DT) and Multilayer Perceptron (MLP), are selected as the machine learning algorithm for Glioma grading. Finally, these three classifiers are used for ensemble classification with a voting mechanism. The performance of hard and soft voting mechanism is also evaluated. Experimental results show that on the dataset BraTS2019, the hard voting mechanism based ensemble learning algorithm achieved the best performance, with the AUC value of 0.933±0.031, the accuracy of 0.886±0.048, the sensitivity of 0.872±0.077, and the specificity of 0.905±0.105. The presented work not only increases the interpretability of glioma grading model, but also ameliorates the grading accuracy.
glioma grading; radiomics; recursive feature elimination; ensemble learning
TP391
A
1001-5132(2021)04-0028-07
2020−10−10.
宁波大学学报(理工版)网址: http://journallg.nbu.edu.cn/
浙江省自然科学基金(LY20H180003);宁波市自然科学基金(T2019A610104); 宁波市公益类科技计划项目(202002N3104).
戴宏(1997-), 男, 江西上饶人, 在读硕士研究生, 主要研究方向: 医学影像. E-mail: 1356913744@qq.com
符冉迪(1971-), 男, 浙江宁波人, 副教授, 主要研究方向: 数字图像处理. E-mail: furandi_nbu@163.com
(责任编辑 史小丽)

