盐碱胁迫下野生大豆CBL-CIPK通路表达分析
魏志园 王 宇 司增志 魏 莱 温晓蕾 乔亚科 李桂兰 张 锴
(1河北科技师范学院农学与生物科技学院,河北秦皇岛 066000;2河北科技师范学院园艺科技学院,河北秦皇岛 066000)
我国是大豆的起源地,保存有丰富的野生大豆(Glycine soja)资源。野生大豆常生长在沿海滩涂、盐碱荒地等环境中,是典型的盐生植物。在野生大豆优质基因利用方面,我国学者通过野生大豆与栽培大豆(Glycine max)杂交育种技术已培育出一些优异的种质资源[1-6]。虽然我国拥有丰富的野生大豆种质资源,但我国对野生大豆的育种利用还不充分。在中美贸易谈判及国内非转基因大豆需求量增加的背景下,进一步充分利用野生大豆种质资源,应用传统技术培育栽培大豆新品种成为解决我国大豆供需矛盾的重要途径。
近年来野生大豆的优异特性尤其是耐盐碱性已成为学者的研究重点。野生大豆对盐碱胁迫的反应比较复杂[7]。肖鑫辉等[8]研究发现,高耐盐碱型野生大豆材料在盐碱胁迫下茎、叶平均Na+和Cl-含量显著低于低耐盐型,但高耐型野生大豆植株茎叶中也存在Na+和Cl-含量高水平和低水平两种类型,推测野生大豆可能存在高耐受性和低吸收性两种耐盐碱机制。此外,研究发现野生大豆可以通过增加超氧化物歧化酶(superoxide dismutase,SOD)、过氧化氢酶(catalase,CAT)及过氧化物酶(peroxidase,POD)等抗氧化酶活性,降低膜脂过氧化物丙二醛(malondialdehyde,MDA)含量来提高自身耐盐性[9-10]。野生大豆具有较强的抗逆能力,近年来学者对其耐盐碱性进行了诸多探索[11-15]。但是,目前野生大豆耐盐碱生理机制,尤其是野生大豆盐碱胁迫下植株体内的离子平衡机制还不清楚,耐盐碱关键基因表达及调控机制尚不明确。
类钙调磷酸酶B 蛋白(calcineurin B-like protein,CBLs)是植物中重要的Ca2+离子感受器。拟南芥CBLs蛋白含有4 个EF 手结构,是Ca2+离子的结合位点,它们的排列顺序和间距高度保守[16-17]。CBLs 蛋白N-端具有豆蔻酰化(myristoylation)和棕榈酰化(palmitoylation)两个脂质修饰位点,介导CBLs 蛋白在胞内的定位。CBLs 互作蛋白激酶(CBL-interacting protein kinase,CIPKs)是一类植物特有的丝氨酸-苏氨酸蛋白激酶,其蛋白的C 端含有天冬酰胺-丙氨酸-苯丙酰胺(NAF)结构域,介导CIPKs 蛋白与CBLs 蛋白的互作;N 端含有丝氨酸/苏氨酸蛋白激酶结构域,具有激酶催化活性[17]。目前研究发现,拟南芥(Arabidopsis)中存在10 个CBLs蛋白和26 个CIPKs 蛋白,栽培大豆(G.max)中存在7个CBLs 蛋白和13 个CIPKs 蛋白[18-20]。拟南芥中CBLs蛋白定位在质膜、液泡膜、细胞质及细胞核中[21-24],而由于CIPKs 蛋白缺少定位信号结构域,在正常状态下CIPKs 定位在细胞质和细胞核中,当CIPKs 与CBLs 蛋白互作形成CBL-CIPK 复合体会重新定位至质膜或液泡膜上[21,25]。
拟南芥AtCBL4 基因(也称salt-overly-sensitive 3,SOS3)可以响应多种逆境,包括盐碱、低钾、干旱和低温等胁迫。在胁迫条件下,植株胞内Ca2+积累,Ca2+可介导AtCBL4 蛋白与AtCIPK24 蛋白(也称SOS2)形成AtCBL4-AtCIPK24 复合物[26],AtCBL4 蛋白N 端豆蔻酰化介导AtCBL4-AtCIPK24 复合物移动到质膜上。AtCBL4-AtCIPK24 复合物可磷酸化质膜上的SOS1(salt-overly-sensitive 1)蛋白[27-28]。SOS1 是定位在质膜上的Na+/H+反向转运蛋白,在盐胁迫下,AtCIPK24可磷酸化SOS1 自抑制结构域中的丝氨酸残基,激活SOS1 的转运活性,导致Na+转运出细胞,这对高盐胁迫下保持细胞Na+平衡,维持细胞渗透压十分重要[29]。另外,AtCBL10 也可与AtCIPK24 互作,形成的AtCBL10-AtCIPK24 复合物移动到液泡膜上,激活AtNHX(vacuolar Na+/H+exchanger)蛋白活性。NHX是存在于液泡膜上的Na+/H+转运蛋白,激活后可使胞质中的Na+转运至液泡中,进一步降低细胞内的Na+浓度。液泡中积累的Na+也可以作为渗透压降低细胞水势,促进植物在盐胁迫下吸水[30]。由此可见,CBLCIPK 信号通路对植物盐胁迫的抗性十分重要[31-32]。
本研究前期对野生大豆材料进行耐盐碱鉴定,筛选到高耐盐碱野生大豆材料2010-12。为进一步揭示野生大豆耐盐碱机制,选择高耐盐碱野生大豆2010-12 和敏感型野生大豆2012-49 为试验材料,在盐碱胁迫处理后0(盐碱胁迫处理后立即取样)、3、10 h 进行转录组分析。鉴于CBL-CIPK 信号通路在植物耐盐碱胁迫中发挥重要作用,且野生大豆CBL-CIPK 信号通路对盐碱胁迫的响应机制尚不清楚,本研究在转录组分析基础上,进一步分析野生大豆中CBL-CIPK 信号通路的蛋白结构、亚细胞定位、磷酸化位点及盐碱胁迫下高耐/敏感野生大豆材料基因表达特点,旨在揭示CBL-CIPK 信号通路基因在野生大豆耐盐碱胁迫中的作用。
1 材料与方法
1.1 试验材料及方法
高耐盐碱野生大豆材料2010-12 和敏感野生大豆材料2012-49,由河北科技师范学院野生大豆课题组提供。
用小刀划破野生大豆种子泥浆膜后,播种于铺满营养土的培养钵中,上覆薄层蛭石。培养钵置于温室(25℃光16 h/15℃暗8 h)。待野生大豆长出真叶后,移栽至装有30 mL 霍格兰培养液的50 mL 离心管中,置于相同光照培养条件的培养箱中,湿度60%,5 d 后进行盐碱胁迫处理。处理液按NaCl ∶Na2CO3(无水)=1 ∶3 质量分数配制[33],调pH 值至8.0,浓度为100 mmol·L-1。处理0(浇灌处理液后立即取样)、3、10 h后分别取两种野生大豆材料的叶片1 g,迅速保存于液氮中,所有样品设3 次生物学重复。
1.2 测序数据来源
提取野生大豆叶片RNA,构建cDNA 文库,送至华大基因(北京)利用BGISEQ-500 平台进行转录组测序。利用Trimmomatic(v0.36)软件对原始数据进行过滤,HISAT2(v2.1.0)软件比对野生大豆参考基因序列。通过华大基因交互系统(https:/ /report.bgi.com/ps/login/login.html)筛选CBL 和CIPK 通路基因,并利用栽培大豆中CBL 和CIPK 通路基因序列与本地转录组数据进行BLAST 比对。
1.3 预测数据来源
通过NCBI(http:/ /www.ncbi.nlm.nih.gov/)获得CBLs和CIPKs基因的蛋白序列,利用MEME(http:/ /alternate.meme-suite.org/tools/meme)在线分析工具进行蛋白结构域预测。利用BUSCA (http:/ /busca.biocomp.unibo.it/)在线分析工具进行蛋白亚细胞定位预测。利用NetPhos 3.1 Server(http:/ /www.cbs.dtu.dk/services/NetPhos/)在线分析工具进行蛋白磷酸化位点预测。
1.4 数据分析
CBLs和CIPKs基因表达量采用FPKM(fregments per kilobase per million)法进行计算,取|log2FoldChange |>1.5 为组间差异显著。利用Heml(v 1.0.3.7)软件对取对数值(log2)的基因表达量进行聚类分析。利用MEGA7 软件对野生大豆和栽培大豆的CBL-CIPK基因进行系统发育分析。
2 结果与分析
2.1 野生大豆CBL-CIPK 通路基因鉴定
对高耐盐碱及敏感野生大豆材料盐碱胁迫处理0、3、10 h 后的转录组测序,结果发现,18 组样品(3 次生物学重复) 共鉴定819.52 M Clean Reads,包括122.91 Gb Clean Bases。通过与已公布的野生大豆(G.soja)基因组比对,鉴定到已知转录本48 875 个,新转录本32 331 个。新转录本中具有蛋白编码潜力的转录本25 036 个,新基因1 780 个。
鉴定到CBL-CIPK 信号通路基因共48 个,其中CBLs基因10 个,CIPKs基因38 个。对筛选到的基因进行KO 注释,发现9 个CBLs基因匹配到K06268(丝氨酸/苏氨酸蛋白磷酸酶2B 调节亚基,serine/threonine-protein phosphatase 2B regulatory subunit)中。30 个CIPKs基因匹配到K07198(5′-AMP 激活蛋白激酶,催化α 亚单位,5′-AMP-activated protein kinase,catalytic alpha subunit)中,14 个匹配到K12761(碳代谢解压蛋白激酶,carbon catabolite-derepressing protein kinase)中(附表1)。

附表1 转录组分析鉴定的野生大豆CBL-CIPK 信号通路基因表达量Enclose Table 1 Gene expression of wild soybean CBL-CIPK proteins indentified by trascriptome analysis
2.2 野生大豆CBL-CIPK 蛋白结构域预测
对野生大豆CBLs 蛋白结构域预测发现,10 个CBLs 蛋白均含有3 个EF 手结构(图1-A)。虽然EF手结构在10 个CBLs 蛋白氨基酸序列的位置不同,但3 个EF 手结构之间的排列及它们之间的距离均一致。分析EF1、EF2 和EF3 蛋白基序发现,10 个EF 手结构氨基酸序列保守程度较高,说明该结构域对CBLs 蛋白发挥功能较重要。
对野生大豆38 个CIPKs 蛋白结构域预测发现,32个CIPKs 蛋白含有ATP 结合结构域(ATP binding domain),36 个含有丝氨酸/苏氨酸蛋白激酶活性结构域(serine/threonine protein kinase active domain,ST),17 个含有天冬酰胺-丙氨酸-苯丙氨酸保守结构域(Asn-Ala-Phe 结构域,NAF 结构域)。从3 个结构域基序的分析结果可知(图1-B),ATP 结合结构域和丝氨酸/苏氨酸蛋白激酶活性结构域高度保守,说明这两个结构域对CIPKs 蛋白较重要。NAF 结构域两端不保守,只在Asn-Ala-Phe 氨基酸位点保守程度较高。除这3 个结构域外,一些CIPKs 蛋白还含有一些保守结构域,丝氨酸/苏氨酸蛋白激酶活性结构域两端的结构域,它们共同介导了CIPKs 的蛋白激酶活性。
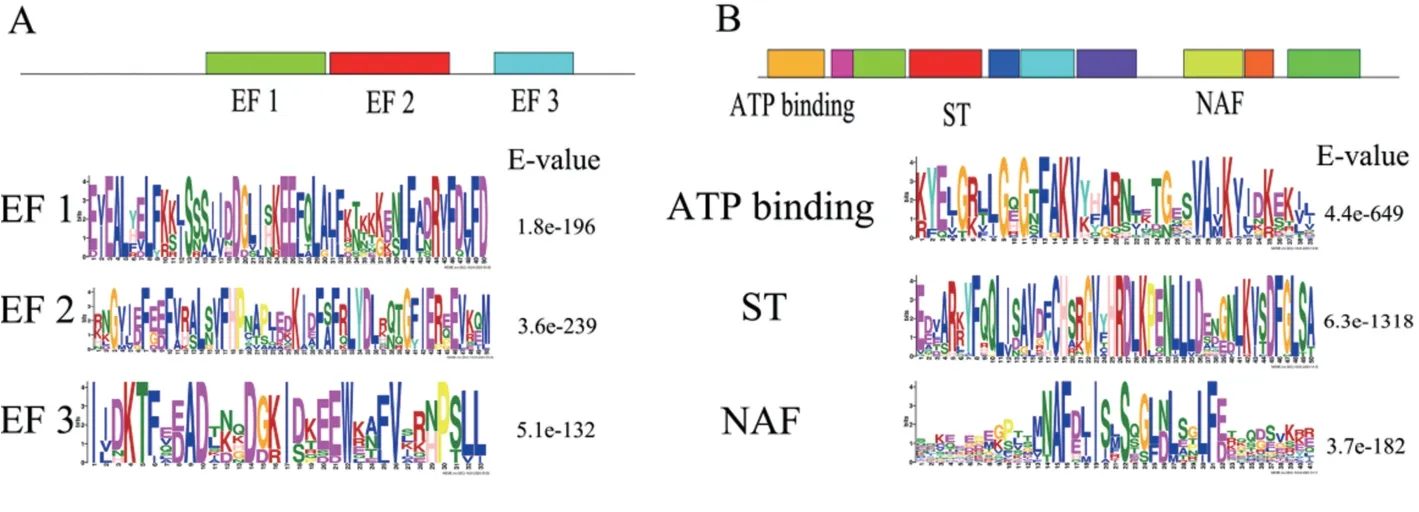
图1 野生大豆CBL-CIPK 蛋白结构域及重要结构域基序预测结果Fig.1 Prediction results of CBL-CIPK proteins domain and domain motif of wild soybean
2.3 野生大豆CBL-CIPK 蛋白亚细胞定位预测
对10 个CBLs 蛋白亚细胞定位预测发现,7 个CBLs蛋白位于细胞核中,1 个CBLs 蛋白位于细胞质中,1 个位于叶绿体中,1 个位于内膜系统上。对38 个CIPKs 蛋白亚细胞定位预测发现,20 个CIPKs 蛋白定位在细胞质中,占52.6%,9 个定位在叶绿体中,占23.6%,6 个定位在细胞核中,占15.7%,另有3 个分别位于细胞器膜、线粒体和内膜系统上,分别均占2.6%(表1)。
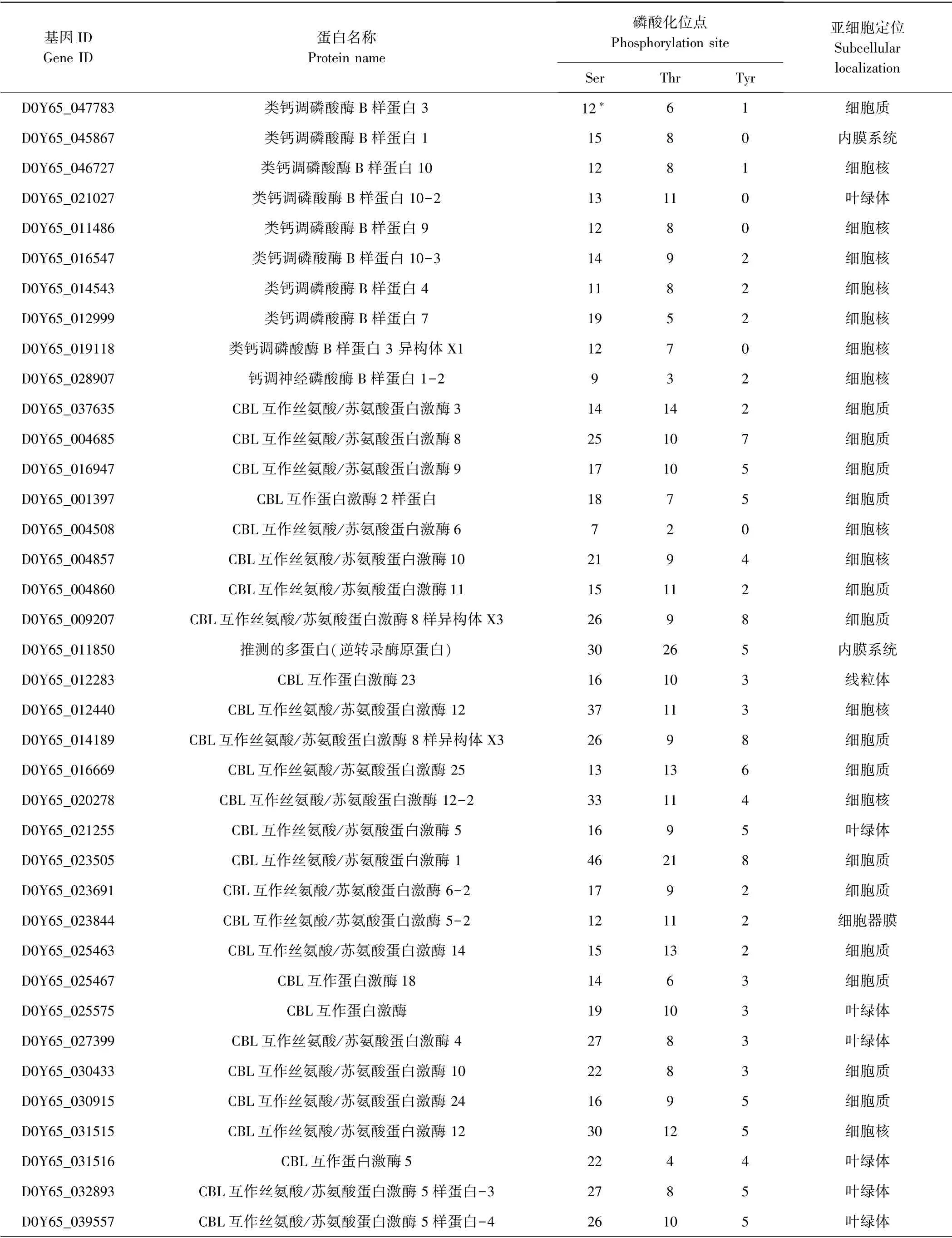
表1 CBL-CIPK 蛋白亚细胞定位及磷酸化位点预测结果Table 1 Subcellular localization and phosphorylation site prediction of CBL-CIPK proteins
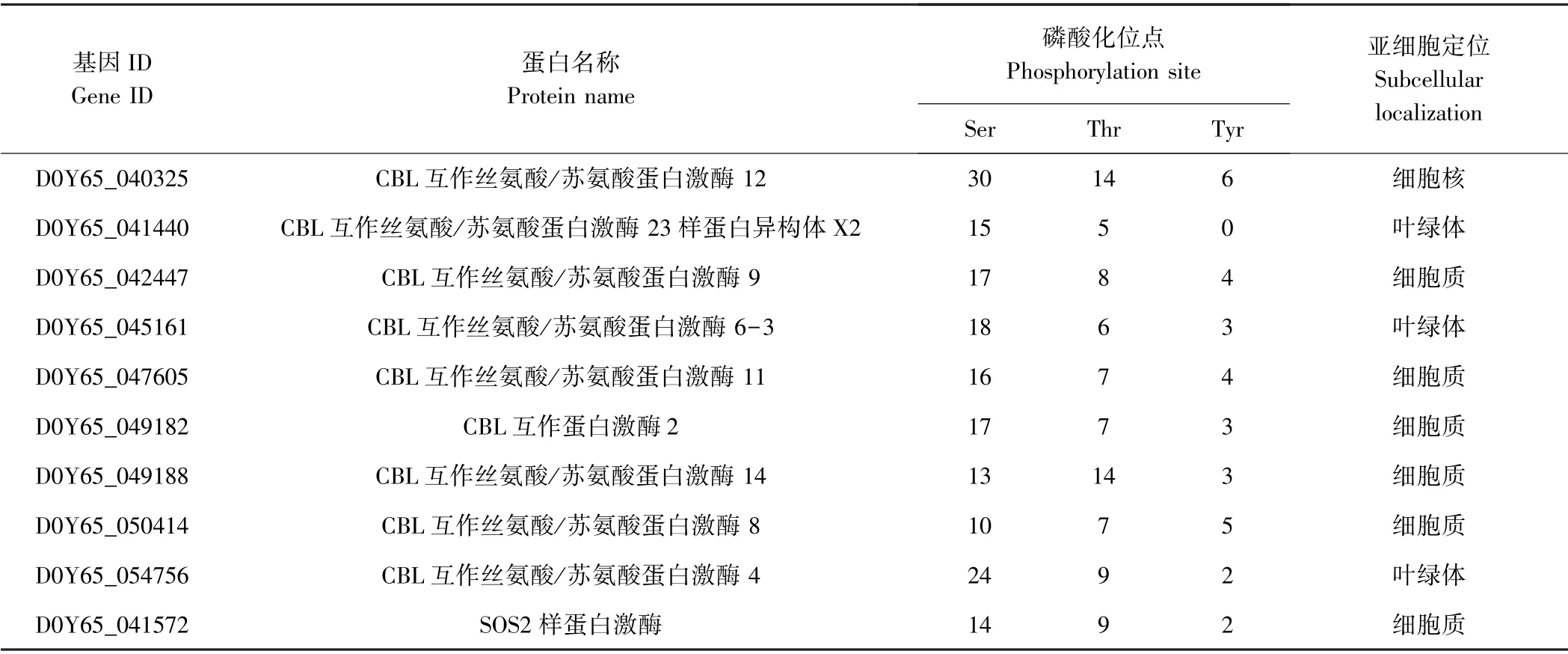
表1(续)
2.4 野生大豆CBL-CIPK 蛋白磷酸化位点预测
对CBL-CIPK 蛋白磷酸化位点预测发现,CBLs 蛋白磷酸化修饰以丝氨酸(Ser)位点最多,占总数的52.3%~73.1%,苏氨酸(Thr)位点次之,占19.2%~45.8%,酪氨酸(Tyr)位点最少,占0~14.3%。CIPKs蛋白磷酸化修饰也以丝氨酸(Ser)位点最多,占40.6%~77.8%,苏氨酸(Thr)位点次之,占13.3%~46.7%,酪氨酸(Tyr)位点最少,占0~22.7%(表1)。
10 个CBLs 蛋白平均每个蛋白发生12.0 次丝氨酸(Ser)位点磷酸化修饰,7.0 次苏氨酸(Thr)位点磷酸化修饰,0.8 次酪氨酸(Tyr)位点磷酸化修饰;38 个CIPKs 蛋白平均每个蛋白发生20.6 次丝氨酸(Ser)位点磷酸化修饰,9.9 次苏氨酸(Thr)位点磷酸化修饰,3.9 次酪氨酸(Tyr)位点磷酸化修饰(表1)。

图2 盐碱胁迫下野生大豆CBLs 蛋白(A)和CIPKs 蛋白(B)基因表达聚类分析Fig.2 The cluster analysis of gene expression of CBLs protein (A) and CIPKs protein (B) in wild soybean under saline-alkali stress
2.5 野生大豆盐碱胁迫下CBL-CIPK 基因表达模式分析
对10 个CBLs基因表达模式进行分析发现,高耐盐碱野生大豆材料GsCBL3(D0Y65_047783)基因在盐碱胁迫处理后3、10 h 的表达量与0 h 相比下调。高耐盐碱野生大豆材料GsCBL1 (D0Y65 _045867) 和GsCBL4(D0Y65_014543)基因在盐碱胁迫处理后10 h的表达量与0、3 h 相比均上调。两个耐性不同的野生大豆材料比较,在盐碱胁迫处理0、10 h 后,GsCBL10(D0Y65_046727)基因在高耐盐碱材料中的表达量比敏感材料上调,其余基因各时间点相比均无差别。
38 个CIPKs基因表达模式分析发现,两个耐性不同的野生大豆材料对比,在盐碱胁迫处理0 h 后,GsCIPK10_like(D0Y65_004857)基因在高耐盐碱野生大豆材料中的表达量比敏感材料上调,其余基因各时间点相比均无差别。
2.6 野生大豆与栽培大豆CBL-CIPK 基因系统发育分析
通过NCBI 检索到栽培大豆(G.max)CBLs基因6个,CIPKs基因15 个。利用MEGA7 软件分别分析野生大豆与栽培大豆CBL家族基因和CIPK家族基因的进化关系。结果发现,野生大豆与栽培大豆CBLs基因亲缘关系较近,野生大豆与栽培大豆的CBL1、CBL3、CBL4、CBL7、CBL10 分别聚为一类,证明其同源性很高。野生大豆与栽培大豆CIPKs基因也高度同源,野生大豆与栽培大豆的CIPK1、CIPK2、CIPK3、CIPK4、CIPK5、CIPK6、CIPK8、CIPK9、CIPK10、CIPK11、CIPK12、CIPK14和CIPK23 分别聚为一类,证明这些基因同源关系很高。但野生大豆GsCIPK5_like 基因与栽培大豆GmCIPK5 亲缘关系较远,而与栽培大豆GmCIPK25 基因亲缘关系较近。栽培大豆GmCIPK21 基因与野生大豆GsCIPK21 亲缘关系较远,而与野生大豆GsCIPK1_like 基因亲缘关系较近(图3)。
3 讨论
Meador 等[34]和Sánchez-Barrena 等[35]指出,拟南芥中CBLs 蛋白含有4 个EF 手结构,这些EF 手结构是CBLs 蛋白结合Ca2+的位置,对其发挥正常功能十分重要。蛋白结构域预测结果表明,野生大豆10 个CBLs 通路蛋白均含有3 个EF 手结构。通过比较拟南芥与野生大豆的EF 手结构,发现拟南芥和野生大豆的EF1 结构相同,野生大豆EF2 序列较长,包括了拟南芥EF2 和EF3 的序列,野生大豆EF3 与拟南芥EF4结构一致。曹齐卫等[36]对黄瓜(Cucumis sativus)CBLs蛋白结构分析也发现其含有3 个EF 手结构。说明野生大豆可能与黄瓜CBLs 蛋白结构相似。CIPKs 蛋白催化结构域有许多保守的区域,其中最重要的是ATP结合结构域和丝氨酸/苏氨酸蛋白激酶活性结构域[37]。这两个结构域分别具有丝氨酸/苏氨酸激酶特异性和酪氨酸激酶的特异性。除此之外,CIPKs 蛋白的NAF 结构域由25 个氨基酸构成,由其保守的天冬酰胺-丙氨酸-苯丙氨酸(Asn-Ala-Phe)残基命名,其功能是与CBLs 蛋白相互作用[21]。本研究通过对野生大豆的CIPKs 蛋白结构域基序分析发现,CIPKs 蛋白保守度最高的结构域为丝氨酸/苏氨酸蛋白激酶活性结构域,其次为ATP 结合结构域,NAF 结构域保守度最低,这与拟南芥中CIPKs 蛋白结构基本一致。
CBLs 蛋白在细胞内的位置受蛋白N-端影响。拟南芥中AtCBL1、AtCBL4、AtCBL5 和AtCBL9 蛋白N-端均含有一小段定位于质膜的N-豆蔻酰化结构域,AtCBL2、AtCBL3、AtCBL6 和AtCBL10 蛋白含有一段延长的N-端结构域,定位在液泡膜上。AtCBL7 和AtCBL8 蛋白由于其N-端结构域突变而定位在细胞质和细胞核中[38]。另外,Oh 等[23]研究指出AtCBL3 定位于细胞质中。由于CIPKs 不含有胞内定位信号,当其独立存在时可能定位在细胞核、细胞质和质膜上。当CIPKs 与CBLs 互作形成CBL-CIPK 复合体后,CIPKs 的胞内定位则由CBLs 蛋白决定[21,25]。如AtCBL4-AtCIPK24 复合物定位于质膜,AtCBL2-AtCIPK1 定位于液泡膜[32]。本研究通过对野生大豆CBL-CIPK 蛋白的亚细胞定位预测发现,GsCBL1-2、GsCBL3 isoform X1、GsCBL4、GsCBL7、GsCBL9、GsCBL10 和GsCBL10-3 定位于细胞核中,这是野生大豆与拟南芥CBLs 蛋白胞内定位的主要差异,代表了野生大豆的特异性。GsCBL3 定位于细胞质中,与拟南芥中AtCBL3 的胞内定位一致。但这仅是蛋白亚细胞定位的预测结果,野生大豆CBL-CIPK 蛋白在胞内的定位还需试验证实。
对高耐盐碱和敏感野生大豆材料在盐碱胁迫下CBL-CIPK基因转录水平进行检测,重点分析胁迫后同一时间点两材料间转录水平差异的基因。结果发现,两个野生大豆材料差异表达基因全部集中在胁迫处理后0 h,且高耐盐碱野生大豆材料中GsCBL10 和GsCIPK10 基因转录水平在胁迫处理后0 h 与敏感材料相比均增加。由于0 h 是胁迫处理后立即取样检测,故推测耐性野生大豆在受到盐碱胁迫后CBL-CIPK基因响应十分迅速。余义和等[39]研究了葡萄(Vitis vinifera)在干旱、低温和盐胁迫下VvCIPK10 基因的表达量,发现VvCIPK10 受到干旱、低温和盐胁迫诱导表达,并且干旱和盐胁迫后2 h 基因表达量即达到峰值。Kim 等[40]研究发现,拟南芥的 AtCBL10 可与AtCIPK24 直接互作并转移至液泡膜上,激活液泡膜上AtNHX1(液泡Na+/H+逆转运子)的活性,可介导胞质中的Na+转运至液泡中,这对增强拟南芥在盐胁迫下的耐性有重要作用。高耐盐碱野生大豆材料中GsCBL10 和GsCIPK10 的上调表达可能受到盐碱胁迫的诱导,这可能与野生大豆对盐碱胁迫的抗性有关,但需要进一步试验证明。
通过对野生大豆与栽培大豆CBL-CIPK基因系统发育分析,发现野生大豆与栽培大豆的CBL-CIPK基因亲缘关系较近。但是,野生大豆与栽培大豆在进化上也存在差异,如野生大豆GsCIPK5_like 基因和栽培大豆GmCIPK5 亲缘关系较远,栽培大豆GmCIPK21 基因与野生大豆GsCIPK21 亲缘关系较远。值得注意的是,转录组分析表明,高耐盐碱野生大豆中GsCIPK5_like 基因在盐碱胁迫后0 h 表达量高达109.88,而敏感材料在同时间点的表达量只有37.12,说明野生大豆GsCIPK5_like 基因受盐碱胁迫的诱导,其在胁迫后表达量迅速上调可能对野生大豆耐盐碱胁迫响应较重要。
4 结论
本研究在野生大豆盐碱胁迫处理转录组分析基础上,鉴定到10 个野生大豆CBLs基因和38 个CIPKs基因。野生大豆CBLs 蛋白均含有3 个EF 手结构,CIPKs 蛋白含有ATP 结合结构域、丝氨酸/苏氨酸蛋白激酶活性结构域和NAF 结构域。CBLs 蛋白主要定位于细胞核中,CIPKs 蛋白定位在细胞质、叶绿体、细胞核、细胞器膜上等。耐性野生大豆CBL-CIPK 通路基因响应盐碱胁迫主要发生在胁迫处理后0 h,推测野生大豆CBL-CIPK基因响应盐碱胁迫十分迅速。野生大豆与栽培大豆CBL-CIPK 通路基因亲缘关系很近,但存在个别亲缘关系较远的基因。

附表1(续)

