基于BSR-Seq对甘蔗脱叶性的研究和生物信息学分析
岳趣,商贺阳,张聘,陈保善,黄有总


摘 要:甘蔗的葉片是制糖过程中杂质的主要来源,育种过程中倾向于选育易脱叶的品种。为了研究甘蔗脱叶性相关的主效、稳定基因,本研究采用BSR-Seq技术,从‘ROC25ב云蔗89-7的F1后代中选取极端难脱叶品系和极端易脱叶品系的+6叶位叶鞘离区,分别构建混池,并和亲本一起进行RNA转录组测序分析,共得到60.46 Gb的有效片段,4个样品的Q30值均在93%以上,GC含量都大于51%,与参考基因组R570比对,比对率都超过41%。4个样品共检测出4085个差异基因,候选区域定位于9号染色体上的4个位置,总长1.40 Mb,注释到基因86个,亲本间注释到的非同义突变15个,挖掘出2个主效差异基因(Sh09_g020620,Sh09_g020080),Sh09_g020080在亲本的表达差异和子代中是同向的,而Sh09_g020620在2个亲本和2个子代混池之间的差异是异向的,表明可能仅Sh09_g020080对易脱叶性有所贡献。选取BSR亚群体中极端易脱叶品系40-159和极端难脱叶品系5-94的+1~+7叶进行荧光定量分析,发现Sh09_g020080基因在单一品系的表达和子代混池的+6叶的表达是同向的。取另一个‘B35-9בCP08-1553的杂交后代个体中的极端易脱叶品系16-226和极端难脱叶品系16-224的+1~+7叶位的叶鞘离区进行荧光定量分析,结果显示Sh09_g020080在2个群体中+4、+5、+6叶位的表达都为易脱叶的品系高于难脱叶品系。注释显示Sh09_g020080具有σ因子活性,参与红光、远红光、蓝光反应。根据理化性质和蛋白质结构,推测Sh09_g020080编码蛋白具有4个保守区域,且具有积累叶绿素的作用。该研究为甘蔗脱叶相关主效候选基因的挖掘提出了新的见解。
关键词:甘蔗;BSR-seq;脱叶;生物信息学分析
中图分类号:S566.1 文献标识码:A
Bioinformatic Analysis and Identification of Genes Contributing to Sugarcane Defoliation via BSR-Seq
YUE Qu1, SHANG Heyang1, ZHANG Pin2, CHEN Baoshan1,3*, HUANG Youzong1,3*
1. College of Agriculture, Guangxi University / State Key Laboratory of Conservation and Utilization of Subtropical Aro-biores?ou-rces, Nanning, Guangxi 530005, China; 2. College of Agronomy, Hebei Agricultural University, Baoding, Hebei 071001, China; 3. Agri-a?n?imal Industrial Development Institute, Guangxi University / Guangxi Key Laboratory of Sugarcane Biology, Nanning, Guangxi 530005, China
Abstract: Sugarcane (Saccharam spp.), a C4 tropical grass mainly for sugar production, is commercially cultivated in over 100 countries. One of the main barriers to mechanical or manual harvesting of whole sugarcane stalk is the high percentage of impurities, mainly the debris of leaves and sheathes that results in increase of harvest time and cost. The most efficient solution for this issue is to develop sugarcane cultivars that are easy to defoliate in sugarcane breeding program. In order to investigate the genes responsible to sugarcane defoliation, BSR-Seq technology was employed in the current study to analyze pools of the leaves at +6 position of extremely hard-to-defoliate and easy-to-defoliate sugarcane plants derived from the cross of ‘ROC25 × ‘Yunzhe 89-7, together with the two parental lines with contrast defoliation phenotypes. A total of 60 Gb of clean reads were obtained and the Q30 values of the four samples were greater than 93% with GC content of 51%. Using R570 genome as reference, the alignment rate was greater than 41%. A total of 4085 differential expressed genes were detected in the 4 samples. The candidate regions were found to locate at 4 loci on chromosome 9, covering a total length of 1.40 Mb in which 86 genes were annotated. There were 15 non-synonymous mutations annotated between the parental lines, and two significant differentially expressed genes (Sh09_g020620, Sh09_g020080) were identified. The expression level of Sh09 g020080 in the easy-to-defoliate genotype was higher than that of the hard-to-defoliate genotype and consistent with the parents but that of Sh09_g020620 was opposite between the offspring pools and the parental lines, suggesting that only Sh09 g020080 may contributing to the phenotype of easy defoliation. Quantitation of Sh09 g020080 gene expression in leaves from position +1 to +7 from the extremely easy-to-defoliate representative accession 40-159 and the extremely hard-to-defoliate accession 5-94 confirmed the BSR-seq results of the two pools. In analysis of another genetic population derived from the cross of ‘B35-9 × ‘CP08-1553, Sh09 g020080 was also found to express in a higher level in the extremely easy-to-defoliate accession 16-226 than in the extremely hard-to-defoliate accession 16-224 in +4 to +6 leaves. Analysis of the deduced Sh09_g020080 protein revealed that this protein has four domains conserved in σ factor-like proteins that may function in in red, far red and blue light reactions and accumulation of chlorophyll. Overall, this study shed a new light into the endeavor of exploring essential genes for defoliation in sugarcane.
Keywords: sugarcane; BSR-Seq; defoliation;
DOI: 10.3969/j.issn.1000-2561.2021.10.013
甘蔗主要分布于热带和亚热带地区[1],在我国主要分布于广西、云南、广东、海南等省(区),是我国主要的糖料作物。脱叶是甘蔗本身所具有的不同于一般落叶现象的性状,难脱叶的甘蔗在制糖过程中会含有大量杂质,对蔗糖的加工造成极大的干扰,影响制糖[2-3],我国糖厂规定甘蔗人工收获的含杂率不能超过1%,而对于机械收获则通常扣杂较高,可见,难脱叶的甘蔗极大影响甘蔗机械收获的质量和人工收获的效率。国外有在成熟期焚烧甘蔗老叶以助脱叶的处理方法,但会产生有毒气体,污染环境[4],同时会影响土壤微生物的活动,对土壤肥力产生不良影响[5-6];另一方面,甘蔗在成熟期叶片的脱落不但不影响产量,适当剥叶还可以提高产量和品质[7]。因此,在甘蔗新品种选育过程中把成熟叶片易脱落的性状作为选育的重要指标具有重要的生产意义。
甘蔗脱叶性状是数量性状,受多基因控制,植物脱落发生于离区[8],对于甘蔗而言,就是叶鞘和茎秆连接的部位。一般认为脱落的过程分成4个阶段:(1)离区的形成;(2)感受到脱落信号;(3)脱落信号的传导;(4)脱落后组织愈合。这4个过程都需要特定基因的表达,其中第2、3阶段在其他植物的叶、花、果等部位的脱落上有不少的研究[9-10]。集群分离分析(bulk segregant analysis, BSA)是QTL技术的一个分支,可以快速地对数量性状的主效基因进行定位,转录组学是研究基因的转录水平以及调控机制规律的技术,Bulked segrant RNA-Seq(BSR-Seq)将BSA技术和转录组学结合在一起,已经应用于小麦抗条锈病基因[11],玉米黄花突变基因的定位[12],大白菜黄化突变基因Brpem2的定位[13],猕猴桃抗寒机制的研究[14],小麦抗白粉病基因PmSESY的定位[15]等。
Li等[16]利用RNA-Seq技术发现了甘蔗脱叶相关基因在植物-病原菌相互作用、应激反应及与脱落酸相关通路中富集,关于甘蔗脱叶方面的研究报道还很少。有报道将甘蔗的脱叶性状分为三级,研究了不同组合选配方式的脱叶遗传情况,发现三级脱叶更易遗传给后代,其次是一级脱叶,最后是二级脱叶,也就是说中间类型的脱叶性状不容易稳定遗传[17]。本研究基于BSR-Seq技术对甘蔗脱叶相关的主效、稳定遗传的候选基因进行了筛选,并利用荧光定量分析对极端材料的+ 1~+7叶叶鞘离区部位进行了不同群体的分析,验证基因是否在不同群体中都具有稳定遗传的效应。通过对稳定遗传基因的生物信息学分析了解该基因的特点,为甘蔗脱叶的分子研究提供理论基础。
1 材料与方法
1.1 材料
甘蔗亲本(‘ROC25ב云蔗89-7)及其杂交后代F1(17 000多个品系)种植于广西大学扶绥基地,‘ROC25表现出相对难脱叶的特点,‘云蔗89-7则表现出相对易脱叶的特点,从F1中挑选极端难脱叶个体(叶鞘与茎秆包裹紧密,只有基部3~4片叶脱落,其他成熟叶鞘难以从茎秆上剥离)113个,易脱叶个体(+7~+10叶位以下全部脱落)116个,取其+6叶叶鞘离区暂时保存于液氮,带回实验室-80 ℃保存,同时将取样品系扩繁,观察1 a,最后筛选出难脱叶品系43个,易脱叶品系48个,选取其中的极端难脱叶品系5-94和极端易脱叶品系40-159的+1~+7叶位的叶鞘保存。取另一杂交群体(‘35-9בCP08-1553)的杂交后代个体中的极端难脱叶品系16-224和极端易脱叶品系16-226的+1~+7叶位的叶鞘用于验证。
主要试剂:RNA-easyTM Isolation Reagent Vazyme Cat(南京诺维赞生物科技股份有限公司),反转录试劑盒TransScript? One-Step gDNA Removal and cDNA Synthesis SuperMix(全式金生物技术有限公司),荧光定量试剂盒ChamQTM Universal SYBR qPCR Master Mix(南京诺维赞生物科技股份有限公司)。
1.2 方法
1.2.1 BSR-Seq建池 RNA提取采用TriZol法,提取后的RNA用Thermo的微量核酸纯度浓度测定仪检测浓度和纯度,利用琼脂糖凝胶电泳检测RNA完整性,各项指标合格后,将易脱叶的43个样品等量混合构建混池T03,难脱叶的48个样品等量混合构建混池T04,母本‘ROC25单个样品存在记为T01,父本‘云蔗89-7单独存在记做T02,最后送北京百迈客生物科技有限公司进行上机分析。
1.2.2 文库构建、组装和质控 利用DNA酶去除RNA样品中的DNA,用带有Oligo(dT)的磁珠富集mRNA,再将其打断成小片段,以小片段为模板合成单链cDNA,将单链扩增为双链,然后对合成的双链进行纯化和修复,对其加A尾,并在两端连接测序接头序列,经过片段大小选择和PCR扩增程序后构建文库[18-19]。用Illunima HiSeq测序平台对原始数据进行分析,去掉接头,过滤低质量的reads,剔除核糖体RNA[20]。
1.2.3 与参考基因组比对和候选区域定位 以甘蔗栽培品种‘R570基因组为参考,利用STAR将过滤后的片段比对到‘R570基因组上,将样品基因组组装到染色体水平。使用GATK[20]软件工具包对样品与参考基因组间及样品之间的单核苷酸多态性(SNP)进行检测,只有比对到参考基因组的唯一unigene才用于SNP挖掘。依靠欧式距离(Euclidean distance, ED)和SNP-index关联分析的方法分别筛选脱叶性状关联的区域,取2种关联方法的交集定位脱叶候选区域。
1.2.4 基因表达量和差异表达 基于负二项分布的基因模型,使用Cufflinks软件的Cuffquant和Cuffnorm组件,对基因和转录本的表达水平进行定量,由于抽取的转录本片段数目与测序数据量,转录本长度,转录表达水平有关,因此需要采用FPKM进行归一化[21],FPKM的计算公式为:
公式中,cDNA Framents表示双端reads数目;Mapped Fragments表示mapped reads总数;Transcript Length表示转录本长度。
借助软件DEGseq检测差异表达基因,以q-value < 0.005且log2 Fold Change > 1作为筛选标准,筛选各个样品中的差异表达基因[22],通过聚类热图进行差异表达基因的可视化分析。利用COG、GO、KEGG、KOG、NR、Pfam等注释数据库,对样本得到的各类基因进行注释。
1.2.5 引物设计和qPCR 荧光定量引物的设计利用Primer Premier 5完成,使用TriZol法提取甘蔗叶鞘RNA。通过qRT-PCR技术检测不同甘蔗叶位的叶鞘中基因的相对表达水平,qRT-PCR反应溶液的配置参照定量试剂盒的说明书操作[23]。以25s基因作为内参,采用两步法在LightCycler?96仪器上运行,为了提高扩增效率,程序设为:95 ℃,30 s;95 ℃,10 s;60 ℃,60 s。记录CT值,以2?ΔΔCT表示样品中基因的表达量。
1.2.6 候选基因的生物信息学分析 使用ORF finder(https://www.ncbi.nlm.nih.gov/orffinder/)对甘蔗脱叶相关的候选序列进行编码区查找,通过NCBI找到与该基因亲缘关系相近的其他物种的同类基因,利用MEGA-X构建进化树,并利用在线工具ITOL(https://itol.embl.de/)对构建的进化树进行处理;运用在线工具ExPASy(https://web. expasy.org/protparam/)分析其理化性质比如相对分子质量、等电点、脂肪族化、不稳定系数等指标;使用在线软件SOPMA(https://npsa-prabi. ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)分析其二级结构、SWISS-MODELR(https:// swissmodel.expasy.org/interactive)分析三级结构、SignalP(http://www.cbs.dtu.dk/services/ SignalP/)分析蛋白的信号肽、Server(http://www. cbs.dtu. dk/services/TMHMM/)预测跨膜结构、Plant-mPloc(http://www.csbio.sjtu.edu.cn/bioinf/ plant-multi/)预测编码蛋白的亚细胞定位情况。
2 结果与分析
2.1 组装和比对
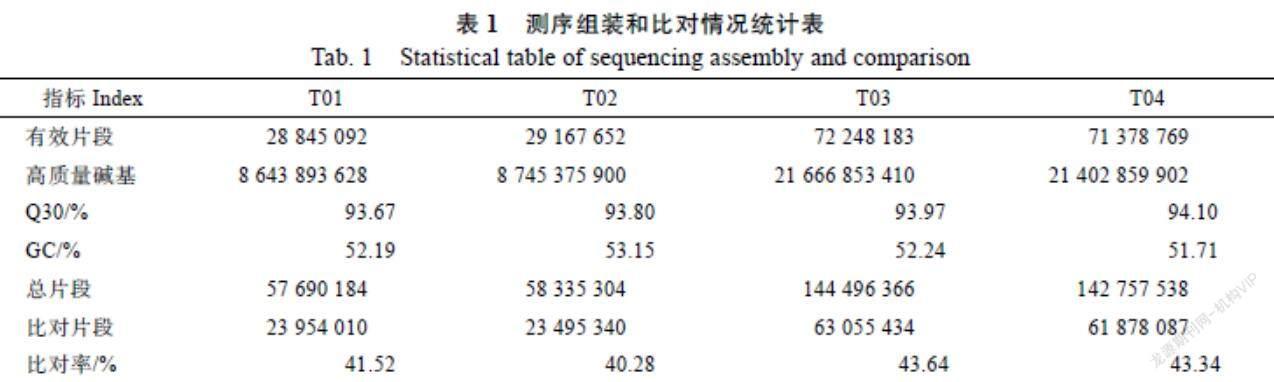
通过Illumina Hiseq 2000測序平台对4个样本进行测序,共产生60.46 Gb clean data,T01、T02、T03、T04的clean data分别为8.64、8.74、21.66、21.40 Gb,Q30值分别为93.67%、93.80%、93.97%、94.10%。GC含量分别为52.19%、53.15%、52.24%、51.71%,共产生的片段数为403 279 392。与参考基因组R570比对,比对上的片段比率分别为41.52%、40.28%、43.64%和43.34%(表1)。
2.2 差异基因表达和候选区域关联分析
对于所检测到的差异基因,样品T01 vs T02中,共有3223个差异基因,其中1621个基因上调,1602个基因下调。样品T03 vs T04中,共有842个差异基因,其中574个基因上调,268个基因下调。欧式距离关联分析得到的候选区域为14个,总长度为4.94 Mb,共包含328个基因,非同义突变SNP位点的基因42个。SNP-index关联分析共得到14个候选区域,总长度2.71 Mb,包含183个基因,非同义突变的基因20个。2种方法取交集得到4个与性状相关的侯选区域,总长度为1.40 Mb,候选区域内亲本之间非同义突变SNP共22个,混池间存在的非同义SNP共57个。候选区域共注释到基因86个,亲本间注释到的非同义突变15个。图1显示,在T01 vs T02差异基因、T03 vs T04差异基因,候选区间的交集中,有2个候选基因在亲本之间和子代混池中都表现出了差异,4个候选基因只在亲本间显示出了差异。
2.3 候选基因定量分析
在亲本之间和子代之间都存在差异的候选基因说明存在遗传效应,很可能与甘蔗脱叶有极其重要的关系。维恩图显示出了Sh09_g020080和Sh09_g020620这2个候选基因满足了各种筛选条件(图2),特别是Sh09_g020080,无论是亲本之间还是子代混池之间都表现出易脱叶的表达量高于难脱叶,很可能存在稳定的遗传差异。Sh09_g020080的注释结果显示,具有σ因子活性,参与红光、蓝光、远红光反应,Sh09_g020620具有半胱氨酸内肽酶的活性,参与蛋白质水解的过程。Sh09_g020080和Sh09_g020620荧光定量引物设计如表2。BSR亚群体中选取难易脱叶品系的+1~+7叶进行荧光定量分析,结果如图3A显示,在+3、+4叶位,易脱叶品系中Sh09_g020620的表达量高于难脱叶品系,而在+1、+2、+5、+6、+7叶位,难脱叶品系的Sh09_g020620表达量高于易脱叶品系,在+7叶位中达到最大值。而对于Sh09_g020080来说,如图3B显示,在+2、+7叶位中,易脱叶品系的表达量相对较高,而在+1、+3、+4、+5、+6叶位中,则易脱叶品系的表达量较高,考虑到Sh09_g020080可能存在稳定的遗传效应,因此选择遗传基础一致的其他亚群体进行定量验证,其结果如图3C,在+1、+2、+3叶位中,难脱叶品系的Sh09_g020080表达量高于易脱叶品系。在+4、+5、+6、+7叶位中,易脱叶品系的Sh09_g020080表达量高于难脱叶品系。综合考虑,Sh09_g020080在难、易脱叶品系中存在稳定的遗传差异,推测很可能在甘蔗脱叶过程中是起着重要的作用。
2.4 甘蔗脫叶相关候选基因的理化性质分析
进化树分析发现Sh09_g020080与编码高粱的σ因子的亲缘关系最近(图4),NCBI中显示,与XP_002440351.1、XP_021302430.1的一致性分别为85.53%和84.43%,可以通过与高粱的σ因子的特性相比较,了解甘蔗中该基因的特性和作用。利用软件分析发现Sh09_g020080 Unigene全长1866 bp,编码621个氨基酸,编码蛋白的相对分子质量为69 533.31 Da。该基因编码蛋白的理论等电点为10.01,据此可以判断为碱性蛋白质,不稳定指数显示为60.05,归为不稳定蛋白[24],平均疏水性为-0.665,为亲水蛋白。信号肽的分析发现有信号肽的可能性仅为0.0009,可以推断出蛋白不具备信号肽的功能,跨膜结构分析显示无跨膜结构,σ因子的作用是与RNA的转录有关,起到和核心酶启动子结合的作用[25],在甘蔗中可能会借助其他物质辅助。亚细胞定位显示该蛋白存在于叶绿体,这和注释结果的情况是一致的,该蛋白的作用是参与红光、远红光、蓝光反应,所在的位置决定了它的功能。二级结构分析发现,该蛋白具有α-螺旋、延伸链、β转角和无规则卷曲,比例分别为47.18%、12.24%、4.83%和35.75%。图5显示,三级结构显示有大量的α-螺旋结构组成一些单独的区域,中心部分也是以α-螺旋为主,这些部位很可能就是Sh09_g020080的保守区域。
3 讨论
无论是机械收获还是人工收获,成熟期甘蔗叶片的自然脱落都可以很大程度地减少成本,降低含杂率。BSA技术广泛使用于各种作物研究,如白菜头型[26]、向日葵耐盐性[27]等。BSR技术已经在小麦[11]、玉米[12]、白菜薹[28]、大白菜[29]等作物上得以应用。本研究利用BSR技术筛选出2个主效候选基因(Sh09_g020620,Sh09_g020080),其中Sh09_g020620在亲本之间的差异状况和子代混池中的差异是异向的,具体表现为:亲本中为易脱叶品种的表达量高于难脱叶,子代混池中则相反。选取BSR亚群体进行单一品系的荧光定量分析,结果显示+6叶位叶鞘的表达和子代混池一致,推测该基因的表达可能受环境或基因互作影响,在遗传过程中其表达量发生了变化。Sh09_g020080在子代混池之间和亲本内却表现出了同向的差异(易脱叶品系的表达量比难脱叶的高),选取BSR亚群体单一品系进行分析,与子代混池的差异状况是一致的。为了进一步验证Sh09_g020080是否具有不同群体的稳定差异遗传效应,选取‘B35-9בCP08-1553的杂交后代个体中的极端易脱叶品系16-226和极端难脱叶品系16-224进行荧光定量分析,整体的表达趋势为易脱叶品系的表达量高于难脱叶,和BSR群体中单一品系的不同叶位整体表达趋势一致。据此,可以初步判断出Sh09_g020080在难易脱叶品系中的差异是稳定存在的。
目前为止,所有研究的陆地植物和藻类的质体都编码了一个类似细菌RNA聚合酶核心亚基的同源物[30]。在真细菌中,必须存在σ因子识别RNA聚合酶核心酶的启动子[25]。候选基因Sh09_g020080的注释显示具有σ因子活性,进化树构建发现Sh09_g020080和高粱中编码σ因子的亲缘关系最近,报道称高粱σ因子的片段大小为2000 bp[30],与甘蔗中该基因(1866 bp)相差不大,高粱σ因子含有4个保守结构域,推测甘蔗σ因子三级结构预测的4个螺旋区域(3个尾部和中心区域)也可能是保守区域的位置。
Sh09_g020080参与红光、蓝光、远红光反应,σ因子是和光合作用相关的,σ因子具有介导叶绿素积累的作用[31],推测Sh09_g020080可能也具有积累叶绿素的作用。根据Sh09_g020080的定量结果,该基因在难易脱叶品系中的表达是有差异的,在易脱叶品系中的整体表达趋势要高于难脱叶,可以推测易脱叶品系有更强的光合作用,相对少量的叶片足够满足光合所需,因此,在成熟期易脱叶品系下部有较多的叶片自动脱落。由于关于甘蔗脱叶的研究较少,而且σ因子的研究大多针对菌类,所以只能推测Sh09_g020080和甘蔗脱叶相关。甘蔗脱叶性状复杂,受较多基因控制,通过本研究可以为甘蔗脱叶研究提供新的见解。
参考文献
[1] Que Y X, Pan Y B, Lu Y H, et al. Genetic analysis of diversity within a Chinese local sugarcane germplasm based on start codon targeted polymorphism[J]. BioMed Research International, 2014, 2014: 468375.
[2] Mazzoli-Rocha F, Magalh?es C B, Malm O, et al. Comparative respiratory toxicity of particles produced by traffic and sugar cane burning[J]. Environmental Research, 2008, 108(1): 35-41.
[3] Cristale J, Silva F S, Zocolo G J, et al. Influence of sugarcane burning on indoor/outdoor PAH air pollution in Brazil[J]. Environmental Pollution, 2012, 169: 210-216.
[4] Mugica-Alvarez V, Rosa S, Figueroa-Lara J, et al. Emissions of PAHs derived from sugarcane burning and processing in Chiapas and Morelos Mexico[J]. Science of the Total Environment, 2015(527/528): 474-482.
[5] Goldemberg J, Coelho S T, Guardabassi P. The sustainability of ethanol production from sugarcane[J]. Energy Policy, 2008, 36(6): 2086-2097.
[6] Souza R A, Telles T S, Machado W, et al. Effects of sugarcane harvesting with burning on the chemical and microbiological properties of the soil[J]. Agriculture Ecosystems & Environment, 2012, 155(2): 1-6.
[7] Gutiérrez-Miceli F A, Morales-Torres R, Espinosa-Casta-?e?da Y D J, et al. Effects of partial defoliation on sucrose accum?ulat?ion, enzyme activity and agronomic parameters in sug?ar cane (Saccharum spp.)[J]. Journal of Agronomy and Crop Science, 2010, 190(4): 256-261.
[8] Sexton R, Roberts J A. Cell biology of abscission[J]. Annual Review of Plant Physiology, 1982, 33(1): 133-162.
[9] Meir S, Philosophhadas S, Sundaresan S, et al. Microarray analysis of the abscission-related transcriptome in the tomato flower abscission zone in response to auxin depletion[J]. Plant Signaling & Behavior, 2011, 154(4): 1929-1956.
[10] Roberts J A, Elliott K A, Gonzalez-Carranza Z H. Abscission, dehiscence, and other cell separation processes[J]. Annual Review of Plant Biology, 2002, 53(1): 131-158.
[11] 李玉荣. BSR-Seq方法定位玉米黄化突变基因[D]. 武汉: 华中农业大学, 2014.
[12] 吴建辉. 基于BSR-Seq和芯片技术的抗条锈基因Yr26候选基因分析及普通小麦成株期抗条锈QTL定位[D]. 杨凌: 西北农林科技大学, 2017.
[13] 赵永慧. 大白菜黄化突变基因Brpem2的定位[D]. 沈阳: 沈阳农业大学, 2019.
[14] Lin M M, Sun S H, Fang J B, et al. BSR-Seq analysis provides insights into the cold stress response of Actinidia arguta F1 populations[J/OL]. BMC Genomics, 2021, 72(22), DOI:10.21203/rs.3.rs-76090/v1.
[15] He H G, Du H N, Liu R K, et al. Characterization of a new gene for resistance to wheat powdery mildew on chromosome 1RL of wild rye Secale sylvestre[J]. Theoretical and Applied Genetics, 2021, 134: 887-896.
[16] Li M, Liang Z X, Zeng Y, et al. De novo analysis of transcriptome reveals genes associated with leaf abscission in sugarcane (Saccharum officinarum L.)[J]. BMC Genomics, 2016, 17(1): 195.
[17] 夏红明, 赵培方, 赵 俊, 等. 甘蔗脱叶性状的遗传力和配合力研究[J]. 湖南农业大学学报(自然科学版), 2017, 43(3): 244-251.
[18] 吴姝青. 基于转录组与代谢组学分析MeJA介导海棠应答O3脅迫的防御机制[D]. 泰安: 山东农业大学, 2020.
[19] 张 震. 基于转录组学和代谢组学研究梨花青苷和原花青苷合成调控机制[D]. 泰安: 山东农业大学, 2020.
[20] 翟楠鑫, 迟 会, 夏玥琳, 等. 海南山栏稻抗旱基因转录组分析[J]. 生物技术通报, 2020, 36(12): 12-20.
[21] Mckenna A, Hanna M, Banks E, et al. The genome analysis Toolkit: a MapReduce framework for analyzing next-gene-ra?tion DNA sequencing data[J]. Genome Res, 2010, 20(9): 1297-1303.
[22] 林苗苗. 基于基因组重测序和BSR-Seq分析的猕猴桃抗寒机制研究[D]. 武汉: 华中农业大学, 2020.
[23] 李聰娜, 刘 峰, 张 旭, 等. 甘蔗类甜蛋白基因ScTLP2和ScTLP3的生物信息学分析及表达[J]. 分子植物育种, 2020, 18(1): 65-73.
[24] 李旭娟, 刘洪博, 林秀琴, 等. 甘蔗KNOX基因(Sckn1)的电子克隆及生物信息学分析[J]. 基因组学与应用生物学, 2015, 34(1): 136-142.
[25] Burgess R R, Travers A A, Dunn J J, et al. Factor stimulating transcription by RNA polymerase[J]. Nature, 1969, 221 (5175): 43-46.
[26] Gu A X, Meng C, Chen Y Q, et al. Coupling Seq-BSA and RNA-Seq analyses reveal the molecular pathway and genes associated with leaf head pattern formation at top region in Chinese cabbage[J]. Frontiers in Genetics, 2017, 8: 176.
[27] 贾秀苹, 卯旭辉, 岳 云, 等. 利用BSA-Seq方法鉴定向日葵耐盐候选基因[J]. 中国油料作物学报, 2018, 40(6): 777-784.
[28] 张文翔. 白菜薹细胞核雄性不育基因的定位[D]. 沈阳: 沈阳农业大学, 2019.
[29] 张亚宁. 大白菜叶面刺毛突变基因Brtrm的精细定位[D]. 沈阳: 沈阳农业大学, 2020.
[30] Kroll D, Streubel M, Westhoff P. A plastid sigma factor sequence from the C4 monocot Sorghum bicolor[J]. Plant Biology, 1999, 1(2): 180-186.
[31] Alameldin H F, Oh S, Hernandez A P, et al. Nuclear - encoded sigma factor 6 (SIG6) is involved in the block of greening response in Arabidopsis thaliana[J]. American Journal of Botany, 2020, 107(2): 329-338.
责任编辑:谢龙莲

