GmNTLs 调控大豆根系响应低磷胁迫的功能研究
李雅雪 盘耀亮 彭光粉 田江 陆星 梁翠月


摘要: 【目的】低磷和铝毒胁迫是酸性土壤中限制作物生产的重要因素。植物NTL 转录因子参与调控多种环境胁迫(包括铝毒胁迫) 的适应性机制,本文探究GmNTLs 调控大豆Glycine max 根系响应低磷胁迫的功能。【方法】通过RT-qPCR 分析大豆15 个GmNTLs 基因在根系响应低磷胁迫的表达模式,进一步构建了GmNTL1/4/7/8/10/12 共6 个GmNTLs 基因的拟南芥超量表达材料,探究GmNTL 成员在拟南芥根系中响应低磷胁迫的功能。【结果】系统进化树及组织表达模式分析结果表明,GmNTLs 家族分3 个亚族,各亚族成员在大豆中组织表达模式不同。RT-qPCR 结果表明,低磷处理12 d 显著提高了GmNTL1/4/7/8/10/12 在大豆根系中的表达。在拟南芥中超量表达不同GmNTL 基因对低磷的响应不同。高磷处理下,超量表达GmNTL4/10/12 拟南芥的鲜质量显著增加;低磷处理时,超量表达GmNTL4显著提高拟南芥鲜质量,而超量表达GmNTL1/12 拟南芥的鲜质量显著降低。同时,仅超量表达GmNTL12拟南芥的主根长显著缩短,而超量表达其他基因对拟南芥植株的主根长无明显影响。【结论】GmNTLs 参与大豆根系对低磷胁迫的响应,该结果可为培育磷高效型大豆品种提供数据支持。
关键词: 大豆;根系;低磷胁迫;GmNTLs;NTL 转录因子
中图分类号: Q945.78;S529文献标志码: A 文章编号: 1001-411X(2023)02-0221-09
NAC(NAM、ATAF1/2、CUC2) 是与植物生长发育和胁迫相关的特异性转录因子[1]。NTL(NACwith transmembrane motif like) 则是蛋白C 端带有跨膜结构域的一类NAC 转录因子[ 2 ]。研究显示,NTL 家族成员在调控生物、非生物胁迫应答和植物发育过程中发挥重要作用[3]。目前多个NTL 的功能被报道,例如,超量表达拟南芥ANAC017/AtNTL7增强了转基因植株对内质网胁迫的耐受性[4];在拟南芥中超量表达ANAC005 抑制了与木质素、形成层、初生壁等生物合成相关的基因的表达,造成植株矮化[ 5 ];在拟南芥中异源表达玉米ZmNTL1、ZmNTL2 和ZmNTL5,可以降低转基因拟南芥对H2O2 的敏感性[6];超量表达AtNTL6 增强转基因拟南芥植株的抗旱能力[7];AtNTL8 在盐胁迫下调控赤霉素(GA) 途径促使种子萌发,同时调控拟南芥的开花时间[8];AtNTL6 受冷胁迫,诱导PR1、PR2 和PR5 等相关抗病基因的表达[9]。
我国南方地区土壤多呈酸性。低磷和铝毒是酸性土壤中长期共存的营养逆境因子,植物为了适应酸性土壤的缺磷和铝毒胁迫,进化出各种协同调控和应答机制,如分泌有机酸、改变根系形态构型等[10-14]。大豆Glycine max 是重要的粮油兼用作物,我国大豆产量不足以满足国内需求,需依赖进口[15]。南方酸性土壤中存在低磷、铝毒胁迫从而限制了大豆的生产[ 1 6 ]。在大豆基因组中鉴定到1 5 个GmNTL 同源基因并对其进行了功能分析,结果表明,超量表达GmNTL1、GmNTL7 等显著提高转基因拟南芥对盐害和干旱的耐受能力[17-19]。前期研究结果表明,GmNTLs 参与了大豆耐铝毒胁迫,超量表达GmNTL4 可能通过调控外排有机酸或细胞壁修饰相关基因来提高转基因拟南芥对铝的耐受性[ 2 0 ]。进一步比较分析野生型(WT) 和超量表达GmNTL4 转基因拟南芥根系的转录组数据,发现一些与根系生长相关的基因,包括RGF2、Extensin、Auxin-responsive protein 等,均受GmNTL4 调控,而一些磷平衡相关基因,包括紫色酸性磷酸酶基因P A P 2 0 ( A t 3 g 5 2 7 8 0 )、酸性磷酸酶基因V S P 1(At5g24780) 和VSP2 (At5g24770) 等,也受GmNTL4调控,但未发现磷转运子相关基因受GmNTL4 调控[20]。据此,我们猜测GmNTLs 可能参与了大豆生长对低磷的适应性机制。因此,本研究首先对大豆基因组中15 个GmNTL 成员进行系统进化树和组织部位表达模式分析,然后利用RT-qPCR 测定该家族成员响应低磷胁迫的表达模式,进一步构建GmNTLs 超量表达转基因拟南芥株系,对其鲜质量和总根长进行分析。
1 材料与方法
1.1 材料
本试验采用的大豆材料为磷高效品种‘粤春03-3(YC03-3);拟南芥材料为哥伦比亚野生型(Col-0)。1.2 大豆GmNTLs 基因家族进化及表达模式预测分析在Phytozome 网站(https://phytozome.jgi.doe.gov/pz/portal.html) 利用BLAST 搜索引擎查找G m N T L s 家族氨基酸序列。使用软件M E G A 7(http://http//www.mega software.net) 以鄰接法构建进化树。利用S o y B a s e 数据库( h t t p s : / / w w w .soybase.org/soyseq/) 获得大豆GmNTLs 家族基因的组织表达数据。
1.3 大豆不同磷浓度处理
大豆磷处理试验在华南农业大学根系生物学研究中心的温室大棚中进行。用砂培萌发种子3~4 d,挑选长势均一的大豆幼苗转移至Hoagland 营养液中进行磷处理:250 μmol·L?1 KH2PO4(高磷) 和5 μmol·L?1 KH2PO4(低磷)。每个处理4 次重复,每次重复4 株苗。收取处理6、12 d 的大豆生理样品,测量主根长,称取鲜质量,同时收取根系样品用液氮速冻后保存在?80 ℃ 冰箱备用。设置5、50 和250 μmol·L?1 KH2PO4 3 个磷浓度,处理12 d,分析不同磷浓度下大豆G m N T L s 家族基因的表达模式。
1.4 植物RNA 的提取、反转录及RT-qPCR
用TRIzol(Omega,美国) 法提取大豆根系RNA;使用HiScript?1st Strand cDNA SynthesisKit 反转录试剂盒(Promega,北京),将其反转录为cDNA 单链。在NCBI 网站(https://www.ncbi.nlm.nih.gov/) 设计GmNTLs 基因及大豆看家基因GmEF1a 的定量PCR 引物(GmNTL1 的正向引物为GTCGTCGAGTGTTACCCACA,反向引物为CGTAGCTCGGTTCAACCTGT;GmNTL4 的正向引物为GCCGTTAGGGTTCCGTTTC,反向引物为GCTCCTTCAAAGTGGGACGATA;GmNTL7 的正向引物为CCCAAGTGCATCAGAAGGCT,反向引物为GCAGTGTCTTTACCTGCTGC;GmNTL8 的正向引物为CAATATGTGCGGAGGAAG,反向引物为G T G A T G A T G A G T A G T T G G A T T ;G m N T L 1 0 的正向引物为T T C T G C C C T A CCGATGAGGA,反向引物为TTTCGCTCTTTGC C A G T T G C ; G m N T L 1 2 的正向引物为CATGGATATTTGCTCACCCTGC,反向引物为AGGCTTGTGCCATTTTCAGC;GmEF1a 的正向引物为TGCAAAGGAGGCTGCTAACT,反向引物为CAGCATCACCGTTCTTCAAA),用实时荧光定量PCR 仪(Thermo Fisher,美国) 测定,相对表达量为目的基因表达量与看家基因表达量的比值。
1.5 大豆GmNTLs 家族基因克隆及重组载体构建
从Phytozome 数据库中获得大豆GmNTLs 家族中GmNTL1、GmNTL4、GmNTL7、GmNTL8、GmNTL10 和GmNTL12 基因的C DS 序列,在NCBI 网站设计其正反向引物,按照一步克隆法连接华南农业大学根系生物学研究中心保存的3 5 S : p T F 1 0 1S 载体。测序正确后,利用试剂盒Hipure Fungal DNA Mini Kit (Magen,广州) 抽取质粒,保存备用。
1.6 超量表达GmNTLs 转基因拟南芥的转化与筛选
将“1.5”获取的6 个GmNTLs 重组载体转化农杆菌GV3101(唯地,上海),使用花序侵染法进行拟南芥转化。获得T0 代拟南芥转基因种子后,取种子用5% (φ) 次氯酸钠溶液进行表面消毒,均匀种植在含0.025% (φ) 除草剂的拟南芥MS 固体萌发培养基中,放置在培养条件为日长12 h、光照4 000 lx、温度23 ℃、相对湿度75%,黑暗12 h、温度21 ℃、相对湿度75% 的人工气候箱(莱福仪器,宁波) 中生长14 d 左右。将正常存活且长出4 片叶子的T1 代存活幼苗移植到蛭石、基质质量比为1∶3 的混合基质中,在12 h 光照/12 h 黑暗条件下生长,待植株叶片较大时取样进行PCR 检测来筛选阳性转基因株系。分株收取阳性T1 代种子重复上述的筛选步骤,获得存活幼苗为T2 代,选取T2 代种子于平板筛选阳性和阴性植株分离比为3∶1 的株系进行T3 代纯合体的筛选。
1.7 转基因拟南芥低磷胁迫处理
将T3 代转基因植株种子和野生型拟南芥种子消毒后播种在MS 固体萌发培养基上,待根长至1 cm左右,挑选长势一致的幼苗移至1 250 μmol·L? 1KH2PO4 的高磷处理、12.5 μmol·L?1 KH2PO4 的低磷处理MS 培养基上,在“1.6”人工气候箱中处理12 d,收样,测定植株鲜质量和主根长。
1.8 数据统计及分析
所有试验数据通过M i c r o s o f t E x c e l2019(Microsoft,美国) 和IBM SPSS StatisticsV26.0(IBM SPSS,美国) 进行处理及统计分析。用TBtools 软件(http://www.tbtools.com/) 绘制表达模式热图。
2 结果与分析
2.1 GmNTLs 蛋白家族进化树及组织表达模式分析
对15 个GmNTLs 家族构建系统进化树,结果显示,GmNTLs 家族分为I、II 和III 3 个亚家族,7 个复制对。其中GmNTL2/12 和GmNTL8/13 2 个复制对属于第I 亚族;GmNTL6/15 和GmNTL7/142 个复制对属于第I I 亚族, G m N T L 1 / 1 1 、G m N T L 3 / 4 和G m N T L 9 / 1 0 3 个复制对及GmNTL5 属于第III 亚族(图1)。
利用SoyBase 数据库对GmNTLs 基因在不同组织部位的表达水平构建表达模式热图,(其中GmNTL5、GmNTL6 和GmNTL9 这3 个基因未被收录),结果如图1 所示。第I 亚家族成员在各组织部位中几乎不表达。第I I 亚族中G m N T L 7 和G m N T L 1 4 在各组织部位表达量都很低;GmNTL15 主要在根和根瘤中表達。第III 亚族中GmNTL1 在根中有较低表达量;GmNTL3 在新叶、根瘤、花后14 d 的荚中表达量较高;GmNTL4 主要在根瘤和花后14 d 的荚中表达,其中在根瘤中的表达量最高。
2.2 不同磷处理时间对大豆生长及GmNTLs 基因家族表达模式的调控
使用水培体系对大豆YC03-3 幼苗进行高磷( 2 5 0 μ m o l · L ? 1 K H 2 P O 4 ) 和低磷( 5 μ m o l · L ? 1KH2PO4) 处理6、12 d 后分别收样。结果显示,高、低磷处理6、12 d 后,高磷处理大豆的地上部生物量要明显高于低磷处理的;而低磷处理大豆的根系要比高磷处理的发达(图2、3)。具体表现为,处理6、12 d 后,与高磷处理相比,低磷处理的地上部干质量显著减少3.6% 和43%(图3A);同时,处理12 d后,低磷处理的根系干质量和总长度分别显著增加5.5% 和39%(图3B、3C)。以上结果表明,低磷处理显著促进大豆根系的生长,植物可以通过根系生长的变化来适应低磷胁迫。
根据系统进化树结果,从GmNTLs 家族的6 个复制对中各挑取1 个基因包括G m N T L 1 、G m N T L 4 、G m N T L 7 、G m N T L 8 、G m N T L 1 0 和GmNTL12,分析不同磷有效性和磷处理时间对大豆GmNTLs 在根系表达水平的影响。结果显示,GmNTLs 家族基因的表达量均在6 或12 d 受低磷胁迫诱导显著上调(图4)。在磷处理6 d 后,低磷条件下, G m N T L 1 、G m N T L 4 、G m N T L 8 和GmNTL12 的表达水平分别比高磷的高1.8、1.8、2.1 和2.1 倍(图4A、4B、4D、4F),GmNTL7/10 与高磷条件下无明显差异(图4C、4E)。磷处理12 d 后,低磷条件下G m N T L 1 、G m N T L 4 、G m N T L 7 、GmNTL8、GmNTL10 和GmNTL12 的表达水平分别比高磷条件下高3.5、5.5、1.5、3.5、1.9 和3.2 倍(图4),这些结果表明GmNTLs 参与了大豆根系适应低磷胁迫过程。
2.3 不同磷处理浓度对大豆根系中GmNTLs 表达模式的影響
从“2.2”结果可知,当磷处理12 d 后,大豆根系中GmNTLs 的表达模式受低磷诱导上调较为显著,因此进一步探究了不同磷浓度(5、50 和250μmol·L?1 KH2PO4) 处理12 d 对大豆根系GmNTLs表达模式的影响。结果表明:随着磷处理浓度的降低,GmNTLs 基因的表达量整体显著上调(图5)。当磷浓度从250 μmol·L?1 下降到50 μmol·L?1 时,除GmNTL10 基因的表达量无明显变化外,其他基因的表达量均随磷浓度降低而显著上调,上调比例介于22.1%~142.6%;而当磷浓度从50 μmol·L?1 进一步下降到5 μmol·L?1 时,6 个GmNTLs 的表达量均显著上调,上调比例介于20.3%~129.7%。以上结果进一步表明GmNTLs 基因参与了大豆根系响应低磷胁迫的过程,但各成员响应程度有差异。
2.4 低磷胁迫对超量表达GmNTLs 拟南芥生长的影响
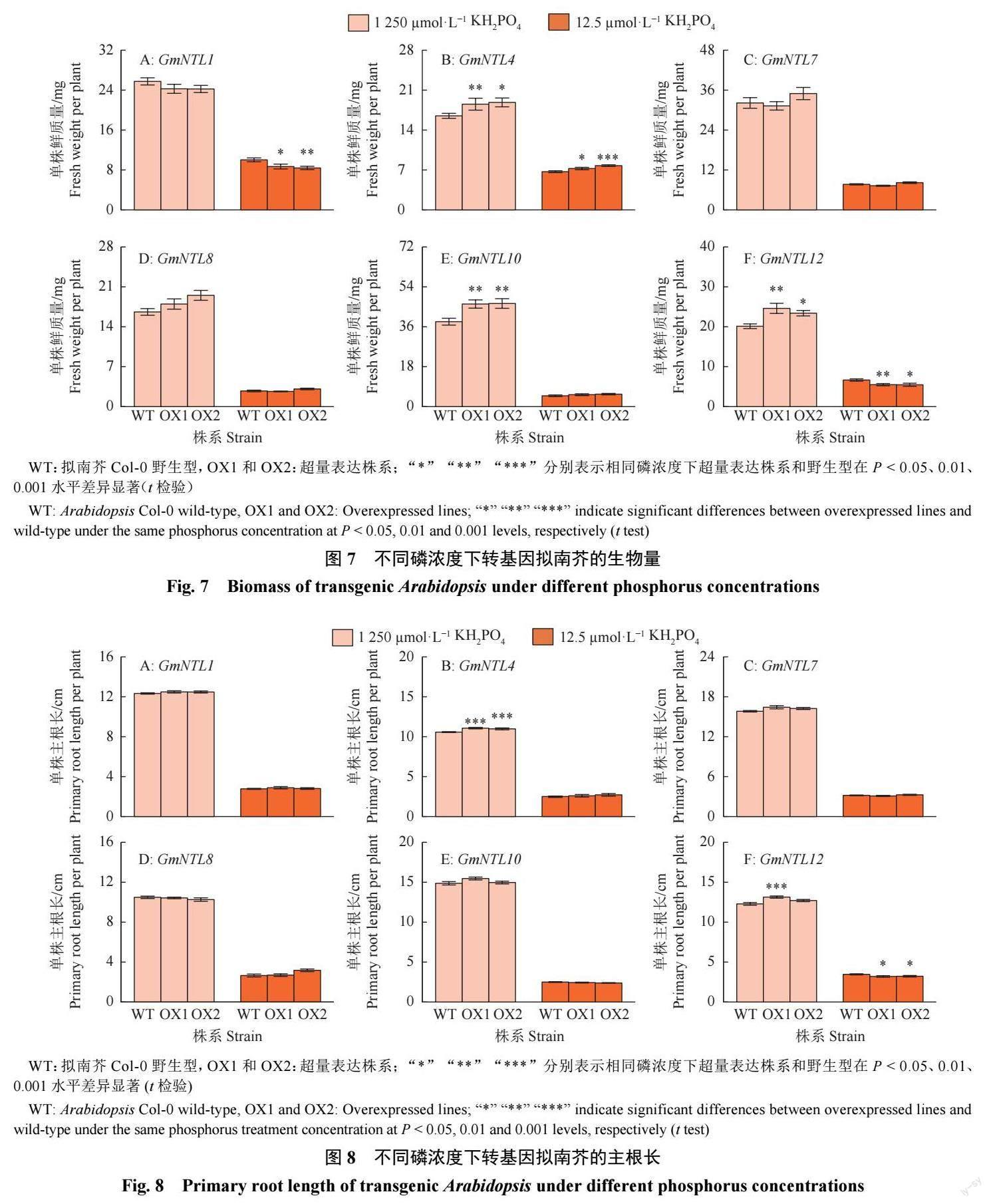
野生型和超量表达GmNTLs 的转基因拟南芥幼苗在高、低磷MS 固体培养基处理12 d 后,分析植株鲜质量和主根长。结果(图6、7、8) 显示,与野生型(WT) 相比,高磷条件下,超量表达GmNTL4、GmNTL10 和GmNTL12 促进了拟南芥植株的生长,其鲜质量分别显著增加11.0%、12.4%, 13.0%、14.4% 和22.2%、16.2%(图7B、7E、7F);低磷条件下,超量表达GmNTL4 拟南芥鲜质量显著增加8.3%、15.9%(图7B),而超量表达GmNTL1 和GmNTL12 拟南芥鲜质量显著减少11.4%、13.3%和17.8%、17.5%(图7A、7F)。与鲜质量相似,在高磷条件下,超量表达GmNTL4 和GmNTL12 植株的主根长显著增加4.7%、3.8% 和7.0%(图8B、8F);在低磷条件下,超量表达GmNTL12 植株的主根长显著减少7 . 7 % 、6 . 8 % ( 图8 F ) 。超量表达GmNTL7 和GmNTL8 基因对转基因拟南芥的生长无明显影响(图7C、7D、8C、8D)。结果表明不同GmNTL 基因成员对拟南芥响应低磷胁迫的功能不同。
3 讨论与结论
植物NTL 转录因子参与干旱、盐害、渗透和铝毒等非生物胁迫[7, 18-23]。酸性土壤中铝毒和低磷胁迫共同存在,植物进化出一系列协同调控应答机制[10-14],Lin 等[20] 研究表明,GmNTLs 参与了大豆耐铝毒胁迫,但是否调控大豆根系响应低磷胁迫还未见报道。本研究通过设置不同磷浓度和不同胁迫时间处理,解析低磷胁迫对GmNTLs 基因在大豆根系中表达水平的调控模式,发现GmNTLs 基因成员都不同程度地受到低磷诱导上调表达,暗示这些GmNTLs基因均在大豆响应低磷胁迫中发挥作用;进一步分析发现,不同基因成员响应模式有所区别,其中GmNTL1、GmNTL4、GmNTL8 和GmNTL12 基因的表达量在低磷处理6、12 d 后显著大幅度上调,而GmNTL7 和GmNTL10 基因的表达量在低磷处理6 d 时无明显变化,只在12 d 后显著上调。不同磷浓度处理的结果也类似,GmNTL10 基因的表达量只在极度缺乏磷时显著上调表达,而其他基因表达量随磷浓度减少不断显著增加,暗示这些GmNTLs基因在大豆适应低磷胁迫中可能起重要的作用。
为进一步验证GmNTLs 在大豆根系响应低磷胁迫中的功能,本研究通过拟南芥的异源表达,构建了超量表达GmNTL1/4/7/8/10/12 6 个基因的拟南芥转基因材料;结果显示,不同GmNTL 基因成员在拟南芥响应低磷胁迫中的功能不同。超量表达GmNTL4 和GmNTL12 基因后,拟南芥转基因株系的鲜质量和主根长与野生型差异显著。例如,高磷条件下,超量表达GmNTL4 的转基因拟南芥鲜质量和主根长均显著高于野生型,但低磷条件下其主根长无明显变化,说明GmNTL4 可能参与了植株地上部生长对磷供应的适应性的调控,但对根系的调控作用不明显。另外, 低磷条件下, 超量表达GmNTL12 的转基因拟南芥鲜质量和主根长均显著低于野生型,而高磷条件下,超量表达GmNTL12 的转基因拟南芥表型与低磷相反。主根缩短是拟南芥根系响应低磷胁迫的重要反应之一[24],低磷诱导的拟南芥主根生长是由生长素信号途径因子TIR1、A U X / I A A 、A t A R F 1 9,以及STOP1、LPR1 和P D R 2 等多基因调控的综合结果[ 2 5 - 2 8 ] 。关于GmNTL12 为什么在不同磷处理下对根系生长的影响不同,其调控机理还有待进一步研究。与GmNTL4相反,超量表达GmNTL1 和GmNTL12 显著降低了转基因株系在低磷条件下的鲜质量,暗示GmNTL1和GmNTL12 可能参与植物适应低磷胁迫的负调控。无论是在高磷还是低磷条件下,超量表达GmNTL7和GmNTL8 基因,对转基因拟南芥的鲜质量和主根伸长均无明显影响。当植物处于逆境时,膜结合转录因子转运到细胞核后发挥功能[29]。超量表达NTLs全长的转基因幼苗与对照之间通常不存在表型差异,但是当去除过表达基因跨膜域后,表型出现明显差异[21, 30];推测超量表达GmNTL7 和GmNTL8 转基因拟南芥在受到低磷胁迫时,可能没有诱导膜释放或膜释放不当,导致这2 个转基因拟南芥与野生型无表型差异。
综上所述,低磷胁迫上调了大豆根系中GmNTL1/4/7/8/10/12 基因的表达,GmNTLs 参与了大豆根系對低磷胁迫的响应。本研究结果可为后续深入研究NTL 在大豆生长发育过程中的功能提供理论依据。
参考文献:
OOKA H, SATOH K, DOI K, et al. Comprehensive analysisof NAC family genes in Oryza sativa and Arabidopsisthaliana[J]. DNA Research, 2003, 10(6): 239-247.
KIM S Y, KIM S G, KIM Y S, et al. Exploring membrane-associated NAC transcription factors in Arabidopsis:Implications for membrane biology in genome regulation[J]. Nucleic Acids Research, 2007, 35(1): 203-213.
赵翠珠, 刘振华, 赵赫, 等. 植物NAC 膜结合转录因子的研究进展[J]. 生命科学, 2012, 24(1): 74-80.
CHI Y H, MELENCION S, ALINAPON C V, et al. Themembrane-tethered NAC transcription factor, AtNTL7,contributes to ER-stress resistance in Arabidopsis[J]. Biochemicaland Biophysical Research Communications,2017, 488(4): 641-647.
ZHAO J, LIU J S, MENG F N, et al. ANAC005 is amembrane-associated transcription factor and regulatesvascular development in Arabidopsis[J]. Journal of IntegrativePlant Biology, 2016, 58(5): 442-451.
WANG D, YU Y, LIU Z, et al. Membrane-bound NACtranscription factors in maize and their contribution to theoxidative stress response[J]. Plant Science, 2016, 250:30-39.
KIM M J, PARK M, SEO P J, et al. Controlled nuclearimport of the transcription factor NTL6 reveals a cytoplasmicrole of SnRK2.8 in the drought-stressresponse[J]. Biochemical Journal, 2012, 448(3): 353-363.
KIM S, LEE A, YOON H, et al. A membrane-boundNAC transcription factor NTL8 regulates gibberellicacid-mediated salt signaling in Arabidopsis seed germination[J]. Plant Journal, 2008, 55: 77-88.
SEO P J, KIM M J, PARK J, et al. Cold activation of aplasma membrane-tethered NAC transcription factor inducesa pathogen resistance response in Arabidopsis[J].Plant Journal, 2010, 61(4): 661-671.
KOCHIAN L V, HOEKENGA O A, PINEROS M A.How do crop plants tolerate acid soils? Mechanisms ofaluminum tolerance and phosphorous efficiency[J]. AnnualReview of Plant Biology, 2004, 55: 459-493.
吴佩, 李浩, 早浩龙, 等. 植物对缺磷和铝毒协同进化应答的分子生理机制[J]. 生物技术通报, 2020, 36(7): 170-181.
LIAO H, WAN H, SHAFF J, et al. Phosphorus and aluminuminteractions in soybean in relation to aluminumtolerance, exudation of specific organic acids from differ- ent regions of the intact root system[J]. Plant Physiology,2006, 141(2): 674-684.
LIANG C, PINEROS M A, TIAN J, et al. Low pH, aluminum,and phosphorus coordinately regulate malate exudationthrough GmALMT1 to improve soybean adaptationto acid soils[J]. Plant Physiology, 2013, 161(3):1347-1361.
CHEN W, TANG L, WANG J, et al. Research advancesin the mutual mechanisms regulating response of plantroots to phosphate deficiency and aluminum toxicity[J].International Journal of Molecular Sciences, 2022, 23(3):1137. doi: 10.3390/ijms23031137.
于芮, 王斌, 王建忠, 等. 我國大豆市场发展现状及建议[J]. 合作经济与科技, 2021(19): 92-93.
刘国选, 陈康, 陆星, 等. 大豆GmPIN2b 调控根系响应低磷胁迫的功能研究[J]. 华南农业大学学报, 2021,42(4): 33-41.
LI S, WANG N, JI D, et al. Evolutionary and functionalanalysis of membrane-bound NAC transcription factorgenes in soybean[J]. Plant Physiology, 2016, 172(3):1804-1820.
向凤宁, 王楠. 大豆威廉姆斯82 中NAC 膜结合转录因子基因GmNTL1 及其应用: CN106167801A[P]. 2018-08-28[2022-04-16].
向凤宁, 王楠. 大豆威廉姆斯82 中NAC 膜结合转录因子基因GmNTL7 及其应用: CN106367423B[P]. 2017-02-01[2022-04-16].
LIN Y, LIU G, XUE Y, et al. Functional characterizationof aluminum (Al)-responsive membrane-bound NACtranscription factors in soybean roots[J]. InternationalJournal of Molecular Sciences, 2021, 22(23): 12854. doi:10.3390/ijms222312854.
PARK J, KIM Y S, KIM S G, et al. Integration of auxinand salt signals by the NAC transcription factor NTM2during seed germination in Arabidopsis[J]. PlantPhysiology, 2011, 156(2): 537-549.
YOON H K, KIM S G, KIM S Y, et al. Regulation of leafsenescence by NTL9-mediated osmotic stress signaling in
Arabidopsis[J]. Molecules and Cells, 2008, 25(3): 438-445.
DUAN M, ZHANG R, ZHU F, et al. A lipid-anchoredNAC transcription factor is translocated into the nucleusand activates glyoxalase I expression during droughtstress[J]. Plant Cell, 2017, 29(7): 1748-1772.
PERET B, CLEMENT M, NUSSAUME L, et al. Rootdevelopmental adaptation to phosphate starvation: Bettersafe than sorry[J]. Trends in Plant Science, 2011, 16(8):442-450.
P?REZ-TORRES C, L?PEZ-BUCIO J, CRUZRAM?REZA, et al. Phosphate availability alters lateralroot development in Arabidopsis by modulating auxinsensitivity via a mechanism involving the TIR1 auxin receptor[J]. Plant Cell, 2008, 20(12): 3258-3272.
SHEN C, WANG S, ZHANG S, et al. OsARF16, a transcriptionfactor, is required for auxin and phosphate starvationresponse in rice (Oryza sativa L. )[J]. Plant Celland Environment, 2013, 36(3): 607-620.
BALZERGUE C, DARTEVELLE T, GODON C, et al.Low phosphate activates STOP1-ALMT1 to rapidly inhibitroot cell elongation[J]. Nature Communications,2017, 8: 15300. doi: 10.1038/ncomms15300.
HAM B K, CHEN J, YAN Y, et al. Insights into plantphosphate sensing and signaling[J]. Current Opinion inBiotechnology, 2018, 49: 1-9.
CHEN Y N, SLABAUGH E, BRANDIZZI F. Membrane-tethered transcription factors in Arabidopsis thaliana:Novel regulators in stress response and development[J]. Current Opinion in Plant Biology, 2008, 11(6):695-701.
LEE S, LEE H J, HUH S U, et al. The Arabidopsis NACtranscription factor NTL4 participates in a positive feedbackloop that induces programmed cell death under heatstress conditions[J]. Plant Science, 2014, 227: 76-83.
【责任编辑 李庆玲】

