花榈木生物碱合成关键酶基因OhpLDC和OhpCAO1的克隆与分析
王佳琦 王欣 邓小梅 吴蔼民





王佳琦,王 欣,鄧小梅,等. 花榈木生物碱合成关键酶基因OhpLDC和OhpCAO1 的克隆与分析[J]. 江苏农业学报,2023,39(8):1646-1657.
doi:10.3969/j.issn.1000-4440.2023.08.004
收稿日期:2022-10-27
基金项目:广东省林业科技创新项目(2017KJCX028)
作者简介:王佳琦(1998-),女,黑龙江大庆人,硕士研究生,主要从事花榈木组学研究。(E-mail)wangjiaqi1990@163.com
通讯作者:吴蔼民,(E-mail)wuaimin@scau.edu.cn
摘要:花榈木是中国传统中药,其代谢产物常用于治疗跌打损伤,具有重要的药用价值,然而目前人们对花榈木的药用化学成分如生物碱合成并不清楚。本研究对花榈木不同组织进行了代谢组和转录组学分析,结果表明花榈木中含有的生物碱大部分属于喹诺里西啶类生物碱(Quinolizidine Alkaloids,QA)。进一步分析发现,QA生物合成的2个关键酶,赖氨酸脱羧酶(LDC) 和铜胺氧化酶(CAO) 在生物碱合成中起重要作用。结合花榈木转录组结果对其编码基因克隆,得到了1 290 bp、2 268 bp CDS序列,分别命名为OhpLDC和OhpCAO1。生物信息学分析结果表明,OhpLDC 和OhpCAO1 编码的蛋白质相对分子质量分别为4.63×104和8.44×104,均无跨膜区域和信号肽。OhpLDC编码的氨基酸序列具有保守的底物结合位点Phe340,分析发现OhpLDC与豆科植物中的LDC基因在进化上具有较近的亲缘关系。 OhpCAO1 编码的氨基酸序列中具有保守结构域“NY-Y”,以及3个组氨酸保守位点,与狭叶羽扇豆的 CAO1 亲缘关系最近。本研究结果为解析花榈木QA的生物合成途径提供了重要基础。
关键词:花榈木;喹诺里西啶类生物碱;赖氨酸脱羧酶;铜胺氧化酶;生物信息学
中图分类号:S792.99 文献标识码:A 文章编号:1000-4440(2023)08-1646-12
Cloning and analysis of key enzyme genes OhpLDC and OhpCAO1 for alkaloid synthesis in Ormosia henryi Prain
WANG Jia-qi WANG Xin DENG Xiao-mei WU Ai-min
(College of Forestry and Landscape Architecture, South China Agricultural University, Guangzhou 510000, China)
Abstract:Ormosia henryi Prain is a traditional Chinese medicine, and its metabolites are commonly used to treat injuries and have important medicinal value. However, the medicinal chemical components of Ormosia henryi Prain, such as alkaloid synthesis, are currently unclear. This study conducted metabolomic and transcriptomic analysis on different tissues of Ormosia henryi Prain, and the results showed that most of the alkaloids contained in Ormosia henryi Prain belonged to quinolizidine alkaloids (QA). Further analysis revealed that the two key enzymes involved in QA biosynthesis, lysine decarboxylase (LDC) and copper amine oxidase (CAO), played important roles in alkaloid synthesis. Based on the transcriptome results of Ormosia henryi Prain, the coding genes were cloned, and two CDS sequences of 1 290 bp and 2 268 bp were obtained, named OhpLDC and OhpCAO1 respectively. The bioinformatics analysis results indicated that the relative molecular weights of the proteins encoded by OhpLDC and OhpCAO1 were 4.63×104 and 8.44×104, and there were no transmembrane regions and signal peptides. The amino acid sequence encoded by OhpLDC had a conserved substrate binding site Phe340. Analysis revealed that OhpLDC had a close evolutionary relationship with LDC genes in leguminous plants. The amino acid sequence encoded by OhpCAO1 had a conserved domain "NY-Y" and three histidine conserved sites. The amino acid sequence encoded by OhpCAO1 had the closest genetic relationship with the CAO1 of narrow-leaved lupin. The results of this study provide an important basis for analyzing the biosynthetic pathway of QA in Ormosia henryi Prain.
Key words:Ormosia henryi Prain;quinolizidine alkaloids;lysine decarboxylase;copper amine oxidase;bioinformatics
花榈木(Ormosia henryi Prain)属于豆科(Leguminosae)蝶形花亚科(Papilionideae)红豆属(Ormosia Jacks.)常绿乔木,属国家二级重点保护树种[1]。其种子、根、茎、皮和叶中都含有可入药的化学成分,具有通经活血的功效。《全国中草药汇编》记述,花榈木常以根、皮、茎及叶入药,其性味归经为辛、温、有毒,具有活血化瘀、祛风、消肿之功[2]。多数红豆属树木可入药,古人常用于治疗跌打损伤、风湿关节炎及无名肿痛等,但具有一定的毒性。迄今为止,国内外学者已从红豆属植物中分离、鉴定出200多种化合物,包括生物碱类、黄酮类、其他类化合物以及挥发油成分等[3]。其中很多生物碱类化合物具有影响中枢神经系统的活性,例如Pouny等[4]发现从红豆树(Ormosia hosiei)分离得到的生物碱对神经元烟碱型乙酰胆碱α4β2受体具有显著亲和力,表明红豆属生物碱在治疗中枢神经系统相关疾病方面具有潜在的应用价值[5-6]。
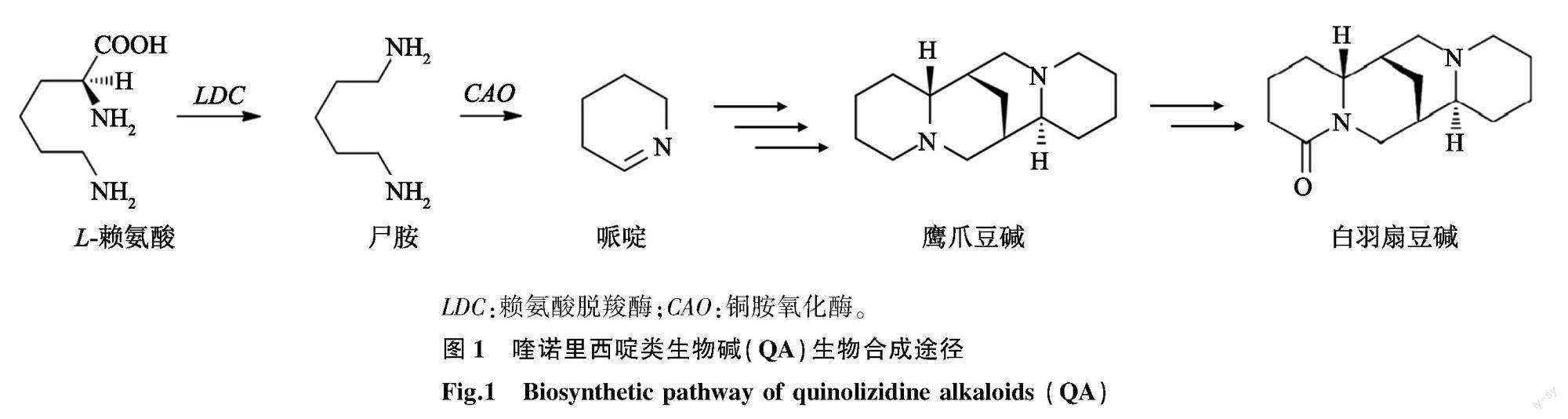
喹诺里西啶类生物碱(Quinolizidine alkaloids,QA)具有广泛生物活性,是一类L-赖氨酸衍生的生物碱,具有170多种化学结构[7],存在于豆科植物槐族(Sophoreae)和染料木族(Genisteae)中[8]。QA具有抗肿瘤、抗病毒和降糖等多种功能[9-10],在农业、医药和化学中都有潜在的应用价值,但人们对QA的生物合成知之甚少[11]。QA的核心生物合成仍然是一个谜,迄今为止只已知赖氨酸脱羧酶(LDC)和铜胺氧化酶(CAO)参与了部分QA生物合成过程[12]。QA生物合成途径的第一步,LDC参与赖氨酸脱羧生成中间产物尸胺(Cadaverine)[13]。而CAO参与了尸胺的脱氨反应,产生所有QA合成的共同途径的中间体哌啶(1-piperideine)[14]。接下来四环QA鹰爪豆碱(Sparteine)的生物合成一直是植物生物化学中未解决的问题(图1)。QA的研究基本集中在豆科植物羽扇豆属的白羽扇豆(Lupinus albus)中,在豆科植物花榈木中还未见有关QA生物合成的报道。
本研究利用代谢组测序数据,发现花榈木中含有大量QA。通过对转录组测序数据进行WGCNA分析,发现与QA生物合成高度相关基因LDC和CAO的转录本。本研究对这2个基因进行克隆,并进行了相关生物信息学分析及其在各组织中表达情况分析。
LDC:赖氨酸脱羧酶;CAO:铜胺氧化酶。
1 材料与方法
1.1 试验材料
试验材料为3年生花榈木,种植于华南农业大学(中国广州)苗圃中。分别采摘花榈木的根、茎、树皮、老叶和新叶,每个部位收集4份样品用于重复试验。在苗圃采摘时,采下的样品立即在液氮中冷冻保鲜。带回实验室后,将样品一部分在-80 ℃下保存用于RNA提取,另一部分在真空下冷冻干燥用于代谢物提取。
1.2 花榈木代谢组分析
冻干的样品用研钵和研杵磨成粉末。每个样品的粉末用80%的HPLC级甲醇提取过夜,其中以1 μmol的白杨素作为内标,每0.1 g样品加入1 ml提取液,于4 ℃下12 000 g离心30 min,将上清液装入样品瓶中,进行超高效液相色谱质谱分析[15]。
1.3 花榈木转录组分析
将采自同一时期的4份花榈木同一组织样品等质量混合,委托北京百迈客云科技有限公司提取RNA并构建测序文库,使用Illumina HiSeq 2000测序平台进行转录组双末端测序。经过Trinity软件组装获得Unigene,并进一步使用HMMER软件[16]与Pfam数据库[17]比对,获得Unigene的注释信息。
1.4 花榈木QA代谢途径关键酶基因筛选
根据参考文献中QA的生物合成代谢通路,结合数据库注释结果,筛选出花榈木转录组中与QA合成相关的Unigenes,以常用的基因表达水平估算方法中FPKM(Fragments per kilobase million)值进行表达量统计,分析相关基因在花榈木不同组织中的表达模式。
1.5 总RNA提取及cDNA合成
将所有收集的样品用液氮磨成粉末,然后用RNAprep pure植物总RNA提取试剂盒[天根生化科技(北京)有限公司产品]提取总RNA。使用1%琼脂糖凝胶进行电泳检查RNA质量,并使用NanoPhotometer分光光度计(美国IMPLEN公司产品)检测RNA的纯度和完整性。用TaKaRa反转录试剂盒[TaKaRa Prime ScriptTM 1st Strand cDNA Synthesis Kit,宝日医生物技术(北京)有限公司产品]将检测合格的总RNA反转录成cDNA。
1.6 基因克隆与测序
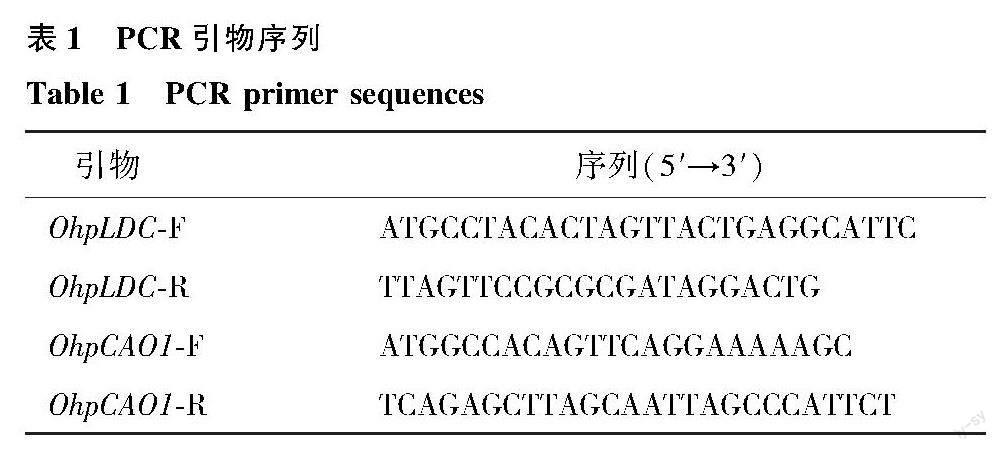
根据前期花榈木转录组数据获得的OhpLDC和OhpCAO1 基因序列,使用Primer Premier 5.0 设计特异引物(表1),以花榈木总cDNA为模板,扩增OhpLDC和OhpCAO1 基因全长。PCR反应程序为98 ℃預变性3 min;98 ℃变性 30 s;55 ℃退火 30 s;72 ℃延伸1.5 min,35个循环;72 ℃复性5 min;12 ℃保存。PCR扩增产物经琼脂糖凝胶电泳检测后,使用DNA回收纯化试剂盒将目的条带切胶回收,将其连接到TOPO-TA克隆载体后转化至大肠杆菌DH5α菌株,挑取阳性单菌落进行PCR验证,无误后送北京擎科生物科技有限公司测序。
1.7 生物信息学分析
将OhpLDC和OhpCAO1 2个基因测序获得的序列结果,利用ORF Finder(https://www.ncbi.nlm.nih.gov/orffinder/)查找开放阅读框(ORF),及其编码的氨基酸序列。利用在线工具Protparam(https://web.expasy.org/protparam/)预测基因编码蛋白质的相对分子质量、氨基酸数目、等电点、不稳定系数等理化性质;利用在线工具DeepTMHMM(https://dtu.biolib.com/DeepTMHMM)和SignalP6.0(https://services.healthtech.dtu.dk/service.php?SignalP-6.0)对OhpLDC和OhpCAO编码的蛋白质进行跨膜结构和信号肽预测。利用Euk-mPLoc 2.0(http://www.csbio.sjtu.edu.cn/bioinf/euk-multi-2/)预测蛋白质亚细胞定位。利用在线工具SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)分析蛋白质二级结构;利用SWISS-MODEL网站(https://swissmodel.expasy.org/)预测蛋白质三级结构。由CLUSTALW网站(https://www.genome.jp/tools-bin/clustalw)软件和ESPript 3(https://espript.ibcp.fr/ESPript/cgi-bin/ESPript.cgi)进行氨基酸多序列比对,并利用MEGA 11.0软件中的邻接法(Neighbor joining,NJ)进行系统进化树分析[18]。
1.8 表达模式分析
使用荧光定量PCR仪分析 OhpLDC和OhpCAO基因在花榈木不同组织中的表达模式。利用Light Cycler 480设备进行qRT-PCR,反应程序设置为95 ℃ 3 s,95 ℃ 5 s,60 ℃ 30 s,40个循环。引物用Primer3Plus(https://dev.primer3plus.com)设计(表2),内参基因是 c71500.graph_c0。每个样品分别进行3次重复,采用2-△Ct方法计算相对基因表达量[19]。
2 结果与分析
2.1 花榈木的代谢组学分析
花榈木是一种豆科木本植物,原产于东南亚和南美洲,具有巨大的药用价值。为了更深入研究花榈木,我们在华南农业大学的田间栽培了一些花榈木,并收取了3年生植物的5个组织部位,即根(HR)、茎(HS)、老叶(HOL)、新叶(HNL)和树皮(HB)。我们对其进行了基于超高效液相色谱-串联质谱的广泛的目标代谢组学分析。在所有样本中有1 535个显著代谢物被记录下来,并通过与KEGG、HMDB和脂质图数据库的映射,对代谢物进行了功能注释。在花榈木中共检测到6种生物碱(表3),其中白羽扇豆碱(Lupanine)、13-羟基羽扇豆碱(13a-Hydroxylupanin)、鹰爪豆碱(Sparteine)、金雀花碱(Cytisine)和乙酰金雀花碱(Acetylcytisine)属于QA,所以本研究主要关注QA的生物合成,详细的二级质谱图如图2所示。
图中0轴以上为数据库中质谱图;0轴以下为花榈木中检测质谱图。
2.2 花榈木的转录组学分析
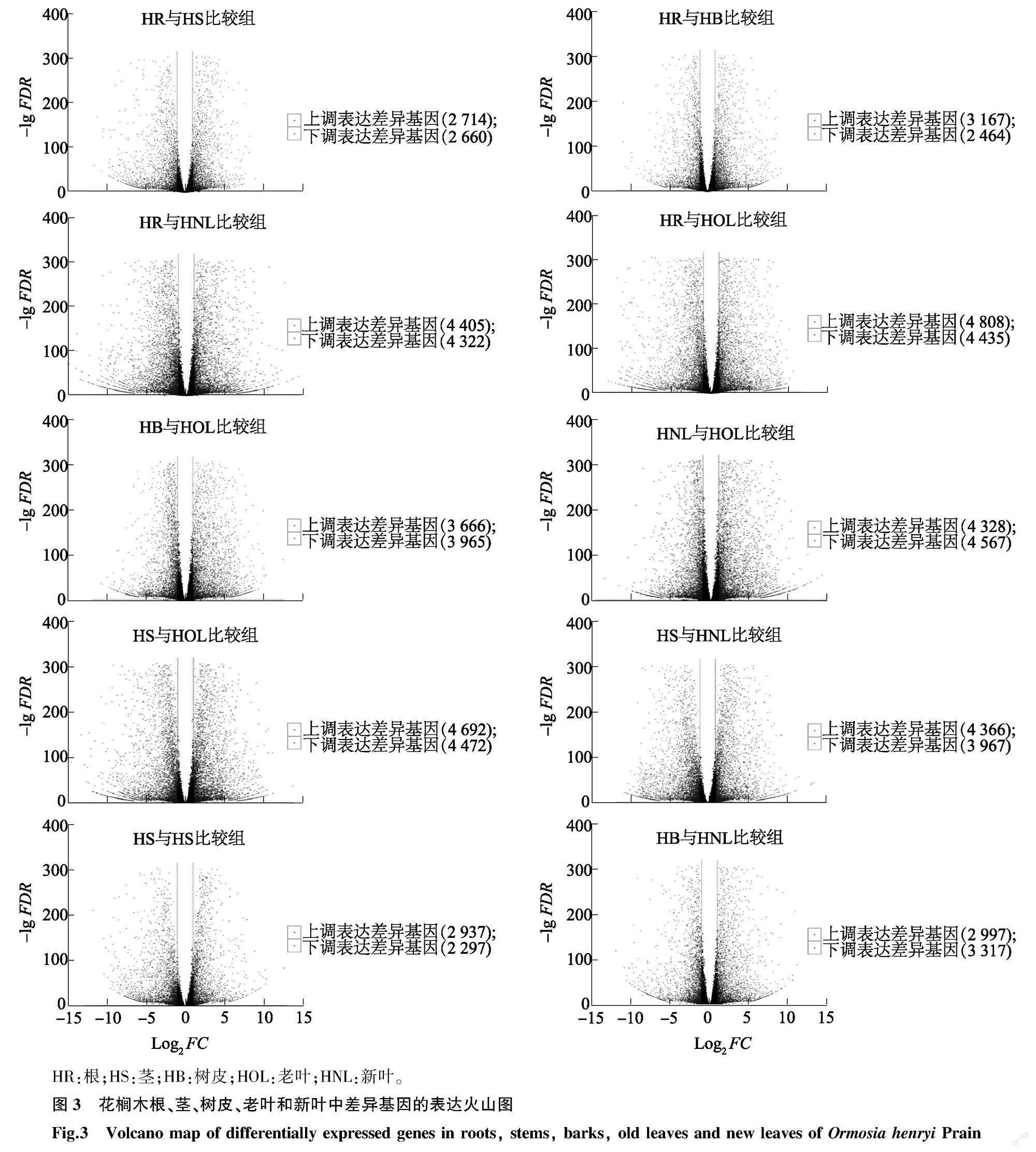
利用Illumina Hiseq 2000平台进行测序,在去除低质量和短的读数后,总共获得了1.9~2.4 Mb的干净读数,共获得96 302个Unigenes。与NR、eggNOG、TrEMBL、Pfam、SwissProt、KEGG、COG、KOG及GO等9个数据库比对,确定CDS有47 809条,并对其进行功能注释。利用R包DESeq2对花榈木5个部位的转录组进行差异基因分析,共建立了10个比较组,分别为HR与HS比较组、HR与HB比较组、HR与HNL比较组、HR与HOL比较组、HB与HOL比较组、HNL与HOL比较组、HS与HOL比较组、HS与HNL比较组、HS与HB比较组和HB与HNL比较组。以FDR≤0.01且FC≥2为筛选条件,10个比较组差异基因数量分别为5 374、5 631、8 727、9 243、7 631、8 895、9 164、8 333、5 234和6 314(图3),一共得到13 840个差异显著基因(Differential gene expression,DEG)。
HR:根;HS:茎;HB:树皮;HOL:老叶;HNL:新叶。
为了研究花榈木中与生物碱合成相关转录本,将所有DEG和生物碱进行了WGCNA分析,DEG共被分为10个模块,发现其中棕色模块基因与喹诺里西啶类生物碱基因(QA)相关性最高,与5种QA的相关性都高于0.9(图4A)。通过KEGG分析,发现棕色模块基因主要富集在代谢过程中,包含一些基础代谢途径淀粉和蔗糖代谢(Starch and sucrose metabolism,编码ID:ko00500)、碳代谢(Carbon metabolism,编码ID:ko01200),还富集到賴氨酸降解(Lysine degradation,编码ID:ko00310)、赖氨酸合成(Lysine biosynthesis,编码ID:ko00300)和吲哚生物碱的生物合成(Indole alkaloid biosynthesis,编码ID:ko00901)等代谢通路(图4B)。分析结果表明花榈木中不同次级代谢物的生物合成可能具有关联性。
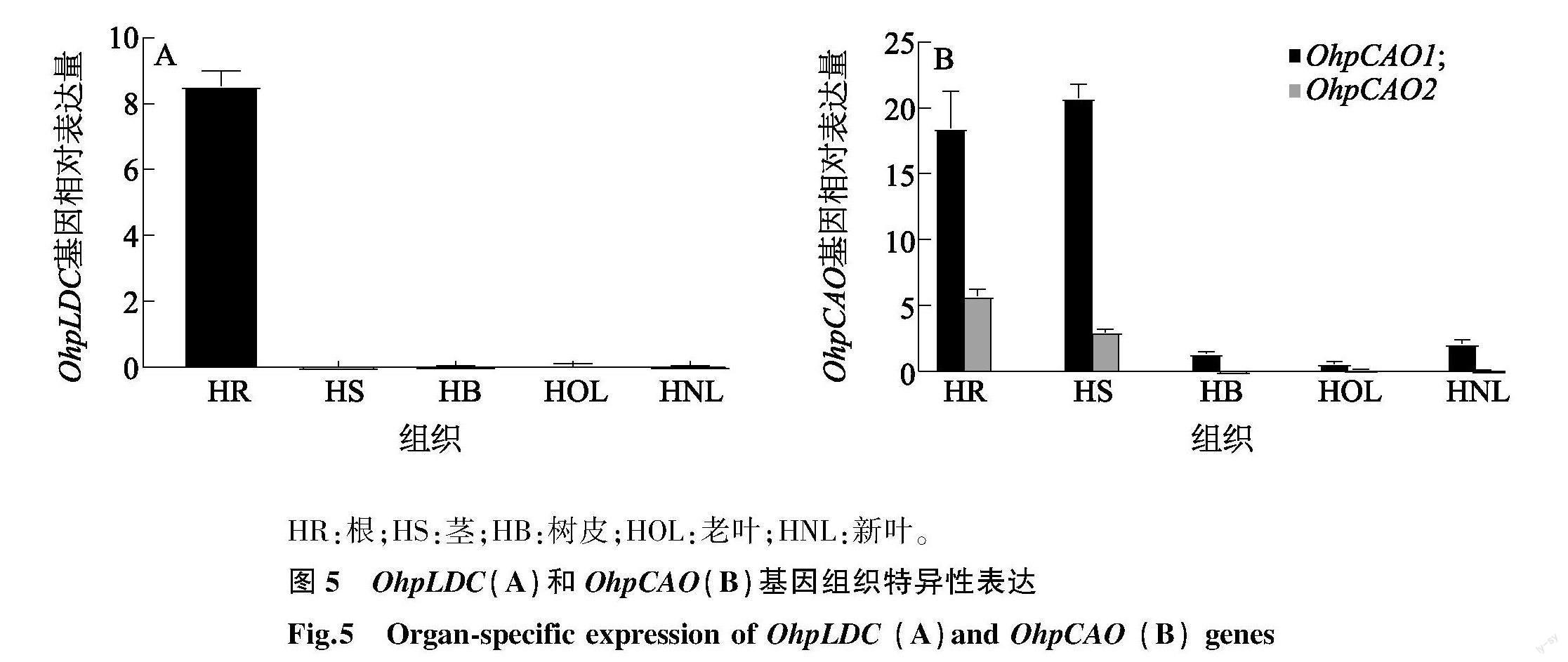
一直以来QA生物合成的前两步是由LDC和CAO催化(图1)。在转录组中发现1个LDC和2个CAO的转录产物,其基因分别命名为OhpLDC、OhpCAO1 和OhpCAO2。根据WGCNA分析发现只有OhpLDC和OhpCAO1 这2个基因在与QA高度相关的棕色模块中,所以本研究鉴定了QA生物合成途径中的2个酶基因OhpLDC和OhpCAO1。OhpLDC和OhpCAO1 在花榈木中不同组织的表达情况有明显差异,都在根中表达量较高(图5A、图5B)。
HR:根;HS:茎;HB:树皮;HOL:老叶;HNL:新叶。
2.3 OhpLDC 和 OhpCAO1 基因克隆与序列分析
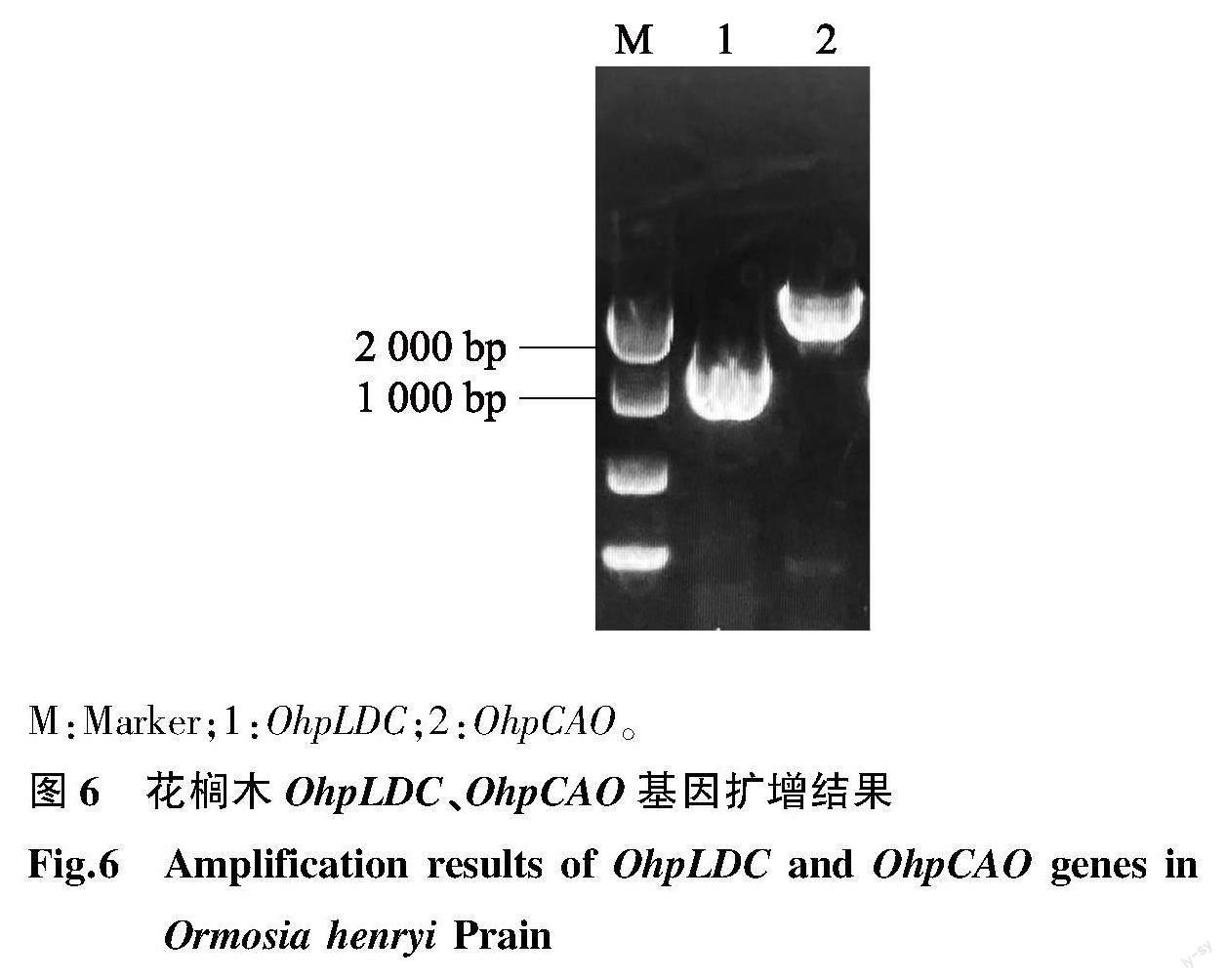
依据花榈木转录组数据获得OhpLDC和OhpCAO1 cDNA序列,并利用ORF Finder在线软件对OhpLDC 和 OhpCAO1 cDNA序列分析,预测OhpLDC含1 290 bp完整的开放阅读框 OhpCAO1 含2 268 bp完整的开放阅读框,分别编码429、755个氨基酸。通过设计特异性引物,进行PCR扩增反应,获得目的基因片段(图6)。将PCR产物回收纯化并重组到TOPO载体中,挑选阳性菌落PCR检测并测序,测序结果与预测结果一致。将2个基因提交到NCBI上利用BLASTP进行序列比对,比对结果显示OhpLDC基因序列与豆科植物Echinosophora koreensis中EkDC(GenBank登录号:AB561139.1)序列高度相似,相似度为87.2% OhpCAO1 基因序列与相思子中ApCAO(GenBank登录号:XM_027491129.1)序列高度相似,相似度为92%。
M:Marker;1:OhpLDC;2:OhpCAO。
2.4 蛋白质理化性质分析
利用Protparam在线软件对OhpLDC和OhpCAO1 基因编码的氨基酸序列进行分析,发现OhpLDC蛋白相对分子质量为4.63×104,分子式为C2 069H3 256N544O618S21,理论等电点(PI)为6.98;OhpCAO1蛋白相对分子质量为8.44×104,分子式为C3 781H5 835N1 053O1 083S34,理论等电点(PI)为6.51。OhpLDC蛋白带负电残基(Asp+Glu)数为42,带正电残基(Arg+Lys)数为42,不稳定系数为41.06。OhpCAO1蛋白带负电残基(Asp+Glu)数为91,带正电残基(Arg+Lys)数为85,不稳定系数为43.63。OhpLDC和OhpCAO1均属于不稳定蛋白质。
利用DeepTMHMM和SignalP6.0在线软件对OhpLDC 和OhpCAO1蛋白进行跨膜结构和信号肽预测,结果表明其氨基酸序列中均无跨膜区域和信号肽序列。利用Euk-mPLoc 2.0在线软件分析,初步判断OhpLDC蛋白定位于线粒体,OhpCAO1蛋白定位于细胞外间质中。
2.5 蛋白质二级结构和三级结构预测
使用SOPMA和PredictProtein在线工具预测OhpLDC和OhpCAO1蛋白二级结构,结果显示OhpLDC和OhpCAO1蛋白二級结构都由α-螺旋(Alpha helix)、β-转角(Beta turn)、延伸链(Extended strand) 和无规则卷曲(Random coil)组成,均无二硫键。其中OhpLDC的α-螺旋为34.50%、β-转角为6.99%、延伸链为18.18%、无规则卷曲为40.33%;OhpCAO1的α-螺旋为17.75%、β-转角为7.15%、延伸链 23.71%、无规则卷曲为51.35%。利用SWISS-MODEL同源建模系统对OhpLDC和OhpCAO1进行同源建模,得到了2个蛋白质的三级结构模型,如图7所示。
A:预测的OhpLDC蛋白三级结构;B:预测的OhpCAO1蛋白三级结构。
2.6 氨基酸序列的同源性及系统进化树分析
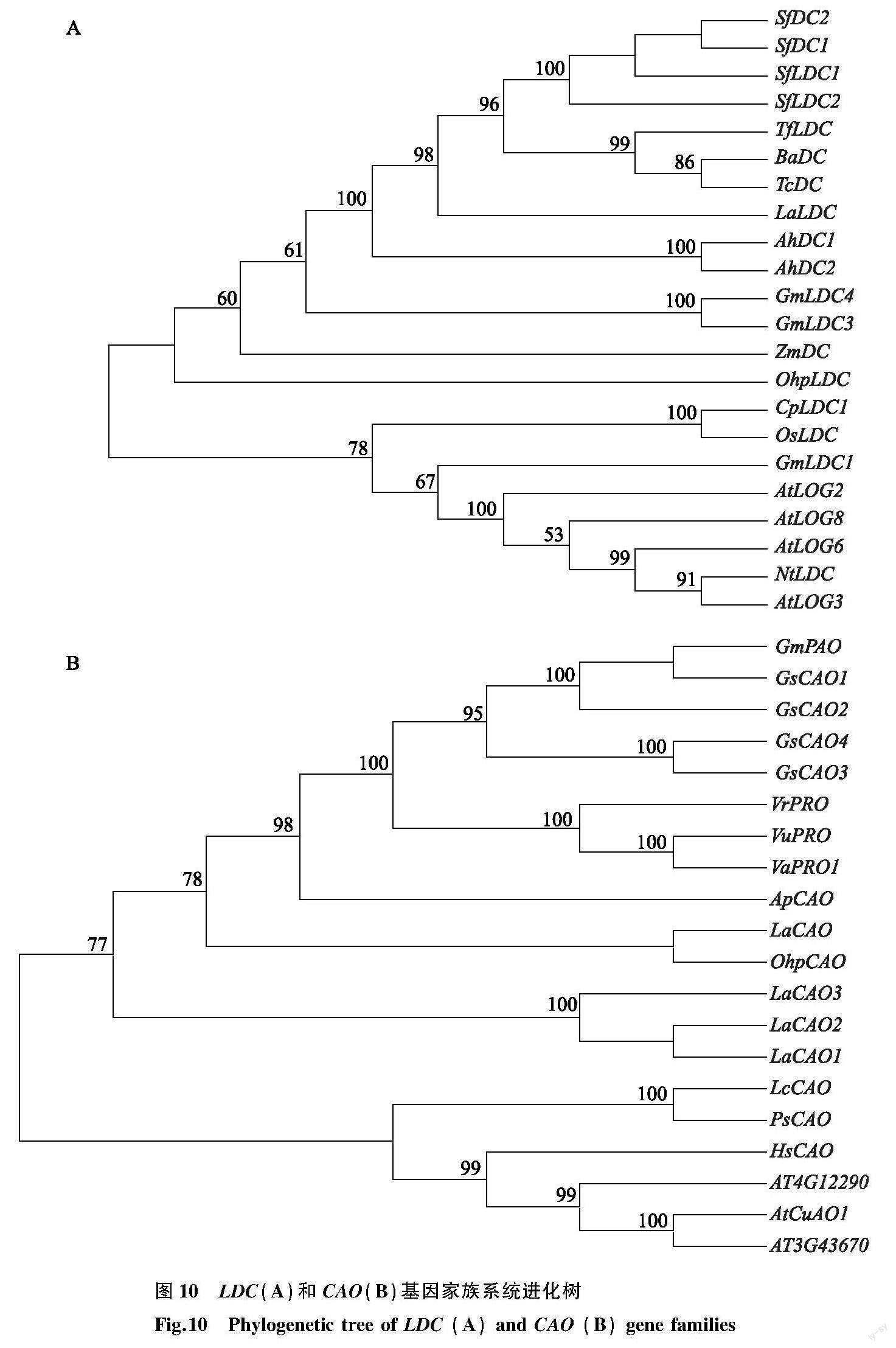
利用NCBI中Blast分析,结果表明花榈木中OhpLDC基因与多个物种的LDC基因高度相似,相似度较高的基因有苦参(Sophora flavescens,GenBank登录号:MW961009.1)、野决明(Thermopsis fabacea,GenBank登录号:AB915698.1)、霍州油菜(Thermopsis chinensis,GenBank登录号:AB647178.1)、蓝花赝靛(Baptisia australis,GenBank登录号:AB647177.1)、落花生(Arachis hypogaea,GenBank登录号:XM_025756574.2)等的赖氨酸/鸟氨酸脱羧酶基因,序列一致性分别为86.51%、84.96%、84.84%、84.24%、77.80%。此外 OhpCAO1 基因与大豆(Glycine soja,GenBank登录号:XM_028389736.1) CAO1 基因的相似度最高,达93.46 %;与相思子(Abrus precatorius,GenBank登录号:XM_027491129.1)、赤豆(Vigna angularis,GenBank登录号:XM_017581226)、熊猫豆(Vigna umbellate,GenBank登录号:XM_047296193.1)、狭叶羽扇豆(Lupinus angustifolius,GenBank登录号:XM_019583671)等的 CAO1 基因也具有较高同源性,序列一致性分别为92.09%、92.24%、93.07%、92.90%。该结果表明OhpLDC和OhpCAO1 是花榈木中赖氨酸脱羧酶和铜胺氧化酶的编码基因。
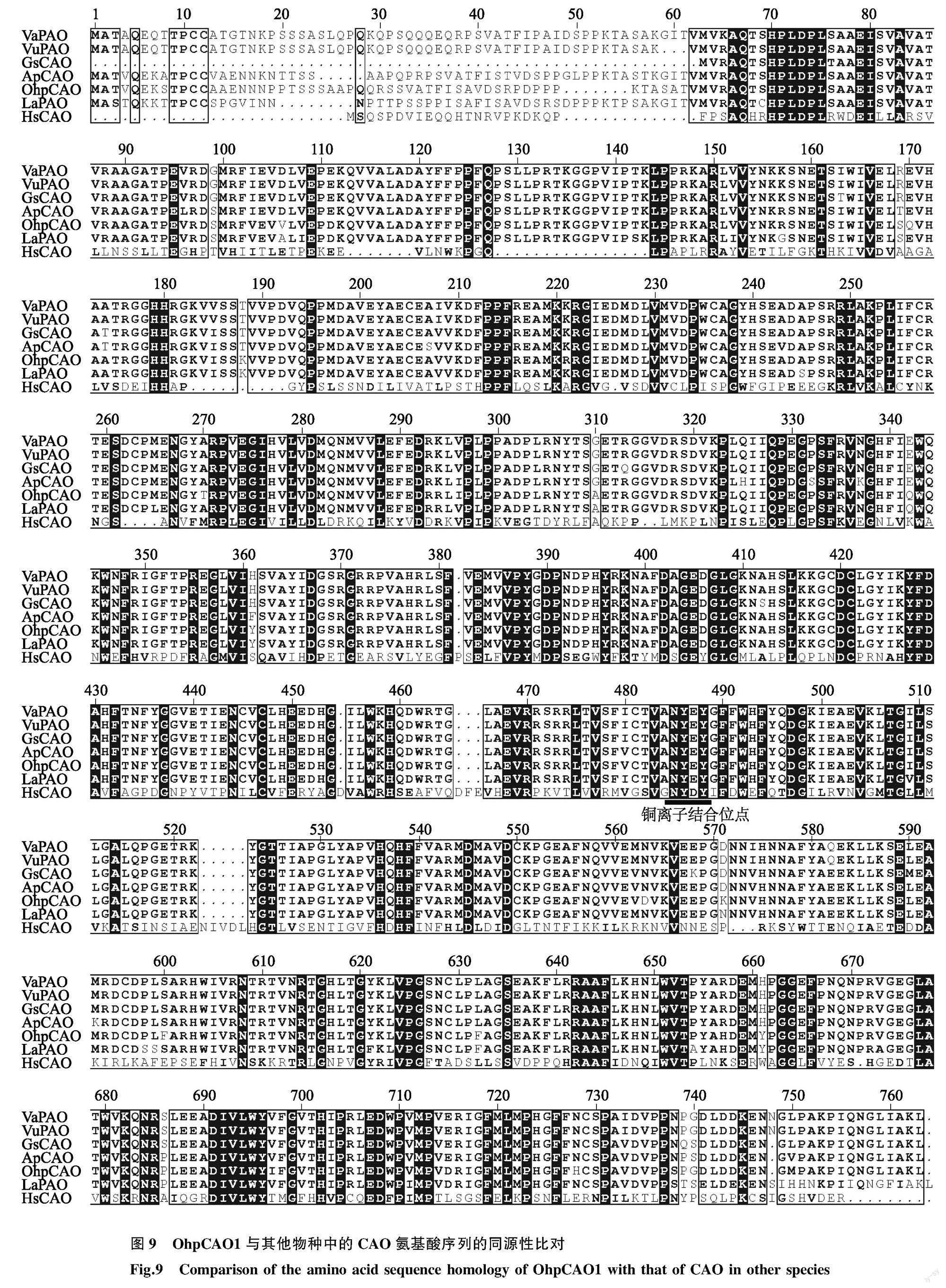
将OhpLDC氨基酸序列与blast结果高度相似的其他植物的赖氨酸脱羧酶氨基酸序列进行比对,结果表明OhpLDC蛋白氨基酸序列中第86到第372位氨基酸与其他植物的赖氨酸脱羧酶具有高度相似性,属于PLP依赖性超家族酶的N端高度保守的PLP结合域和QA植物中保守的C端底物结合位点Phe340(图8)。OhpCAO1氨基酸序列与blast结果同源性较高的几个物种的CAO氨基酸序列进行同源性比对,结果发现与豆科植物的CAO1氨基酸序列第62到第763位氨基酸的相似性非常高,与其他物种的CAO氨基酸序列也相似,发现1个CAO的保守结构“NY-Y”,以及3个组氨酸保守位点(图9)。
为了分析花榈木中OhpLDC和OhpCAO1 与其他物种相关基因的进化关系,采用MEGA 11.0将OhpLDC与多个物种的LDC基因一起构建进化树,并将 OhpCAO1 与多个物种的 CAO1 基因一起构建进化树。结果发现OhpLDC与豆科类植物中的LDC基因亲缘关系较近,表明编码这些赖氨酸脱羧酶的基因在进化上具有较近的亲缘关系。并发现 OhpCAO1 与狭叶羽扇豆的 CAO1 基因亲缘关系最近(图10)。
3 讨 论
QA通常出现在豆科中[20],但也在藜科(Chenopodiaceae)、小檗科(Berberidaceae)、毛茛科 (Ranunculaceae)和茄科(Solanaceae)中被发现[7]。花榈木属于豆科蝶形花亚科植物,对花榈木代谢组进行分析,结果表明花榈木也生成QA,并检测到了白羽扇豆碱、13-羟基羽扇豆碱、鹰爪豆碱、金雀花碱和乙酰金雀花碱5个QA。QA是植物生物碱的一个重要家族,但核心生物合成途径一直未被阐明,目前人们只发现了2个QA生物合成的关键催化酶,赖氨酸脱羧酶(LDC)和铜胺氧化酶(CAO)[21],通过对花榈木转录组数据分析,发现了这2个关键酶的存在,并成功克隆了花榈木的编码赖氨酸脱羧酶和铜胺氧化酶基因,分别命名为OhpLDC、OhpCAO1。对OhpLDC、OhpCAO1 编码的蛋白质氨基酸序列分析,发现OhpLDC属于PLP依赖性超家族酶,含有高度保守的PLP结合域和植物中QA保守的底物结合位点Phe340。Bunsupa等[22]研究结果表明,不同物种LDC蛋白氨基酸序列中的His340到Phe344是LDC与底物结合催化的关键位点。系统进化树分析结果表明,OhpLDC与大豆、苦参和狭叶羽扇豆等豆科植物的LDC基因聚为一类,表明花榈木中OhpLDC基因与这些植物中LDC基因具有共同的起源,也表明LDC具有催化赖氨酸脱羧生成尸胺的酶活性[23],为后续QA的合成提供尸胺。
此外本研究发现 OhpCAO1 在产生QA的植物中进化是比较保守的,含有一个保守结构“NY-Y”,以及3个组氨酸保守位点,是一种活性酶[24]。在系统进化树中发现,花榈木与狭叶羽扇豆亲缘关系最近,表明花榈木的铜胺氧化酶与狭叶羽扇豆的铜胺氧化酶有相似的特性[25]。Yang等[21]发现LaCAO对尸胺具有较高的亲和力,可以将尸胺氧化为1-哌啶,推测 OhpCAO1 也可能催化花榈木中QA生物合成的第二步,为后续QA的合成提供1-哌啶。
本试验克隆了花榈木中QA代谢中相关的2个酶基因OhpLDC、OhpCAO1 ,它们具有组织特异性,在根中表达量较高。与之相对应,通过代谢组学分析发现部分QA是在根中含量较高,表明OhpLDC、OhpCAO1 可能是花榈木根中参与QA生物合成的2个重要基因,QA在根中可能发挥重要的生物学功能。因此,花榈木的编码赖氨酸脱羧酶和铜胺氧化酶基因的克隆和分析對QA生物合成的分子调控具有重要意义,为进一步研究花榈木QA合成与代谢提供了基础,对豆科植物中的木本物种的研究具有重要意义。
参考文献:
[1] 桂 平,龙 鹏. 珍稀树种花榈木研究进展[J]. 贵州农业科学,2021,49(7):98-106.
[2]陈坚波,潘春燕,张慧芳,等. 花榈木中毒急救1例[J]. 浙江中医杂志,2014,49(6):456.
[3]张琳婧,周文娟,倪 林,等. 红豆属植物化学成分及其药理活性研究进展[J]. 中草药,2021,52(14):4433-4442.
[4]POUNY I, BATUT M, VENDIER L, et al. Cytisine-like alkaloids from Ormosia hosiei Hemsl.& E.H. Wilson[J]. Phytochemistry,2014,107:97-101.
[5]SU B, HWANG B Y, CHAI H, et al. Activity-guided fractionation of the leaves of Ormosia sumatrana using a proteasome inhibition assay[J]. Journal of Natural Products,2004,67(11):1911-1914.
[6] XU H, QIU Y, CHEN J, et al. Chemical constituents and their activities from the seeds of Ormosia hosiei[J]. Natural Product Communications,2019,14(7). DOI:10.1177/1934578X19859977.
[7]ROBERTS M F, WINK M. Alkaloids biochemistry ecology and medicinal applications[M]. New York:Plenum Press,1999.
[8]BUNSUPA S, KATAYAMA K, IKEURA E, et al. Lysine decarboxylase catalyzes the first step of quinolizidine alkaloid biosynthesis and coevolved with alkaloid production in Leguminosae[J]. The Plant Cell,2012,24(3):1202-1216.
[8]LIANG L, WANG X, ZHANG X, et al. Sophoridine exerts an anti-colorectal carcinoma effect through apoptosis induction in vitro and in vivo[J]. Life Sciences,2012,91(25/26):1295-1303.
[10]YANG Y, XIU J, ZHANG X, et al. Antiviral effect of matrine against human enterovirus 71[J]. Molecules,2012,17(9):10370-10376.
[11]SHANG X F, MORRIS-NATSCHKE S L, YANG G Z, et al. Biologically active quinoline and quinazoline alkaloids part Ⅱ[J]. Medicinal Research Reviews,2018,38(5):1614-1660.
[12]YANG T, NAGY I, MANCINOTTI D, et al. Transcript profiling of a bitter variety of narrow-leafed lupin to discover alkaloid biosynthetic genes[J]. Journal of Experimental Botany,2017,68(20):5527-5537.
[13]SACCHETTI A, ROSSETTI A. Synthesis of natural compounds based on the[3,7]-diazabicyclo[3.3.1]nonane (bispidine)core[J]. European Journal of Organic Chemistry,2021,2021(10):1491-1507.
[14]GOLEBIEWSKI W M, SPENSER I D. Biosynthesis of the lupine alkaloids Ⅱ. Sparteine and lupanine[J]. Canadian Journal of Chemistry,1988,66(7):1734-1748.
[15]UM Y, JIN M L, LEE D Y, et al. Functional characterization of the β-amyrin synthase gene involved in platycoside biosynthesis in Platycodon grandiflorum[J]. Horticulture, Environment, and Biotechnology,2017,58(6):613-619.
[16]EDDY S R. Profile hidden Markov models[J]. Bioinformatics,1998,14(9):755-763.
[17]PUNTA M, COGGILL P C, EBERHARDT R Y, et al. The Pfam protein families database[J]. Nucleic Acids Research,2012,40(1):290-301.
[18]赵松子,黄建建,高 伟. 油茶氧化鲨烯环化酶基因序列分析[J]. 江西林业科技,2014(1):1-5, 9.
[19]LIU Y, YIN Q, DAI B, et al. The key physiology and molecular responses to potassium deficiency in Neolamarckia cadamba[J]. Industrial Crops and Products,2021,162:113260.
[20]MANCINOTTI D, FRICK K M, GEU-FLORES F. Biosynthesis of quinolizidine alkaloids in lupins:mechanistic considerations and prospects for pathway elucidation[J]. Natural Production Report,2022,39(7):1423-1437.
[21]YANG T, NAGY I, MANCINOTTI D, et al. Transcript profiling of a bitter variety of narrow-leafed lupin to discover alkaloid biosynthetic genes[J]. Journal of Experimental Botany,2017,68(20):5527-5537.
[22]BUNSUPA S, YAMAZAKI M, SAITO K. Quinolizidine alkaloid biosynthesis:recent advances and future prospects[J]. Frontiers in Plant Science,2012,3:239.
[23]李賽男,王雯静,张蓓蓓,等. 蛇足石杉中赖氨酸脱羧酶基因的克隆表达及功能鉴定[J]. 药学学报,2022,57(11):3437-3445.
[24]商二磊,隋正红,刘 源,等. 链状亚历山大藻铜胺氧化酶基因的克隆和表达分析[J]. 中国海洋大学学报(自然科学版),2018,48(11):79-86.
[25]KAMPHUIS L G, HANE J K, NELSON M N, et al. Transcriptome sequencing of different narrow-leafed lupin tissue types provides a comprehensive uni-gene assembly and extensive gene-based molecular markers[J]. Plant Biotechnology Journal,2015,13(1):14-25.
(责任编辑:成纾寒)

