静电排斥表面诱导溶菌酶分子站立
白 姝 李 浩 张 麟
(天津大学化工学院生物工程系,天津300072; 天津大学系统生物工程教育部重点实验室,天津300072)
1 引言
基因工程技术的飞速发展推动重组蛋白质药物在微生物体内的大规模生产.1-3但是,重组蛋白质在原核细胞中的高水平表达往往导致其发生错误的折叠和聚集,4形成被称为包含体(inclusion body)的无活性聚集体,需要通过蛋白质复性过程使其恢复天然构型和生物学活性.5,6因此,蛋白质复性技术开发和理论完善对蛋白质药物生产具有重要意义.7
蛋白质复性的最大挑战就是创造合适的复性条件,最大程度地抑制聚集体生成,从而提高复性收率.8而现阶段重组蛋白质复性仍存在收率低,操作条件较为严苛等问题.近期,Wang等9通过实验研究发现,带有正电荷的离子交换介质Q和DEAE Sepharose FF可以显著促进带正电的溶菌酶复性,而带有负电荷的离子交换介质SP和CM Sepharose FF可以显著促进带负电的牛血清白蛋白复性,并推断与蛋白质带有同种电荷的离子交换介质可以诱导蛋白质在介质表面形成定向排列.此定向排列促使蛋白质之间的静电排斥力增大,从而抑制蛋白质的聚集而促进复性.但是,通过实验研究很难直接证实此机理.而分子动力学(MD)模拟10,11能够直接提供系统的微观结构信息,并能通过数学统计等手段计算系统的宏观性质,已经成为与理论研究和实验研究并行互补的科学研究手段,广泛用于蛋白质相关领域的研究.12迄今为止,已有大量研究者通过分子模拟研究界面吸引诱导的小分子13,14和蛋白质15-23吸附行为,但很少研究界面排斥作用诱导的蛋白质空间分布.
因此,本文将构建静电排斥表面模型以模拟带同种电荷的离子交换介质,通过分子动力学模拟考察蛋白质与静电排斥表面之间的相互作用过程,展示蛋白质在静电排斥表面上的空间取向和构象转换,考察表面所带电荷数的影响规律,揭示同电荷离子交换介质辅助蛋白质复性的微观机理,推动蛋白质在荷电表面折叠和分子相互作用研究.
2 模拟方法
2.1 模拟体系构建
根据蛋白质数据库(http://www.rcsb.org/pdb/)中的晶体衍射结构(PDB ID:3LYZ)构建溶菌酶的全原子模型,24如图1A所示.溶菌酶由129个残基组成,pH=7时带8个正电荷(包括17个带正电荷的残基和9个带负电荷的残基).
本文构建静电排斥表面模型以模拟同电荷离子交换介质,包括基质和配基两部分(参比体系只含基质).基质由正六边形网格结构的平板表示,其中正六边形的边长为0.152 nm.配基的原子组成参照商业化介质的化学结构(https://www.gelifesciences.com/)确定,其键长、键角和二面角及其力常数等结构参数由PRODRG 2.5(http://davapc1.bioch.dundee.ac.uk/cig-bin/prodrg_beta)25生成,而原子的部分电荷通过量子化学计算和类似结构比对调整确定,标示于图1B中.其后,将配基连接到基质,根据实验条件调整配基密度为0.22 mmol·mL-1,如图1C所示.

图1 溶菌酶和静电排斥表面的全原子模型Fig.1 All-atom models of the lysozyme and the surface with electrostatic repulsion
构建模拟体系时,首先将静电排斥表面放入6.84 nm×6.91 nm×18.00 nm的长方体盒子底部(参比体系的盒子高度为10.00 nm).然后将1个溶菌酶分子放置于表面上方,其质心距离表面约2 nm.进而添加水分子,并添加适量反离子使模拟体系呈电中性.最后将模拟体系放入6.84 nm×6.91 nm×100 nm的长方体盒子中心进行模拟.本文共构建4个模拟体系,包括静电排斥表面体系(标记为Q),参比体系(标记为control),介质所带电荷增大30%(标记为1.3Q)和减小25%(标记为0.75Q,体系高度为12 nm)的体系,其具体组成列于表1.
2.2 分子动力学模拟
分子动力学模拟利用GROMACS 4.0.5程序(http://www.gromacs.org/)26,27完成,使用GROMOS96 43A1力场.28模拟采用NVT系综,通过v-rescale温度偶联法29控制温度为298.15 K.采用半步蛙跳法积分,步长为2 fs.静电相互作用计算采用PME(particle-mesh Ewald)算法,其截断距离设为1.2 nm.范德华作用计算采用cut-off算法,其截断半径设为1.2 nm.近邻原子列表截断半径设为1.2 nm.粒子运动的初始速度根据模拟温度298.15 K下的Maxwell分布产生.对模拟盒子施加x,y,z的三个方向的周期性边界条件.
本文采用最速下降法进行能量最小化,能量阀值设置为2000 kJ·mol-1.然后,进行20 ns的分子动力学模拟,得到模拟轨迹用于后续分析.为了保证模拟结果的准确性,本文中对体系Q执行3次平行模拟.所有模拟均使用曙光TC2600刀片服务器完成.文中蛋白质构象图利用RASMOL软件30绘制.
2.3 数据分析方法
2.3.1 蛋白质空间取向分析
本文定义角度θ为蛋白质的偶极和z坐标轴之间的夹角,以定量描述蛋白质在静电排斥表面上的空间取向.当θ=0°时,蛋白质的偶极垂直于静电排斥表面(平行于+z坐标轴).当θ为90°或-90°时,蛋白质的偶极平行于静电排斥表面(偶极指向+x方向定义为正).通过自编程序计算蛋白质的θ值随模拟时间的变化.

表1 模拟体系的参数Table 1 Parameters of simulation systems
2.3.2 分子间势能分析
利用GROMACS软件包的g_energy程序进行势能分析,包括Lennard-Jones(LJ)势能(标记为ELJ)和库仑势能(标记为EC).
2.3.3 二级结构分析
为了描述MD模拟过程中蛋白质的构象转化,利用Define Secondary Structure of Proteins(DSSP)方法31及GROMACS软件包的do_dssp程序完成其二级结构分析.
2.3.4 均方根偏差计算
利用GROMACS软件包的g_rms程序计算溶菌酶分子相比于其天然结构的均方根偏差(RMSD),如式(1)所示.RMSD值越小,表明溶菌酶天然结构保持更好.
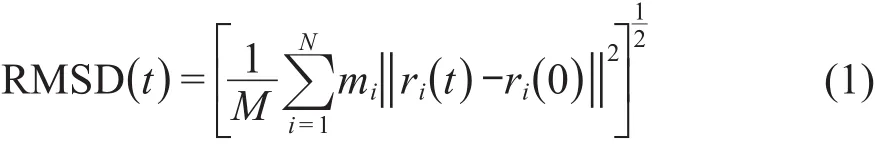
2.3.5 回转半径计算
利用GROMACS软件包的g_gyrate程序计算溶菌酶的回转半径(Rg)以描述其结构紧密程度,如式(2)所示.Rg值越小,表明蛋白质的结构越紧密.
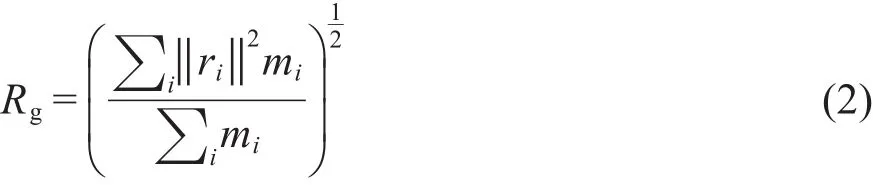
2.3.6 自由能表面图
参照文献20,32计算蛋白质处于特定θ和zcom的概率分布P(θ,zcom),其中zcom为蛋白质质心的z坐标值.通过模拟轨迹采样和自编程序计算完成.
3 结果与讨论
3.1 溶菌酶空间取向分析
首先通过系统快照(snapshots)直观分析模拟体系的微观构象随模拟时间的变化,包括溶菌酶的构象,空间取向以及位置等,如图2所示.此处,着重分析溶菌酶的偶极取向,偶极已放大以便清晰显示,在图中以棒状标出,其偶极方向由红色指向蓝色(印刷版中由黑色指向灰色).注意,此处仅显示一条典型轨迹结果,其余平行模拟轨迹的分析结果一致,故略去.
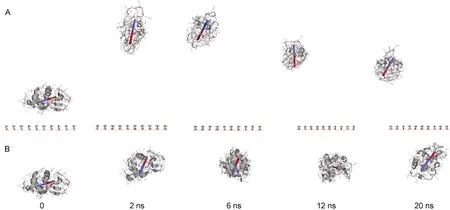
图2 溶菌酶在静电排斥表面上的构象随模拟时间变化图Fig.2 Snapshots of lysozyme over the surface with electrostatic repulsion as a function of simulation time
图2A显示,溶菌酶的初始位置靠近静电排斥表面,其偶极方向接近平行方向,溶菌酶含有丰富的α-螺旋和β-折叠等二级结构.其后,蛋白质受到表面的排斥作用而远离.2 ns时,蛋白质已经远离表面并呈现“站立”姿势,其偶极方向接近垂直方向.此时蛋白质整体结构被拉伸,其二级结构部分丧失而转变为相对柔性的卷曲结构.6 ns时,蛋白质依然远离表面,其偶极方向有轻微偏转,但依然接近垂直方向,即蛋白质一直保持“站立”姿势.其后,由于蛋白质远离表面,且偶极旋转会进一步降低表面对其的排斥作用,蛋白质可以再次靠近表面.例如,12 ns时,蛋白质略微靠近表面,而其偶极旋转到几乎完全垂直的方向,表明蛋白质靠近表面时,静电相互作用增强利于诱导形成“站立”姿势.但同时也需注意,20 ns时,蛋白质进一步接近表面,但其偶极反而偏离垂直方向,表明蛋白质的取向还受到其他因素,如溶剂的影响,存在一定波动.
在参比体系(图2B)中,由于表面呈电中性,和蛋白质之间不存在静电相互作用.因此,蛋白质一直保持在表面附近,在溶剂作用下存在一定的移动和转动,但其偶极方向变化并无明显规律.同时,在参比体系中,蛋白质的二级结构一直很好保持,表明蛋白质自身结构在溶液中比较稳定.
因此,微观构象分析表明,静电排斥表面推动蛋白质远离.在此过程中,表面诱导蛋白质逐渐“站立”,使其偶极保持在垂直于表面的方向.尽管蛋白质的偶极方向存在一定的摆动,但总体而言都接近垂直方向,即蛋白质基本保持“站立”姿势.
进而考察角度θ(定义和计算流程见2.3.1节)的变化以定量描述蛋白质在静电排斥表面作用下的空间取向分布及其变化情况,同时通过蛋白质质心的z坐标值定量描述蛋白质与表面的相对位置变化,如图3所示.此处,体系Q的数据为3次平行模拟结果的平均值.
在静电排斥表面上,蛋白质的θ值从初始的-105°迅速变化到0°左右,即蛋白质从“平躺”姿势迅速转变为“站立”姿势.同时其zcom迅速增大,表明蛋白质迅速远离表面.相比而言,zcom变化更缓慢,即其变化速率小于θ的变化速率,表明蛋白质在受到排斥而远离表面的过程中即已形成“站立”姿势.在2 ns附近时,蛋白质的zcom呈现峰值,此时蛋白质的θ在0°附近波动,但都维持在-60°到60°的区间内,表明蛋白质在远离表面时,由于静电排斥减弱,蛋白质的偶极方向存在一定的摆动.其后,蛋白质的zcom值略微降低,在3.0到5.5 ns期间呈现平台期,其zcom值维持在8 nm,而此时蛋白质的θ值基本维持在0°,表明蛋白质依然很好维持“站立”姿势.此后,蛋白质略微接近表面,而θ值的变化幅度增大,但依然维持在0°附近.到12 ns后,蛋白质的θ值变化幅度进一步增大,表明蛋白质从远离静电排斥表面的位置逐渐靠近表面时,由于静电排斥作用增强,蛋白质的偶极方向需要重新调整以达到新的平衡状态.其概率分布(图3B)表明,蛋白质的zcom主要介于5到10 nm之间,表明蛋白质受到排斥而远离表面,其θ角主要分布于-60°到60°之间,确证蛋白质形成“站立”姿势.

图3 溶菌酶空间取向和位置随模拟时间变化图Fig.3 Orientation and the location of lysozyme as a function of simulation time
在参比体系中,蛋白质一直维持在表面附近,其zcom值变化较小,可以认为蛋白质只是在其初始位置附近波动而并无明显移动(图3C的统计结果给出明确证据).在模拟初始阶段,蛋白质的θ值也无明显变化,只是在其初始值附近波动.2.5 ns后蛋白质的θ值发生明显变化,但分析表明都是无规的转动导致,从图2的系统快照中也可得到直观证明.15 ns后,蛋白质的偶极方向又恢复到初始状态并存在轻微波动.
因此,θ值和zcom值随模拟时间的变化确证蛋白质在静电排斥表面作用下迅速远离,而在远离过程中其偶极旋转,形成特定的“站立”姿势.此结果直接验证实验推测,表明蛋白质确实在静电排斥表面形成定向排布,有助于增强蛋白质之间的静电排斥作用而抑制溶菌酶聚集.但同时需注意,蛋白质和表面间的距离,以及溶剂均影响蛋白质的偶极方向使其存在一定的摆动.
然后,通过蛋白质和表面间相互作用势能分析,考察蛋白质形成特定“站立”姿势的关键作用力,如图4所示.此处,体系Q的数据为3次平行模拟结果的平均值.

图4 溶菌酶和介质间的LJ势能和库仑势能随模拟时间变化图Fig.4 Lennard-Jones(LJ)and Coulomb potential energies between lysozyme and resin as a function of simulation time
仅在模拟初始阶段观察到静电排斥表面和蛋白质之间存在很小的LJ势能,约-1.2 kJ·mol-1.其后,LJ势能持续为0,这是因为蛋白质被排斥远离表面(图3)而LJ势能是短程相互作用,随距离迅速衰减.而蛋白质和表面之间存在很大的静电相互作用势能,接近140 MJ·mol-1.静电相互作用势能随模拟时间迅速减小,到2 ns附近时达到最小值83 MJ·mol-1,这是因为蛋白质在静电排斥作用下迅速远离表面(图3),使静电势能迅速降低.其后,随着蛋白质再次靠近表面(图3),蛋白质和表面间的静电相互作用势能再次上升,最终达到105 MJ·mol-1.而在参比体系中,蛋白质与表面之间的LJ势能和静电作用势能都基本为0,因而表面对蛋白质并无作用,蛋白质呈现溶液主体行为,与图2和图3的结果完全吻合.
因此,通过势能分析可以确证,表面通过静电排斥作用驱动蛋白质远离并诱导其形成特定“站立”姿势.为验证此结论,本文还构建带负电的静电表面,发现带正电的溶菌酶分子在静电吸引作用下直接吸附于负电表面(数据未显示).如果通过调整溶液pH值使溶菌酶也带负电(如pH=14),则溶菌酶同样在静电排斥作用下迅速远离,再次确证静电排斥作用是关键驱动力.但同时也发现,pH=14时,溶菌酶中只有带负电的氨基酸残基而无带正电的氨基酸残基,无法形成明显偶极而导致无法形成特定“站立”姿势(数据未显示),表明正负电荷的不均匀分布是特定“站立”姿势形成的关键因素.
3.2 溶菌酶构象分析
利用DSSP方法确定蛋白质二级结构随模拟时间的变化,如图5所示.结果表明,溶菌酶的主要二级结构是α-螺旋,间杂零星β-折叠结构.参比体系中,各颜色条带随模拟进行都比较稳定.例如,10号残基、30号残基和95号残基处的α-螺旋在模拟过程中一直保持完好,而110号残基处的α-螺旋结构存在短暂消失的情况,但很快重新生成,且一直维持到模拟终点,表明蛋白质的二级结构得到很好保持.
但在静电排斥表面体系中,各颜色条带均存在消失或增加的反复过程,总体呈现不连续分布状态,表明蛋白质的二级结构存在明显变化.其变化细节表明蛋白质中多处的α-螺旋结构丧失.参照图2的系统快照分析,在静电排斥表面作用下,蛋白质中带正电荷的残基受到排斥而试图远离表面,但带负电荷的残基受到吸引而试图靠近表面,因此蛋白质受力不均而被轻微拉伸,其内部原子相对位置的变化造成其二级结构的破坏.从图5中也可看出,带电残基处的二级结构变化最为剧烈.例如,天然溶菌酶中ARG5-LYS13形成α-螺旋结构,在参比体系中此结构很好保持(图5B).而在静电排斥表面体系中,由于ARG5和LYS13均带电,受到表面的影响而导致此螺旋结构不稳定,甚至整体螺旋结构被破坏(图5A).同时需注意,随着蛋白质位置和偶极方向的调整,蛋白质构象也存在细微调整过程.
因此,计算蛋白质的RMSD和Rg值模拟时间的变化,以通过结构偏差程度及其整体结构紧密程度(详见2.3节中数据分析方法描述)定量考察蛋白质的构象变化,如图6所示.此处数据为3次平行模拟结果的平均值.
参比体系中,蛋白质的RMSD值在模拟开始时轻微增大,表明蛋白质在溶液环境中存在细微结构调整,其后则一直在0.10 nm上下波动.蛋白质的Rg值随模拟时间轻微降低,然后维持在1.35 nm上下波动.二者的数值都很小且波动幅度也很小,表明参比体系中蛋白质并无明显构象变化,与图2的系统快照和图5的二级结构分析结果一致.
而在静电排斥表面体系中,蛋白质的RMSD值在模拟初始阶段即迅速增大,并进而维持在0.5 nm上下波动且持续到模拟终点,表明蛋白质确实在模拟初始的排斥远离过程中受到很大的结构扰动,使其结构明显偏离天然态.蛋白质的Rg值在模拟初始阶段也迅速上升,高于参比体系,表明蛋白质结构确实被拉伸而使其整体结构松散.其后,Rg值很快下降到与参比体系相当,进而继续下降并稳定在1.30 nm上下波动,其稳定值低于参比体系中的Rg值.综合RMSD值和Rg值的变化表明蛋白质被排斥远离表面后,由于距离增大(图3)导致静电排斥力减弱(图4),蛋白质受到外界干扰减小,开始构象调整,呈现整体塌缩的变化趋势.然而,蛋白质的最终结构仍然偏离天然结构,但其偏差数值并不大,表明其最终骨架结构还能较好保持,与图2的系统快照分析结果一致.因而,静电排斥表面对溶菌酶的分子结构有一定扰动,尽管整体骨架结构变化不大,也可能造成其活性丧失.
因此,综合分析二级结构,RMSD和Rg值的结果表明,参比体系中,蛋白质的构象很好保持.而在静电排斥表面体系中,蛋白质由于受力不均,存在先拉伸后塌缩的变化过程,其二级结构遭到破坏,但主体骨架结构较好保持.
3.3 表面所带电荷数的影响分析
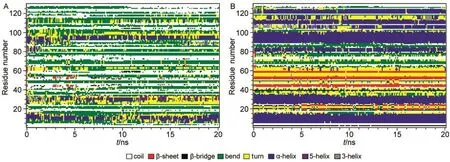
图5 分子动力学模拟中溶菌酶分子的二级结构演化Fig.5 Secondary structure evolution of lysozyme during the molecular dynamics simulation
上述结果表明,静电排斥表面能够诱导蛋白质形成“站立”姿势,而静电排斥作用是主要驱动力.因此,表面所带电荷数对蛋白质的空间取向应该具有重要影响.本文构建电荷数增大的静电排斥表面模型(1.3Q)以及电荷数减小(0.75Q)的模型,分析其与蛋白质的相互作用及其对蛋白质的空间取向和构象变化的影响,如图7、8、9所示.
图7显示,在体系1.3Q中,蛋白质的θ值一直维持在0°上下波动,且波动幅度很小,表明表面所带电荷数增大使其静电排斥作用增强,蛋白质能够更好形成并保持“站立”姿势.蛋白质的zcom值迅速增大,到6 nm处增大变缓,最终维持在10 nm上下波动.其概率分布(图7B)表明,蛋白质的θ角更集中于-60°到60°之间.因此,相比于体系Q(图3),蛋白质的排斥远离过程更快且最终稳定距离更远,但其“站立”姿势也更稳定,表明表面所带电荷数增大确实有利于蛋白质的“站立”姿势形成和稳定.
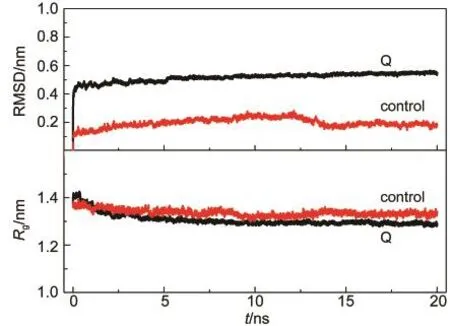
图6 溶菌酶中Cα原子的RMSD和Rg随模拟时间变化图Fig.6 RMSD and the Rgvalues for Cαatoms of lysozyme as a function of simulation time
而在体系0.75Q中,蛋白质的θ值主要维持在-50°上下波动且波动幅度很大.在17至20 ns期间,蛋白质远离表面,其θ值发生明显变化,在-120°至-60°区间内波动,即偶极平行于表面,蛋白质呈现“平躺”姿势.蛋白质的zcom值到1.6 ns时增大到5.7 nm,变化比体系1.3Q和体系Q中缓慢.其后,zcom值降低,到5 ns时再次上升,最终达到7.2 nm.因此,体系0.75Q中,模拟终点时zcom值小于体系1.3Q的情况,表明静电排斥减弱,蛋白质稳定位置更靠近表面.但需要注意的是,虽然体系0.75Q中模拟终点的zcom值大于体系Q,但其概率分布(图7C)表明,在zcom值较大区域蛋白质的θ角偏离-60°到60°区域,表明蛋白质此时并不存在“站立”姿势.而其主要分布区域是在zcom=5 nm处,此时其θ=-60°,表明体系0.75Q中,蛋白质也形成“站立”取向,但趋势更弱.
图8显示,在1.3Q和0.75Q两体系中,蛋白质和表面之间的LJ势能都基本为0,与体系Q相同,表明在静电排斥表面体系中,蛋白质和表面间距离较远,而LJ势能为短程相互作用,随距离增大迅速衰减.而静电相互作用很强,在两体系中均呈现迅速下降,然后趋于平稳的变化趋势,分别稳定在83和19 MJ·mol-1.势能变化表明蛋白质的位置和空间取向变化受静电排斥作用调控,且受到表面所带电荷数影响.表面电荷数增大,蛋白质的平衡位置更远离表面,且利于“站立”姿势的形成和维持.
图9显示,在1.3Q和0.75Q两体系中,蛋白质的RMSD值均随模拟进行而迅速上升,然后分别在0.70和0.15 nm上下波动.而两体系中蛋白质的Rg值略微不同,体系1.3Q中Rg值高于初始天然态,且存在显著增大再回落的过程(如2.6 ns时).而体系0.75Q中Rg值基本保持在天然态的Rg值.结合RMSD和Rg值分析表明,随着所带电荷数增大,表面对蛋白质的结构扰动加剧.反之,电荷数降低的表面对蛋白质的结构扰动减弱,使蛋白质能够更好保持其天然结构.
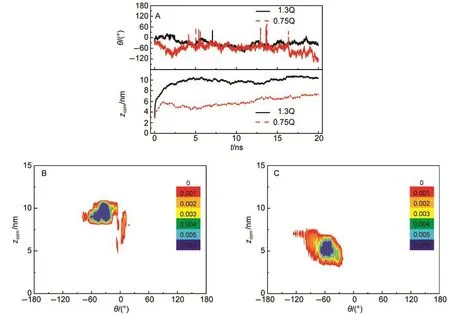
图7 介质电荷数对溶菌酶的空间取向和位置的影响Fig.7 Effects of charge number of resin on the orientation and location of lysozyme
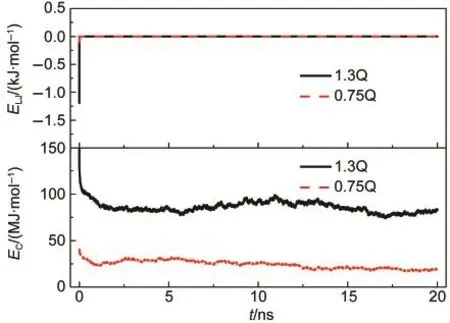
图8 介质电荷数对溶菌酶和介质间相互作用势能的影响Fig.8 Effects of charge number of resin on the interaction energies between the lysozyme and the resin
4 结论

图9 介质电荷数对溶菌酶构象的影响Fig.9 Effects of charge number of resin on the conformation of lysozyme
以同电荷离子交换介质促进溶菌酶等蛋白质复性的实验结果为背景,通过静电排斥表面模拟同电荷离子交换介质,构建溶菌酶在静电排斥表面体系中的全原子模型,利用分子动力学模拟考察蛋白质在表面上的空间取向和构象变化,并通过相互作用势能分析深入剖析静电排斥表面的作用机理和细节.结果表明,蛋白质在表面的静电排斥作用下迅速远离,在此过程中逐渐形成其偶极垂直于表面的“站立”姿势.此定向排布有助于增强蛋白质之间的静电排斥作用而抑制溶菌酶聚集.但是,由于蛋白质中带正电荷的残基和带负电荷的残基受力方向相反,导致蛋白质结构拉伸而破坏其二级结构.研究发现表面所带电荷数对蛋白质的空间取向和构象变化有显著影响.电荷数增大使得表面对蛋白质的静电排斥作用增强,能够诱导蛋白质更快更好地形成“站立”姿势.而表面所带电荷数降低,则不利于蛋白质维持“站立”姿势.同时,本文的模拟中尚有四个影响因素需要在后续研究中考虑.其一,模拟体系中的大量溶剂分子会影响蛋白质的构象和空间取向调整.其二,本文研究的排斥性界面不同于吸附性界面.吸附性界面作用下,蛋白质靠近界面使得相互作用增强,因而利于形成明显的空间取向分布.而排斥性界面作用下,蛋白质远离界面,使得相互作用减弱,不利于蛋白质形成明显空间取向,造成排斥性界面研究的困难.其三,限于计算量和模拟体系复杂度的考虑,本文仅模拟单个溶菌酶分子,能够确证静电排斥表面诱导蛋白质形成定向排布,但无法直接阐释表面对蛋白质分子间相互作用的影响,将在后续研究中开展.其四,本文直接用晶体结构建模计算,后续研究中可以先通过一定时间的模拟获得溶菌酶在“自由”状态下的构象以消除溶液条件和力场的影响.总体而言,本文通过分子动力学模拟完整阐释蛋白质在静电排斥表面上的空间取向和构象变化过程,考察此过程的微观细节,并能确定表面电荷数等参数的影响规律,将有助于推动蛋白质在荷电表面折叠和分子相互作用研究.
(1) Durocher,Y.;Butler,M.Curr.Opin.Biotech.2009,20,700.doi:10.1016/j.copbio.2009.10.008
(2)Manning,M.C.;Chou,D.K.;Murphy,B.M.;Payne,R.W.;Katayama,D.S.Pharm.Res.2010,27,544.doi:10.1007/s11095-009-0045-6
(3) Ferrer-Miralles,N.;Domingo-Espin,J.;Corchero,J.L.;Vazquez,E.;Villaverde,A.Microb.Cell.Fact.2009,8,17.doi:10.1186/1475-2859-8-17
(4) Fink,A.L.Fold.Des.1998,3,R9.
(5) Middelberg,A.R.Trends Biotechnol.2002,20,437.doi:10.1016/S0167-7799(02)02047-4
(6) Baldwin,R.L.J.Mol.Biol.2007,371,283.doi:10.1016/j.jmb.2007.05.078
(7)Chen,Y.W.;Ding,F.;Nie,H.F.;Serohijos,A.W.;Sharma,S.;Wilcox,K.C.;Yin,S.Y.;Dokholyan,N.V.Arch.Biochem.Biophys.2008,469,4.doi:10.1016/j.abb.2007.05.014
(8) Lu,D.;Liu,Z.Annu.Rep.Prog.Chem.,Sect.C 2010,106,259.doi:10.1039/b903487k
(9)Wang,G.;Dong,X.;Sun,Y.Biotechnol.Bioeng.2011,108,1068.doi:10.1002/bit.23038
(10) Karplus,M.Biopolymers 2003,68,350.
(11) Karplus,M.;McCammon,J.A.Nat.Struct.Biol.2002,9,646.doi:10.1038/nsb0902-646
(12) Bai,H.J.;Lai,L.H.Acta Phys.-Chim.Sin.2010,26,1988.[白红军,来鲁华.物理化学学报,2010,26,1988.]doi:10.3866/PKU.WHXB20100725
(13)Zhang,X.R.;Wang,W.C.Acta Phys.-Chim.Sin.2002,18,680.[张现仁,汪文川.物理化学学报,2002,18,680.]doi:10.3866/PKU.WHXB20020803
(14) Xiang,Z.H.;Wang,W.C.;Cao,D.P.Scientia Sinica Chimica 2012,42,235.[向中华,汪文川,曹达鹏.中国科学:化学,2012,42,235.]doi:10.1360/032011-297
(15) Kubiak-Ossowska,K.;Mulheran,P.A.Langmuir 2010,26,15954.doi:10.1021/la102960m
(16) Ravichandran,S.;Madura,J.D.;Talbot,J.J.Phys.Chem.B 2001,105,3610.doi:10.1021/jp010223r
(17) Carlsson,F.;Hyltner,E.;Arnebrant,T.;Malmsten,M.;Linse,P.J.Phys.Chem.B 2004,108,9871.doi:10.1021/jp0495186
(18)Slater,G.W.;Holm,C.;Chubynsky,M.V.;de Haan,H.H.;Dube,A.;Grass,K.;Hickey,O.A.;Kingsburry,C.;Sean,D.;Shendruk,T.N.;Nhan,L.X.Electrophoresis 2009,30,792.doi:10.1002/elps.v30:5
(19) Chandrasekhar,I.;Kastenholz,M.;Lins,R.D.;Oostenbrink,C.;Schuler,L.D.;Tieleman,D.P.;van Gunsteren,W.F.Eur.Biophys.J.Biophy.2003,32,67.
(20) Zhang,L.;Zhao,G.F.;Sun,Y.J.Phys.Chem.B 2009,113,6873.doi:10.1021/jp809754k
(21) Zhang,L.;Zhao,G.F.;Sun,Y.J.Phys.Chem.B 2010,114,2203.doi:10.1021/jp903852c
(22)Kang,K.;Lu,D.N.;Liu,Z.Chin.J.Chem.Eng.2012,20,284.
(23) Shen,L.M.;Lin,D.Q.;Mei,L.H.;Yao,S.J.Chinese Journal of Bioprocess Engineering 2006,4,29.[沈立民,林东强,梅乐和,姚善泾.生物加工过程,2006,4,29.]
(24) Diamond,R.J.Mol.Biol.1974,82,371.doi:10.1016/0022-2836(74)90598-1
(25)Schuttelkopf,A.W.;vanAaltern,D.M.F.Acta Crystallogr.D 2004,60,1355.doi:10.1107/S0907444904011679
(26) Berendsen,H.J.;van der Spoel,D.;van Drunen,R.Comput.Phys.Commun.1995,91,43.doi:10.1016/0010-4655(95)00042-E
(27) Lindahl,E.;Hess,B.;van der Spoel,D.J.Mol.Model.2001,7,306.
(28)Mackerell,A.D.J.Comput.Chem.2004,25,1584.
(29) Bussi,G.;Donadio,D.;Parrinello,M.J.Chem.Phys.2007,126,014101.doi:10.1063/1.2408420
(30) Sayle,R.;Milnerwhite,E.Trends Biochem.Sci.1995,20,374.doi:10.1016/S0968-0004(00)89080-5
(31) Kabsch,W.;Sander,C.Biopolymers 1983,22,2577.
(32)Li,W.F.;Zhang,J.;Wang,J.;Wang,W.J.Am.Chem.Soc.2008,130,892.doi:10.1021/ja075302g
——《势能》

