利用TALENs和手工克隆技术高效获得GHR基因敲除巴马猪
李飞达,李勇,刘欢,张欢欢,刘楚新,张兴举,窦红伟,杨文献,杜玉涛
1. 深圳华大基因研究院,深圳 518083;
2. 深圳华大方舟生物技术有限公司,深圳 518083;
3. 深圳动物基因组辅助育种工程实验室,深圳 518083
利用TALENs和手工克隆技术高效获得GHR基因敲除巴马猪
李飞达1,3,李勇1,2,3,刘欢1,3,张欢欢1,3,刘楚新1,3,张兴举1,3,窦红伟2,杨文献2,杜玉涛1,2,3
1. 深圳华大基因研究院,深圳 518083;
2. 深圳华大方舟生物技术有限公司,深圳 518083;
3. 深圳动物基因组辅助育种工程实验室,深圳 518083
DNA编辑技术是基因靶向修饰技术的研究热点,已广泛应用于生物医学和农业研究。然而,传统基因打靶技术存在效率低、成本高、工作量大等缺点,其应用受到了极大的限制。文章利用最近发展起来的新型人工核酸酶——转录激活因子样效应物核酸酶(Transcription activator-like effector nuclease, TALENs)介导的基因组定点修饰技术,通过构建特异识别猪生长激素受体(GHR)基因的TALENs表达载体,共转染巴马胎猪成纤维细胞系,酶切鉴定G418抗性克隆细胞株的基因修饰效率为46.2%,其中2个为双等位基因敲除的克隆细胞株。以双等位基因敲除的克隆细胞株为核供体,利用手工克隆技术制备GHR-KO巴马猪克隆胚,第6 d囊胚率为43.5%,654枚体外发育的囊胚移植6头受体母猪,共获得10头存活的GHR-KO巴马仔猪,其中7头为双等位基因敲除。体重检测结果显示,第 20周龄的 GHR-KO巴马猪体重仅为对照巴马猪的 50%。研究结果表明,TALENs和手工克隆技术能够高效制备出基因敲除大动物。GHR-KO巴马猪的成功制备为研究猪GHR基因生理功能以及人类侏儒症分子机理提供了重要模型,也证明该技术比传统基因打靶和克隆技术具备更简捷、快速、高效,更易于在生物医学和农业基因靶向修饰研究中推广。
基因敲除;TALENs;手工克隆;GHR-KO
自20世纪80年代末诞生靶向性基因敲除小鼠以来[1,2],经过 20多年的发展,基因打靶技术逐渐成为科研工作者选择性修改基因组和探讨基因功能的必备工具。由于传统的基因敲除技术依靠细胞自身的DNA断裂修复机制,打靶效率极其低下,人们一直在设法寻求新的提高基因打靶效率的方法。人工构建的核酸内切酶——锌指核酸酶(Zinc-finger nucleases, ZFNs)能够识别并切割靶DNA序列,造成DNA双链断裂,从而启动细胞损伤修复机制,从而大大提高基因敲除效率。然而设计并构建一个剪切活性高、特异性好的ZFNs需要很大的工作量,且很多基因位点难以找到合适的ZFN靶点,大大限制了ZFNs的应用[3~5]。
近几年发展起来的靶向基因编辑技术——转录激活因子样效应物核酸酶(Transcription activator-like effector nuclease, TALENs)技术,不仅具备ZFNs相似的特异性识别和切割靶DNA区域,还具有易于构建、特异性高、细胞毒性低等特点[3,5~8],至今已成功应用于动物、植物、微生物等多个物种[3,5,9]。
小型猪在解剖、代谢、生理方面,如心血管系统、消化系统、皮肤系统、骨骼发育、营养代谢等,与人类极为相似,是人类比较医学中较好的实验动物材料[5,10]。生长激素受体(Growth hormone receptor, GHR)是一种由单一基因编码的跨膜蛋白,是细胞因子受体超家族成员之一。GHR在动物生长发育及新陈代谢中发挥重要作用,其功能缺失会导致动物生长发育迟缓[11,12]。猪GHR基因位于16号染色体上,包含10个外显子,全长16.14 kb,cDNA为1925 bp,共编码 638个氨基酸,包括胞外域(生长激素结合域)245个氨基酸、跨膜域30个氨基酸和胞内域345个氨基酸[13]。胞外区缺失是GHR基因功能缺失的主要原因[14~16],GHR基因胞外区包含 1~6号外显子。因此,本研究利用TALENs特异性识别并剪切GHR基因胞外编码区2号外显子,形成GHR胞外区突变,导致 GHR功能缺失,通过手工克隆技术制备GHR-KO巴马猪,研究GHR基因在调控猪生长发育中的生理作用,并为人类 Laron综合症的分子机制研究提供小型猪疾病动物模型。
1 材料和方法
1.1 材料
细胞培养相关耗材为BD Falcon公司产品,相关试剂为Life technologies公司产品,手工克隆耗材为Nunc公司产品。巴马胎猪成纤维细胞系为本实验室保存,内切酶为NEB公司产品,PCR相关试剂为TAKARA公司产品。
1.2 方法
1.2.1 TALENs位点设计
利用在线软件设计TALENs位点(https://tale-nt. cac.cornell.edu/node/add/talen)。猪GHR基因TALENs位点为5′-TCCTTGTCAGAGCATCTCAGAGTCTGCAG AGAGTTCATCCAGGCCTAGAGA- 3′,其中GHR-TALEN1识别5′-TCCTTGTCAGAGCATCTC-3′,GHR-TALEN2识别5′-TCATCCAGGCCTAGAGA-3′的反向互补序列,中间间隔区下划线为PstⅠ酶切位点,用于检测TALENs剪切活性。
1.2.2 TALENs载体构建
本研究采用的是Bedell等[17]和Doyon等[18]所采用的GoldyTALENs载体结构,即截短型的TALE蛋白,包含136个氨基酸残基的N-端和63个氨基酸残基的 C-端,并在 C-端与 FokⅠ核酸酶融合(在FokⅠ上分别突变3个氨基酸,上下游分别为FokⅠ-ELD和FokⅠ-KKR)。N端连接上NLS序列,N端与C端之间加2个IIS型BsmBⅠ酶切位点用于各RVD模块的连接。将设计好的GoldyTALENs序列进行全序列合成(上海捷瑞生物工程有限公司)。各RVD模块(addgene,1000000024)通过Golden Gate方法连接到上游 pTAL-ELD(GHR-TALEN1)和下游pTAL-KKR (GHR- TALEN2)表达终载体中[19,20]。TALENs载体构建好后测序鉴定,测序正确的GHR-TALEN1和GHR- TALEN2质粒用无内毒素质粒提取试剂盒(OMEGA, D6950-01)提取备用。
1.2.3 TALENs质粒活性验证
复苏PK15细胞系到6孔板,在含10%FBS的DMEM培养液中培养,待细胞长至约 60%密度时,按照1 μg GHR-TALEN1+1 μg GHR-TALEN2+6 μL Lipo2000(Invitrogen, 11668-027)的用量共转染猪PK15细胞系,转染48 h后,0.25%胰酶(Life technologies, 25200056)消化收取细胞,按照Tissue DNA Kit (OMEGA,D3396-02)步骤提取DNA进行活性鉴定,即将提取的DNA利用鉴定引物进行扩增,鉴定引物为GHR2-F(5′-TTCATGTTTCTGGGCTGTG-3′)和GHR2-R(5′-ACTTGTTTGCTTGCTGTGC-3′),扩增条件为95℃ 2 min;95℃ 30 s,60℃ 30 s,72℃ 30 s,共32个循环;最后再72℃延伸10 min。扩增产物进行PstI酶切检测GHR位点的突变情况,PCR片段大小为452 bp,酶切片段大小为279 bp+173 bp。
1.2.4 GHR-KO细胞株的获得及鉴定
转染用 GHR-TALEN1、GHR-TALEN2及 pcDNA3.1(+)空质粒的提取严格按照无内毒素质粒提取试剂盒(OMEGA, D6950-01)操作说明进行,AhdⅠ酶切 pcDNA3.1(+)空质粒,按照 Cycle-Pure Kit(OMEGA, D6492-02)步骤回收目的片段,线性化的pcDNA3.1(+)空质粒用于细胞转染后的 G418抗性筛选。转染前24 h,将巴马胎猪成纤维细胞接种到6 cm皿中,加含15%FBS(Hyclone,sv30087.02)的DMEM培养液(Gibco11965-084)培养、待细胞长至 90%密度时,0.05%胰酶(Life technologies,25300-054)消化,血细胞计数板计数,细胞用量为0.5~1×106,质粒用量为2 μg GHR-TALEN1+2 μg GHR-TALEN2+800 ng线性化pcDNA3.1(+)质粒,混匀,按照AmaxaTMBasic NucleofectorTMKit(Lonza,VPI-1002)步骤操作,利用Lonza核转仪核转细胞,选择 U-023核转程序,核转完成后,轻轻将细胞转移到 6孔板中。细胞长至80%~90%密度时,按10%密度接种到10 cm板中,待培养细胞培养到 30%~40%密度时,加入含 500 μg/mL G418的培养液进行筛选,直到出现明显的单克隆为止。挑取单克隆到 48孔板中,长满后传至24孔板中,长满后1传2,一个用于提取细胞总DNA,一个用于后续实验。
按照Tissue DNA Kit(OMEGA,D3396-02)提取细胞总DNA,用GHR2-F/R引物进行PCR,PCR产物用PstⅠ酶切检测GHR突变情况,酶切结果只有单条带的为双敲细胞系,测序鉴定基因序列突变情况。
1.2.5 手工克隆和移植
按照已报道的手工克隆的方法进行克隆[21~24],即先获得卵巢,收集卵子,并将其在改良的TL-Hepes-PVA缓冲液中清洗后,放入培养板中,在培养箱内进行未成熟卵细胞体外成熟(in vitro maturation, IVM)培养,得到成熟卵子。然后,将所得到的成熟卵子在显微镜下用小刀片去核,以便获得成熟去核卵母细胞。挑取单个筛选获得的阳性GHR-KO转基因细胞(包含利用大白猪胎儿成纤维细胞和巴马耳源成纤维细胞制备的转基因细胞系),利用融合仪电激将其融入到成熟去核卵母细胞中,制成重构胚。重构胚放入培养箱内,培养5~6 d,形成成熟囊胚。囊胚体外移植代孕母猪受体,妊娠114 d之后,小猪出生。
1.2.6 GHR-KO巴马猪的鉴定
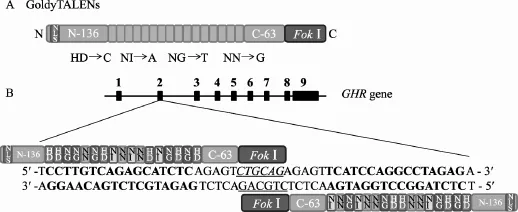
图1 TALENs定向剪切GHR基因结构示意图A:GoldyTALENs结构示意图;B:TALENs定向剪切GHR基因位点示意图。
剪耳钳剪取2周龄仔猪耳朵样品约30 mg,按照Tissue DNA Kit(OMEGA,D3396-02)步骤提取总DNA,按照1.2.3方法进行鉴定,同时每2周同一时间点对空腹小猪进行体重测定。
1.2.7 统计学分析
采用双尾T检验,比较GHR敲除巴马猪与野生型巴马猪的体重差异。
2 结果与分析
2.1 TALENs载体构建及活性鉴定
本研究所采用的是 Bedell等[17]和 Doyon等[18]所采用的GoldyTALENs载体结构,即N端保留136个氨基酸残基,C端保留63个氨基酸残基,中间为RVDs模块序列。C端连接突变3个氨基酸的FokⅠ序列(上游为FokⅠ-ELD,下游为FokⅠ-KKR)。同时在N端加上NLS识别序列,如图1A。模块HD、NI、NG、NN分别识别C、A、T、G碱基[25]。在GHR基因2号外显子上设计上下游TALEN识别位点,模块序列分别为5′-HDHDNGNGNNNGHDNINNNINNHDNINGHDNGHD-3′和 5′-HDNGHDNGNINNNNHDHDNGNNNNNINGNNNI-3′,中间5′-CTGCAG-3′为PstⅠ酶切位点,如图1B。
将构建好的GHR-TALEN1和GHR-TALEN2质粒转染PK15细胞系,转染48 h后收取细胞提取基因组DNA,用GHR2-F/R引物进行PCR扩增,PstⅠ酶切检测GHR位点突变情况,结果见图2,经过PstⅠ酶切后,GHR-TALENs组与 EGFP对照组相比有未切开条带,说明GHR-TALEN能剪切GHR基因位点。
2.2 GHR-KO细胞的筛选

图2 GHR-TALENs质粒活性鉴定图1:GHR-TALENs;2:EGFP;3:PCR产物。
GHR-TALEN1、GHR-TALEN2和线性化pcDNA3.1(+)质粒共转巴马胎猪成纤维细胞系,G418筛选获得173个抗性克隆(图3),提取克隆细胞总DNA,进行PCR扩增,按照活性鉴定的方法检测各抗性克隆GHR位点的突变情况。经酶切鉴定,单等位基因敲除克隆78个,双等位基因敲除克隆2个,基因修饰效率为 46.2%(80/173),如图 4所示。其中,GHR-16和GHR-148细胞克隆获得的扩增条带未能被切开,为双等位基因敲除细胞株(图4A),Sanger测序结果显示,GHR-16删除 11 bp碱基,GHR-148删除9 bp碱基(图4B)。
2.3 手工克隆获得GHR-KO阳性猪
将双等位基因敲除细胞株GHR-16作为核供体,与去核的卵母细胞结合,2次融合后,分别进行 6次手工克隆操作,共得到1773个重构胚,平均囊胚率为43.5%±2.1%(表1),重构胚体外培养到第6 d (图5),移植到发情的受体母猪子宫内,怀孕约114 d后,出生12头巴马仔猪,其中10头活仔,2头死胎(图6)。
图3 转染得到的抗性克隆A:GHR-TALENs转染后巴马胎猪成纤维细胞系;B:GHR-TALENs转染巴马胎猪成纤维细胞系后筛选的抗性克隆。
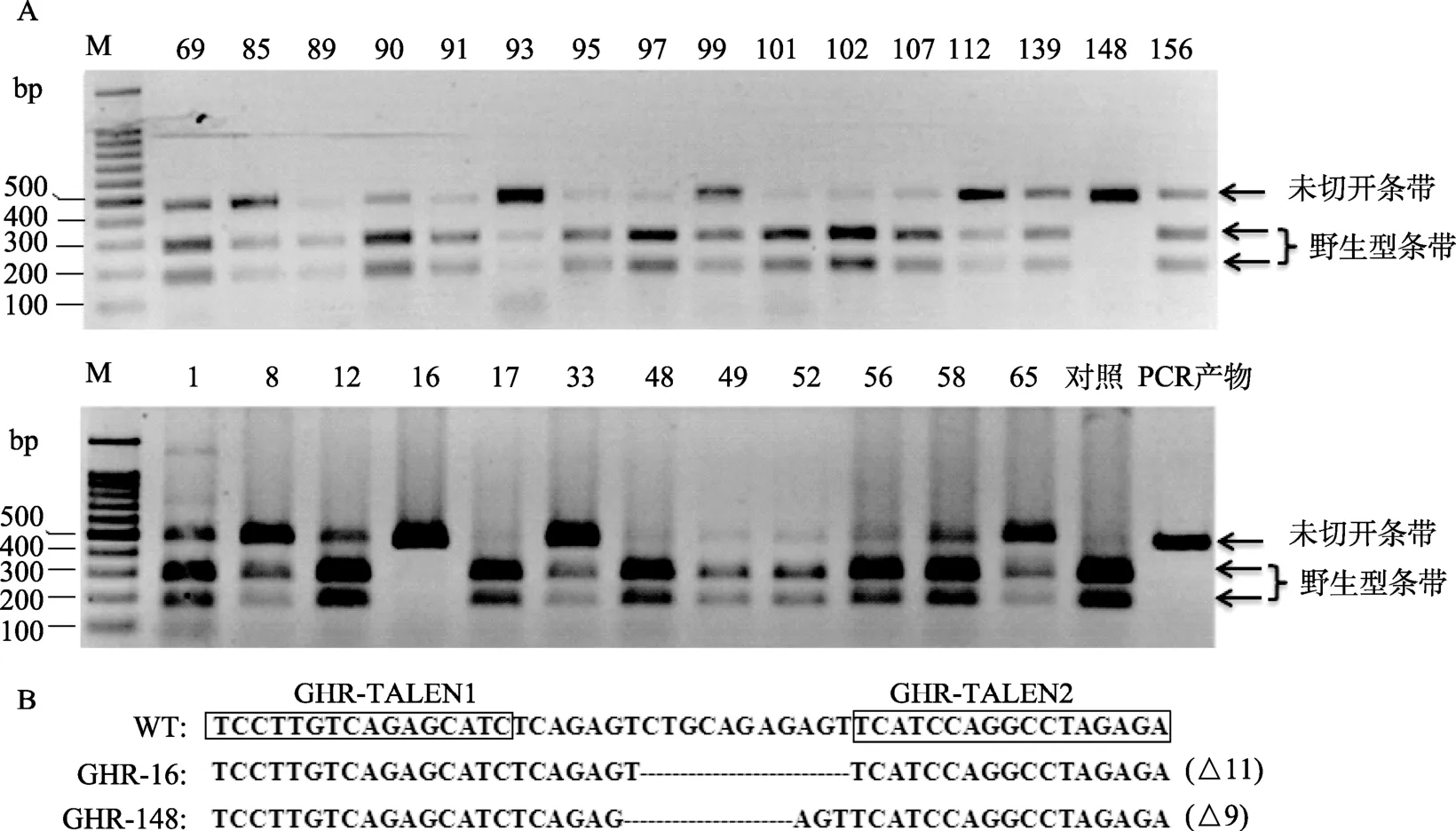
图4 细胞克隆鉴定A:GHR-TALENs部分抗性克隆PstⅠ酶切电泳鉴定图;B:双等位基因突变克隆细胞系测序结果。
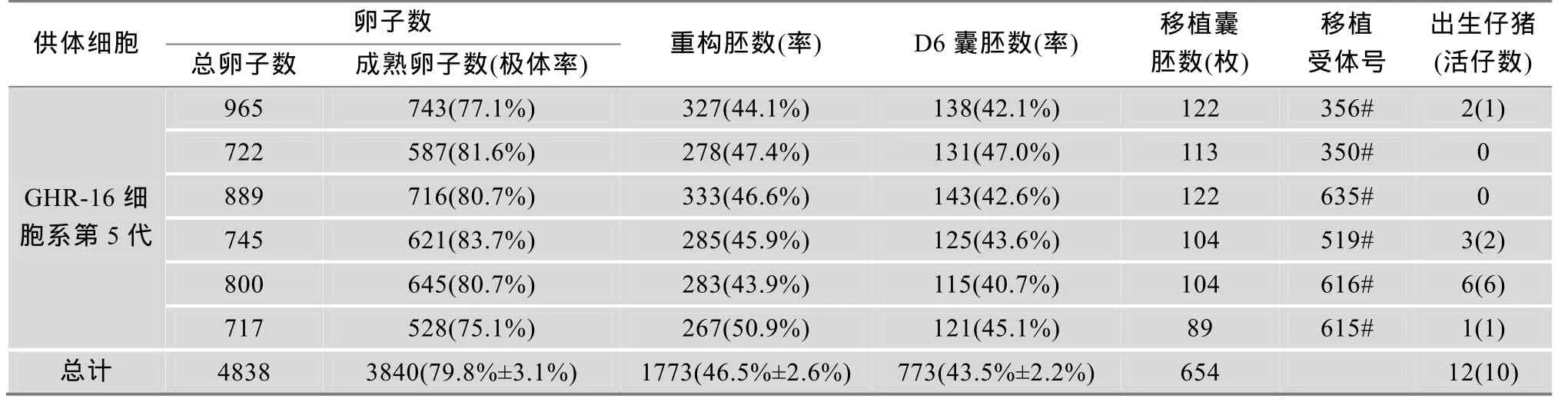
表1 巴马猪转基因细胞的核移植结果
2.4 GHR-KO小猪鉴定
剪取10头2周龄巴马仔猪约30 mg耳朵组织,提取基因组DNA,PCR扩增后PstⅠ酶切和测序鉴定GHR突变情况,从电泳结果可以看出,10只存活巴马仔猪中有7只为GHR双等位基因敲除仔猪,测序鉴定7头缺失11 bp碱基,3头没有发生碱基突变(图7)。
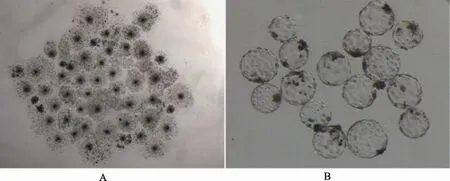
图5 巴马猪COCs体外成熟与体外发育A:体外成熟的巴马猪COCs;B:D6发育的囊胚。
对GHR-KO和野生型巴马猪从出生到20周龄的体重进行动态测定比较,每2周测定一次。测定结果显示,与对照相比,除了第8周差异显著(P<0.05)外,其他周龄GHR-KO巴马猪体重都极显著低于野生型(P<0.01)。GHR-KO巴马猪第6周开始生长缓慢,第20周时,体重仅为野生型的50%。如图8所示。
3 讨 论

图6 出生1周的巴马仔猪
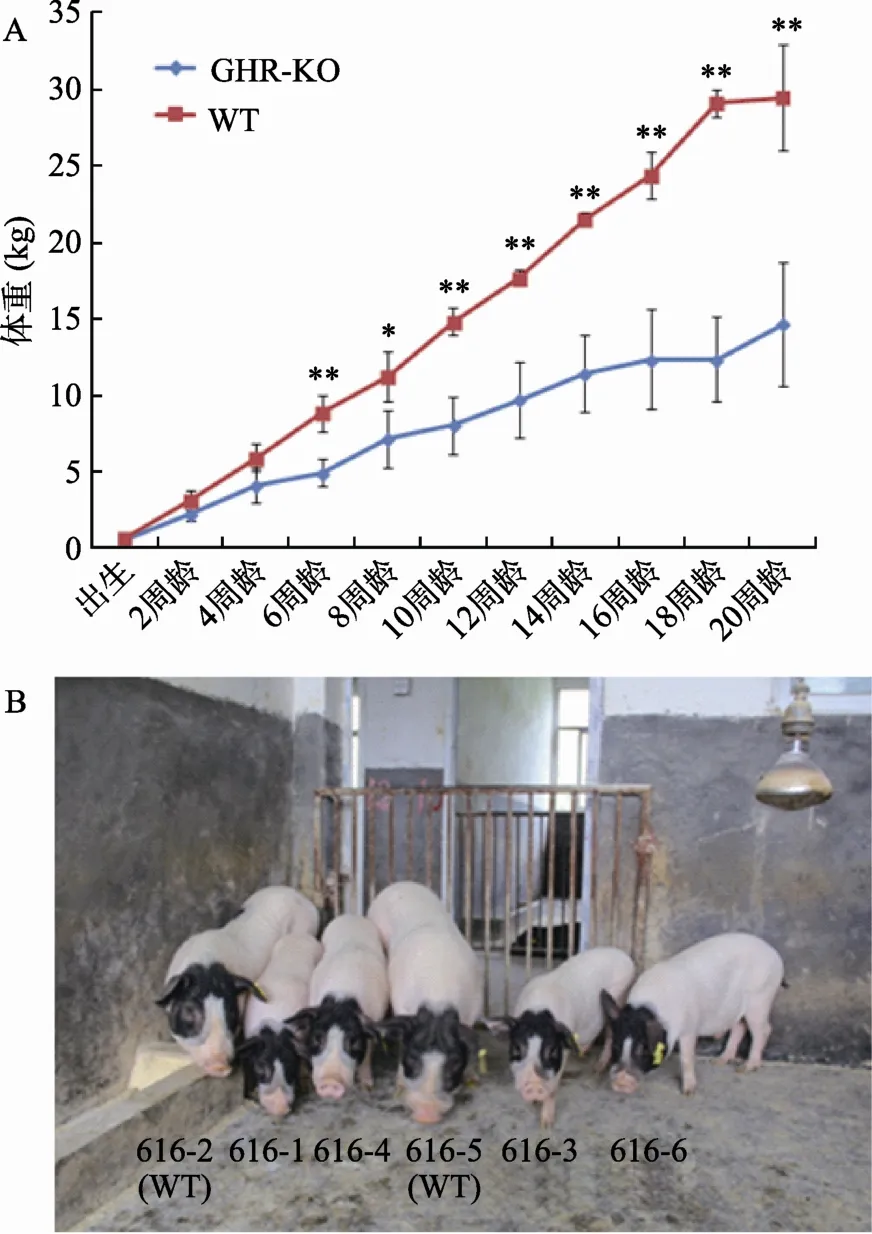
图8 GHR-KO巴马猪体重指标监测结果A:GHR-KO巴马猪生长曲线。*表示差异显著(P<0.05),**表示差异极显著(P<0.01)。B:5月龄GHR-KO巴马猪与对照猪。
传统打靶技术效率低,应用大大受限。ZFN技术实验设计复杂、成本高,且特异性不高[4],而TALEN技术实验简单,周期短,识别序列特异性高,剪切效率高及脱靶效率低等特点[19],至今已经被成功应用于动物、植物和物微生物在内的多个物种[20]。本实验室根据Bedell等[17]和Doyon等[18]结果改造的TALEN表达骨架和突变FokⅠ酶(ELD/KKR),构建的GHR靶向修饰TALENs质粒,转染并筛选获得细胞克隆,酶切鉴定基因靶向修饰效率为 46.2%,与Carlson等[6]、Tan等[9]报道的效率相当,但未达到Xin等[5]所报道的89.5%的修饰效率。Xin等[5]通过电穿孔方法将靶向修饰 GGTA1基因的 TALENs质粒和pcDNA3.1质粒共转染到巴马胎猪成纤维细胞系,获得了187个GGTA1突变的细胞克隆,占总209个克隆的89.5%。基因靶向修饰效率的差异可能是不同的基因序列、位置以及细胞转染效率所致,相同基因在不同细胞中的基因修饰效率存在差异[26],相同细胞中不同的基因剪切效率同样存在差异[6,27~29]。
与传统的体细胞核移植技术相比,手工克隆技术具有成本低、操作简单、效率高等特点,特别适合规模化应用及推广[21,23]。本研究利用手工克隆生产的GHR-KO囊胚,囊胚效率平均高达43.5%。这可能是手工克隆的过程中融合了2个去核卵细胞胞质,能提供更多营养物质[21]。本研究中敲除猪的出生存活率达 83.3%(出生活仔/出生仔猪总数),比Zhang等[23]的 75%、Lai等[30]的 35.7%和 Pan等[31]的 70%要高。本研究结果表明,与传统基因打靶和克隆技术相比,TALENs和手工克隆技术能够更简捷、快速、高效制备出基因敲除大动物,更易于在农业和生物医学基因靶向修饰研究中推广应用。
实验动物在人类疾病研究中发挥着重要作用,长期以来啮齿类动物由于其制作方法简便、实验成本低、实验周期短等特点而被人们常用作实验动物,然而由于其个体大小、生理学和病理学特征等与人类有较大差异,往往不能获得满意的结果。小型猪解剖、生理学和免疫学特征与人类极其相似,是人类比较医学中良好较好的实验动物材料[10],如囊胚性纤维症疾病克隆猪模型[32,33]、阿尔茨海默病猪模型[34]及生物节律紊乱猪模型[22]、皮肤炎症猪模型[35]、动脉粥样硬化病猪模型[36]。本文通过基因工程及手工克隆方法高效获得GHR-KO巴马猪,每2周进行体重监测,与对照相比,GHR-KO巴马猪生长迟缓,发育受阻,与Zhou等[37]建立的GHR-KO敲除小鼠出生后出现的生长阻滞相似。通过敲除小鼠建立的首个 Laron综合征哺乳动物模型,虽然能够模拟人类 Laron综合征的病理特征[38,39],但是仍有较多的差异点,如人类 Laron综合征病人在婴儿期会患低血糖症,而Laron小鼠的血糖正常[40]。Laron综合征儿童胰岛素水平很高,表现出胰岛素抵抗,而Laron综合征小鼠胰岛素水平很低[41,42]。因此,本研究希望通过敲除巴马猪GHR基因,获得跟人类更加相近的Laron综合征猪模型。未来将会对GHR-KO巴马猪做更深入的研究,如测定血液中的GH、IGF1蛋白含量高低、GH-GHR通路中GHR基因下游各基因的表达模式等,以期获得可推广的疾病模型。
[1] Thomas KR, Capecchi MR. Site-directed mutagenesis by gene targeting in mouse embryo-derived stem cells. Cell, 1987, 51(3): 503-512.
[2] Mansour SL, Thomas KR, Capecchi MR. Disruption of the proto-oncogene int-2 in mouse embryo-derived stem cells: a general strategy for targeting mutations to non-selectable genes. Nature, 1988, 336(6197): 348-352.
[3] 沈延, 肖安, 黄鹏, 王唯晔, 朱作言, 张博. 类转录激活因子效应物核酸酶(TALEN)介导的基因组定点修饰技术. 遗传, 2013, 35(4): 395-409.
[4] 肖安, 胡莹莹, 王唯晔, 杨志苖, 王展翔, 黄鹏, 佟向军,张博, 林硕. 人工锌指核酸酶介导的基因组定点修饰技术. 遗传, 2011, 33(7): 665-683.
[5] Xin J, Yang HQ, Fan NN, Zhao BT, Ouyang Z, Liu ZM, Zhao Y, Li XP, Song J, Yang Y, Zou QJ, Yan QM, Zeng YZ, Lai LX. Highly efficient generation of GGTA1 biallelic knockout inbred mini-pigs with TALENs. PLoS ONE, 2013, 8(12): e84250.
[6] Carlson DF, Tan W, Lillico SG, Stverakova D, Proudfoot C, Christian M, Voytas DF, Long CR, Whitelaw CBA, Fahrenkrug SC. Efficient TALEN-mediated gene knockout in livestock. Proc Natl Acad Sci USA, 2012, 109(43): 17382-17387.
[7] Boch J, Bonas U. Xanthomonas AvrBs3 family-type III effectors: discovery and function. Annu Rev Phytopathol, 2010, 48(1): 419-436.
[8] Lei Y, Guo X, Liu Y, Cao Y, Deng Y, Chen X, Cheng CHK, Dawid IB, Chen Y, Zhao H. Efficient targeted gene disruption in Xenopus embryos using engineered transcription activator-like effector nucleases (TALENs). Proc Natl Acad Sci USA, 2012, 109(43): 17484-17489.
[9] Tan W, Carlson DF, Lancto CA, Garbe JR, Webster DA, Hackett PB, Fahrenkrug SC. Efficient nonmeiotic allele introgression in livestock using custom endonucleases. Proc Natl Acad Sci USA, 2013, 110(41): 16526-16531.
[10] Luo YL, Li J, Liu Y, Lin L, Du YT, Li ST, Yang HM, Vajta G, Callesen H, Bolund L, Sørensen CB. High efficiency of BRCA1 knockout using rAAV-mediated gene targeting: developing a pig model for breast cancer. Transgenic Res, 2011, 20(5): 975-988.
[11] David A, Hwa V, Metherell LA, Netchine I, Camacho-Hübner C, Clark AJL, Rosenfeld RG, Savage MO. Evidence for a continuum of genetic, phenotypic, and biochemical abnormalities in children with growth hormone insensitivity. Endocr Rev, 2011, 32(4): 472-497.
[12] Savage MO, Attie KM, David A, Metherell LA, Clark AJ, Camacho-Hubner C. Endocrine assessment, molecular characterization and treatment of growth hormone insensitivity disorders. Nat Clin Pract Endocrinol Metab, 2006, 2(7): 395-407.
[13] Cioffi JA, Wang X, Kopchick JJ. Porcine growth hormone receptor cDNA sequence. Nucleic Acids Res, 1990, 18(21): 6451-6451.
[14] Laron Z. Growth hormone insensitivity (Laron syndrome). Rev Endocr Metab Disord, 2002, 3(4): 347-355.
[15] López-Bermejo A, Buckway CK, Rosenfeld RG. Genetic defects of the growth hormone-insulin-like growth factor axis. Trends Endocrinol Metab, 2000, 11(2): 39-49.
[16] Galli-Tsinopoulou A, Nousia-Arvanitakis S, Tsinopoulos I, Bechlivanides C, Shevah O, Laron Z. Laron syndrome. Laron syndrome. First report from Greece. Hormones (Athens), 2003, 2(2): 120-124.
[17] Bedell VM, Wang Y, Campbell JM, Poshusta TL, Starker CG, Krug RG, 2nd, Tan WF, Penheiter SG, Ma AC, Leung AYH, Fahrenkrug SC, Carlson DF, Voytas DF, Clark KJ, Essner JJ, Ekker SC. In vivo genome editing using a highefficiency TALEN system. Nature, 2012, 491(7422): 114-118.
[18] Doyon Y, Vo TD, Mendel MC, Greenberg SG, Wang JB, Xia DF, Miller JC, Urnov FD, Gregory PD, Holmes MC. Enhancing zinc-finger-nuclease activity with improved obligate heterodimeric architectures. Nat Methods, 2010, 8(1): 74-79.
[19] Cermak T, Doyle EL, Christian M, Wang L, Zhang Y, Schmidt C, Baller JA, Somia NV, Bogdanove AJ, Voytas DF. Efficient design and assembly of custom TALEN and other TAL effector-based constructs for DNA targeting. Nucleic Acids Res, 2011, 39(12): e82.
[20] 沈延, 黄鹏, 张博. TALEN 构建与斑马鱼基因组定点突变的实验方法与流程. 遗传, 2013, 35(4): 533-544.
[21] 张鹏, 杨珍珍, 窦红伟, 李伟杭, 律波, Lars B, 杜玉涛,谭萍萍, 马润林. 利用改进的手工克隆技术生产转 GFP基因猪克隆胚胎. 遗传, 2011, 33(5): 527-532.
[22] Liu H, Li Y, Wei Q, Liu CX, Bolund L, Vajta G, Dou HW, Yang WX, Xu Y, Luan J, Wang J, Yang HM, Staunstrup NH, Du YT. Development of transgenic minipigs with expression of antimorphic human cryptochrome 1. PLoS ONE, 2013, 8(10): e76098.
[23] Zhang P, Zhang Y, Dou H, Yin J, Chen Y, Pang X, Vajta G, Bolund L, Du Y, Ma RZ. Handmade cloned transgenic piglets expressing the nematode fat-1 gene. Cell Reprogram, 2012, 14(3): 258-266.
[24] Fan NN, Chen JN, Shang ZC, Dou HW, Ji GZ, Zou QJ, Wu L, He LX, Wang F, Liu K, Liu N, Han JY, Zhou Q, Pan DK, Yang DS, Zhao BT, Ouyang Z, Liu ZM, Zhao Y, Lin L, Zhong CM, Wang QL, Wang SQ, Xu Y, Luan J, Liang Y, Yang ZZ, Li J, Lu CX, Vajta G, Li ZY, Ouyang HS, Wang HY, Wang Y, Yang Y, Liu ZH,Wei H, Luan ZD, Esteban MA, Deng HK, Yang HM, Pei DQ, Li N, Pei G, Liu L, Du YT, Xiao L, Lai LX. Piglets cloned from induced pluripotent stem cells. Cell Res, 2013, 23(1): 162-166.
[25] Miller JC, Tan SY, Qiao GJ, Barlow KA, Wang JB, Xia DF, Meng XD, Paschon DE, Leung E, Hinkley SJ, Dulay GP, Hua KL, Ankoudinova I, Cost GJ, Urnov FD, Zhang HS, Holmes MC, Zhang L, Gregory PD, Rebar EJ. A TALE nuclease architecture for efficient genome editing. Nat Biotechnol, 2011, 29(2): 143-148.
[26] Hockemeyer D, Wang H, Kiani S, Lai CS, Gao Q, Cassady JP, Cost GJ, Zhang L, Santiago Y, Miller JC, Zeitler B, Cherone JM, Meng XD, Hinkley SJ, Rebar EJ, GregoryPD, Urnov FD, Jaenisch R. Genetic engineering of human ES and iPS cells using TALE nucleases. Nat Biotechnol, 2011, 29(8): 731-734.
[27] Reyon D, Tsai SQ, Khayter C, Foden JA, Sander JD, Joung JK. FLASH assembly of TALENs for high-throughput genome editing. Nat Biotechnol, 2012, 30(5): 460-465.
[28] Cade L, Reyon D, Hwang WY, Tsai SQ, Patel S, Khayter C, Joung JK, Sander JD, Peterson RT, Yeh JRJ. Highly efficient generation of heritable zebrafish gene mutations using homo- and heterodimeric TALENs. Nucleic Acids Res, 2012, 40(16): 8001-8010.
[29] Moore FE, Reyon D, Sander JD, Martinez SA, Blackburn JS, Khayter C, Ramirez CL, Joung JK, Langenau DM. Improved somatic mutagenesis in zebrafish using transcription activator-like effector nucleases (TALENs). PLoS ONE, 2012, 7(5): e37877.
[30] Lai LX, Kang JX, Li RF, Wang JD, Witt WT, Yong HY, Hao YH, Wax DM, Murphy CN, Rieke A, Samuel M, Linville ML, Korte SW, Evans RW, Starzl TE, Prather RS, Dai YF. Generation of cloned transgenic pigs rich in omega-3 fatty acids. Nat Biotechnol, 2006, 24(4): 435-436.
[31] Pan DK, Zhang L, Zhou YR, Feng C, Long C, Liu X, Wan R, Zhang J, Lin AX, Dong EQ, Wang SC, Xu HG, Chen HX. Efficient production of omega-3 fatty acid desaturase (sFat-1)-transgenic pigs by somatic cell nuclear transfer. Sci China Life Sci, 2010, 53(4): 517-523.
[32] Rogers CS, Hao YH, Rokhlina T, Samuel M, Stoltz DA, Li YH, Petroff E, Vermeer DW, Kabel AC, Yan ZY, Spate L, Wax D, Murphy CN, Rieke A, Whitworth K, Linville ML, Korte SW, Engelhardt JF, Welsh MJ, Prather RS. Production of CFTR-null and CFTR-DeltaF508 heterozygous pigs by adeno-associated virus-mediated gene targeting and somatic cell nuclear transfer. J Clin Invest, 2008, 118(4): 1571-1577.
[33] Rogers CS, Stoltz DA, Meyerholz DK, Ostedgaard LS, Rokhlina T, Taft PJ, Rogan MP, Pezzulo AA, Karp PH, Itani OA, Kabel AC, Wohlford-Lenane CL, Davis GJ, Hanfland RA, Smith TL, Samuel M, Wax D, Murphy CN, Rieke A, Whitworth K, Uc A, Starner TD, Brogden KA, Shilyansky J, McCray PB, Jr., Zabner J, Prather RS, Welsh MJ. Disruption of the CFTR gene produces a model of cystic fibrosis in newborn pigs. Science, 2008, 321(5897): 1837-1841.
[34] Kragh PM, Nielsen AL, Li J, Du YT, Lin L, Schmidt M, Bøgh IB, Holm IE, Jakobsen JE, Johansen MG, Purup S, Bolund L, Vajta G, Jorgensen AL. Hemizygous minipigs produced by random gene insertion and handmade cloning express the Alzheimer's disease-causing dominant mutation APPsw. Transgenic Res, 2009, 18(4): 545-558.
[35] Staunstrup NH, Madsen J, Primo MN, Li J, Liu Y, Kragh PM, Li R, Schmidt M, Purup S, Dagnæs-Hansen F, Svensson L, Petersen TK, Callesen H, Bolund L, Mikkelsen JG. Development of transgenic cloned pig models of skin inflammation by DNA transposon-directed ectopic expression of human beta1 and alpha2 integrin. PLoS ONE, 2012, 7(5): e36658.
[36] Al-Mashhadi RH, Sørensen CB, Kragh PM, Christoffersen C, Mortensen MB, Tolbod LP, Thim T, Du YT, Li J, Liu Y, Moldt B, Schmidt M, Vajta G, Larsen T, Purup S, Bolund L, Nielsen LB, Callesen H, Falk E, Mikkelsen JG, Bentzon JF. Familial hypercholesterolemia and atherosclerosis in cloned minipigs created by DNA transposition of a human PCSK9 gain-of-function mutant. Sci Transl Med, 2013, 5(166): 166ra1.
[37] Zhou Y, Xu BC, Maheshwari HG, He L, Reed M, Lozykowski M, Okada S, Cataldo L, Coschigamo K, Wagner TE, Baumann G, Kopchick JJ. A mammalian model for Laron syndrome produced by targeted disruption of the mouse growth hormone receptor/binding protein gene (the Laron mouse). Proc Natl Acad Sci USA, 1997, 94(24): 13215-13220.
[38] Kopchick JJ, Laron Z. Is the Laron mouse an accurate model of Laron syndrome? Mol Genet Metab, 1999, 68(2): 232-236.
[39] List EO, Coschigano KT, Kopchick JJ. Growth hormone receptor/binding protein (GHR/BP) knockout mice: a 3-year update. Mol Genet Metab, 2001, 73(1): 1-10.
[40] Laron Z, Pertzelan A, Karp M, Keret R, Eshet R, Silbergeld A. Laron Syndrome-a unique model of IGF-1 deficiency. // Pediatric and adolescent endocrinology. Vol. 24. New York: Karger, 1993, 24: 3.
[41] Laron Z, Avitzur Y, Klinger B. Insulin Resistance in Laron Syndrome (Primary Insulin-like Growth Factor-I (IGF-I) Deficiency) and Effect of IGF-I Replacement Therapy. J Pediatr Endocrinol Metab, 1997, 10(Suppl.): 105-116.
[42] Coschigano KT, Clemmons D, Bellush LL, Kopchick JJ. Assessment of growth parameters and life span of GHR/BP gene-disrupted mice. Endocrinology, 2000, 141(7): 2608-2613.
(责任编委: 任 军)
Production of GHR double-allelic knockout Bama pig by TALENs and handmade cloning
Feida Li1,3, Yong Li1,2,3, Huan Liu1,3, Huanhuan Zhang1,3, Chuxin Liu1,3, Xingju Zhang1,3, Hongwei Dou2, Wenxian Yang2, Yutao Du1,2,3
1. BGI-Shenzhen, Shenzhen 518083, China;
2. BGI ARK Biotechnology Co., Ltd, Shenzhen 518083, China;
3. Shenzhen Engineering Laboratory for Genomics-Assisted Animal Breeding, Shenzhen 518083, China
Abstract:DNA editing techniques for targeted genome modification have witnessed remarkable advances and been widely used in various organisms. However, traditional gene targeting and cloning method has been shown to be low efficient, time-consuming and expensive for generating knockout animals, especially for big animals. Here we report the generation of site-specific genome modified pig with the newly developed artificially engineered sequence-specific endonucleases (transcription activator-like effector nuclease, TALENs) and handmade cloning (HMC) methods. First, we constructed the porcine GHR-knockout vector according to TALENs kit protocol. To obtain the nuclear donor, the fetal fibroblast cell of Bama (BM) pig were transfected with GHR-knockout vector in G418 selection medium. We collected 173 cell for further positive identification which showed that 46.2% (78/173) of the clones were GHR-knockout cell strains. We chose one bi-allelic knockout cell strain as nuclear donor to produce reconstructed embryos by HMC. It was shown that the blastocyst rate was 43.5% at the 6thday in vitro, then 654 HMC-blastocysts were transplanted to uterus of six recipient sows. Finally, a total of 10 live offspring were delivered including 7 bi-allelic knockout piglets. Fibroblasts were obtained from ear biopsies for GHR knockout detection. The body weight of the piglets was measured consecutively, and it was found that the GHR-/-pigs were only 50% smaller than that of the controls at the 20thweek. In conclusion, our results indicate that TALENs and HMC technology can rapidly and efficiently produce knockout animals for agricultural and biomedical research.
gene knockout; TALENs; handmade cloning; GHR-KO
2014-02-27;
2014-04-22
农业部动物转基因重大专项课题(编号:2014ZX0801007B)和深圳市生物产业发展专项资金(编号:JC201005260182A)资助
李飞达,硕士,助理研究员,专业方向:动物基因工程。E-mail:lifeida@genomics.cn
杜玉涛,博士,副教授,研究方向:克隆与基因工程。E-mail: duyt@genomics.cn
10.3724/SP.J.1005.2014.0903
时间: 2014-7-30 11:23:54
URL: http://www.cnki.net/kcms/detail/11.1913.R.20140730.1123.001.html

