DNA甲基化和去甲基化的研究现状及思考
邓大君
北京大学肿瘤医院/研究所, 北京 100142
DNA甲基化和去甲基化的研究现状及思考
邓大君
北京大学肿瘤医院/研究所, 北京 100142
DNA甲基化通过调节基因转录、印记、X染色体灭活和防御外源性遗传物质入侵等, 在细胞分化、胚胎发育、环境适应和疾病发生发展上发挥重要作用, 是当前表观遗传学研究的热点领域之一。文章介绍了在过去几年中TET介导的DNA羟甲基化及其在早期胚胎发育中的作用, DNA主动去甲基化及其与被动去甲基化的关系, DNA甲基化建立及其与组蛋白修饰、染色质构象、多梳蛋白和非编码RNA结合等关系方面的重要研究进展和存在的问题以及DNA甲基化的转化应用前景。
DNA甲基化; 去甲基化; 表观遗传学; 稳态; 转化研究
DNA甲基化是指DNA序列中的腺嘌呤(A)或胞嘧啶(C)碱基在甲基化转移酶的催化下与甲基发生共价结合, 可在细胞分裂过程中传递给子细胞的表观遗传现象。由DNA腺嘌呤甲基化酶(DNA adenine methylase, DAM)催化形成的 O6-甲基腺嘌呤(6mA)是一种CTA G序列复制后维持甲基化, 在细菌表观遗传过程中发挥作用, 具体功能包括染色体复制、错配修复、毒力基因表达控制、外源性基因防御等[1,2]。近年发现 DNA腺嘌呤甲基化还参与细菌细胞周期控制[3]。细菌DNA腺嘌呤甲基化的具体过程及功能已经研究的比较清楚, 本文不做进一步叙述。存在于高等生物细胞内的 C5-甲基胞嘧啶(5mC)由一组DNA甲基转移酶(DNA methyltransferase, DNMT)催化形成, 包括从头甲基化和DNA复制后维持甲基化两种形成途径。在成年脊椎动物体细胞中主要发生在胞嘧啶-鸟嘌呤(G)二核苷酸(CpG)位点上; 在植物细胞和动物胚胎干细胞还可发生在非CpG位点上。胞嘧啶甲基化在多细胞生物的细胞分化和环境适应方面发挥重要作用, 具体功能包括X染色体灭活、基因印记、基因长效沉默、细胞分化、外源基因防御、异物代谢等, 是比较活跃的研究领域之一, 近年取得了诸多研究进展。本文简要综述了DNA去甲基化的机制和 DNA甲基化建立及维持研究的主要进展, 并对存在的几个关键问题进行了讨论。
1 DNA主动去甲基化及其机制
DNA胞嘧啶甲基化是一种共价修饰, 化学性质稳定。5mC有两种形成途径:DNA半保留复制后由UHRF1(Ubiquitin-like, containing PHD and RING finger domains, 1)招募DNMT1, 催化半甲基化DNA双链中未甲基化链的维持甲基化(Maintenance methylation); 由DNMT3a和DNMT3b催化未甲基化DNA双链的从头甲基化(de novo methylation)。DNA去甲基化也存在被动去甲基化和主动去甲基化两种途径。细胞可通过抑制DNMT1表达或催化活性来阻断 DNA的维持甲基化, 在细胞分裂过程中稀释/降低基因组中甲基化胞嘧啶的密度, 实现被动去甲基化(图 1A)。最近日本学者报道, 这种被动去甲基化机制是小鼠胚胎发育过程中原生殖细胞(Primordial germ cell)去除基因组亲本DNA甲基化的关键机制[4]。在高等脊椎动物卵子受精后, 经过 4次卵裂, 除印记基因外, 基因组DNA将完全去甲基化, 形成全能的胚胎干细胞(Embryonic stem cell, ESC)。在诱导多能干细胞(Induced pluripotent stem cell, iPSC)形成过程中也存在类似的完全去甲基化现象。卵裂过程中如果只存在被动去甲基化, 经过4次分裂的ESC基因组中至少还应该保留1/16的甲基化胞嘧啶, 无法实现完全去甲基化。显然, 在胚胎干细胞中还存在其他去甲基化的机制。
胞嘧啶脱氨基酶能够催化未甲基化和甲基化胞嘧啶脱氨基, 分别形成尿嘧啶(U)和胸腺嘧啶(T)。甲基化胞嘧啶的脱氨基效率明显高于未甲基化者。胞嘧啶脱氨基是脊椎动物进化过程中基因组发生 CpG抑制(CpG suppression)的主要原因。在肿瘤细胞基因组中, 大部分点突变都是CpG位点的胞嘧啶转换成胸腺嘧啶(C:G→T:A)[5,6]。由于脱氨基形成的尿嘧啶或胸腺嘧啶与互补链上的鸟嘌呤不配对, 在正常情况下大部分将在尿嘧啶或者胸腺嘧啶 DNA糖苷酶等的催化下, 按碱基切除修复(BER)方式修复。通过这种胞嘧啶脱氨基-碱基切除修复途径, 细胞的确有实现DNA主动去甲基化的可能。在受到感染、辐射等致癌物打击后, 宿主靶器官/组织的胞嘧啶脱氨基酶(包括APOBEC和AID等)含量会迅速上升, 全基因组同步低甲基化。尚不清楚这种脱氨基酶活性的升高是否为全基因组低甲基化发生的关键环节。Popp等[7]研究表明, AID缺乏小鼠的原生殖细胞全基因组甲基化水平升高, 但是胚胎和胎盘组织的总甲基化水平无差别, 提示AID可能仅在原生殖细胞主动去甲基化过程中发挥作用。AID是胞嘧啶脱氨基酶家族的一员, 在离体条件下能够使单链DNA胞嘧啶脱氨基, 在 B细胞中高表达, 促进编码免疫球蛋白IgM的基因转化成编码IgG的基因。该基因遗传缺陷将导致常染色体隐性遗传病——免疫缺陷综合征(无IgG、IgA、IgE, 对细菌感染高度易感)或Ⅱ型高IgM综合征 (HIGM2)。在脊椎动物细胞基因组中还存在许多其他DNA胞嘧啶脱氨基酶。不同的胞嘧啶脱氨基酶有不同的DNA序列偏好, 如AID偏好WRC Y(W=A或T; R=嘌呤; Y=嘧啶), APOBEC偏好胞嘧啶串(Cs)上的末位胞嘧啶, APOBEC1偏好 TC位点, APOBEC3DC偏好WC 位点, 却未发现偏好CpG位点的胞嘧啶脱氨基酶。尽管甲基化胞嘧啶的脱氨基效率明显高于未甲基化者, 胞嘧啶脱氨基-碱基切除修复途径是否为脊椎动物细胞实现CpG位点主动去甲基化的主要途径尚缺乏定论。
2009年美国的两个研究小组同时发现脑组织和ESC DNA中存在5-羟甲基胞嘧啶(hmC), 证明TET1 (Ten-eleven translocation methylcytosine dioxygenase 1)能够催化DNA中5mC氧化成为hmC(图2), 推测hmC的功能之一是参与DNA主动去甲基化[8,9]。2011年徐国良等与美国学者同步报道, TET1和TET2基因还能够使 hmC进一步氧化, 形成 5-醛基胞嘧啶(fC)和5-羧基胞嘧啶(caC)[10,11]; 在离体条件下DNA中的caC可被胸腺嘧啶DNA糖苷酶切除, 用siRNA敲减该酶含量后ESC中caC含量升高, 推测它参与了caC的碱基切除修复过程(图1B)。TET3基因在小鼠受精卵去除雄原核 DNA甲基化标记过程中也能发挥作用[12]。Williams等[13]也报道, 在哺乳动物ESC基因组中广泛存在TET1蛋白结合和hmC。值得注意的是, Hackett等[14]报道TET1和TET2蛋白催化的hmC形成与小鼠原始生殖细胞分裂过程平行, 在去除亲本 DNA甲基化印记过程中发挥作用, 提示TETs介导的DNA主动去甲基化与细胞分裂过程存在密切关系。TET1和TET2可与干细胞因子NANOG相互结合, 共同在诱导小鼠细胞获得多能性过程中发挥作用[15]。
尽管斑马鱼等低等脊椎动物的精子 DNA的甲基化水平与其体细胞相同, 在胚胎发育早期, 雄源性 DNA的总甲基化水平仍然维持不变[16~18], 这与小鼠等哺乳动物胚胎发育早期基因组存在普遍的去甲基化明显不同。相反, 斑马鱼在卵子成熟过程中就完成了全基因组去甲基化, 而小鼠卵子在受精卵分裂(卵裂)过程中才能够完成全基因组去甲基化, 尽管这种去甲基化在卵子成熟过程中就开始了[19]。这些结果说明, 在低等脊椎动物与哺乳动物之间, 与 ESC获得全能性相关的基因组去甲基化方式存在明显差别。
那么, DNA主动去甲基化与被动去甲基化之间是否存在内在联系呢?在常规的碱基切除修复过程中需要切除一小段核苷酸序列(2~6 bp), 而 CpG位点的甲基化在DNA双链中是对称性的。如果DNA双链上同一甲基化CpG位点发生同步氧化和碱基切除修复, 将导致 DNA双链断裂, CpG位点密集(如CpG岛和Alu元件及重复序列LINE)的DNA甚至可能碎片化(图 1B)。如果以细胞分裂过程中形成的半甲基化DNA为切除修复底物, 则可避免这种DNA断裂或碎片化。如上所述, 被动去甲基化和TET氧化5mC均在原生殖细胞DNA去甲基化过程中发挥作用[14]。笔者认为全基因组TET主动去甲基化存在与DNA被动去甲基化配合完成的可能(图1C)。

图1 5-甲基胞嘧啶的被动(A)、主动(B)、被动伴主动(C)去甲基化模式
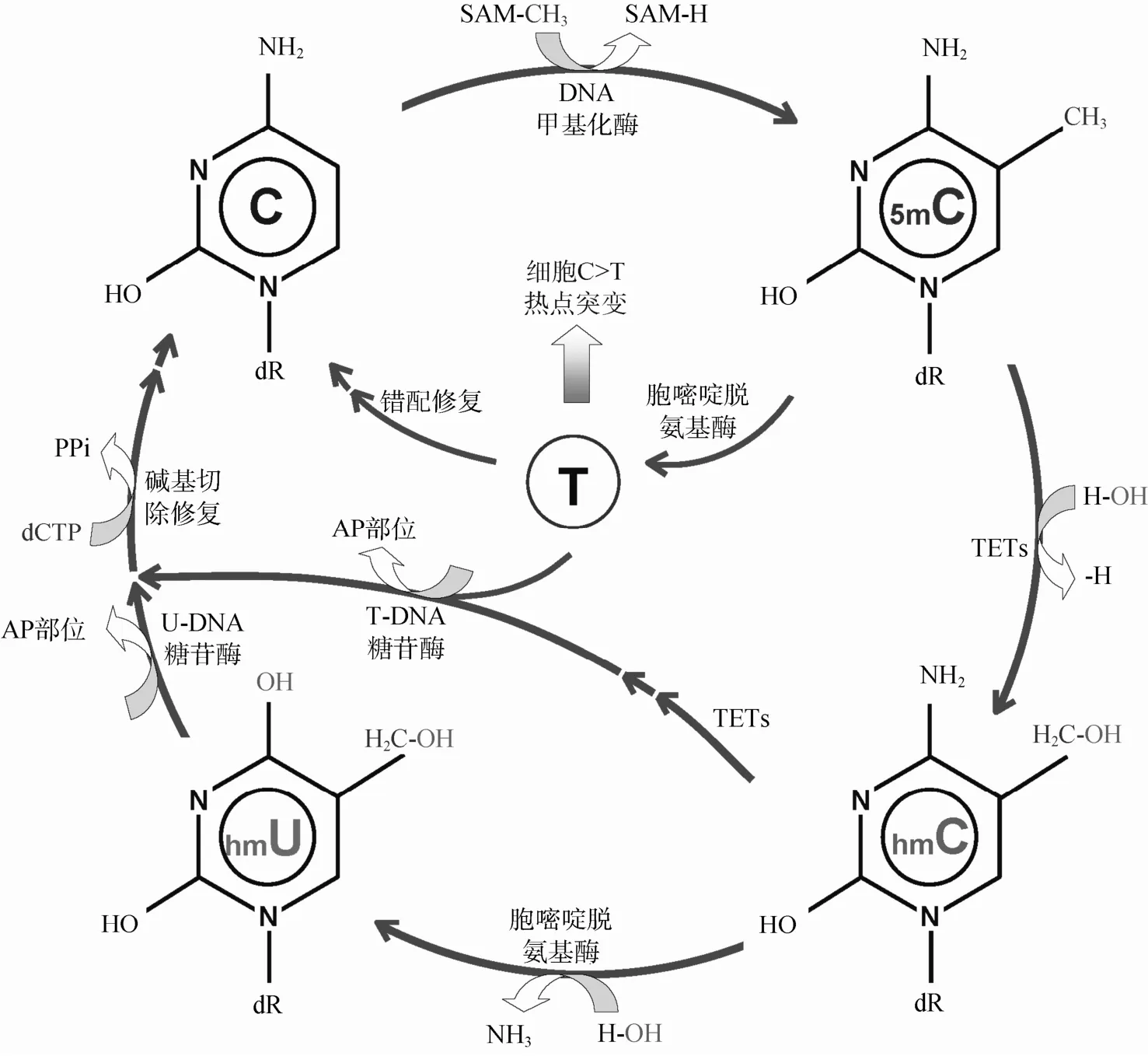
图2 DND胞嘧啶的修饰与去修饰过程
在缺乏细胞分裂相关的被动去甲基化的条件下, DNA是否能够实现主动去甲基化呢?2012年沈哲鲲等[20]报道, 在非细胞体系中, DNMT3a和DNMT3b能够直接切除双链 DNA胞嘧啶中的羟甲基, 而DNMT1无此作用。DNMTs基因在ESC中基本不表达, 在分化的细胞(如胚球和衍生的体细胞)中表达,在肿瘤细胞中高表达。DNMTs是否的确参与这些分化细胞的主动去甲基化过程还有待研究。在植物细胞去甲基化方面, 2012年Qian等[21]发现植物细胞内的乙酰化酶IDM1可以与甲基化的DNA结合, 催化该区域的组蛋白发生乙酰化, 创造环境使5mC糖苷酶发挥功能, 从而使DNA发生去甲基化。
hmC不仅参与原生殖细胞和ESC去甲基化, 而且还可不进一步氧化, 稳定地存在于成体组织细胞中。在不同成年组织的细胞中, hmC的分布与核小体组蛋白H3K27me3存在高度的相关性[22]。hmC在成年组织的细胞基因组内为什么不会象在ESC中那样进一步氧化?其功能尚未明确。在小鼠脑组织中, hmC可以较高的丰度稳定存在[8]; 在斑马鱼和人体细胞老化过程中, 其体细胞基因组中临近CpG岛周围区域(CpG island shores)的甲基化水平不断降低[23];在胰腺肿瘤基因组中hmC的含量不断降低, 可聚集在原癌基因DNA上[24]。
最近研究表明, hmC的形成还受环境因素影响,二甲基亚砜(DMSO)处理会显著促进培养细胞 hmC含量[25]; 还原型维生素C处理可明显提高小鼠ESC TET1的催化活性和 hmC水平[26]; 苯巴比妥处理能够迅速诱发大鼠肝脏基因组hmC的分布改变[27]。这些结果提示hmC的稳定性比5mC低, 对环境因素更加易感, 与环境适应基因的甲基化状态改变关系更密切。
2 DNA甲基化状态的建立和维持
基因转录起始点(TSS)周围 CpG岛甲基化是基因长期稳定沉默的标志, 在细胞分化过程中可能发挥关键作用。哺乳动物的胚泡(Blastocyst)内完全去甲基化的ESC按照不同的分化方向, 在DNMT3a和3b的催化下发生有序的从头甲基化, 建立细胞分化相关甲基化谱。从头甲基化和去甲基化现象还广泛存在于机体正常适应环境和疾病的发生发展过程中,建立环境适应相关甲基化谱。在DNMTs高表达的细胞中(例如肿瘤细胞), DNA中的CpG位点的默认状态到底是非甲基化的还是甲基化的?肿瘤抑制基因的甲基化失活是肿瘤组织中的常见现象, 一般认为这种失活的机制是这些基因的转录抑制复合物招募了DNMTs, 导致CpG岛甲基化。然而, 这一理论不能解释为什么在体细胞基因的躯干部位(Gene body)70%以上的散在CpG位点都是甲基化的。
目前已经发现了多种转录基因维持去甲基化状态的机制。长链非编码RNA(lncRNA)不仅可通过竞争性结合miRNA或PcG蛋白等途径来发挥正向调节基因表达的作用[28~35], 而且还可通过与 DNMT1结合来阻断其甲基化活性, 维持目的基因DNA的去甲基化状态[36]。H3K4me3是基因存在转录活性的标志, H3K4me1则是H3K4me3形成的基础。2009年Hu等[37]发现未甲基化的H3K4是DNMT3L的结合区, 能够促进DNMT3L-DNMT3a复合物的从头甲基化作用。未甲基化的H3K4还能够诱导DNMT3a蛋白的构象变化, 提高其甲基化酶活性; DNMT3a的甲基化活性与H3K4me1的含量存在负相关关系[38]。近年来, 包括ENCODE研究在内的各种高分辨率的转录因子结合组和DNA甲基化组研究揭示, 活性基因转录起始点附近序列是转录复合物的稳定结合区,在这些区域染色质中普遍存在甲基化的H3K4, DNA则呈低甲基化状态[39]。基因组中 DNA的甲基化与核小体的分布亦存在高度的相关性。在人胃癌发生过程中, p16基因CpG岛甲基化具有以核小体为基本单元脉冲式扩展的特征[40]。在降低组蛋白甲基化酶 G9A和 SUV39H1蛋白水平和基因启动子区H3K9me2/3水平后, 启动子区结合的 HP1和DNMT1减少[41]。Ficz等[42]用hmC特异性抗体对全基因组进行hMeDIP-测序分析, 发现hmC大部分分布于小鼠胚胎干细胞常染色质区。Sun等[43]用AbaSI酶切-测序法发现, hmC主要分布在小鼠ESC基因组的CTCF结合区, 特别是存在H3K4me1的区域, 而不是在H3K27ac富集区。在骨髓间质干细胞中也存在高水平的hmC[44]。种种迹象表明, 在DNMT1充分表达的条件下, DNA中大部分CpG位点的默认状态是甲基化的。在基因常转录的区域存在活跃的DNA去甲基化现象。上述现象说明, 基因的转录产物及其相关的组蛋白修饰在维持 DNA去甲基化状态方面发挥着关键作用。一旦基因处于非转录状态,则将失去这些去甲基化机制的保护, 发生甲基化。
我们最近的工作表明, 尽管肿瘤细胞中 p16基因的甲基化和非甲基化状态非常稳定, 仍然伴有低水平的局部甲基化和羟甲基化及去甲基化, 这种局部甲基化变化不稳定地存在于每个细胞中, 说明该基因的甲基化状态是以稳态(Homeostasis)的模式维持(待发表)。因此, 有必要对甲基化状态稳态维持是否具有普遍性、稳态调控的网络构成等开展进一步研究, 以了解表观遗传的本质。
3 结语与展望
人体内存在 200多种不同类型的细胞, 不仅各自有不同的细胞分化相关DNA甲基化谱, 而且还有随着生存环境因素和年龄的变化而变化的适应相关DNA 甲基化谱。显然, 与基因组相比较, 人体内DNA甲基化组等表观遗传组更加复杂多样, 研究工作尤其艰巨。DNA基因组序列完全相同的人 ESC如何分化成不同的细胞?DNA甲基化如何与其他表观遗传网络协调工作?这些网络是怎样有效适应不同生存环境因素变化的?这些网络在疾病发生过程中究竟发挥了什么作用?哪些能够用于疾病的预防、诊断和治疗?这些都是仍然有待阐明的热点问题。表观遗传现象极其复杂, 一方面需要杰出的科学家们继续创造性地工作, 以揭示DNA甲基化等表观遗传网络启动、扩展、维持、去除的基本规律, 另一方面还需要投入相当的资源, 发展灵敏可靠的低成本单细胞组学分析技术, 剖析DNA甲基化等在影响疾病发生、评价环境因素安全、增加农作物产量和抗病能力中的作用及机制, 研究其潜在应用价值。随着深度测序成本的降低和各种数据挖掘软件的出现, 动物体内不同类型细胞之间和不同物种之间的比较表观遗传组学的研究可望蓬勃发展, DNA甲基化的进化形成机制有望得到揭示。
与直接检测基因的表达产物 RNA和蛋白质不同, 基因转录启始点周围CpG岛甲基化状态反映的是基因表达状态的长期稳定变化, 在各种条件下保存的样品中均能够稳定存在。此外, 甲基化和非甲基化的CpG岛可分别使用甲基化和非甲基化特异性技术检测, 检测方法极其灵敏。这些使得CpG岛甲基化变异具备成为理想的生物学标志物的各项条件,尤其是在检测杂合性组织中存在的少数细胞时能够发挥特殊作用[46]。目前欧洲已经批准用循环性SEPT9甲基化筛查结肠癌, 本课题组用 p16甲基化早期预测上皮异型增生癌变诊断试剂也在开发中;用循环型 MGMT甲基化预测甲基脲类化疗药物敏感性开始用于临床试验; DNA甲基化阻断剂脱氧杂氮胞苷已经批准用于骨髓异型增生综合征的临床治疗, 开始向其他瘤种扩展[45]。我国是疾病资源和人力资源大国, 当前掀起的转化医学研究高潮使得DNA甲基化与疾病关系研究也在全国各地不断展开,近期有望涌现一批有临床用途的DNA甲基化产品。
[1] Marinus MG, Casadesus J. Roles of DNA adenine methylation in host-pathogen interactions: mismatch repair, transcriptional regulation, and more. FEMS Microbiol Rev, 2009, 33(3): 488–503.
[2] Chatti A, Landoulsi A. The DNA-methylation state regulates virulence and stress response of Salmonella. C R Biol, 2008, 331(9): 648–654.
[3] Collier J. Epigenetic regulation of the bacterial cell cycle. Curr Opin Microbiol, 2009, 12(6): 722–729.
[4] Saya Kagiwada, Kazuki Kurimoto, Takayuki Hirota, Masashi Yamaji and Mitinori Saitou. Replication-coupled passive DNA demethylation for the erasure of genome imprints in mice. EMBO J, 2012, 32: 340–353.
[5] Pleasance ED, Cheetham RK, Stephens PJ, McBride DJ, Humphray SJ, Greenman CD, Varela I, Lin ML, Ordóñez GR, Bignell GR, Ye K, Alipaz J, Bauer MJ, Beare D, Butler A, Carter RJ, Chen L, Cox AJ, Edkins S, Kokko-Gonzales PI, Gormley NA, Grocock RJ, Haudenschild CD, Hims MM, James T, Jia M, Kingsbury Z, Leroy C, Marshall J, Menzies A, Mudie LJ, Ning Z, Royce T, Schulz-Trieglaff OB, Spiridou A, Stebbings LA, Szajkowski L, Teague J, Williamson D, Chin L, Ross MT, Campbell PJ, Bentley DR, Futreal PA, Stratton MR. A comprehensive catalogue of somatic mutations from a human cancer genome. Nature, 2010, 463(7278): 191–196.
[6] Alexandrov LB, Nik-Zainal S, Wedge DC, Aparicio SA, Behjati S, Biankin AV, Bignell GR, Bolli N, Borg A, Børresen-Dale AL, Boyault S, Burkhardt B, Butler AP, Caldas C, Davies HR, Desmedt C, Eils R, Eyfjörd JE, Foekens JA, Greaves M, Hosoda F, Hutter B, Ilicic T, Imbeaud S, Imielinsk M, Jäger N, Jones DT, Jones D, Knappskog S, Kool M, Lakhani SR, López-Otín C, Martin S, Munshi NC, Nakamura H, Northcott PA, Pajic M, Papaemmanuil E, Paradiso A, Pearson JV, Puente XS, Raine K, Ramakrishna M, Richardson AL, Richter J, Rosenstiel P, Schlesner M, Schumacher TN, Span PN, Teague JW, Totoki Y, Tutt AN, Valdés-Mas R, van Buuren MM, van 't Veer L, Vincent-Salomon A, Waddell N, Yates LR, Australian Pancreatic Cancer Genome Initiative, ICGC Breast Cancer Consortium, ICGC MMML-Seq Consortium, ICGC PedBrain, Zucman-Rossi J, Futreal PA, McDermott U, Lichter P, Meyerson M, Grimmond SM, Siebert R, Campo E, Shibata T, Pfister SM, Campbell PJ, Stratton MR. Signatures of mutational processes in human cancer. Nature, 2013, 500(7463): 415–421.
[7] Popp C, Dean W, Feng S, Cokus SJ, Andrews S, Pellegrini M, Jacobsen SE, Reik W. Genome-wide erasure of DNA methylation in mouse primordial germ cells is affected by AID deficiency. Nature, 2010, 463: 1101–1105.
[8] Kriaucionis S, Heintz N. The nuclear DNA base 5-hydroxymethylcytosine is present in purkinje neurons and the brain. Science, 2009, 324(5929): 929–930.
[9] Tahiliani M, Koh KP, Shen Y, Pastor WA, Bandukwala H, Brudno Y, Agarwal S, Iyer LM, Liu DR, Aravind L, Rao A. Conversion of 5-methylcytosine to 5-hydroxymethylcytosine in mammalian DNA by MLL partner TET1. Science, 2009, 324(5929): 930–935.
[10] He YF, Li BZ, Li Z, Liu P, Wang Y, Tang Q, Ding J, Jia Y, Chen Z, Li L, Sun Y, Li X, Dai Q, Song CX, Zhang K, He C, Xu GL. Tet-mediated formation of 5-carboxylcytosine and its excision by TDG in mammalian DNA. Science, 2011, 333(6047): 1303–1307.
[11] Ito S, Shen L, Dai Q, Wu SC, Collins LB, Swenberg JA, He C, Zhang Y. Tet proteins can convert 5-methylcytosine to 5-formylcytosine and 5-carboxylcytosine. Science, 2011, 333(6047): 1300–1303.
[12] Gu TP, Guo F, Yang H, Wu HP, Xu GF, Liu W, Xie ZG, Shi L, He X, Jin SG, Iqbal K, Shi YG, Deng Z, Szabó PE, Pfeifer GP, Li J, Xu GL. The role of Tet3 DNA dioxygenase in epigenetic reprogramming by oocytes. Nature, 2011, 477(7366): 606–610.
[13] Williams K, Christensen J, Pedersen MT, Johansen JV, Cloos PA, Rappsilber J, Helin K. TET1 and hydroxymethylcytosine in transcription and DNA methylation fidelity. Nature, 2011, 473(7347): 343–348.
[14] Hackett JA, Sengupta R, Zylicz JJ, Murakami K, Lee C, Down TA, Surani MA. Germline DNA demethylation dynamics and imprint erasure through 5-hydroxymethylcytosine. Science, 2013, 339(6118): 448–452.
[15] Costa Y, Ding J, Theunissen TW, Faiola F, Hore TA, Shliaha PV, Fidalgo M, Saunders A, Lawrence M, Dietmann S, Das S, Levasseur DN, Li Z, Xu M, Reik W, Silva JCR, Wang J. NANOG-dependent function of TET1 and TET2 in establishment of pluripotency. Nature, 2013, 495(7441): 370–374.
[16] Jiang L, Zhang J, Wang JJ, Wang L, Zhang L, Li G, Yang X, Ma X, Sun X, Cai J, Zhang J, Huang X, Yu M, Wang X, Liu F, Wu CI, He C, Zhang B, Ci W, Liu J. Sperm, but not oocyte, DNA methylome is inherited by zebrafish early embryos. Cell, 2013, 153(4): 773–784.
[17] Potok ME, Nix DA, Parnell TJ, Cairns BR. Reprogramming the maternal zebrafish genome after fertilization to match the paternal methylation pattern. Cell, 2013, 153(4): 759–772.
[18] Almeida RD, Loose M, Sottile V, Matsa E, Denning C, Young L, Johnson AD, Gering M, Ruzov A. 5-hydroxymethyl-cytosine enrichment of non-committed cells is not a universal feature of vertebrate development. Epigenetics, 2012, 7(4): 383–389.
[19] Smith ZD, Chan MM, Mikkelsen TS, Gu H, Gnirke A, Regev A, Meissner A. A unique regulatory phase of DNA methylation in the early mammalian embryo. Nature, 2012, 484(7394): 339–344.
[20] Chen CC, Wang KY, Shen CK. The mammalian de novo DNA methyltransferases DNMT3A and DNMT3B are also DNA 5-hydroxymethylcytosine dehydroxymethylases. J Biol Chem, 2012, 287(40): 33116–33121.
[21] Qian W, Miki D, Zhang H, Liu Y, Zhang X, Tang K, Kan Y, La H, Li X, Li S, Zhu X, Shi X, Zhang K, Pontes O, Chen X, Liu R, Gong Z, Zhu JK. A histone acetyltransferase regulates active DNA demethylation in Arabidopsis. Science, 2012, 336: 1445–1448.
[22] Haffner MC, Pellakuru LG, Ghosh S, Lotan TL, Nelson WG, De Marzo AM, Yegnasubramanian S. Tight correlation of 5-hydroxymethylcytosine and Polycomb marks in health and disease. Cell Cycle, 2013, 12(12): 1835–1841.
[23] Shimoda N, Izawa T, Yoshizawa A, Yokoi H, Kikuchi Y, Hashimoto N. Decrease in cytosine methylation at CpG island shores and increase in DNA fragmentation during zebrafish aging. Age (Dordr), 2014, 36(1): 103–115.
[24] Bhattacharyya S, Yu Y, Suzuki M, Campbell N, Mazdo J, Vasanthakumar A, Bhagat TD, Nischal S, Christopeit M, Parekh S, Steidl U, Godley L, Maitra A, Greally JM, Verma A. Genome-wide hydroxymethylation tested using the HELP-GT assay shows redistribution in cancer. Nucleic Acids Res, 2013, 1(16): e157.
[25] Thaler R, Spitzer S, Karlic H, Klaushofer K, Varga F. DMSO is a strong inducer of DNA hydroxymethylation in pre-osteoblastic MC3T3-E1 cells. Epigenetics, 2012, 7(6): 635–651.
[26] Blaschke K, Ebata KT, Karimi MM, Zepeda-Martínez JA, Goyal P, Mahapatra S, Tam A, Laird DJ, Hirst M, Rao A, Lorincz MC, Ramalho-Santos M. Vitamin C induces Tet-dependent DNA demethylation and a blastocyst-like state in ES cells. Nature, 2013, 500(7461): 222–226.
[27] Thomson JP, Hunter JM, Lempiäinen H, Müller A, Terranova R, Moggs JG, Meehan RR. Dynamic changes in 5-hydroxymethylation signatures underpin early and late events in drug exposed liver. Nucleic Acids Res, 2013, 41(11): 5639–5654.
[28] Hansen TB, Jensen TI, Clausen BH, Bramsen JB, Finsen B, Damgaard CK, Kjems J. Natural RNA circles function as efficient microRNA sponges. Nature, 2013, 495(7441): 384–388.
[29] Yap KL, Li SD, Muñoz-Cabello AM, Raguz S, Zeng L, Mujtaba S, Gil J, Walsh MJ, Zhou MM. Molecular interplay of the noncoding RNA ANRIL and methylated histone H3 lysine 27 by polycomb CBX7 in transcriptional silencing of INK4a. Mol Cell, 2010, 38(5): 662–674.
[30] Gupta RA, Shah N, Wang KC, Kim J, Horlings HM, Wong DJ, Tsai MC, Hung T, Argani P, Rinn JL, Wang Y, Brzoska P, Kong B, Li R, West RB, van de Vijver MJ, Sukumar S, Chang HY. Long non-coding RNA HOTAIR reprograms chromatin state to promote cancer metastasis. Nature, 2010, 464(7291): 1071–1076.
[31] Huarte M, Guttman M, Feldser D, Garber M, Koziol MJ, Kenzelmann-Broz D, Khalil AM, Zuk O, Amit I, Rabani M, Attardi LD, Regev A, Lander ES, Jacks T, Rinn JL. A large intergenic noncoding RNA induced by p53 mediates global gene repression in the p53 response. Cell, 2010, 142: 409–419.
[32] Zhao J, Ohsumi TK, Kung JT, Ogawa Y, Grau DJ, Sarma K, Song JJ, Kingston RE, Borowsky M, Lee JT. Genome-wide identification of polycomb-associated RNAs by RIP-seq. Mol Cell, 2010, 40: 939–953.
[33] Guttman M, Rinn JL. Modular regulatory principles of large non-coding RNAs. Nature, 2012, 482(7385): 339–346.
[34] Poliseno L, Salmena L, Zhang J, Carver B, Haveman WJ, Pandolfi PP. A coding-independent function of gene and pseudogene mRNAs regulates tumour biology. Nature, 2010, 465(7301): 1033–1038.
[35] Tay Y, Kats L, Salmena L, Weiss D, Tan SM, Ala U, Karreth F, Poliseno L, Provero P, Di Cunto F, Lieberman J, Rigoutsos I, Pandolfi PP. Coding-independent regulation of the tumor suppressor PTEN by competing endogenousmRNAs. Cell, 2011, 147(2): 344–357.
[36] Di Ruscio A, Ebralidze AK, Benoukraf T, Amabile G, Goff LA, Terragni J, Figueroa ME, De Figueiredo Pontes LL, Alberich-Jorda M, Zhang P, Wu M, D'Alò F, Melnick A, Leone G, Ebralidze KK, Pradhan S, Rinn JL, Tenen DG. DNMT1-interacting RNAs block gene-specific DNA methylation. Nature, 2013, 503(7476): 371–376.
[37] Hu JL, Zhou BO, Zhang RR, Zhang KL, Zhou JQ, Xu GL. The N-terminus of histone H3 is required for de novo DNA methylation in chromatin. Proc Natl Acad Sci USA, 2009, 106(52): 22187–22192.
[38] Li BZ, Huang Z, Cui QY, Song XH, Du L, Jeltsch A, Chen P, Li G, Li E, Xu GL. Histone tails regulate DNA methylation by allosterically activating de novo methyltransferase. Cell Res, 2011, 21(8): 1172–1181.
[39] The ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome. Nature, 2012, 489(7414): 57–74.
[40] Lu ZM, Zhou J, Wang X, Guan Z, Bai H, Liu ZJ, Su N, Pan K, Ji J, Deng D. Nucleosomes correlate with in vivo progression pattern of de novo methylation of p16 CpG islands in human gastric carcinogenesis. PLoS ONE, 2012, 7(4): e35928.
[41] Wu LP, Wang X, Li L, Zhao Y, Lu S, Yu Y, Zhou W, Liu X, Yang J, Zheng Z, Zhang H, Feng J, Yang Y, Wang H, Zhu WG. Histone deacetylase inhibitor depsipeptide activates silenced genes through decreasing both CpG and H3K9 methylation on the promoter. Mol Cell Biol, 2008, 28(10): 3219–3235.
[42] Ficz G, Branco MR, Seisenberger S, Santos F, Krueger F, Hore TA, Marques CJ, Andrews S, Reik W. Dynamic regulation of 5-hydroxymethylcytosine in mouse ES cells and during differentiation. Nature, 2011, 473(7347): 398–402.
[43] Sun Z, Terragni J, Borgaro JG, Liu Y, Yu L, Guan S, Wang H, Sun D, Cheng X, Zhu Z, Pradhan S, Zheng Y. High-resolution enzymatic mapping of genomic 5-hydroxymethylcytosine in mouse embryonic stem cells. Cell Rep, 2013, 3(2): 567–576.
[44] Ruzov A, Tsenkina Y, Serio A, Dudnakova T, Fletcher J, Bai Y, Chebotareva T, Pells S, Hannoun Z, Sullivan G, Chandran S, Hay DC, Bradley M, Wilmut I, De Sousa P. Lineage-specific distribution of high levels of genomic 5-hydroxymethylcytosine in mammalian development. Cell Res, 2011, 21(9): 1332–1342.
[45] Deng DJ, Liu ZJ, Du YT. Epigenetic alterations as cancer diagnostic, prognostic, and predictive biomarkers. Adv Genet, 2010, 71: 125–176.
(责任编委: 朱卫国)
DNA methylation and demethylation: current status and future perspective
Dajun Deng
Peking University Cancer Hospital and Institute, Beijing 100142, China
DNA methylation plays important roles in cell differentiation, embryonic development, host adaptations to environmental factors, and pathogenesis through regulation of gene transcription and imprinting, X-inactivation, and defense of foreign genetic material invasion, is currently one of the hottest research fields on epigenetics. In the past few years, a number of important findings on DNA methylation have been achieved. These findings include discovery of TETs-catalyzed cytosine hydroxymethylation and its functions in the early embryonic development; the relationship between active and passive DNA demethylation; establishment and maintenance of DNA methylation patterns and their associations with histone modifications, chromatin configuration, polycomb group proteins and non-coding RNA bindings. DNA methylation has become a new potential biomarker and therapy target.
DNA methylation; demethylation; epigenetics; homeostasis; translational research
2014-01-07;
2014-01-27
国家自然科学基金项目(编号:30921140311,31261140372)资助
邓大君,教授,研究方向:肿瘤病因学和DNA甲基化研究。E-mail:dengdajun@bjmu.edu.cn
10.3724/SP.J.1005.2014.0403
时间: 2014-3-3 12:41:25
URL: http://www.cnki.net/kcms/detail/11.1913.R.20140303.1241.001.html

