蒺藜苜蓿叶绿体密码子偏好性分析
杨国锋,苏昆龙,赵怡然,宋智斌,孙娟
(1.青岛农业大学经济草本植物应用研究所,山东 青岛 266109;2.青岛农业大学生命科学学院,山东省高校植物生物技术重点实验室,山东 青岛266109)
蒺藜苜蓿叶绿体密码子偏好性分析
杨国锋1,2*,苏昆龙1,2,赵怡然1,宋智斌1,2,孙娟1
(1.青岛农业大学经济草本植物应用研究所,山东 青岛 266109;2.青岛农业大学生命科学学院,山东省高校植物生物技术重点实验室,山东 青岛266109)
本文对蒺藜苜蓿叶绿体基因组全序列密码子进行分析,筛选出50条CDS(coding DNA sequence)利用CodonW软件进行分析其密码子使用模式。结果显示,蒺藜苜蓿叶绿体基因组密码子第3位碱基GC含量为26.9%,即第3位密码子富含A和U,ENC值在37.11~51.91之间密码子偏好性较弱。相对同义密码子使用度分析显示RSCU值大于1的密码子有23个,其中以A和U为结尾20个。中性绘图分析显示GC12与GC3的相关系数为0.341,相关性不显著,回归系数为0.4843;单基因ENC比值多分布在-0.05~0.05,即大部分基因ENC值离ENC期望值较近;对应性分析,第一轴显示了12.50%的差异为主要影响因素,第一轴与ENC和GC3的相关系数分别为0.091和-0.092,均相关不显著。综合这几项分析发现蒺藜苜蓿叶绿体基因组密码子偏好性主要受到突变的影响,但是并不是唯一的影响因素,其他因素对密码子偏好性也可能有一定的影响。最终通过高表达优越密码子方法确定得出UUA、UUG、CCU等23个密码子为最优密码子,为之后对外源基因进行改造,提高其在叶绿体中的表达效率奠定了基础。
蒺藜苜蓿;叶绿体;密码子偏好性;最优密码子
遗传信息的传递是生物进行生命活动最主要环节,在这一活动中密码子扮演着重要的角色,编码不同氨基酸密码子的准确识别是保证遗传信息正确表达的关键。密码子具有兼并性,即同一氨基酸能由一个或多个密码子所对应,这样的密码子称为同义密码子(synonymous codon)。研究发现,密码子的使用存在不均等的现象,即有的密码子使用频率高于其他同义密码子的使用,这一现象称为密码子偏好性(codon usage bias)。密码子偏好性是在生物长期进化过程中所形成的,不同的物种间密码子使用的偏好性不同。由于密码子偏好性的存在常导致外源基因在宿主中表达量降低[1]。研究密码子的使用模式对探究物种的进化模式和提高外源基因的表达具有重要意义。
关于密码子偏好性形成的机制,最早Grantham等[2]提出的“多基因假说”认为在长期的系统发育进化过程中,不同的基因组存在不同的密码子使用模式,暗示环境的影响(突变压力)是密码子偏好性形成的主要影响因素。Ikemura[3]发现高表达基因更倾向于使用一些特定的密码子,这与tRNA的丰富度相对应,并提出选择压力通过优化翻译效率和精确度来影响同义密码子的使用频率。近年来的研究发现,不同物种间密码子偏好性的差异并非由单一因素决定。Sharp和Li[4]发现密码子的使用情况与同义替换率有关,Olejniczak和Uhlenbeck[5]通过对单细胞组织进行研究发现密码子的使用情况与tRNA的丰富度有关系。同时密码子亲水性、DNA复制起止位点[6]、基因长度[7]、翻译准确性选择[8-10]和表达水平[11]等因素对密码子偏好性也产生影响。
与核基因组相比叶绿体基因组密码子使用模式的研究相对落后些,目前数据库中完整的叶绿体基因序列数据相对有限,仅有玉米(Zeamays)、水稻(Oryzasativa)、文心兰(Oncidium)、银白杨(Populusalba)等植物的完整叶绿体基因组数据。苜蓿(Medicago)为一年生或多年生草本植物,是重要的豆科牧草。随着基因测序技术的发展,蒺藜苜蓿(Medicagotruncatula)为研究豆科植物的尤其是苜蓿属的模式植物[12],其叶绿体基因组的测序已经完成,但是关于苜蓿叶绿体密码子使用模式的研究还未见报道。本研究通过对蒺藜苜蓿叶绿体基因组密码子使用模式以及影响密码子使用模式的主要因素进行分析,为以后苜蓿叶绿体的研究和应用奠定基础。
1 材料与方法
1.1材料
2014年10月从GenBank下载完整的蒺藜苜蓿叶绿体基因组(登录编号:NC_003119.6)。共下载到不含重复的76条CDS(coding DNA sequence),由于短序列不能正确地计算有效密码子数[13],研究需去除其中长度小于300 bp的CDS。本研究去除26条长度小于300 bp的CDS,剩余50条CDS用于后面的数据分析。
1.2方法
在分析时去除不编码任何氨基酸的终止密码子UAA、UGA和UAG,还有色氨酸的唯一密码子UGG和蛋氨酸的唯一密码子AUG,这些密码子不存在偏好性。使用DNAMAN将50条DNA序列编码成可在CodonW软件中运行的.dat格式文件,运行CodonW软件对基因序列进行分析得到相应数据。对所得到的数据进行整理,使用SPSS及Excel进行下文所述的相关作图及分析。对序列进行3次重复分析,每次分析均得到相同的实验结果。
1.2.1相对同义密码子使用度分析 相对同义密码子使用度(relative synonymous codon usage, RSCU)表示该密码子的实际使用值与理论使用值之间的比值[14]。当RSCU<1时,表示该密码子的使用频率低于其他同义密码子;RSCU>1时,则表示该密码子的使用频率高于同义密码子;RSCU=1表示该密码子没有偏好性。
1.2.2中性绘图分析 统计密码子3个位置的GC含量,第1,2,3位的GC含量分别表示为GC1、GC2、GC3,GC12表示GC1和GC2的平均值。中性绘图分析以GC12为纵坐标,以GC3为横坐标作图。通过分析密码子的第1,2位和第3位碱基组成的相关性,研究密码子的偏好性影响因素。当GC12与GC3之间显著相关,说明3个位置上的碱基组成无差异,密码子的使用受突变的影响。当GC12与GC3相关性不显著,回归系数接近0,说明第1,2位和第3位碱基组成不同,基因组GC含量高度保守,密码子的使用更多地受选择影响[15]。
1.2.3ENC-plot绘图分析 有效密码子数(effective number of codon,ENC)反映的是密码子偏离随机选择的程度[16],是反映同义密码子非均衡使用偏好程度的重要指标,通常高表达基因其密码子偏好程度较大,因此ENC取值较小;低表达基因含有较多种类的稀有密码子偏好性较弱,ENC值也较大。ENC-plot绘图分析以ENC为纵坐标,GC3为横坐标作图。该分析主要检测碱基组成对密码子偏好性的影响,绘图中的标准曲线表示密码子偏好性仅由碱基组成决定基因位置,标准曲线计算方法如下:
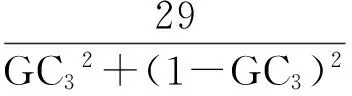
基因沿标准曲线分布或落在标准曲线附近表示该基因密码子偏好性仅受突变影响,基因落在标准曲线下方较远的位置表示该基因密码子偏好性受到选择的影响。
1.2.4PR2-plot绘图分析 PR2偏倚分析(PR2-bias plot analysis)是为了避免密码子第3位碱基A与T和C与G之间突变不平衡,根据偏倚规则(parity rule 2,PR2),如果两条互补链间不存在任何突变或选择效应上的偏倚,那么碱基含量上应该有A=T和C=G。对由4个同义密码子编码的氨基酸的各个密码子第3位A、T、C、G的情况进行分析,计算每个基因A3/(A3+T3)和G3/(G3+C3),分别作纵坐标和横坐标作图,图中中心点表示A=T且C=G,其余的点由中心点向该点发出的矢量表示了该基因的偏倚程度和方向[17]。
1.2.5对应性分析 对应性分析(correspondence analysis)是一种多元相依的变量统计分析技术,通过分析由定性变量构成的交互汇总表来揭示变量间的联系,可以揭示同一变量的各个类别之间的差异,以及不同变量各个类别之间的对应关系。为探究不同基因在密码子用法上的变异趋势,基于RSCU值进行对应性分析,CodonW软件会将样本中所有基因分布到一个58维(58个同义密码子)的向量空间中,其中每个点就代表一个同义密码子。密码子的用法特点通过图中每个点之间的位置体现出来。通过探究基因向量及基因间的值的变异情况,推测出密码子偏好性的主要影响因素。第一轴表现最大差异的密码子使用变化,随后的副轴变化量逐渐降低。McInerney[18]指出,两个主轴间基因的分散代表了主要的密码子变化趋势。将表现差异最大的第一轴和第二轴分别作为横纵坐标作图,图中点代表不同基因,根据图中点的分布情况判断基因密码子的使用模式。
1.2.6最优密码子的确定 最优密码子(the most preferred codon)的选择,以ENC为偏好性标准,两极各选10%的基因,分别建成高低偏性库,取两库ΔRSCU>0.08的密码子[19]。
本研究所使用的软件包括DNAMAN 6.0、CodonW 1.4.2、SPSS 19和Excel 2013。
2 结果与分析
2.1密码子组成分析
使用CodonW软件对各基因编码序列进行分析(表1)。原则上ENC的取值范围为20~61,ENC值大小反映的密码子偏性的强弱,当ENC为20时,表示同义密码子完全偏倚;为61时,表示同义密码子没有偏倚;按照惯例以35作为偏性强弱的区分标准[18],表中ENC取值范围在37.11~51.91之间,并且大部分大于45,所以蒺藜苜蓿叶绿体基因密码子偏性较弱。密码子第3位GC的平均含量为26.9%,明显低于前两位45.5%和36.8%的GC含量,说明密码子第3位碱基组成多为A和U。
GC1、GC2、GC3、GCall、ENC和密码子数目N之间的关联分析见表2。GCall与GC1、GC2、GC3之间的相关性均达到了极显著水平,GC1与GC2显著相关,GC3与GC1、GC2均未达到显著水平,说明密码子的第1位与第2位的碱基组成比较相似,但是与第3位不同。ENC与密码子第1、第2位置GC含量相关但是相关性不显著,与密码子第3位的GC含量显著相关。ENC与密码子数目N之间未达到显著水平,说明密码子数目对ENC的影响很弱,即排除了基因序列过短对密码子偏好性的影响。
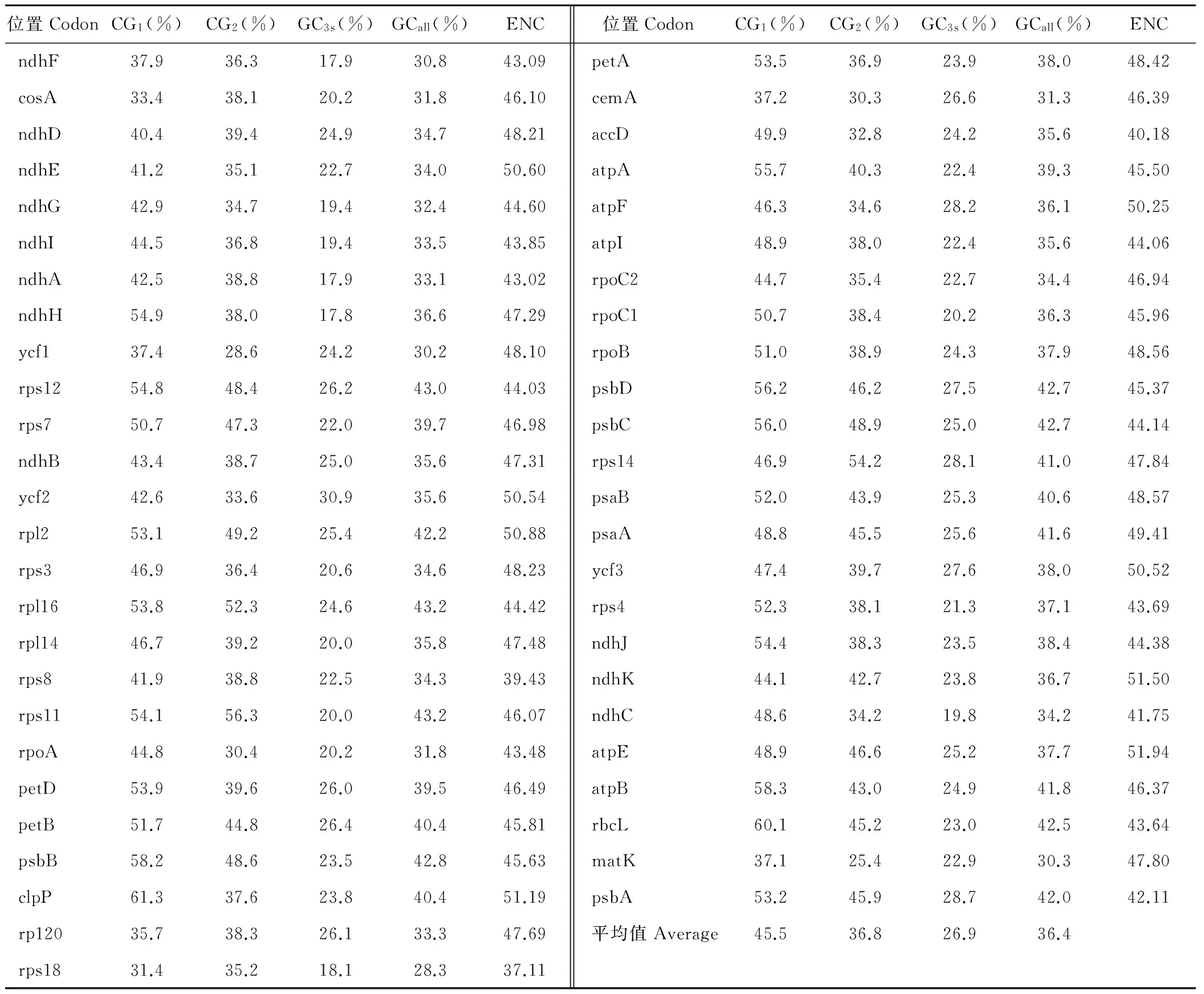
表1 蒺藜苜蓿叶绿体基因组不同位置的GC含量Table 1 GC content of different positions of codon in chloroplast of M.truncatula
各种氨基酸的RSCU值分析见表3。从表中可以看出,RSCU值大于1的主要是以U和A为结尾的密码子,密码子以U和A为结尾的出现频率比较高,说明这些为基因组偏爱密码子。相反以C和G为结尾的密码子出现频率比较低,这些是基因组的非偏爱密码子。
2.2中性绘图分析
苜蓿叶绿体基因组各基因中性绘图分析见图1,可以看出GC12的取值范围在0.178~0.309之间,GC3取值范围在0.321~0.552之间。图中各基因未落在对角线上或沿对角线分布,都在对角线上方分布。GC12与GC3的相关系数为0.341,相关性不显著,回归系数(即图中斜率)为0.4843,说明第一、二位和第三位碱基组成无差异,蒺藜苜蓿叶绿体基因组密码子的使用更多地受突变的影响。
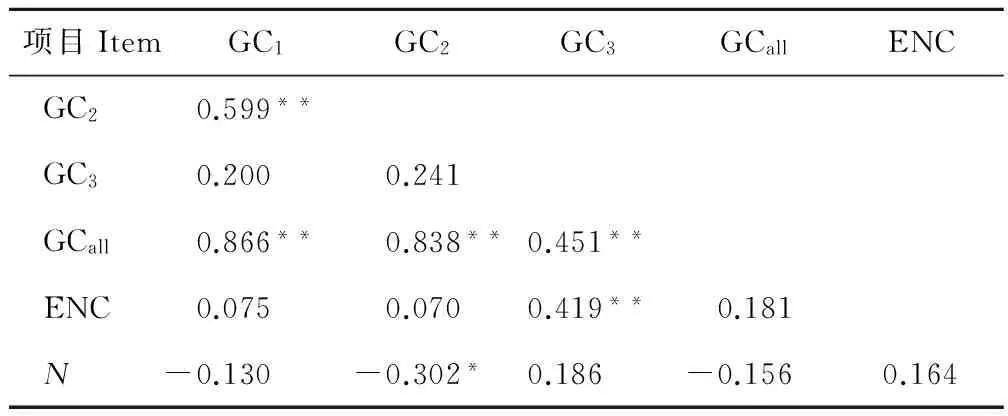
表2 各基因相关性分析Table 2 Correlation analysis of each genes related parameters
** 在0.01水平上显著相关;* 在0.05水平上显著相关。
** Significant correlation atP<0.01; *Significant correlation atP<0.05.

表3 蒺藜苜蓿各氨基酸相对同义密码子使用度Table 3 RSCU analysis of protein coding region in M.truncatula
2.3ENC-plot绘图分析
ENC与GC3的关联分析见图2,由图可以看出部分基因位点沿着标准曲线分布或者落在标准曲线附近,这些基因的实际ENC值与预期ENC值接近,部分基因位点落在期望值的下方。为了更直接地显示实际ENC值和预期ENC值之间的差异,计算了ENC比值即(预期ENC值-实际ENC值)/预期ENC值,统计ENC比值频数,从而显示出有多少基因距离期望值更近。从表4中可以看出大多数的ENC比值分布在-0.05~0.05之间,这一范围内实际ENC值与预期ENC值相差较小,因此蒺藜苜蓿叶绿体基因组密码子偏好性与GC3的差异相关。所以蒺藜苜蓿叶绿体基因组密码子偏好性更多地受突变的影响,受选择地影响相对较弱一些。
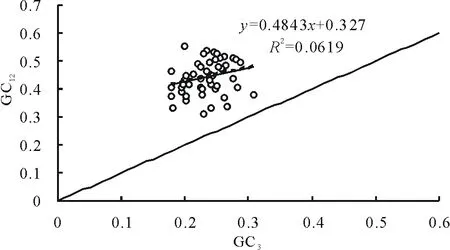
图1 中性绘图分析
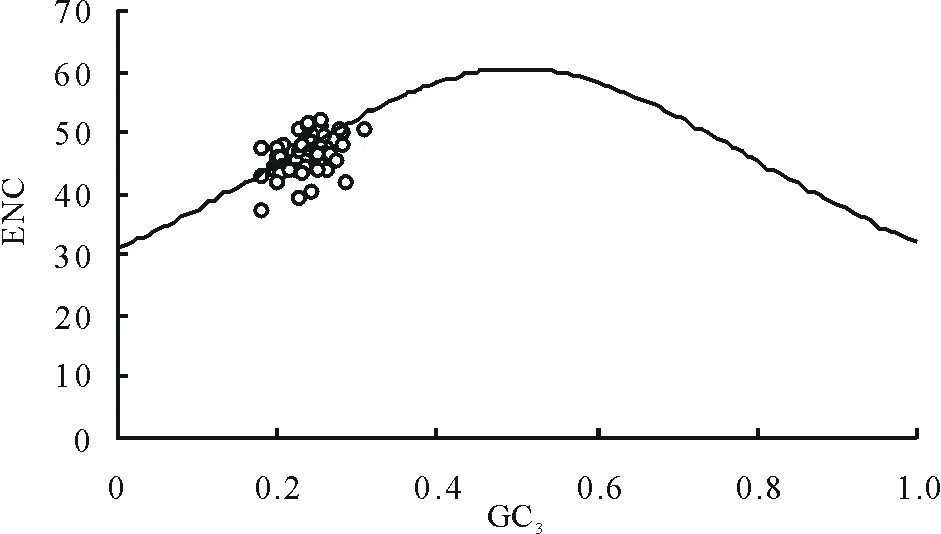
图2 ENC-plot绘图分析
2.4PR2-plot绘图分析
通过PR2-plot绘图分析了各基因部分氨基酸的嘌呤(A和G)嘧啶(T和C)的关系。由图3可以看出大部分基因分布在平面图的右下方,说明T的使用频率高于A,G的使用频率高于C。若完全受到突变的影响,A和T以及G和C的使用频率应当相等,因此蒺藜苜蓿叶绿体基因组密码子使用模式不仅是受到突变的影响,还受其他因素的影响。
2.5对应性分析
基于RSCU的对应性分析中第1向量轴显示了12.50%的差异,另外3个轴分别显示了9.01%,8.89%,8.23% 的差异,前4轴的累积差异38.63%,说明第1轴是影响密码子叶绿体基因组密码子偏好性的主要因素。第1轴与ENC和GC3的相关系数分别为0.091和-0.092,均相关不显著,说明GC3并不是造成蒺藜苜蓿叶绿体基因组密码子偏好性的主要因素,暗示影响密码子使用模式的因素并不单一。以第1轴为横坐标,第2轴为纵坐标,按照基因功能将基因分布于平面上(图4)。编码核糖体蛋白基因和遗传系统基因分布相对比较集中,说明这些基因的密码子使用模式相近;而其他基因则分布比较分散,说明这些密码子的使用模式存在差异。
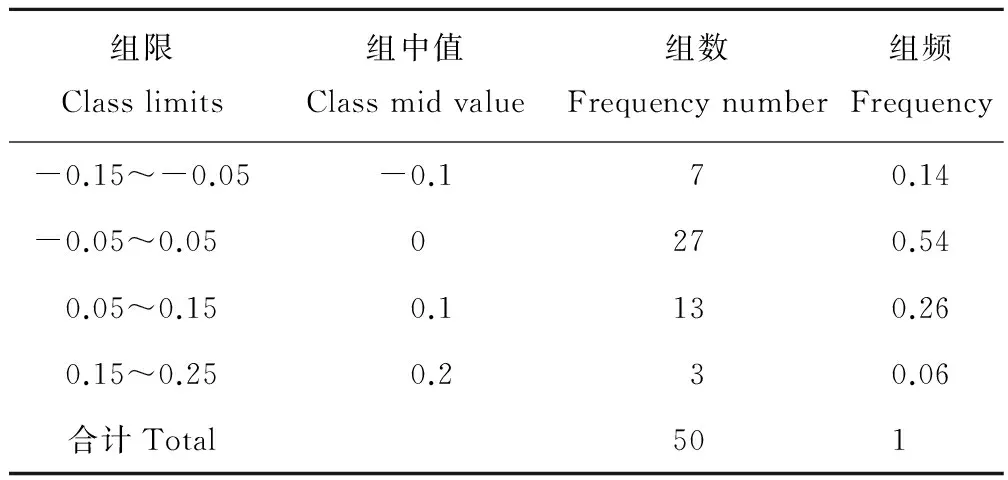
表4 ENC比值频数分布Table 4 Distribution of ENC ration
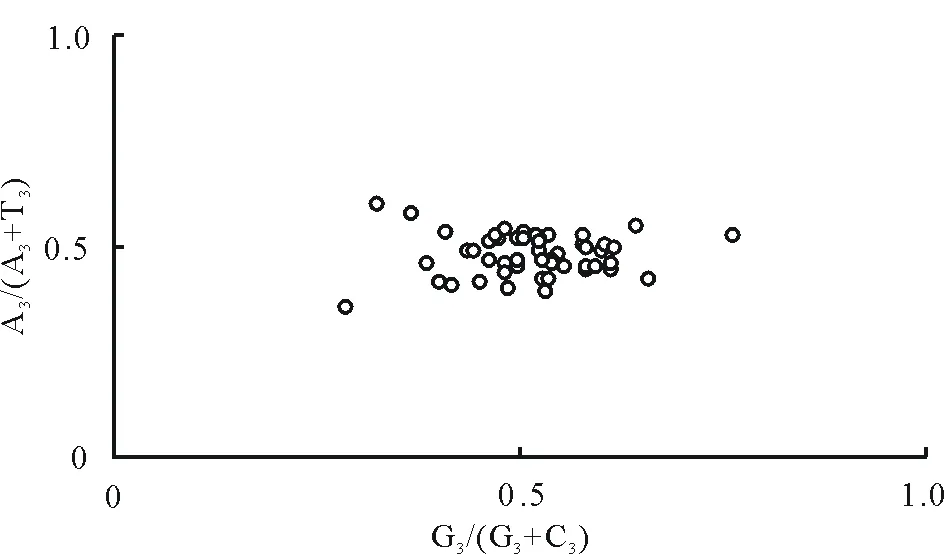
图3 PR2-plot绘图分析
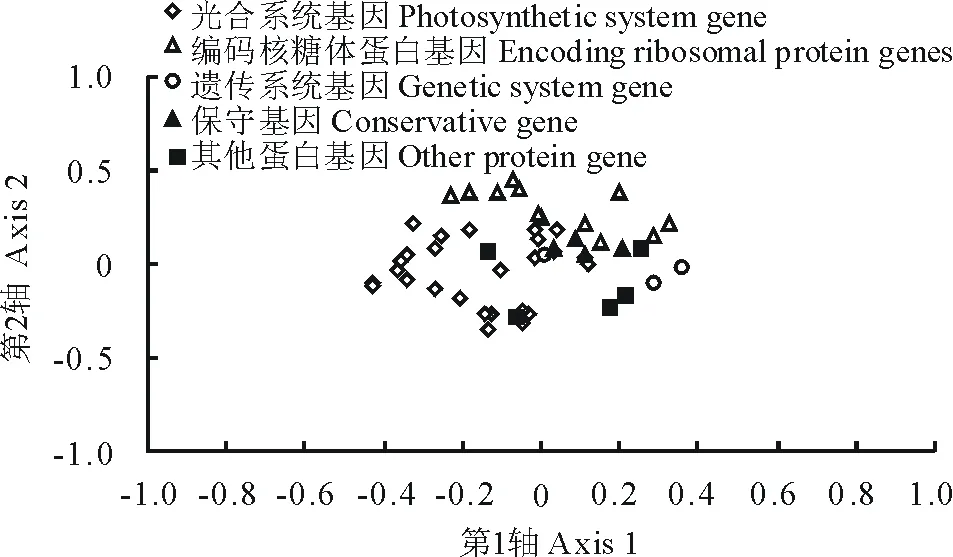
图4 基于RSCU的对应性分析
2.6最优密码子分析
按ENC值的大小对各基因进行排序,两极各选取5个基因构建高表达基因库和低表达基因库,并对两个库中密码子分别计算RSCU值,计算ΔRSCU值(表5),表中带星号的为最优密码子,其中可以看出ΔRSCU>0.5的均为以A和U为结尾的密码子。
3 讨论
密码子偏好性是由多个因素共同作用的结果,其中中性选择影响和方向突变影响是两个主要因素,哪一个因素为主导因素是目前的关注热点。密码子的碱基组成是由核苷酸突变和回复突变的结果,是对密码子使用最普遍的影响[19-21],尤其是密码子第3位的改变通常不会引起编码氨基酸的改变,第3位上的碱基突变受到的选择压力比较小,因此研究第3位碱基的碱基组成对研究密码子偏好性有重要意义。通过对第3位密码子的GC含量计算发现,第3位密码子富含A和T。本研究通过对蒺藜苜蓿叶绿体基因组中的基因部分氨基酸的密码子进行PR2-plot绘图分析发现,密码子第3位碱基T的使用频率高于A,G的使用频率高于C。尚明照等[22]以及Zhang等[23]分别对陆地棉(Gossypiumhirsutum)和小麦(Triticumaestivum)叶绿体基因组进行研究,发现密码子第3位碱基T的使用频率高于A,C的使用频率略高于G;张月荣[24]对23种禾本科植物叶绿体密码子进行分析发现密码子第3位碱基C和G使用频率相当,T的使用频率高于A。这表明不同植物的叶绿体基因组的碱基组成存在一定的差异,暗示影响密码子偏好性的因素也不相同。
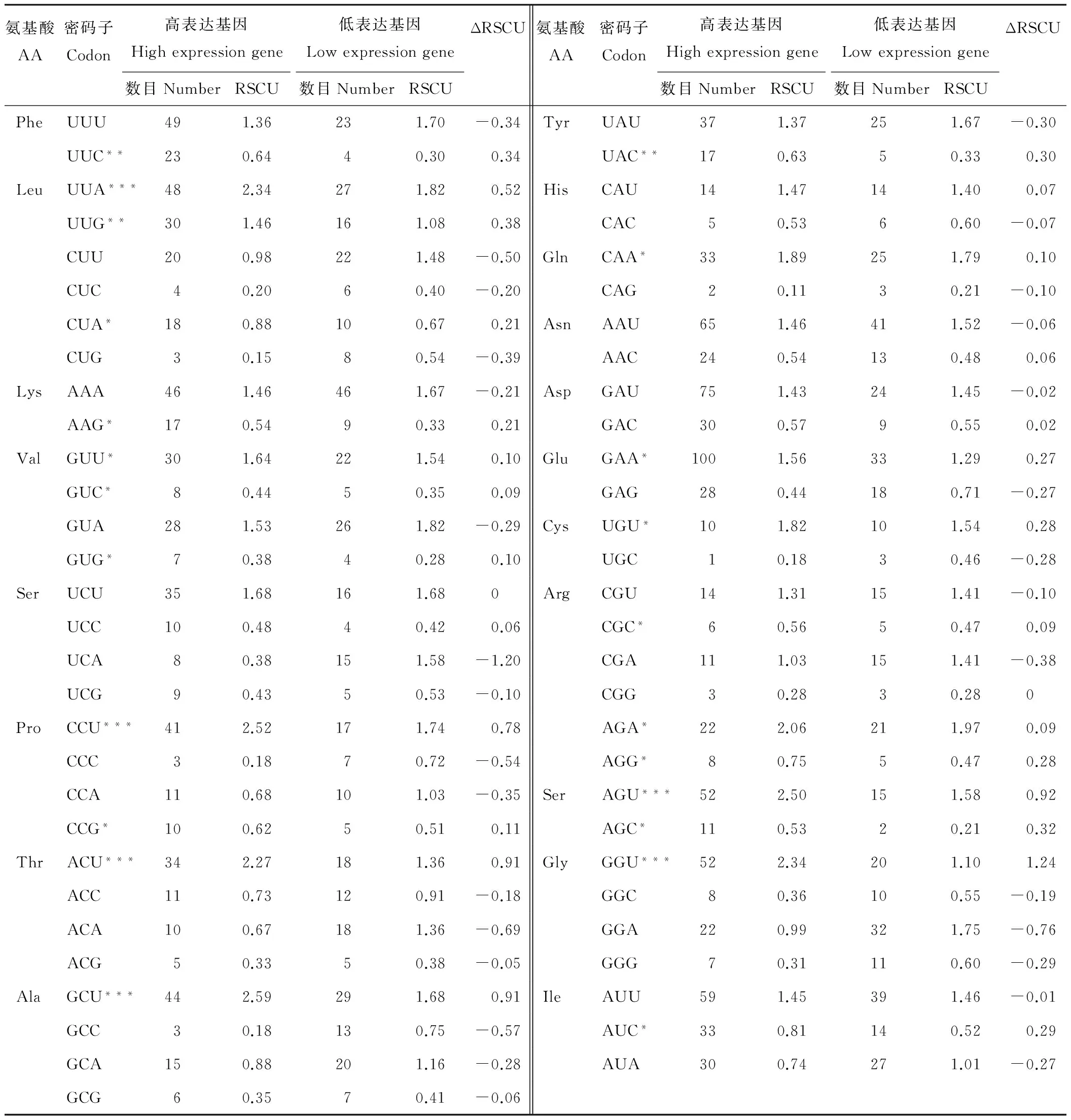
表5 蒺藜苜蓿叶绿体基因组最优密码子确定Table 5 Preferred codons in chloroplast genome of M.truncatula
*表示ΔRSCU>0.08,**表示ΔRSCU>0.3,***表示ΔRSCU>0.5。
* mean ΔRSCU>0.08,** mean ΔRSCU>0.3,*** mean ΔRSCU>0.5.
结合中性绘图分析、ENC-plot绘图分析以及对应性分析,发现蒺藜苜蓿叶绿体基因组密码子偏好性主要受到突变的影响,但是并不是唯一的影响因素,其他因素也对密码子偏好性造成影响。Zhou等[25]在对拟南芥(Arabidopsisthaliana)、杨树等植物的研究中指出,叶绿体基因组密码子偏好性主要受到突变影响,其他因素也对其产生影响,本研究结果与之相同。但是续晨等[26]对蝴蝶兰(Phalaenopsisaphrodite)叶绿体密码子偏好性进行研究,发现其密码子偏好性主要受到碱基差异和自然选择的影响。因此可以看出不同植物密码子偏好性的影响因素不同,但是影响植物密码子偏好性的因素都不是单一的,是多因素综合的结果。
本研究采用高表达优越密码子方法确定最优密码子,选出了23个最优密码子,确定的最优密码子中第3位碱基为A、U、C、G的分别5,7,6,5个,但是ΔRSCU>0.5的均以A和U为结尾。这与前人对水稻[27]、银白杨[28]等植物的研究结果中叶绿体基因组中密码子偏好NNA、NNU型相符合。
4 结论
蒺藜苜蓿叶绿体基因组密码子偏好性受突变的影响比较大,但与此同时也受到其他因素的影响,密码子偏好性是多因素影响的结果。确定了23个最优密码子,为以后通过外源基因密码子改造后转入苜蓿叶绿体中提高其表达量[29]。
[1] Wu X M, Wu S F, Ren D M,etal.The analysis method and progress in the study of codon bias.Hereditas, 2007, 29(4):420-426.
[2] Grantham R, Gautier C, Gouy M.Codon frequencies in 119 individual genes confirm consistent choices of degenerate bases according to genome type.Nucleic Acids Research, 1980, 8(9):1893-1912.
[3] Ikemura T.Codon usage and tRNA content in unicellular and multicellular organisms.Molecular Biology and Evolution, 1985, 2(1):13-34.
[4] Sharp P M, Li W H.The rate of synonymous substitution in enterobacterial genes is inversely related to codon usage bias.Molecular Biology and Evolution, 1987, 4(3):222-230.
[5] Olejniczak M, Uhlenbeck O C.tRNA residues that have coevolved with their anticodon to ensure uniform and accurate codon recognition.Biochimie, 2006, 88(8):943-950.
[6] Deschavanne P, Filipski J.Correlation of GC content with replication timing and repair mechanisms in weakly expressedE.coligenes.Nucleic Acids Research, 1995, 23(8):1350-1353.
[7] Sun Z, Ma L, Murphy R,etal.Analysis of codon usage onWolbachiapipientiswMel genome.Science in China Series C:Life Sciences, 2009, 39(10):948-953.
[8] Xia X H.Mutation and selection on the anticodon of tRNA genes in vertebrate mitochondrial genomes.Gene, 2005, 345(1):13-20.
[9] Zalucki Y M, Power P M, Jennings M P.Selection for efficient translation initiation biases codon usage at second amino acid position in secretory proteins.Nucleic Acids Research, 2007, 35(17):5748-5754.
[10] Huang Y, Koonin E V, Lipman D J,etal.Selection for minimization of translational frame shifting errors as a factor in the evolution of codon usage.Nucleic Acids Research, 2009, 37(20):6799-6810.
[11] Hiraoka Y, Kawamata K, Haraguchi T,etal.Codon usage bias is correlated with gene expression levels in the fission yeastSchizosaccharomycespombe.Genes to Cells, 2009, 14(4):499-509.
[12] Zhang J, Song L L, Guo D L,etal.Genome-wide identification and investigation of the MADS-box gene family inMedicagotruncatula.Acta Prataculturae Sinica, 2014, 23(6):233-241.
[13] Rosenberg M S, Subramanian S, Kumar S.Patterns of transitional mutation biases within and among mammalian genomes.Molecular Biology and Evolution, 2003, 20(6):988-993.
[14] Sharp P M, Li W H.The codon adaptation index-a measure of directional synonymous codon usage bias, and its potential applications.Nucleic Acids Research, 1987, 15(3):1281-1295.
[15] Sueoka N.Directional mutation pressure and neutral molecular evolution.Proceedings of the National Academy of Sciences of the United States of America, 1988, 85(8):2653-2657.
[16] Wright F.The “effective number of codons” used in a gene.Gene, 1990, 87(1):23-29.
[17] Sueoka N.Near homogeneity of PR2-Bias fingerprints in the human genome and their implications in phylogenetic analyses.Journal of Molecular Evolution, 2001, 53(4-5):469-476.
[18] McInerney J O.Replicational and transcriptional selection on codon usage inBorreliaburgdorferi.Proceedings of the National Academy of Sciences of the United States of America, 1998, 95(18):10698-10703.
[19] Yang H J, Liu G S, Zhang S T,etal.Codon usage bias studies and cluster analysis on chloroplastic gene in tobacco.Acta Tabacaria Sinica, 2012, 18(2):37-43.
[20] Jiang Y, Deng F, Wang H L.An extensive analysis on the global codon usage pattern of baculoviruses.Archives of Virology, 2008, 153(12):2273-2282.
[21] Sueoka N, Kawanishi Y.DNA G+C content of the third codon position and codon usage biases of human genes.Gene, 2000, 261(1):53-62.
[22] Shang M Z, Liu F, Hua J P,etal.Analysis on codon usage of chloroplast genome ofGossypiumhirsutum.Scientia Agricultura Sinica, 2011, (2):245-253.
[23] Zhang W J, Zhou J, Li Z F,etal.Comparative analysis of codon usage patterns among mitochondrion, chloroplast and nuclear genes inTriticumaestivumL.Journal of Integrative Plant Biology, 2007, 49(2):246-254.
[24] Zhang Y R.Analysis of Codon Usage Patterns of the Chloroplast Genomes in the Poaceae Family and RNA Editing Sites in the Chloroplast Transcripts ofAgreartinaadenophorum[D].Yangling:Northwest A&F University, 2013.
[25] Zhou M, Long W, Li X.Patterns of synonymous codon usage bias in chloroplast genomes of seed plants.Forest Study of China, 2008, 11(4):235-242.
[26] Xu C, Ben A L, Cai X N.Analysis of synonymous codon usage in chloroplast genome ofPhalaenopsisaphroditesubsp.Formosana.Molecular Plant Breeding, 2011, 8(5):945-950.
[27] Liu Q P, Xue Q Z.Codon usage in the chloroplast genome of rice (OryzasativaL.ssp.japonica).Acta Agronomica Sinica, 2004, 30(12):1220-1224.
[28] Zhou M, Long W, Li X.Analysis of synonymous codon usage in chloroplast genome ofPopulusalba.Journal of Forestry Research, 2008, 19(4):293-297.
[29] Zhang X M, Xin W, Wang S F,etal.Optimizing the codon usage of HIV-1 gag gene according to the codon bias of vaccinia virus improves the gag gene expression.Chinese Journal of Virology, 2005, 21(3):210-216.
参考文献:
[1] 吴宪明, 吴松锋, 任大明.等.密码子偏性的分析方法及相关研究进展.遗传, 2007, 29(4):420-426.
[7] 孙铮, 马亮, Murphy R, 等.Wolbachia pipientis wMel 基因组水平上的密码子使用分析.中国科学C辑:生命科学, 2009, 39(10):948-953.
[12] 张军, 宋丽莉, 郭东林, 等.MADS-box 基因家族在蒺藜苜蓿的全基因组分析.草业学报, 2014, 23(6):233-241.
[19] 杨惠娟, 刘国顺, 张松涛, 等.烟草叶绿体密码子的偏好性及聚类分析.中国烟草学报, 2012, 18(2):37-43.
[22] 尚明照, 刘方, 华金平, 等.陆地棉叶绿体基因组密码子使用偏性的分析.中国农业科学, 2011, (2):245-253.
[24] 张月荣.禾本科叶绿体基因组密码子使用模式及紫茎泽兰叶绿体RNA编辑分析[D].杨凌:西北农林科技大学, 2013.
[26] 续晨, 贲爱玲, 蔡晓宁.蝴蝶兰叶绿体基因组密码子使用的相关分析.分子植物育种, 2010, 8(5):945-950.
[27] 刘庆坡, 薛庆中.粳稻叶绿体基因组的密码子用法.作物学报, 2004, 30(12):1220-1224.
[29] 张相民, 辛伟, 王世峰, 等.按痘苗病毒优势密码子改造HIV-1 gag基因提高表达水平的研究.病毒学报, 2005, 21(3):210-216.
Analysis of codon usage in the chloroplast genome ofMedicagotruncatula
YANG Guo-Feng1,2*, SU Kun-Long1,2, ZHAO Yi-Ran1, SONG Zhi-Bin1,2, SUN Juan1
1.InstituteofEconomicHerbPlants,QingdaoAgriculturalUniversity,Qingdao266109,China; 2.CollegeofLifeSciences,QingdaoAgriculturalUniversity,KeyLabofPlantBiotechnologyinUniversitiesofShandongProvince,Qingdao266109,China
The complete nucleotide sequence of the chloroplast genome ofMedicagotruncatulawas investigated.Fifty CDS (coding DNA sequences) selected from the chloroplast genome sequence ofM.truncatula, were analyzed using CodonW software.The results show that the third codon position was rich in A and U.ENC ranged from 37.1 to 51.9 meaning that the codon bias was weak.There were 23 codons with relative synonymous codon usage greater than 1 and 20 codons ending with A and T.ENC-plot analysis showed that GC3was not correlated with GC12; ENC ratio’s of most genes ranged from -0.05 to 0.05.In the correspondence analysis of the first group of four axes, the first axis showed 10.3% variation.The correlation coefficients for axis 1 between ENC and GC3were 0.091 and -0.092 respectively (not significant).Synonymous codon usage bias was found, mainly due to the effect of mutation pressure, but there were other factors.In addition, analysis of the high expression codons enabled 23 to be affirmed as the “optimal codons” as UAA, UUG, CCU.The results provide evidence for molecular modification of exogenous genes to increase the expression efficiency inM.truncatulachloroplasts.
Medicagotruncatula; chloroplast; codon usage bias; codon usage bias
10.11686/cyxb2015016
http://cyxb.lzu.edu.cn
2015-01-12;改回日期:2015-03-12
现代农业产业技术体系(CARS-35),公益性行业项目(200903060)和国家自然科学基金青年科学基金项目(31200906)资助。
杨国锋(1977-),男,山东青岛人,在读博士。
*通信作者Corresponding author.E-mail:yanggf@qau.edu.cn
杨国锋, 苏昆龙, 赵怡然, 宋智斌, 孙娟.蒺藜苜蓿叶绿体密码子偏好性分析.草业学报, 2015, 24(12):171-179.
YANG Guo-Feng, SU Kun-Long, ZHAO Yi-Ran, SONG Zhi-Bin, SUN Juan.Analysis of codon usage in the chloroplast genome ofMedicagotruncatula.Acta Prataculturae Sinica, 2015, 24(12):171-179.

