基于Miseq高通量测序技术的古襄阳酒窖泥细菌多样性评价
杨小丽,尚雪娇,余海忠,刘文汇,杨少勇,郭 壮*
(1.湖北文理学院 食品科学技术学院 鄂西北传统发酵食品研究所,湖北 襄阳 441053;2.湖北古襄阳酒业有限公司,湖北 襄阳 441100)
作为我国销量最大的白酒类型,浓香型白酒是以粮谷为主要原料,经传统固态法发酵、蒸馏、陈酿和勾兑而成[1]。浓香型白酒的发酵主要在窖池中完成,窖泥微生物区系的形成和演变很大程度上决定了产品的质量[2],因而对窖泥中微生物的群落结构进行解析则显得尤为重要。目前国内学者常采用传统微生物学手段[3-5]和以变性梯度凝胶电泳技术(denaturing gradient gel electrophoresis,DGGE)为代表的指纹图谱技术[6-8]揭示窖泥中微生物的多样性。然而传统微生物学手段依赖于纯培养技术,对于多数营养要求苛刻、严格厌氧的微生物分离较为困难,难以获取微生物群落更为完整全面的信息[9]。虽然指纹图谱技术具有无需培养的技术优势,但仍存在无法实现样品间平行分析和图谱条带信息量低的缺陷[10]。
以Illumina MiSeq为代表的第二代高通量测序技术具有通量高、准确率高和试验周期短的特点,可在分类学地位“属”水平上全面客观地揭示目标环境中微生物群落信息[11]。LIU M等[12]分别采用DGGE和MiSeq技术对泸州老窖5年与100年窖泥中的真菌微生物进行了研究,结果发现DGGE仅检测到了12个菌属,而Miseq技术可以检测到111个菌属。湖北省是国内白酒生产与消费的重要省份,2016年白酒产量逾90万kL,销售收入近800亿元,白酒生产企业近450家[13]。作为省域副中心城市的襄阳市亦提出了力争襄阳白酒产业过百亿元,打造鄂酒除稻花香和白云边之外的湖北白酒“第三极”的行业发展目标。但是目前关于襄阳地区浓香型白酒窖泥微生物多样性评价的报道尚少。
本研究采用Miseq高通量测序技术,对4份采集自湖北古襄阳酒业窖泥的细菌多样性进行了解析,同时结合传统微生物学手段对其中蕴含的乳酸菌菌株进行了分离鉴定,通过本项目的实施以期为华中地区浓香型白酒窖泥微生物的多样性研究提供参考。
1 材料与方法
1.1 材料与试剂
窖泥:采集自湖北古襄阳酒业窖泥车间;E.Z.N.A.RSoil DNA Kit试剂盒:美国OMEGA公司;10×聚合酶链式反应(polymerase chain reaction,PCR)缓冲液和DNA聚合酶:宝生物工程(大连)有限公司;FastPfuFly脱氧核糖核酸(deoxyribonucleic acid,DNA) 聚合酶、5×Trans StartTMFastPfu Buffer和三磷酸脱氧核糖核苷酸(deoxyribonucleoside triphosphate,dNTPs)Mix:北京全式金生物技术有限公司;MRS琼脂培养基:北京陆桥技术股份有限公司;十六烷基三甲基溴化铵、异戊醇、乙醇、氯仿、乙二胺四乙酸二钠、碳酸钙、三羟甲基氨基甲烷、饱和酚、十二烷基硫酸钠和醋酸钠:国药集团化学试剂有限公司;引物338F/806R(其中正向引物前端加入7个核苷酸标签)和引物27F/1495R:由武汉天一辉远生物科技有限公司合成。
1.2 仪器与设备
DYY-12电泳仪:北京六一仪器厂;5810R台式高速冷冻离心机:德国Eppendorf公司;vetiri梯度基因扩增仪:美国AB公司;ND-2000C微量紫外分光光度计:美国Nano Drop公司;FluorChem FC3化学发光凝胶成像系统:美国Fluor Chem公司;Miseq高通量测序平台:美国Illumina公司;DG250厌氧工作站:英国DWS公司;ECLIPSE Ci生物显微镜:日本Nikon公司;R920机架式服务器:美国DELL公司。
1.3 实验方法
1.3.1 样品采集及DNA提取
从湖北古襄阳酒业有限公司窖泥车间的4个窖龄为2年的窖池中分上中下3个部位进行样品采集,4个窖池同时建造且深度均为2.2 m,取样时从每个窖壁的上层(距地面20 cm)、中层(距地面110 cm)和窖底3个位置各取100 g窖泥。将同一窖池来源的窖泥混合均匀后装入无菌采样袋中低温运送回实验室,编号分别为GXY1、GXY2、GXY3和GXY4。采用E.Z.N.A.RSoilDNAKit试剂盒对窖泥中微生物宏基因组DNA进行提取。
1.3.2 细菌16S rDNA序列V4-V5区扩增及高通量测序
扩增体系为:DNA模板10 ng,5 μmol/L正向和反向引物各0.8 μL,5 U/μL DNA聚合酶0.4 μL,2.5 mmol/L dNTPs mix2μL,10×PCR缓冲液4μL,体系用ddH2O补充至20μL。扩增条件为:95℃、3 min,95 ℃、30 s,55 ℃、30 s,72、℃ 45 s,35个循环,72℃、10 min。PCR产物检测合格后,上海美吉生物医药科技有限公司使用Miseq PE300平台进行高通量测序。
1.3.3 序列质控
将双端序列进行拼接后,根据核苷酸标签(barcode)信息将拼接好的序列划分到各样品,同时去除各条序列中的barcode和引物。在拼接过程中序列需满足以下条件[14]:
1)重叠区≥10 bp;
2)最大错配比率≤0.2;
3)7个barcode碱基不存在错配;
4)引物碱基错配数≤2 bp。
1.3.4 生物信息学分析
使用QIIME(v1.7.0)分析平台[15]按照以下流程进行生物信息学分析:
1)使用PyNAST[16]将序列对齐;
2)采用UCLUST算法[17],根据100%和97%的相似性将对齐后的序列进行归并,并建立分类操作单元(operational taxonomic units,OTU);
3)使用ChimeraSlayer软件[18]进行嵌合体检查,去除含有嵌合体的OTU,同时定义存在所有4个窖泥样品中的OTU为核心OTU;
4)从去除嵌合体的OTU中选取代表性序列,使用RDP(Ribosomal Database Project,Release 11.5)[19]和Greengenes(Release 13.8)[20]数据库在门、纲、目、科和属水平明确其分类学地位,同时计算该OTU在各样品中的相对含量。若隶属于某一门、属或OTU的样品在所有样品中的平均相对含量>1.0%,则将其定义为优势门、属或优势OTU,若某一OTU在所有样品中均存在则将其定义为核心OTU。
5)进一步从去除嵌合体的OTU中选取代表性序列,使用FastTree软件[21]绘制系统发育进化树,并对香农指数(Shannon index)和超1指数(Chao1 index)等α多样性指数进行计算。
1.3.5 核酸登录号
Miseq高通量测序数据提交至MG-RAST数据库(http://metagenomics.anl.gov/),ID号为mgp83537。
1.3.6 乳酸菌的分离鉴定
采用10倍梯度将窖泥样品倍比稀释后,分别涂布于含有CaCO3的MRS琼脂培养基平板中,于厌氧条件下(85%N2,5%CO2及10%H2)37℃培养48 h后挑取有透明圈的且形态不同的菌落进行分离纯化,30%甘油-80℃保藏备用。参照武俊瑞等[22]的方法使用十六烷基三甲基溴化铵(hexadecyl trimethyl ammonium bromide,CTAB)法提取疑似乳酸菌分离株的基因组DNA并进行16SrDNA序列扩增,检测合格的PCR产物送南京金斯瑞生物科技有限公司进行序列测定。反馈回的序列在GenBank数据库中进行基本局部相似性比对搜索工具(BLAST)同源性比对分析,继而构建系统发育树,进而明确乳酸菌的系统发育地位。
1.3.7 数据统计学分析
使用Mega5.0软件绘制系统发育树;使用在线软件Venny 2.1(http://bioinfogp.cnb.csic.es/tools/venny/index.html)绘制维恩(Venn)图;其他图均使用Origin 8.6软件绘制。
2 结果与分析
2.1 序列丰富度和多样性分析
本研究采集的4个浓香型窖泥样品共产出140 750条高质量16S rDNA序列,平均每个样品产出35 188条。去除引物和barcode后的序列长度分布图如图1所示。

图1 序列长度分布图Fig.1 Distribution diagram of sequence length
由图1可知,去除引物和barcode后,测序序列长度主要集中在440~459 bp,占到序列总数的84.92%,另有13.98%的序列长度集中在420~439bp。使用PyNAST将序列对齐,共有1466条序列因比对失败而被剔除,因而共有139284条序列进行OTU划分。4个窖泥样品16S rDNA V4-V5区序列测序情况及各分类地位数量如表1所示。
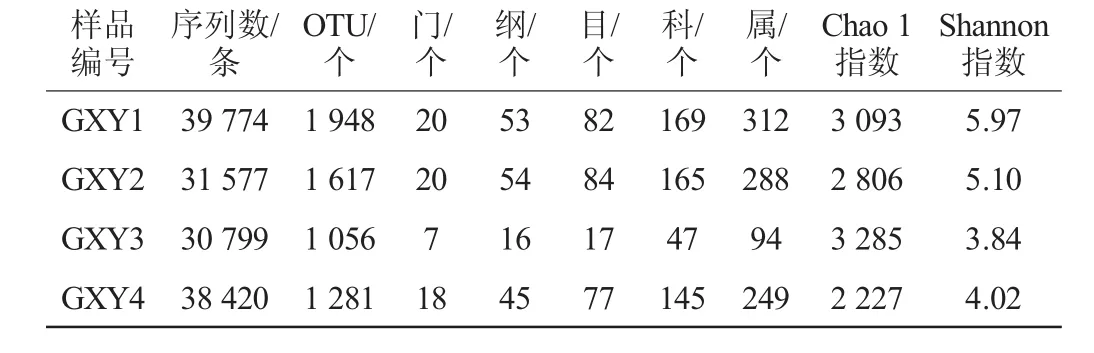
表1 样品16S rDNA测序情况及各分类地位数量Table 1 16S rDNA sequencing and number of taxonomical status ofsamples
由表1可知,根据100%的相似性进行UCLUST时共得到了139284条代表性序列,继而根据97%相似性划分得到了4 772个OTU,采用ChimeraSlayer检测到965个OTU存在嵌合体,去除嵌合体后还剩余3 807个OTU,每个样品平均1 476个。在OTU划分的基础上,本研究将序列鉴定为21个门、59个纲、96个目、199个科和425个属,其中仅有0.24%和6.14%的序列不能鉴定到门和属水平。由表1亦可知,在4个窖泥样品中GXY3的Chao 1指数最高而GXY1的Shannon指数最大,由此可见,GXY3窖池中细菌微生物的丰富度最大而GXY1窖泥细菌多样性最高。
2.2 基于各分类学地位窖泥细菌相对含量的分析
2.2.1 主要细菌在门水平上的相对含量分析
古襄阳浓香型白酒窖泥样品中平均相对含量>1.0%的细菌门如图2所示。
由图2可知,在门水平上,窖泥中的细菌类群主要隶属于硬壁菌门(Firmicutes)、变形菌门(Proteobacteria)、放线菌门(Actinobacteria)和拟杆菌门(Bacteroidetes),其平均相对含量分别为92.02%、2.92%、1.83%和1.28%。

图2 窖泥中优势细菌门相对含量的比较分析Fig.2 Comparative analysis on the content of the dominant bacteria in pit mud samples in phyla level
2.2.2 主要细菌在属水平上的相对含量分析
古襄阳浓香型白酒窖泥样品中平均相对含量>1.0%的细菌属如图3所示。

图3 窖泥中优势细菌属相对含量的比较分析Fig.3 Comparative analysis on the content of the dominant bacteria in pit mud samples in genus level
由图3可知,窖泥中细菌主要隶属于乳酸杆菌属(Lactobacillus)、梭菌属(Clostridium)、芽孢杆菌属(Bacillus)和高温放线菌属(Thermoactinomyces),其平均相对含量分别为77.16%、3.70%、1.64%和1.04%。由此可见,窖泥中细菌类群主要由隶属于硬壁菌门(Firmicutes)的4个属构成,累计相对含量为83.53%。
2.2.3 基于OTU水平的Venn图分析
本研究进一步在OTU水平对窖泥中细菌菌群的构成进行了解析,基于OTU水平的Venn图如图4所示。
由图4可知,通过两步UCLUST划分,本研究共得到3 807个OTU,虽然核心OTU有241个,仅占OTU总数的6.33%,但其包含了118 950条序列,占所有质控后合格序列数的87.25%。此外,在4个样品中分别出现3次、2次和1次的OTU分别有420个、532个和2 614个,分别占OTU总数的11.03%、13.97%和68.66%,其包含的序列数分别为11839条、2071条和3468条,分别占所有质控后合格序列数的8.68%、1.52%和2.54%。由此可见,虽然有些浓香型窖池较之其他窖池可能含有一些较为独特的细菌种系型,但其平均含量仅为0.001%。

图4 基于OTU水平的Venn图Fig.4 Venn diagram based on OTU level
2.2.4 核心OTU相对含量热图及系统发育树构建
本研究进一步绘制了相对含量>1.0%的核心OTU在各窖泥样品中相对含量的热图,并依据各OUT的相对含量建立了左侧和上方的聚类树,结果如图5所示。

图5 相对含量大于1.0%的核心OTU在各窖泥样品中相对含量的热图Fig.5 Heat map of core OTU in pit mud samples with the relative abundance more than 1.0%
由图5可知,在241个核心OTU中,平均相对含量>1.0%的OTU有8个,其中7个隶属于Lactobacillus,1个隶属于Thermoactinomycetaceae。8个OTU的累计平均相对含量为71.31%,其中均隶属于Lactobacillus的OTU662和OTU792的平均相对含量为35.01%和16.99%。进一步选取上述8个核心OTU中的代表性序列构建了系统发育树,其结果如图6所示。

图6 相对含量大于1.0%OTU的系统发育树Fig.6 Phylogenetic tree of OTU with relative abundance more than 1.0%
由图6可知,OTU792、OTU662、OTU4536、OTU2192、OTU2891、OTU2828和OTU2357中的代表性序列可以与耐酸乳杆菌(Lactobacillusacetotolerans)形成聚类,而OTU4559虽然可被鉴定到Thermoactinomycetaceae,但无法将其与某一种细菌形成聚类。
2.3 窖泥中乳酸菌的分离及鉴定
本研究从4个浓香型白酒窖泥样品中共分离了12株菌株,所有菌株在含有1%碳酸钙的MRS固体培养基中均可形成透明圈,革兰氏染色均为阳性,过氧化氢酶试验均为阴性,初步鉴定该12株菌为乳酸菌。将12株菌进行16S rDNA扩增产物测序后,在NCBI数据库中将序列结果进行了相似性比对,并构建了乳酸菌菌株和参考菌株的系统发育树,如图7所示。

图7 乳酸菌菌株和参考菌株的系统发育树Fig.7 Phylogenetic tree of lactic acid bacteria and reference strains
由图7可知,12株菌与副干酪乳杆菌(Lactobacillus paracasei)NBRC15889和副干酪乳杆菌副干酪亚种(Lactobacillusparacaseisubsp.paracasei)JCM8130形成了一个类群,且所有乳酸菌与对应的参考菌株同源性均达到了99%以上。由此可见,本研究分离出的乳酸菌均隶属于L.paracasei,但仅采用16S rDNA序列同源性比对无法明确其“亚种”分类学地位。
3 结论
本研究采用Miseq高通量测序技术,对4份采集自湖北古襄阳酒业窖泥的细菌多样性进行了解析,结果显示窖泥中细菌主要由隶属于Firmicutes的Lactobacillus、Clostridium、Bacillus和Thermoactinomyces构成,经分离鉴定发现其中的乳酸菌均为副干酪乳杆菌(Lactobacillus paracasei)。

