海岛棉H268PPR蛋白家族基因鉴定及SNP/InDel分析
李枝玲 孔祥军 郑杰 韦美玲 李斌 周瑞阳

摘要:【目的】通過对海岛棉不育系H276A及其育性恢复材料H268进行三角状五肽重复蛋白(PPR)家族基因鉴定及比对分析,获得序列存在差异的PPR基因,为后续育性恢复基因(Rf)的发掘提供理论基础。【方法】通过生物信息学分析海岛棉PPR基因,并对其进行亚细胞定位预测与功能注释分析;采用Illumina HiSeq 4000高通量测序技术对海岛棉H276A及H268对PPR蛋白家族基因进行转录组测序,将筛选获得的PPR蛋白与其他植物具有育性恢复的PPR蛋白进行氨基酸序列同源比对并构建系统发育进化树。【结果】鉴定获得912个PPR基因,其中有846个在海岛棉H276A和H268间存在序列差异,且分别存在7226个SNP差异位点和301个InDel差异位点,其中作用于线粒体的PPR基因有2143个SNP差异位点和134个InDel差异位点。转录组测序结果显示,共检测到表达基因数量为68197个,其中已知基因56311个、新基因11886个,可满足后续数据分析。GO功能注释结果显示,PPR基因主要富集于生物学过程,富集于分子功能的PPR基因最少。50.0% PPR蛋白定位于线粒体,29.0% PPR蛋白定位于叶绿体,0.9% PPR蛋白定位于细胞核,20.0% PPR蛋白定位于胞外,0.1% PPR蛋白定位于细胞质膜。xp_016708712(LOC107923023)和xp_016710013(LOC107924193)与多个具有育性恢复功能的PPR蛋白氨基酸序列相似性达33%,说明二者亲缘关系较近,且亚细胞定位于线粒体。【结论】xp_016708712和xp_016710013 2个PPR蛋白可能与海岛棉不育系H276A育性恢复相关,可用于发掘海岛棉CMS的Rf基因。
关键词: 海岛棉;PPR蛋白家族基因;转录组测序;生物信息学分析;SNP;InDel
中图分类号: S562 文献标志码: A 文章编号:2095-1191(2020)11-2614-12
Identification and SNP/InDel analysis of H268 PPR protein family genes in Gossypium barbadense
LI Zhi-ling, KONG Xiang-jun, ZHENG Jie, WEI Mei-ling, LI Bin, ZHOU Rui-yang*
(College of Agriculture,Guangxi University, Nanning 530004, China)
Abstract:【Objective】Through the identification of pentatricopeptide repeat protein PPR protein family genes and comparative analysis of gene sequence between male sterile line H276A and fertility restoration material H268 of Gossy-pium barbadense,the PPR protein coding genes with sequence difference was obtained. It provided a theoretical reference for the further mining of fertility restoration(Rf) genes. 【Method】The PPR genes of G. barbadense were identified by bioinformatics,and subcellular location prediction and gene function annotationanalysis were performed,then further high-throughput sequencing technology was used to identify the PPR protein family genes in G. barbadense H276A and H268 . Homologous alignment in amino acid sequencewas performed between the identified PPR protein and PPR protein with fertility restore genes,then the phylogenetic tree was constructed. 【Result】912 PPR protein coding genes were identified,846 among them were different in sequence between H276A and H268,7226 SNP differential loci and 301 InDel differential loci were obtained in total. Among them, PPR genes acting on mitochondria contained 2143 SNP differential loci and 134 InDels differential loci. Transcriptome sequencing results showed that a total of 68197 genes were detected,including 56311 known genes and 11886 new genes,which could meet the needs of subsequent data analysis. The results of GO functional classification showed that PPR genes were mainly enriched in biological processes and the least in molecular functions. 50.0% of PPR protein was located in mitochondria,29.0% in chloroplast,0.9% in nucleus,and the remaining 20.1% in plasma membrane and extracellular structure, 0.1 in cellular branch membrane.The results of homology alignment showed thatboth of the homology between xp_016708712(LOC107923023),xp_016710013(LOC107924193) and several identified PPR protein amino acids with fertility restoration function were more than 33%,which indicated that they were closely related ,and both of them were located in mitochondria. 【Conclusion】The PPR protein xp_016708712 and xp_016710013 maybe related to the fertility restoration of the male sterile line H276A. It can be used to explore the Rf gene of CMS in G. barbadense.
Key words: Gossypium barbadense; PPR protein family; transcriptome sequencing; bioinformatics analysis; SNP; InDel
Foundation item: National Natural Science Foundation of China(31571719)
0 引言
【研究意义】细胞质雄性不育(Cytoplasmic male sterility,CMS)是广泛存在于高等植物中的一种自然现象,表现为母体遗传、花粉败育和雌蕊正常,其育性可被大多数育性恢复基因(Rf)恢复(Liu et al.,2011),且这些基因编码三角状五肽重复蛋白(Pentatricopeptide repeat,PPR),是三系配套进行杂交育种的生物学基础。利用CMS系进行杂交制种,无需人工去雄,不仅节省人力物力,还可提高杂交种子的纯度(刘加周,2008)。迄今为止,已从玉米(Cui et al.,1996)、高粱(Klein et al.,2001;Jordan et al.,2010)、矮牵牛(Bentolila et al.,2002)、水稻(Akagi et al.,2004;Huang et al.,2012)、萝卜(Desloire et al. 2003;Wang et al.,2008;Yasumoto et al.,2009)和辣椒(Yeong et al.,2010)等植物中克隆獲得Rf基因。棉花是我国重要的经济作物,具有显著的杂种优势,但棉花Rf基因尚有待发掘。因此,研究海岛棉育性恢复材料H268的PPR蛋白基因,对棉花优良恢复系选育、杂种优势利用及Rf基因筛选均具有重要意义。【前人研究进展】目前,在已鉴定的Rf基因中,除玉米Rf2基因及水稻Rf2和Rf17基因(Fujii and Toriyama,2009;Itabashi et al.,2011)外,其他已知的Rf基因均编码PPR蛋白。PPR蛋白家族广泛存在于植物中,且大多数定位于线粒体和叶绿体,以35个简并氨基酸为重复单位串连排列而成,对细胞质基因表达具有重要调控作用(Small et al.,2004;丁安明,2014)。与基因组相比,转录组具有时间和空间的限定,在不同的生长时期及环境下,同一细胞的基因表达情况存在差异(王莎,2013)。基于基因表达差异,利用Illumina高通量测序平台的转录组测序技术能在单核苷酸水平下对任意物种的整体转录活动进行检测,在分析转录本结构和表达水平的同时,还能发现未知转录本和稀有转录本,精确识别可变剪切位点及编码序列单核苷酸多态性(cSNP),提供更全面的转录组信息(刘楚霄等,2016)。与传统的芯片杂交平台相比,转录组测序无需预先针对已知序列设计探针,即可对任意物种的整体转录活动进行检测,可提供更精确的数字化信号、更高的检测通量及更广泛的检测范围,是目前深入研究转录组复杂性的强大工具(Trapnell et al.,2010)。单核苷酸多态性(SNP)是指基因组中单个碱基变异所引起的遗传多态性,其变异类型主要包括转换、颠换、缺失和插入。单碱基突变可导致蛋白发生变异,从而影响生命体有关性状的生物学功能(唐立群等,2012)。插入缺失标记(InDel)通常指两亲本全基因组中的差异,相对另一亲本而言,其中一个亲本的基因组中存在一定数量的核苷酸插入或缺失(王莎,2013)。InDel广泛分布于基因组中,适用于全基因组关联分析、高密度遗传图谱的构建及图位克隆等研究。随着不同棉花种质资源数据库的增加,可通过比较基因组学分析挖掘种间SNP/InDels变异情况(徐相波等,2006)。【本研究切入点】PPR蛋白主要定位于高等植物的线粒体中,通过对不育基因转录本进行剪接修饰,进而调控不育基因的表达,致使育性得以恢复。由于基因编码区内的SNP/InDels可能会导致编码氨基酸的种类发生变化,从而影响其功能。但目前鲜见有关海岛棉H268 PPR基因家族鉴定及SNP/InDel分析的研究报道。【拟解决的关键问题】采用生物信息学软件对海岛棉基因组进行PPR蛋白家族基因鉴定分析,并运用高通量转录组测序技术对海岛棉不育系H276A和育性恢复材料H268进行转录组测序,筛选出差异表达的PPR基因和作用于线粒体的PPR基因,为后续育性的鉴别提供候选基因。
1 材料与方法
1. 1 试验材料
海岛棉CMS系H276A是本课题成员周瑞阳教授于2014年通过花粉管通道法创制的新型不育材料(刘冬梅,2014);海岛棉H268是在以不育系H276A为母本进行测交过程中发现的育性恢复材料(陈平,2018)。以上2种材料均种植于广西大学农学院棉花种植基地,于开花期选取直径3~5 mm的花蕾剥离花药,液氮处理后于-80 ℃保存,用于转录组测序。
1. 2 PPR蛋白家族基因鉴定
下载海岛棉的基因组(https://cottonfgd.org/)构建本地数据库。使用HMMER 3.0和PPR蛋白家族的保守结构域Pfam(PF01535)的隐马尔可夫模型(HMM)对海岛棉基因组功能注释的蛋白序列进行对比检索,获得海岛棉预测候选PPR蛋白序列。此外,利用HMMER 3.0中的hmmbuild构建海岛棉PPR蛋白的隐马尔可夫模型,并搜索海岛棉蛋白数据库(E<1e-5)搜索鉴定出具有海岛棉PPR蛋白结构域的蛋白。同时,利用在线工具CDD、InterPro和PFAM数据库对获得的PPR蛋白进行鉴定,筛选出具有PPR结构域序列的PPR蛋白用于后续分析。
1. 3 转录组测序
采用CTAB法提取海岛棉H268花蕾的总RNA。使用Agilent 2100型核酸生物分析仪、NanoDrop核酸分析仪和1%琼脂糖凝胶电泳检测其浓度及完整性。取3 μg总RNA样品,适温变性打开其二级结构,用携带Oligo(dT)的磁珠富集mRNA,将mRNA打断成小片段,然后加入cDNA第一链合成剂反转录合成cDNA第一链,再配制双链合成反应体系合成双链cDNA。修复双链cDNA末端,并在3'末端加上A碱基,使接头与cDNA连接。最后使用Agilent 2100 Bioanalyzer检测文库的插入片段范围,并以Illumina HiSeq 4000测序平台进行高通量测序。
1. 4 生物信息学分析
利用SoftBerry(http://linux1.softberry.com/)对鉴定得到的PPR蛋白进行亚细胞定位预测,利用ProtParam(https://web.expasy.org/protparam/)预测海岛棉PPR蛋白的理化性質。在Gene Ontology(GO)数据库中对鉴定获得的海岛棉PPR蛋白进行功能注释。根据基因组注释信息,利用EditPlus鉴定筛选出作用于线粒体的PPR基因。结合转录组测序获得的SNP/InDel注释信息对作用于线粒体的PPR基因进行SNP/InDel分析,筛选出含SNP/InDel差异位点的基因。
1. 5 SNP位点克隆验证
根据候选SNP位点,通过NCBI数据库查到该位点前后各100 bp碱基序列作为模版,利用Premier 5.0设计特异性引物(表1)。为鉴定SNP位点的准确性,从作用于线粒体且有SNP差异的基因中随机挑选10个基因进行克隆,胶回收纯化后连接至pEASY?-Blunt Cloning Vector载体上,将连接产物转化Trans1-T1感受态细胞,菌液涂布于含有氨苄青霉素的LB固体培养基上,37 ℃培养12 h,挑取阳性菌落。每基因至少取5个阳性克隆送至深圳华大基因股份有限公司测序。用DNAMAN 6.0将供试材料(H276A和H268)的基因测序结果与基因组编码区序列(CDS)进行多重比对,通过序列比对找出SNP位点。
1. 6 系统发育进化分析
基于Poisson校正模型,利用极大似然法推导出海岛棉PPR蛋白家族基因的演化历史。利用邻接法(Neighbor-joining)和BioNJ算法构建系统发育进化树:基于JTT模型估算的成对距离矩阵,选择具有较高对数似然值的拓扑,自动得到启发式搜索的初始树,并按比例绘制,分支长度以每个站点的替换数量来度量,分析涉及47个氨基酸序列,所有站点覆盖率低于95%的位置均被淘汰。使用MEGA 7.0对相似性较高的10个氨基酸序列与其他植物中具有育性恢复功能的PPR蛋白氨基酸序列构建系统发育进化树(Kumar et al.,2016)。
2 结果与分析
2. 1 PPR蛋白家族基因鉴定及理化性质分析结果
使用隐马尔可夫模型搜索含PPR蛋白家族保守结构域的蛋白,并结合生物信息学分析鉴定出PPR基因,共获得1269个PPR基因,剔除重复序列后最终获得912个PPR基因(表2)。理化性质预测结果显示,PPR蛋白的氨基酸残基数为77~1476个,分子量为8.66~16.69 kD,理论等电点(pI)为4.68~10.08,总平均亲水性为-0.784~0.411,不稳定系数为17.02~60.28,脂肪系数为65.92~115.89。由图1可知,68.0% PPR蛋白的pI属于碱性范围,富含碱性氨基酸;63.0% PPR蛋白为亲水蛋白,以亲水性氨基酸居多;70.0% PPR蛋白的稳定性较好;94.0% PPR蛋白为脂溶性蛋白,以脂溶性氨基酸居多,具有较好的脂溶性(图1)。
2. 2 海岛棉H268转录组测序结果
采用Illumina HiSeq 4000测序平台对海岛棉H268的2个不同单株进行转录组测序,结果(表3)显示平均每单株样品获得8.38 Gb的转录组数据,原始数据(Raw reads)均为61530000条,经过滤处理后分别获得55771950和55958762条高质量序列(Clean reads)。再以Q20值(≥95.00%)作为测序结果评价标准,结果显示,2个单株样品的Q20值均大于98.00%,说明测序结果可靠。将Clean reads比对至陆地棉基因组,发现2个单株样品的总比对率分别为86.72%和86.71%,比对到参考序列唯一位置的比对率分别为63.79%和64.67%,说明样品间比对率无明显差异,表明样品间的数据具有可比性。此外,测序结果还显示,共检测到表达基因数量为68197个,其中已知基因56311个、新基因11886个,可满足后续的数据分析。
2. 3 PPR基因家族的生物信息学分析结果
GO功能注释结果(图2)显示,PPR基因主要富集于生物学过程,其次是细胞组分,以富集于分子功能的PPR基因最少。在生物学过程方面,富集于生物过程的PPR基因最多,其次是细胞过程、代谢过程和含核碱化合物代谢过程,此外,部分PPR基因富集于胚胎发育、授粉及非生物刺激反应等;在细胞组分方面,富集于质体的PPR基因最多,其次是细胞、细胞质、线粒体和细胞成分,还有少部分PPR基因富集于细胞膜,可能PPR蛋白与信号转导、胁迫响应有关;在分子功能方面,富集于RNA结合和核酸结合的PPR基因最多,富集于结合、结构分子活性和结合活性的PPR基因相对较少。海岛棉PPR蛋白家族的亚细胞定位预测结果表明,50.0% PPR蛋白定位于线粒体,29.0% PPR蛋白定位于叶绿体,0.9% PPR蛋白定位于细胞核,20.0% PPR蛋白定位于胞外,剩余的0.1% PPR蛋白定位于细胞质膜(图3)。
2. 4 PPR基因SNP/InDel分析结果
将海岛棉H268转录组数据与H276A和H276B的转录组数据(孔祥军,2017)进行比对,获得含SNP和InDel差异位点的基因,分别为835567和120995个,通过与鉴定获得的912个PPR基因比对筛选出含SNP差异位点的PPR基因845个,含InDel差异位点的PPR基因220个(表4)。结合基因组注释信息,利用EditPlus进行比对鉴定,共获得作用于线粒体的PPR蛋白基因484个。结合转录组注释信息筛选具有SNP和InDel差异且作用于线粒体的PPR基因,其中含SNP差异位点的基因445个,含InDel差异位点的基因104个(表4)。
2. 5 SNP克隆验证结果
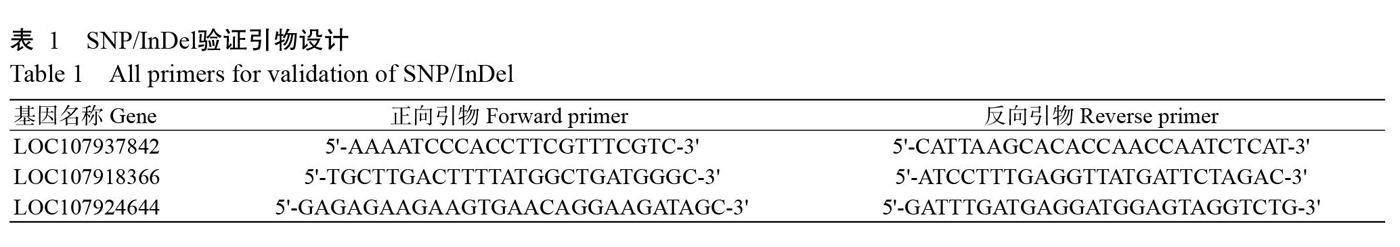
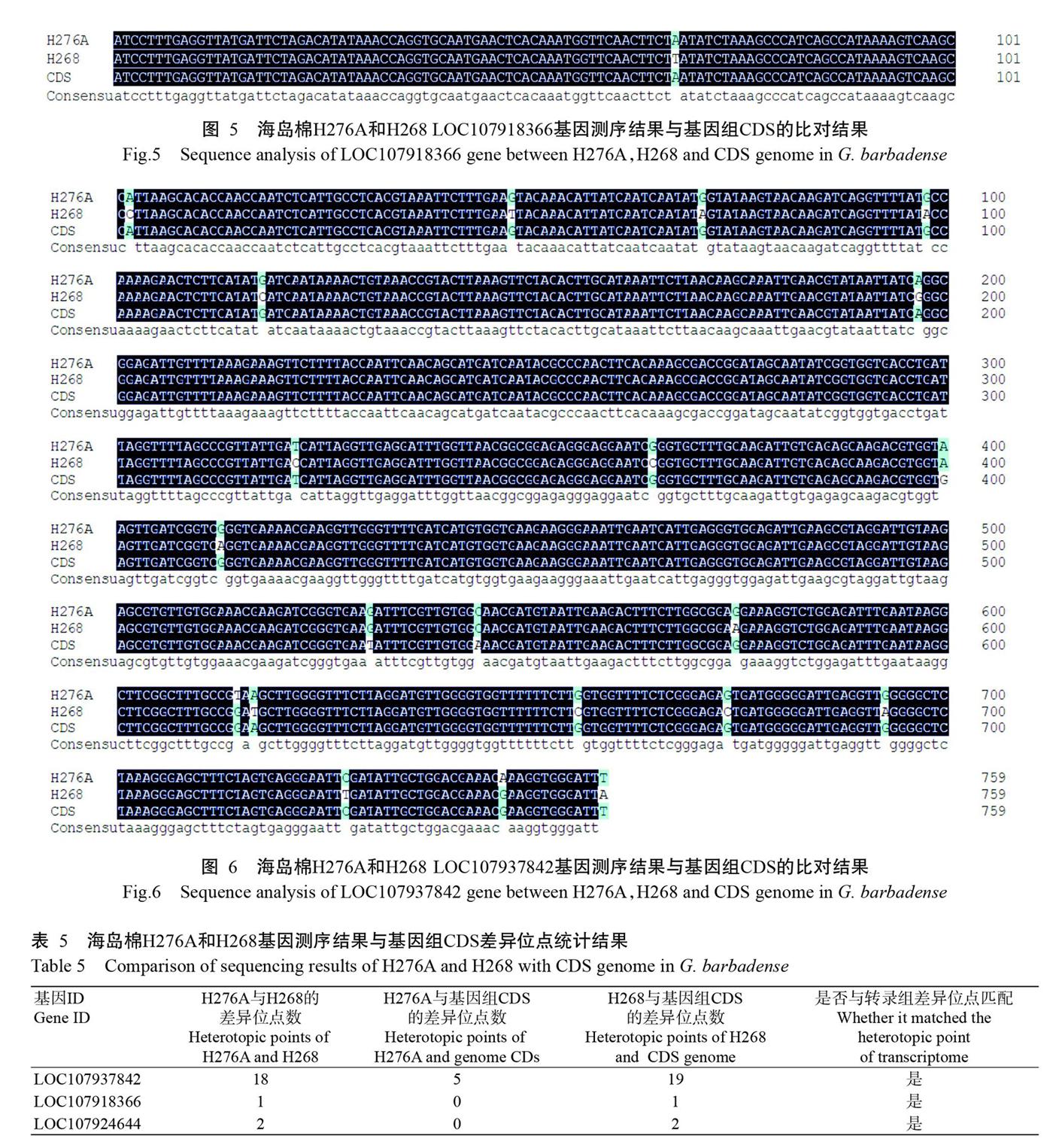
剔除假阳性克隆后获得3组可靠的测序结果,将基因测序结果与基因组CDS进行比对,结果(表5)表明,在海岛棉H276A与H268的基因测序结果比对中,LOC107924644和LOC107918366基因分别含有2和1个SNP差异位点,以LOC107937842基因SNP差异位点数最多,为18个(图4);在海岛棉H276A的基因测序结果与基因组CDS比对中,LOC107918366和LOC107924644基因不存在SNP差异位点,LOC- 107937842基因有5个SNP差异位点(图5);在海岛棉H268的基因测序结果与基因组CDS比对中,LOC107924644、LOC107918366和LOC107937842基因均存在SNP差异位点,其中,LOC107937842基因SNP差异位点最多,为19个(图6)。经检验所有差异位点与转录组测序所得位点匹配,进一步验证测序结果的可靠性。
2. 6 海岛棉PPR基因家族育性恢复基因候选分析
在NCBI和EMBL网站搜索植物中已鉴定的具有育性恢复功能的PPR蛋白氨基酸序列,然后与鉴定获得的PPR蛋白氨基酸序列构建系统发育进化树(图7),结果发现,同一物种间进化关系较近,不同物种间分化程度较高,说明PPR基因家族在不同物种间存在一定差异;xp_016708712和xp_016710013与多个具有育性恢复功能的氨基酸序列亲缘关系较近,且亚细胞定位预测均定位于线粒体,推测二者具有一定的育性恢复作用。通过DNAMAN 6.0进行比对,结果发现在鉴定获得的PPR蛋白氨基酸序列中,与其他植物中具有育性恢复功能的PPR蛋白氨基酸序列相似性25%以上的共有10个,其中,xp_016708712(LOC107923023)与xp_016710013(LOC107924193)与多个具有育性恢复功能的PPR蛋白氨基酸序列相似性达33%。
3 讨论
3. 1 PPR基因家族鉴定
随着不同物种基因组测序的完成,PPR基因家族成员鉴定也取得长足进展。目前,除了模式植物烟草(丁安明,2014)、拟南芥(Liu et al.,2016b)和水稻(Chen et al.,2018)外,番茄(丁安明等,2014)、谷子(Liu et al.,2016a)、甘蓝型油菜(谭晖和官春云,2017)、小麦(郭彩娟等,2019)等物种的PPR基因家族也被鉴定,且研究发现PPR基因能参与线粒体和叶绿体基因转录后加工、调控雄性不育相关基因表达、参与胚胎形成和逆境防御等(丁安明等,2013)。本研究对海岛棉PPR基因家族进行鉴定,剔除重复序列后共获得912个PPR基因,成员数量较多,与丁安明(2014)、郭彩娟等(2019)研究结果相似,表明PPR基因家族成员众多。
Lahmy等(2000)通过荧光蛋白定位试验和Wes-tern boltting证实拟南芥P67定位于叶绿体。Lurin等(2004)通过软件分析和荧光蛋白定位均證实大部分拟南芥PPR蛋白定位于线粒体或叶绿体。Schmitz-Linneweber等(2006)利用RNA免疫沉淀技术证实玉米PPR4蛋白定位于叶绿体。丁安明等(2014)研究发现,60% PPR蛋白定位于线粒体和叶绿体。本研究亚细胞定位预测结果显示,79.0%PPR蛋白定位于线粒体和叶绿体,与上述前人研究结果相似,故推测PPR基因参与线粒体和叶绿体基因转录后加工。此外,研究发现PPR蛋白具有RNA结合活性,推测其参与RNA加工,可与靶RNA的特异位点直接发生相互作用(丁安明等,2014);拟南芥P67与玉米CRP1均具有RNA结合活性(Lahmy et al.,2000);水稻OsPPR1参与叶绿体RNA加工及质体早期生物合成(Gothandam et al.,2005)。本研究的GO功能注释结果显示,较多PPR蛋白具有核酸结合活性和蛋白结合活性,还参与核碱化合物代谢过程、生物进程、代谢和细胞过程等,且部分PPR蛋白还参与胚胎发育、授粉及非生物刺激反应等生物过程;在细胞组分方面,富集于质体的PPR基因最多,还有少部分PPR基因富集于细胞膜,可能PPR蛋白与信号转导、胁迫响应有关,与丁安明等(2014)对绒毛状烟草PPR蛋白的GO注释结果基本一致。
3. 2 SNP/InDel分析
在功能基因的图位克隆中常需高密度的分子标记,但目前公布的各种分子标记密度均未达到令人满意的程度。InDel和SNP分子标记是新型的分子标记类型,基本可满足精细定位目的基因的需求。InDel和SNP分子标记经济实用、特异性高、稳定性好(曹立超等,2014)。本研究通过转录组测序获得作用于线粒体的PPR基因中分别有2143个SNP差异位点和134个InDel差异位点,为后续海岛棉CMS育性恢复基因的发掘提供了重要依据。
3. 3 作用于线粒体的PPR蛋白及育性恢复
PPR基因家族作为一类重要的恢复基因,在植物的生长发育和细胞器基因的表达调控等多种生物学过程中发挥至关重要的作用(Zhao et al.,2018)。已有研究表明,雄性植株含有Rf基因,能拮抗细胞质线粒体中的CMS诱导基因(CMS-inducing gene)。在胞质雄性不育系统中,CMS诱导基因能表达产生疏水的CMS特异性多肽(CMS-specific pelypeptide),Rf基因编码的PPR蛋白能调控CMS诱导基因的表达水平,通过降低或抑制不育相关基因的转录,降低有毒蛋白的水平,从而促使CMS植株恢复育性(Bentolila et al.,2002;刘天骄等,2017)。含有异常开放阅读框(ORF)的线粒体RNA会导致雄性不育,Rf基因所表达的PPR蛋白能与这些线粒体RNA相结合而抑制其表达(Kazama et al.,2008;Uyttewaal et al.,2008)。含16个PPR基序的萝卜恢复基因Rfo(Rfk1),虽对线粒体基因ORF125转录本的含量无影响,但影响其蛋白翻译或翻译后的加工过程(Desloire et al.,2003;Kazama and Toriyama,2003);含有14个PPR基序的矮牵牛育性恢复基因Rf-PPR592可能通过影响线粒体基因PCF转录本的表达量,而在一定程度上减少PCF蛋白含量(Mili and Pind-Roma,2003;Koizuka et al.,2003);水稻BT型恢复基因Rf-1具有16个PPR基序,参与线粒体基因atp6转录本的表达(Alfonso et al.,2004;Komori et al.,2004)。PPR基因可改变与胞质不育相关基因的表达水平,通过阻止线粒体诱导基因编码蛋白的表达以恢复花粉育性(徐相波等,2006)。郭彩娟等(2019)通过与已报道的水稻育性恢复基因进行同源比对,结果获得候选23个PPR基因,并结合结实率等重要农艺性状表型分析初步确定4个PPR基因可能参与调控二系杂交小麦杂交种的育性恢复进程。本研究的同源比对分析结果表明,在鉴定获得的PPR蛋白氨基酸序列中,与植物已鉴定具有育性恢复功能的PPR蛋白氨基酸序列相似性在25%以上的共有10个,其中xp_016708712和xp_016710013与多个具有育性恢复作用的氨基酸序列亲缘关系较近,且亚细胞定位预测均定位于线粒体,故推测二者具有一定的育性恢复作用。
4 结论
xp_016708712和xp_016710013 2个PPR蛋白可能与海岛棉不育系H276A育性恢复相关,可用于发掘海岛棉CMS的Rf基因。
参考文献:
曹立超,樊双虎,张春义,陈茹梅. 2014. 大刍草渐渗系雌穗表型统计及SNP分子标记开发[J]. 种子,33(4):42-47. [Cao L C,Fan S H,Zhang C Y,Chen R M. 2014. Female spike phenotypic statistics of teosint introgression and development of SNP markers[J]. Seed,33(4):42-47.]
陈平. 2018. 陆地棉细胞质雄性不育系J-4A育性恢复基因的遗传与分子标记[D]. 南宁:广西大学. [Chen P. 2018. Genetic and molecular markers of fertility restorer gene of cytoplasmic male sterile line J-4A of upland cotton[D]. Nanning:Guangxi University.]
丁安明,李凌,屈旭,孙亭亭,陈雅琼,宗鹏,李尊强,龚达平,孙玉合. 2014. 番茄 PPR基因家族的鉴定与生物信息学分析[J]. 遗传,36(1):77-84. [Ding A M,Li L,Qu X,Sun T T,Chen Y Q,Zong P,Li Z Q,Gong D P,Sun Y H. 2014. Genome-wide identification and bioinformatic analysis of PPR gene family in tomato[J]. Genetics,36(1):77-84.]
丁安明,屈旭,孙玉合. 2013. 植物PPR蛋白家族研究进展[J]. 中国农学通报,30(9):218-224. [Ding A M,Qu X,Sun Y H. 2013. Research progress of plant PPR protein family[J]. Chinese Agricultural Science Bulletin,30(9):218-224.]
丁安明. 2014. 番茄与烟草PPR基因家族分析及育性相关基因功能研究[D]. 青島:中国农业科学院. [Ding A M. 2014. PPR gene family analysis and fertility related gene function of tomato and tobacco[D]. Qingdao:Chinese Aca-demy of Agricultural Sciences.]
郭彩娟,公杰,刘永杰,赵昌平,高世庆,吴华伟. 2019. 全基因组小麦PPR基因家族鉴定及表达分析[J]. 生物技术通报,35(8):1-8. [Guo C J,Gong J,Liu Y J,Zhao C P,Gao S Q,Wu H W. 2019. Genome-wide bioinformatics identification of PPR gene family and expression profiles ana-lysis in wheat[J]. Biotechnology Bulletin,35(8):1-8.]
孔祥军. 2017. 海岛棉H276A雄性不育的细胞学与分子生物学基础研究[D]. 南宁:广西大学:73-76. [Kong X J. 2017. Studies on cytology and molecular biology sterility line H276A in Gossypium hirsutum[D]. Nanning: Guangxi University:73-76.]
刘楚霄,李响,陈玲玲. 2016. 全转录组水平研究长非编码RNA方法学进展[J]. 生命的化学,36(6):745-752. [Liu C X,Li X,Chen L L. 2016 .Methods for genome-wide characterization of long noncoding RNAs[J]. Chemistry of Life,36(6):745-752.]
刘冬梅. 2014. 棉花转基因雄性不育种质创新及不育相关基因的挖掘[D]. 南宁:广西大学. [Liu D M. 2014. Innovation of transgenic male sterility germplasm and excavation of sterility related genes in cotton[D]. Nanning:Guangxi University.]
刘加周. 2008. 玉米C型细胞质雄性不育系及其保持系花药SSH文库构建[D]. 雅安:四川农业大学. [Liu J Z. 2008. Construction of anther SSH library of C-type cytoplasmic male sterile line and its maintainer[D]. Yaan:Si-chuan Agricultural University.]
刘天骄,程大友,孟祥雯,崔杰,代翠红,刘巧红. 2017. PPR家族与植物育性恢复基因的研究进展[J]. 中国甜菜糖业,(1):20-22. [Liu T J,Cheng D Y,Meng X W,Cui J,Dai C H,Liu Q H. 2017. Research progress of PPR family and plant fertility restoration gene[J]. China Beet & Su-gar,(1):20-22.]

