生菜LsPHYB可变剪接体的克隆与高温诱导表达模式
隋心意,赵小刚,陈鹏宇,李亚灵,温祥珍
生菜可变剪接体的克隆与高温诱导表达模式
隋心意,赵小刚,陈鹏宇,李亚灵,温祥珍
山西农业大学园艺学院,山西晋中 030801
【】光敏色素B(phytochrome,PHYB)是光和温度的受体。通过克隆光敏色素B基因()可变剪接体并分析其在高温诱导下的表达模式,探究可变剪接体在生菜响应环境高温中的生物学功能,为培育耐热性生菜提供理论依据。采用生物信息学方法在生菜的基因组数据库搜索获得的cDNA序列的相关信息;对克隆得到的3个可变剪接体、和进行多序列比对、可变剪接方式分析及系统进化树分析;通过在线软件预测PHYB1、PHYB2和PHYB3蛋白分子量、等电点和亲水性、疏水性等蛋白质理化性质,并通过生物信息学软件预测三者的二级结构、三级结构和保守结构域;采用荧光定量PCR(qRT-PCR)检测、和在高温处理后的表达特征。克隆获得的生菜的3个可变剪接体、和的CDS长度分别为3 509、3 877和2 690 bp,编码氨基酸长度分别为1 094、960和853 aa。其中发生可变3′端位点和外显子跳跃类型可变剪接,发生选择性保留polyA尾和内含子保留型可变剪接,发生外显子跳跃类型可变剪接。保守结构域分析表明PHYB2的N端缺少PAS和PHY功能域;PHYB3的N端缺少PAS和PHY功能域,C端缺少HisKA功能域;系统进化树分析表明,3个可变剪接体聚为一支。qRT-PCR分析表明在高温处理第1天,的表达量最高;在高温处理第5—9天,的表达量高于和;在高温处理第11天,的表达量高于和,处理11 d内三者表达量达到峰值的时间不同。高温下生菜的转录本存在3个可变剪接体和。在高温处理前期高表达,分别在高温处理中期、后期高表达,推测生菜3个可变剪接体在抗高温胁迫中发挥不同的作用。
生菜;;可变剪接体;高温胁迫;响应
0 引言
【研究意义】近年来,全球气候变暖趋势显著,高温成为影响植物生长发育的重要因素[1]。生菜(L.)性喜冷凉,最适宜的生长温度为15—20℃,高温会导致其叶片变薄、节间伸长,直接影响商品价值[2]。前人研究表明[3],PHYB是光和温度的受体。本研究在克隆时发现其在高温下存在可变剪接,探究可变剪接体在响应高温中的功能对生菜的越夏栽培以及热品种的培育具有重要的理论和实践意义。【前人研究进展】可变剪接(alternative splicing,AS)是植物响应高温胁迫的重要转录后调控方式[4-6],直接决定蛋白的结构和功能[7]。可变剪接是指一个前体信使RNA通过不同的剪接方式形成多个成熟mRNA亚型的过程[8],主要有外显子跳跃(skipped exon,SE)、内含子保留(retain intron,RI)、可变5′端位点(alternative 5′ splice site,A5′SS)、可变3′端位点(alternative 3′ splice site,A3′SS)、外显子互斥(mutually exclusive exon,MEE)、可变启动子(alternative promoter)和可变polyA(alternative polyA)7种类型[9]。前人研究表明,多种植物均通过基因的可变剪接来响应高温胁迫[10-13]。番茄()[14]中发现热激蛋白HSPs(heat shock proteins)和热激转录因子HSFs(heat shock transcription factors)在高温下发生RI和SE类型的可变剪接产生短截的蛋白来响应高温。水稻(L.)干旱应答元件结合蛋白()在高温下通过产生新的可变剪接体来应对高温胁迫[15]。甘蓝(L.)在高温胁迫下会产生2种可变剪接体增加对转录调控的复杂性,以此提高甘蓝的耐热性[16]。AIROLDI等[17]研究发现拟南芥()中()在高温下通过增加非功能型的剪接变体来促进拟南芥提前开花,以此来响应环境高温。POSE等[18]研究表明高温下拟南芥()通过增加可变剪接体的表达,促进与SVP(short vegetative phase)相互作用,激活开花启动子()与(),进而促使其花期提前,避开高温的不利影响。YAN等[19]研究发现高温下拟南芥通过可变剪接产生大量microRNA 400初级转录本促进种子萌发和细胞伸长,进而促进植物在高温下的生长发育。综上所述,植物通过可变剪接改变蛋白的功能来增强对高温的适应性。【本研究切入点】前人研究[20-21]发现在拟南芥中光敏色素B(phytochrome B,PHYB)不仅可以整合光信号,还可以整合温度信号,生菜中PHYB是否也作为温度感受器参与生菜对环境高温的感知?此外,已有多个基因的可变剪接被鉴定并证实可以对高温胁迫做出响应,生菜中是否也通过可变剪接响应高温胁迫?【拟解决的关键问题】本研究以热敏性生菜为试验材料,对的可变剪接体进行克隆,鉴定其序列特征及可变剪接情况,分析其基因结构并检测在高温处理过程中的表达特征,比较其蛋白的结构、性质和功能,旨在初步明确可变剪接在高温下发挥的功能,为后续研究生菜耐热分子机制提供理论依据。
1 材料与方法
试验材料生菜样品的采集于2020年10—11月在山西农业大学园艺实验中心进行;基因克隆试验和基因表达试验于2020年12月至2021年4月在山西农业大学园艺实验中心进行。
1.1 试验材料及处理
供试材料为生菜(L.)品种‘GRAND RAPIDS TBR’,购买于Seminis公司。选取颗粒饱满、无病虫害的种子播种于装有基质(草炭﹕蛭石=1﹕1)的6 cm×6 cm的花盆中,于光照培养箱内(光照强度30 000 lx,光照/黑暗时间为16 h/8 h,相对湿度50%)培养。待植株长至6叶1心后,将其分为常温组和高温组进行为期11 d的处理,每组包含9株。其中高温组生长条件为:光照条件下35℃,黑暗条件下25℃,常温组生长条件则分别为25℃和15℃。分别于第1、3、5、7、9和11天对处理1 h后的生菜取样,采集中部叶片混合后作为一个重复,每个处理重复3次。样品采集后用锡纸包裹并立即用液氮速冻,储存于-80℃冰箱备用。
1.2 总RNA提取和cDNA的合成
取适量材料在液氮中研磨,按照RNAprep pure植物总RNA提取试剂盒提取总RNA。采用Prime ScriptTMRT reagent Kit反转录试剂盒将RNA反转录为第一链互补链DNA(cDNA)。反转录的反应条件及程序均按照Prime ScriptTMRT reagent Kit和Prime ScriptTMRT reagent Kit with gDNA Eraser试剂盒说明书进行,以上试剂均购自Takara公司。
1.3 LsPHYB可变剪接体的克隆
从生菜基因组数据库(https://lgr.genomecenter. ucdavis.edu)中搜索得到的序列信息,通过premier6.0设计克隆引物。-F:5′-AACCAACA CCTCAAATCCACC-3′;-R:5′-CCAGACCAG AATCTCATTCCTAT-3′,以上述引物为模板,通过PCR法克隆目的基因。
PCR的反应体系为:2×PfuMasterMix 25.0 µL,灭菌ddH2O 50 µL,模板cDNA 1 µL,上、下游引物各0.5 µL。PCR扩增程序为95℃预变性5 min;94℃变性30 s,50℃退火30 s,72℃延伸30 s,30个循环;最后72℃延伸10 min。按照凝胶回收试剂盒的说明书,进行目的片段的回收,置于-20℃保存备用。将纯化后PCR产物连接到T载体,转入DH5α感受态细胞中,随后进行阳性克隆鉴定。
通过qRT-PCR技术分析在高温处理过程中的表达特征。根据克隆得到的序列,设计qRT-PCR引物。-F:5′-GCATAAGAAAGT GCACAACAGCC-3′和-R:5′-CGACGAACCT GGAAGAATGG-3′,用生菜作为内参基因,引物序列为F:5′-GTGAGTGAAGAAGGGCAATG-3′和R:5′-AGTGAATTGGTTTCGAGAGC-3′,目标基因与生菜一起扩增。根据2-∆∆CT法用Excel软件对获得的数据进行相对表达量分析[22]。
1.4 可变剪接体的生物信息学分析
通过比对3个可变剪接体的序列与NCBI中的CDS序列,得到基因内含子和外显子的剪接与保留情况。使用IBS1.0.3软件对3个可变剪接体的基因结构进行绘制;使用ExPASy- ProtParam(https://web.expasy.org/protparam/)分析蛋白的理化性质和组成成分;采用ExPASy-ProtScale(https://web.expasy.org/protscale/)进行蛋白的亲疏水性分析;利用SMART(http://smart.embl-heidelberg.de/)在线程序预测蛋白的保守结构域;利用SignalP 4.0 Server(http://www.cbs.dtu.dk/services/SignalP-5.0/)对蛋白有无信号肽进行预测;利用TMHMM(http://www. cbs.dtu.dk/services/TMHMM/)对蛋白的跨膜区域进行预测;借助网站SOPMA(https://npsa-prabi.ibcp.fr/cgi- bin/npsa_automat.pl?page=npsa_sopma.html)进行蛋白的二级结构分析;使用SWISS-MODEL(https:// swissmodel.expasy.org)在线预测PHYB蛋白的三级结构;利用MEGA7.0软件构建系统进化树;利用在线软件Cell-PLoc 2.0(http://www.csbio.sjtu.edu.cn/bioinf/ Cell-PLoc-2)和Softberry(http://www.softberry.com)对该蛋白质的亚细胞定位进行预测。
2 结果
2.1 LsPHYB的克隆与可变剪接分析
基于NCBI数据库中生菜基因(Gene ID: 111911925)的mRNA序列信息,序列比对发现克隆得到的3个不同转录本均是的可变剪接体,将其分别命名为、和,根据比对结果绘制各转录本的基因结构图(图1)。NCBI数据库中CDS长度为3 569 bp,由4个外显子和3个内含子构成。与相比,CDS长度为3 509 bp,第3个外显子由于发生A3′SS类型的可变剪接而缺少了一段大小为29 bp的片段,第4个外显子由于发生SE类型的可变剪接而缺失;CDS长度为3 877 bp,第2和第3内含子发生RI类型的可变剪接,此外polyA尾也被保留;3 CDS长度为2 690 bp,第3个外显子和第4个外显子由于发生SE类型的可变剪接而缺失。
2.2 LsPHYB可变剪接体的生物信息学分析
2.2.1 理化性质分析 通过ExPASy-ProtParam分析发现,PHYB1、PHYB2和PHYB3蛋白的氨基酸长度分别为1 094、960和853 aa;理论等电点分别为5.78、5.69和5.72,均属于酸性蛋白质;3个PHYB总平均亲水性(GRAVY)分别为-0.123、-0.139和-0.115,均为负值,表明三者均为亲水性蛋白;不稳定系数分别为46.97、48.24和47.25,均大于40,表明三者均为不稳定性蛋白。PHYB均无典型的信号肽区域,无跨膜螺旋结构域,均不属于分泌蛋白。利用ExPASy- ProtScale预测PHYB的亲水性、疏水性发现,三者的氨基酸疏水值最高峰均在第348位,分值为2.822;亲水值最低峰均在第543位,分值为-3.311(图2)。
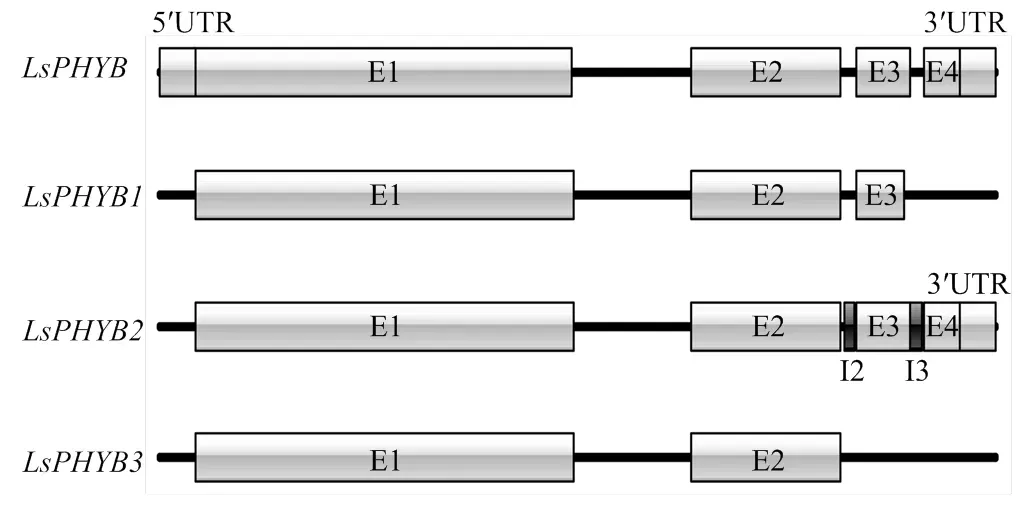
黑色线段表示内含子,浅灰色方框表示外显子;深灰色方框表示可变剪接保留的内含子
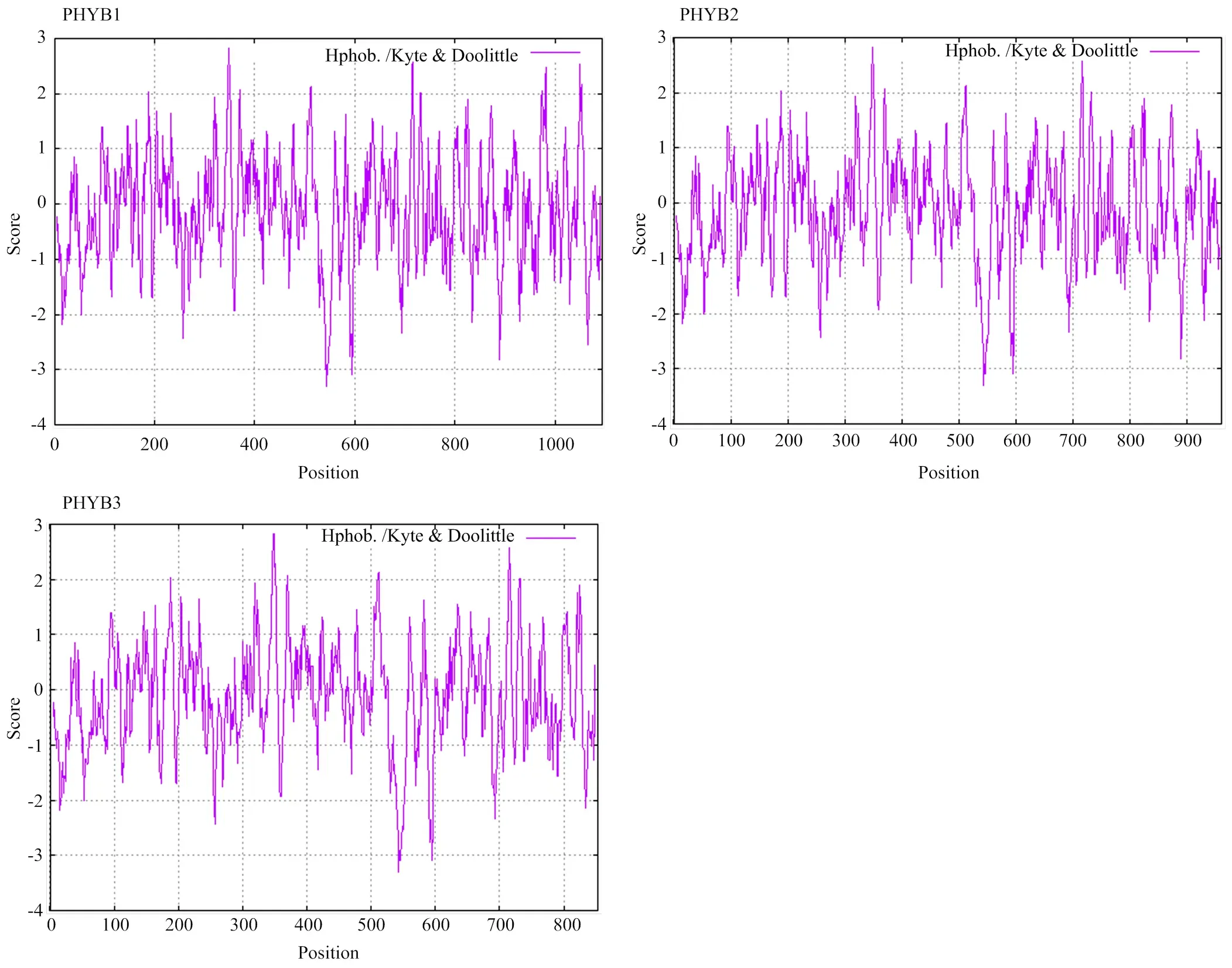
图2 PHYB1、PHYB2和PHYB3蛋白亲水性、疏水性分析
2.2.2 二级和三级结构分析 3种PHYB蛋白序列均由-螺旋(-helix)、延伸链(extended strand)、无规则卷曲(random coil)和-转角(-turn)组成。其中PHYB2蛋白所含-螺旋比例最高,为49.06%;PHYB3蛋白所含无规则卷曲、延伸链和-转角比例均最高,分别为35.76%、13.72%和5.63%(图3)。3个PHYB蛋白的三维结构整体十分相似,但是三者在C端上存在差异。
2.2.3 保守结构域分析 PHYB1蛋白结构完整,即N端具有Per-Arnt-Sim(PAS)、cGMP磷酸二酯酶-腺苷酸环化酶-FhlA(cGMP phosphodiesterase-adenylylcyclase- FhlA,GAF)和PHY特异性(PHY-specific,PHY)结构域,C端具有2个连续的PAS和组氨酸激酶相关(histidine kinase A,HisKA)结构域;与PHYB1蛋白相比,PHYB2蛋白的N端缺少PAS和PHY功能域;PHYB3蛋白不仅N端缺少PAS和PHY功能域,而且C端还缺少HisKA功能域(图4)。
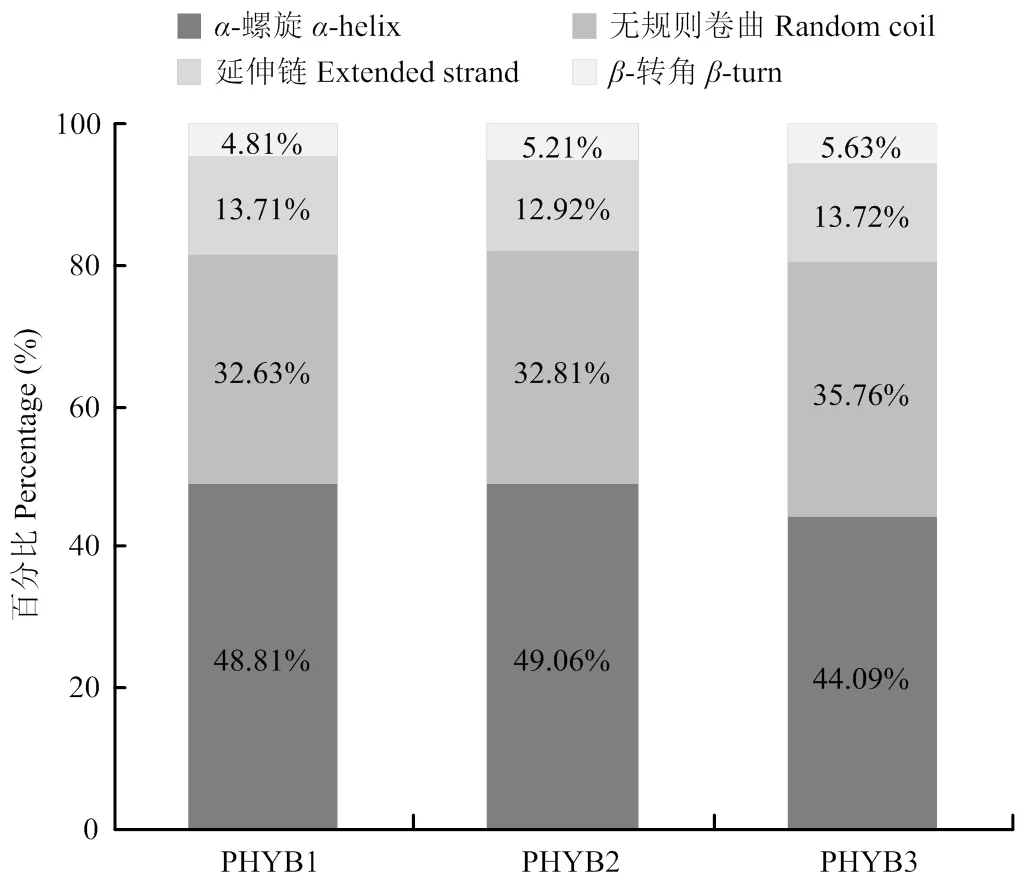
图3 PHYB1、PHYB2和PHYB3蛋白的二级结构特征
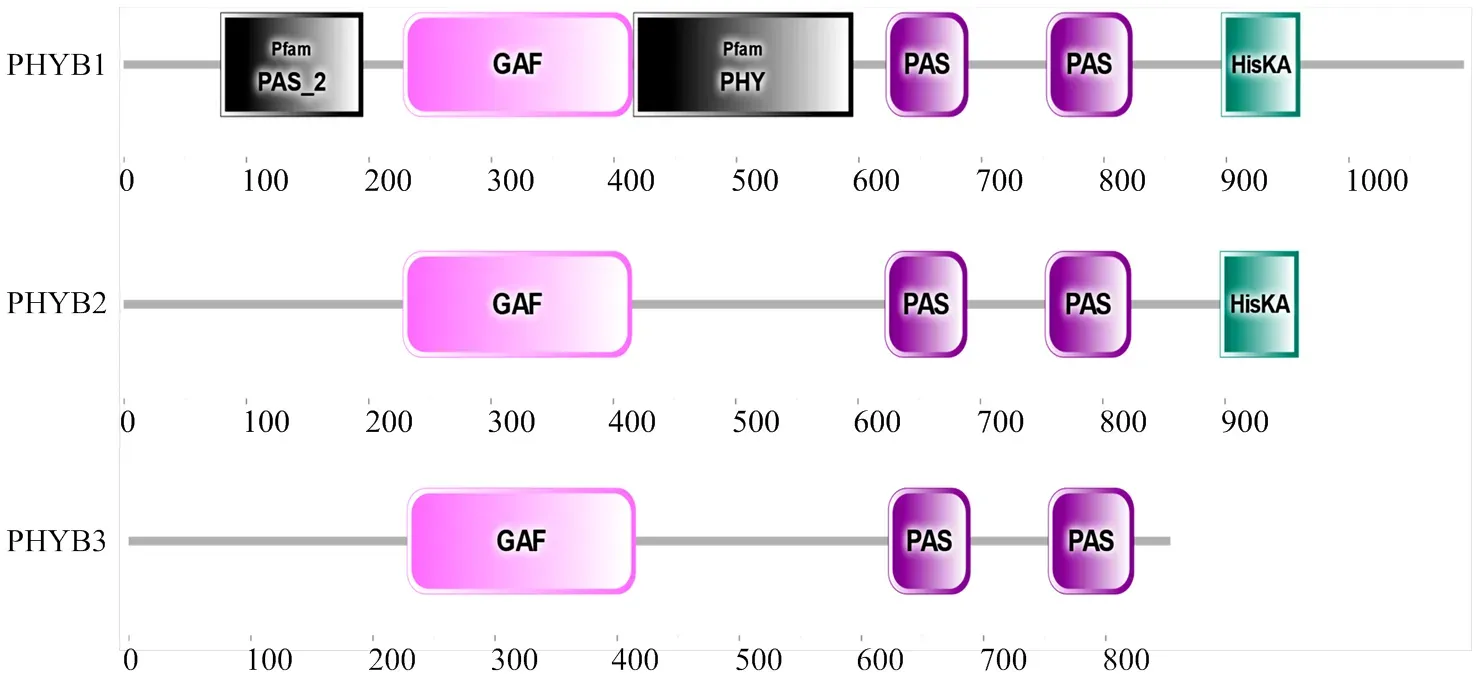
图4 PHYB1、PHYB2和PHYB3蛋白结构域示意图
2.2.4 系统进化分析 利用MEGA7.0将3种PHYB氨基酸序列与向日葵(,NC_ 035433.2)、烟草(,NW_015887446.1)、拟南芥(,NC_003071.7)、甘蓝(L.,NC_027752.1)、芜菁(,NC_024799.2)、番茄(,NC_015442.3)、马铃薯(,NW_ 006239191.1)和菠菜(,NW_018932796.1)进行序列比对并构建系统进化树。结果表明LsPHYB的3个可变剪接体首先聚为一个分支,与同为菊科的向日葵进化距离最近,与十字花科的拟南芥、甘蓝和芜菁进化距离较近;与茄科的烟草、番茄和马铃薯进化距离较远;与藜科的菠菜进化距离最远(图5)。
2.3 LsPHYB可变剪接体在高温处理下的表达特征分析
与对照相比,高温处理下的表达量均高于对照,且在处理后第1、3、5和9天的表达量均显著高于对照,第11天时表达量最高,为常温下表达量的5倍。与对照相比,在处理第5、7和11天的表达量显著高于常温,第5天时表达量最高,为常温下表达量的4.3倍。与对照相比,在第3和11天高温下的表达量均显著低于对照;在第1、5、7和9天均高于对照,且在第1、5和9天差异显著;第1天时表达量最高,为常温下表达量的1.75倍。综上,处理11 d内,三者表达量达到最大峰值的时间不同,高温处理第1天,的表达量最高;高温处理第5—9天,的表达量高于和;高温处理第11天,的表达量高于和(图6)。
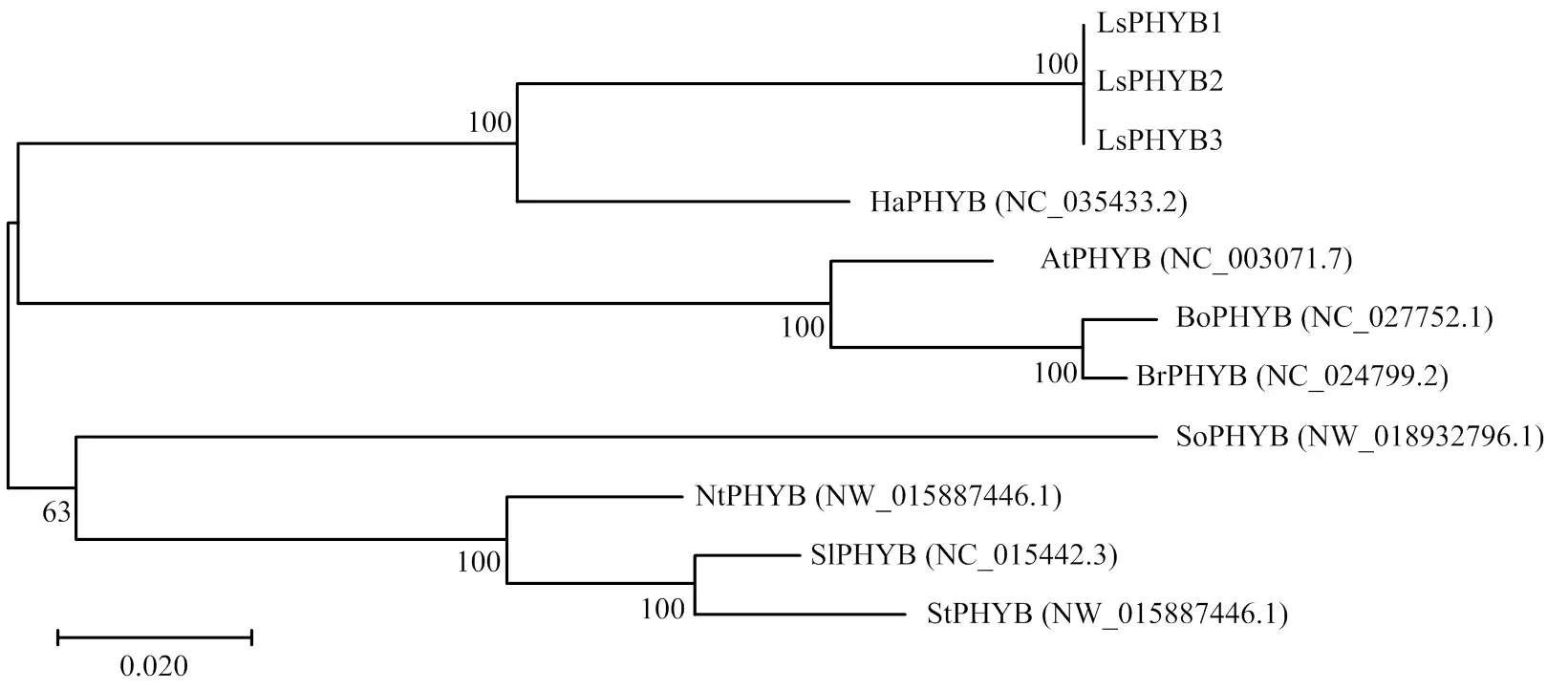
Ha:向日葵Helianthus annuus,NC_035433.2;Nt:烟草Nicotiana tabacum,NW_015887446.1;At:拟南芥Arabidopsis thaliana,NC_003071.7;Bo:甘蓝Brassica oleracea,NC_027752.1;Br:芜菁Brassica rapa,NC_024799.2;Sl:番茄Solanum lycopersicum,NC_015442.3;St:马铃薯Solanum tuberosum,NW_006239191.1;So:菠菜Spinacia oleracea,NW_018932796.1
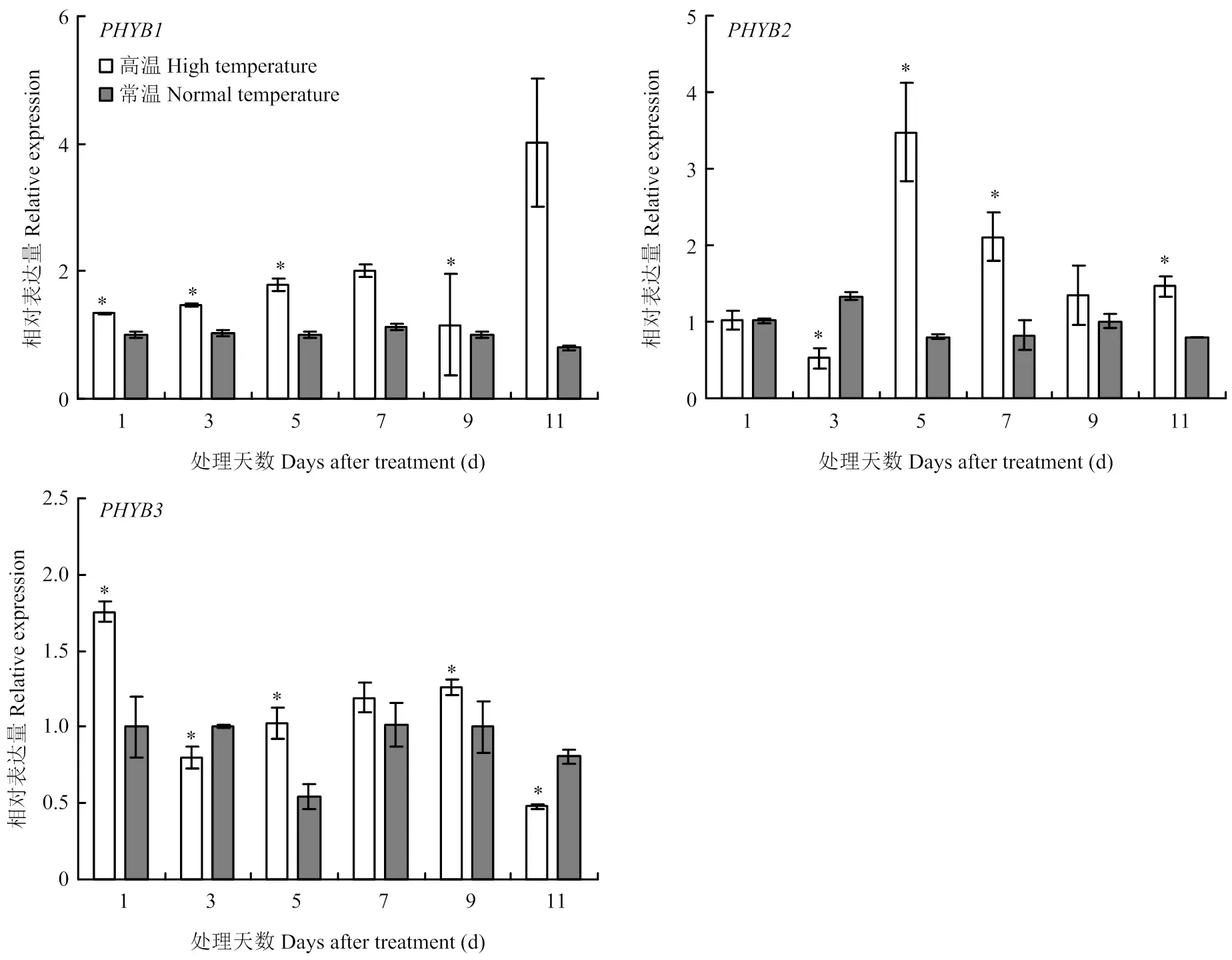
*表示差异显著(P<0.05) * indicate significant difference (P<0.05)
3 讨论
3.1 LsPHYB可变剪接体序列分析
可变剪接可能会导致蛋白的结构和功能发生变化,例如,A3′SS和RI可导致转录本插入提前终止密码子(premature termination codon,PTC),具有PTC的转录本可被翻译成短截的蛋白质[23];SE类型可变剪接会因缺少必须的结构域而形成非功能性蛋白[24]。因此,可变剪接会改变PHYB1、PHYB2和PHYB3蛋白的功能域,从而导致三者的结构和功能发生变化。
蛋白质的功能与结构密切联系,结构域的差异会导致其功能活性方面发生较大的变化[25]。通过比较三者保守结构域发现,PHYB2和PHYB3的N端均缺少PAS和PHY功能域。PAS和PHY功能域可促进无活性的红光吸收型Pr(phytochrome R-absorbing form)和具有生物活性的远红光吸收型Pfr(phytochrome FR-absorbing isomer)之间的转化并维持Pfr的稳定性[26-28]。因此,推断高温下缺少PAS和PHY功能域的PHYB2和PHYB3不能从Pr转化为Pfr,进而无法从细胞质移向细胞核并与核内转录因子相互作用[29]。此外,PHYB3蛋白C端还缺少HisKA功能域,前人研究表明HisKA功能域可通过与下游转录因子相互作用来参与光信号转导[30],并且发现去除HisKA功能域后可加速从Pfr到Pr的热转换,降低了活性Pfr的数量[20,31],推测高温下生菜通过可变剪接改变PHYB2和PHYB3蛋白的功能。
常温条件下照光后,从Pr向Pfr转化时,PHY结构域由延伸链变为-螺旋,使PHY结构域与GAF结构域接触[32]。二级结构分析发现,高温下PHYB3的延伸链比例升高,推测延伸链比例升高将减少PHY结构域与GAF结构域的接触,导致PHYB3在高温下主要以Pr形式存在,影响PHYB与下游转录因子的相互作用,进而提高拟南芥对高温的适应性[33]。
3.2 LsPHYB可变剪接体在高温胁迫下的功能分析
目前,关于表达特征的研究主要集中在响应光周期特点及在不同光照条件下组织特异性表达方面[34],而在响应温度的表达特征方面的研究较少。本研究中,生菜3个可变剪接体表达量达到峰值的时间不同,缺少PAS、PHY和HisKA功能域蛋白亚型的在高温处理第1天表达量最高,之后逐渐降低;而功能域完整的和产生缺乏PAS和PHY功能域蛋白亚型的在高温处理中后期高表达。LIU等[35]研究发现黄瓜赤霉素代谢关键酶Gibberellin 2-beta-dioxygenase8(CsGA2ox8)在强光下通过可变剪接产生“无功能”转录本CsGA2ox8.2来维持幼苗生长所必须的内源赤霉素浓度。WU等[36]研究发现百合(spp.)在高温下通过产生可变剪接体增强对盐胁迫和高温胁迫的耐受性。因此,推测的3个剪接体在生菜响应环境高温过程中发挥着重要功能。
前人研究表明PAS、PHY和HisKA功能域的缺失将导致PHYB无法进入细胞核[37-38],而PHYB的核定位对其发挥生物学功能具有重要作用[39]。另外,有研究表明可变剪接可以通过改变转录本编码蛋白的亚细胞定位影响其生物学功能[40-41],是否通过可变剪接改变蛋白的细胞定位来影响其功能?缺乏功能域的PHYB2和PHYB3蛋白能否正常进入细胞核内发挥作用?这些都有待后续深入研究。
4 结论
克隆了生菜的全长cDNA序列,该基因存在3个可变剪接体、和,分别编码功能域完整的PHYB1蛋白,缺失PAS和PHY功能域的PHYB2蛋白和缺失PAS、PHY和HisKA功能域的PHYB3蛋白。在高温处理前期高表达,和分别在高温处理中、后期高表达,推测生菜3个可变剪接体在抵抗高温胁迫中发挥不同的作用。
[1] LIPPMANN R, BABBEN S, MENGER A, DELKER C, QUINT M. Development of wild and cultivated plants under global warming conditions. Current Biology, 2019, 29(24): R1326-R1338. doi: 10.1016/j.cub.2019.10.016.
[2] HAO J H, ZHANG L L, LI P P, SUN Y C, LI J K, QIN X X, WANG L, QI Z Y, XIAO S, HAN Y Y, LIU C J, FAN S X. Quantitative proteomics analysis of lettuce (L.) reveals molecular basis-associated auxin and photosynthesis with bolting induced by high temperature. International Journal of Molecular Sciences, 2018, 19(10): 2967.
[3] HALLIDAY K J, DAVIS S J. Light-sensing phytochromes feel the heat. Science, 2016, 354(6314): 832-833. doi: 10.1126/science.aaj1918.
[4] LIN J Y, ZHU Z Q. Plant responses to high temperature: a view from pre-mRNA alternative splicing. Plant Molecular Biology, 2021, 105(6): 575-583. doi: 10.1007/s11103-021-01117-z.
[5] CAPOVILLA G, PAJORO A, IMMINK R G, SCHMID M. Role of alternative pre-mRNA splicing in temperature signaling. Current Opinion in Plant Biology, 2015, 27: 97-103. doi: 10.1016/j.pbi.2015. 06.016.
[6] STAIGER D, BROWN J W S. Alternative splicing at the intersection of biological timing, development, and stress responses. The Plant Cell, 2013, 25(10): 3640-3656. doi: 10.1105/tpc.113.113803
[7] 曾纪晴, 张明永. 选择性剪接在植物逆境相关基因表达调控中的作用. 植物生理学通讯, 2006, 42(6): 1005-1014.
ZENG J Q, ZHANG M Y. The role of alternative splicing in the regulation of plant stress-associated gene expression. Plant Physiology Communications, 2006, 42(6): 1005-1014. (in Chinese)
[8] GILBERT W. Why genes in pieces? Nature, 1978, 271(5645): 501.
[9] KEREN H, LEV-MAOR G, AST G. Alternative splicing and evolution: diversification, exon definition and function. Nature Reviews Genetics, 2010, 11(5): 345-355. doi: 10.1038/nrg2776.
[10] KANNAN S, HALTER G, RENNER T, WATERS E R. Patterns of alternative splicing vary between species during heat stress. AoB PLANTS, 2018, 10(2): ply013. doi: 10.1093/aobpla/ply013.
[11] CHANG C Y, LIN W D, TU S L. Genome-wide analysis of heat-sensitive alternative splicing in. Plant Physiology, 2014, 165(2): 826-840. doi: 10.1104/pp.113.230540.
[12] HAYNES J G, HARTUNG A J, HENDERSHOT J D, PASSINGHAM R S, RUNDLE S J. Molecular characterization of the B' regulatory subunit gene family ofprotein phosphatase 2A. European Journal of Biochemistry, 1999, 260(1): 127-136. doi: 10.1046/j.1432- 1327.1999.00154.x.
[13] KINOSHITA S, KANEKO G, LEE J H, KIKUCHI K, YAMADA H, HARA T, ITOH Y, WATABE S. A novel heat stress-responsive gene in the marine diatomcompressum encoding two types of transcripts, a trypsin-like protease and its related protein, by alternative RNA splicing. European Journal of Biochemistry, 2001, 268(17): 4599-4609.
[14] KELLER M, HU Y J, MESIHOVIC A, FRAGKOSTEFANAKIS S, SCHLEIFF E, SIMM S. Alternative splicing in tomato pollen in response to heat stress. DNA Research, 2016, 24(2): 205-217. doi: 10.1093/dnares/dsw051.
[15] MATSUKURA S, MIZOI J, YOSHIDA T, TODAKA D, ITO Y, MARUYAMA K, SHINOZAKI K, YAMAGUCHI-SHINOZAKI K. Comprehensive analysis of rice DREB2-type genes that encode transcription factors involved in the expression of abiotic stress- responsive genes. Molecular Genetics and Genomics, 2010, 283(2): 185-196. doi: 10.1007/s00438-009-0506-y.
[16] LEE S S, JUNG W Y, PARK H J, LEE A, KWON S Y, KIM H S, CHO H S. Genome-wide analysis of alternative splicing in an inbred cabbage (L.) line ‘HO’ in response to heat stress. Current Genomics, 2018, 19(1): 12-20.
[17] AIROLDI C A, MARY M, BRENDAN D.MAFis regulated by temperature-dependent splicing and represses flowering at low temperatures in parallel with FLM. PLoS ONE, 2015, 10(5): e0126516. doi: 10.1371/journal.pone.0126516.
[18] POSE D, VERHAGE L, OTT F, YANT L, MATHIEU J, ANGENENT G C, IMMINK R, SCHMID M. Temperature-dependent regulation of flowering by antagonistic FLM variants. Nature, 2013, 503(7476): 414-417. doi:10.1038/nature12633.
[19] YAN K, LIU P, WU C G, YANG G D, XU R, GUO Q H, HUANG J G, ZHENG C C. Stress-induced alternative splicing provides a mechanism for the regulation of microRNA processing in. Molecular Cell, 2012, 48(4): 521-531. doi: 10.1016/j.molcel. 2012.08.032.
[20] LEGRIS M, KLOSE C, BURGIE E S, ROJAS C C R, NEME M, HILTBRUNNER A, WIGGE P A, SCHÄFER E, VIERSTRA R D, CASAL J J. Phytochrome B integrates light and temperature signals in. Science, 2016, 354(6314): 897-900. doi: 10.1126/science. aaf5656.
[21] JAEHOON J, MIRELA D, CORNELIA K, SUROJIT B, DAPHNE E, GAO M J, KHAN K A, BOX M S, VARODOM C, SANDRA C, MANOJ K, ALASTAIR G, LOCKE J C W, EBERHARD S, JAEGER K E, WIGGE P A. Phytochromes function as thermosensors in. Science, 2016, 354(6314): 886-889. doi: 10.1126/science. aaf6005.
[22] LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-∆∆Ctmethod.Methods, 2001, 25(4): 402-408.
[23] 冯雅岚, 熊瑛, 张均, 原佳乐, 蔡艾杉, 马超. 可变剪切在植物发育和非生物胁迫响应中的作用. 核农学报, 2020, 34(1): 62-70. doi: 10.11869/j.issn.100-8551.2020.01.0062.
FENG Y L, XIONG Y, ZHANG J, YUAN J L, CAI A S, MA C. Role of alternative splicing in plant development and abiotic stress responses. Journal of Nuclear Agricultural Sciences, 2020, 34(1): 62-70. doi: 10.11869/j.issn.100-8551.2020.01.0062. (in Chinese)
[24] 卢欢欢, 邓琴霖, 吴梦丹, 王志敏, 魏大勇, 王鹤冰, 向华丰, 张洪成, 汤青林. 可变剪接调控植物开花的作用机制进展. 生物工程学报, 2021, 37(9): 2991-3004. doi: 10.13345/j.cjb.200628.
LU H H, DENG Q L, WU M D, WANG Z M, WEI D Y, WANG H B, XIANG H F, ZHANG H C, TANG Q L. Mechanisms of alternative splicing in regulating plant flowering: A review. Chinese Journal of Biotechnology, 2021, 37(9): 2991-3004. doi: 10.13345/j.cjb.200628. (in Chinese)
[25] 石国良, 武强, 杨念婉, 黄聪, 刘万学, 钱万强, 万方浩. 苹果蠹蛾几丁质脱乙酰基酶2的基因克隆、表达模式和分子特性. 中国农业科学, 2021, 54(10): 2105-2117. doi: 10.3864/j.issn.0578-1752.2021. 10.007.
SHI G L, WU Q, YANG N W, HUANG C, LIU W X, QIAN W Q, WAN F H. Gene cloning, expression pattern and molecular characterization of chitin deacetylase 2 in. Scientia Agricultura Sinica, 2021, 54(10): 2105-2117. doi: 10.3864/j.issn. 0578-1752.2021.10.007. (in Chinese)
[26] FANKHAUSER C. The phytochromes, a family of red/far-red absorbing photoreceptors.Journal of Biological Chemistry, 2001, 276(15): 11453-11456.
[27] BAE G, CHOI G. Decoding of light signals by plant phytochromes and their interacting proteins. Annual Review of Plant Biology, 2008, 59: 281-311. doi: 10.1146/annurev.arplant.59.032607.092859.
[28] 张媛媛. 光敏色素的结构及其信号调控机制. 湖北农业科学, 2020, 59(4): 5-10. doi:10.14088/j.cnki.issn0439-8114.2020.04.001.
ZHANG Y Y. Structure and signal regulation mechanism of phytochrome. Hubei Agricultural Sciences, 2020, 59(4): 5-10. doi: 10.14088/j.cnki.issn0439-8114.2020.04.001. (in Chinese)
[29] KLOSE C, VICZIÁN A, KIRCHER S, SCHÄFER E, NAGY F. Molecular mechanisms for mediating light-dependent nucleo/cytoplasmic partitioning of phytochrome photoreceptors. The New Phytologist, 2015, 206(3): 965-971.
[30] BURGIE E S, BUSSELL A N, WALKER J M, DUBIEL K, VIERSTRA R D. Crystal structure of the photosensing module from a red/far-red light-absorbing plant phytochrome. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(28): 10179-10184.
[31] BURGIE E S, BUSSELL A N, LYE S H, WANG T, HU W M, MCLOUGHLIN K E, WEBER E L, LI H L, VIERSTRA R D. Photosensing and thermosensing by phytochrome B require both proximal and distal allosteric features within the dimeric photoreceptor. Scientific Reports, 2017, 7(1): 13648. doi: 10.1038/s41598-017- 14037-0.
[32] BURGIE E S, ZHANG J R, VIERSTRA R D. Crystal structure ofphytochrome in the photoactivated state reveals a cascade of structural rearrangements during photoconversion. Structure, 2016, 24(3): 448-457. doi: 10.1016/j.str.2016.01.001.
[33] XU D Q. Multifaceted roles of PIF4 in plants. Trends in Plant Science, 2018, 23(9): 749-751. doi: 10.1016/j.tplants.2018.07.003.
[34] 吴发强. 大豆光敏色素基因的克隆和功能研究[D]. 北京: 中国农业科学院, 2011.
WU F Q. Cloning and functional study of soybean phytochrome genes [D]. Beijing: Chinese Academy of Agricultural Sciences, 2011. (in Chinese)
[35] LIU B, ZHAO S, LI P L, YIN Y L, NIU Q L, YAN J Q, HUANG D F. Plant buffering against the high-light stress-induced accumulation of CsGA2ox8 transcripts via alternative splicing to finely tune gibberellin levels and maintain hypocotyl elongation. Horticulture Research, 2021, 8(1): 170-180.
[36] WU Z, LIANG J H, WANG C P, DING L P, ZHAO X, CAO X, XU S J, TENG N J, YI M F. Alternative splicing provides a mechanism to regulate LlHSFA3 function in response to heat stress in lily. Plant Physiology, 2019, 181(4): 1651-1667. doi: 10.1104/pp.19.00839.
[37] CHEN M, TAO Y, LIM J, SHAW A, CHORY J. Regulation of phytochrome B nuclear localization through light-dependent unmasking of nuclear-localization signals. Current Biology, 2005, 15(7): 637-642. doi: 10.1016/j.cub.2005.02.028.
[38] MATSUSHITA T, MOCHIZUKI N, NAGATANI A. Dimers of the N-terminal domain of phytochrome B are functional in the nucleus. Nature, 2003, 424(6948): 571-574.
[39] HUQ E, AL-SADY B, QUAIL P H. Nuclear translocation of the photoreceptor phytochrome B is necessary for its biological function in seedling photomorphogenesis. The Plant Journal, 2003, 35(5): 660-664.
[40] KOO S C, YOON H W, KIM C Y, MOON B C, CHEONG Y H, HAN H J, LEE S M, KANG K Y, KIM M C, LEE S Y, CHUNG W S, CHO M J. Alternative splicing of thegene generates three transcript variants showing differential subcellular localizations. Biochemical and Biophysical Research Communications, 2007, 360(1): 188-193. doi: 10.1016/j.bbrc.2007.06.052.
[41] HE Z S, XIE R, ZOU H S, WANG Y Z, ZHU J B, YU G Q. Structure and alternative splicing of a heat shock transcription factor gene, MsHSF1, in. Biochemical and Biophysical Research Communications, 2007, 364(4): 1056-1061.
Cloning of Alternative Splice Variants ofin Lettuce and Its Expression Patterns Under Heat Stress
SUI XinYi, ZHAO XiaoGang, CHEN PengYu, LI YaLing, WEN XiangZhen
Horticulture College, Shanxi Agricultural University, Jinzhong 030801, Shanxi
【】Phytochrome B (PHYB) is a receptor for both light and temperature. In this study, the biological functions of alternative splice variants ofin lettuce in response to high environmental temperatures were investigated through cloning them and analyzing their expression patterns, so as to provide a theoretical basis for breeding heat-tolerant lettuce.【】The cDNA sequences ofwere searched from the genomic database of lettuce by bioinformatics method. The clonedandwere subjected to multi-sequence alignment, alternative splice variants form analysis and phylogenetic analysis. Protein properties, such as molecular weight, theoretical isoelectric point, hydrophilicity and hydrophobicity, were predicted by online software. Secondary structure, tertiary structure and conserved domains were analyzed by bioinformatics software. Three alternative splice variants were characterized for expression after high temperature treatment by RT-PCR. 【】There were three alternative splice variants ofobtained by cloning, namely,and, with their CDS lengths of 3 509, 3 877 and 2 690 bp, which encoded 1 094, 960 and 853 amino acids, respectively. Alternative splice forms ofwere alternative 3′ splice site and skipped exon. Alternative splice forms ofwere alternative polyA and retain intron. An alternative splice form ofwas skipped exon. Conserved structural domain analysis showed that the N-terminal of PHYB2 lacked the PAS and PHY domains. The N-terminal of PHYB3 lacked the PAS and PHY domains, and its C-terminal lacked the HisKA domain. Phylogenetic analysis showed that three alternative splice variants were clustered into a clade.qRT-PCR analysis showed that the expression ofwas the highest at the first day of high temperature treatment;had higher expression thanandat days 5-9 of high temperature treatment; at day 11 of high temperature treatment, the expression ofwas higher than that ofand. The three alternative splice variants peaked at different times during the 11 days of high temperature treatment. 【】There were three alternative transcript variants of, named,andB3.expression was the highest in the early stage of high temperature treatment,in response to high temperature stress.
lettuce;; alternative splice variants; heat stress; response

10.3864/j.issn.0578-1752.2022.09.011
2021-09-06;
2021-12-04
山西省重点研发计划(201803D221001-1)
隋心意,E-mail:sui821793836@163.com。赵小刚,E-mail:1185178867@qq.com。隋心意和赵小刚为同等贡献作者。通信作者温祥珍,E-mail:330821473@qq.com
(责任编辑 赵伶俐)

