原核生物转录调控研究技术及进展
陈臣 黄芝阳 于海燕 袁海彬 田怀香
(上海应用技术大学香料香精技术与工程学院,上海 201418)
继Francois Jacob和Jacques Monod在操纵子学说中提出基因转录调控的概念之后,科学家开始意识到转录调控在原核生物调控中的重要性。1995年实现了第一个原核生物的全基因组测序[1],随着测序技术不断地进步,大量完整的基因组序列快速出现[2-3],尤其是人类基因组计划的顺利进行,使得研究的焦点由结构基因组学过渡为功能基因组学[4],而基因转录调控正是功能基因组学的一个重要内容。
基因的调控可在多个层次上进行,包括基因水平、转录水平、转录后水平、翻译水平和翻译后水平的调控。与真核生物相比,原核生物的调控主要发生在转录水平,根据调控机制的不同可分为负转录调控和正转录调控。由于调控的关键步骤主要是在启动子的转录阶段,所以对启动子和转录进行研究具有重要的意义。启动子附近存在一段特定序列能够结合称为转录因子(transcription factors,TFs)的蛋白质,结合RNA聚合酶并由此起始基因转录[5],转录因子在调节细菌中的基因表达方面起核心作用,它通过与位于启动子区的结合位点(transcription factor binding sites,TFBSs)相结合,从而激活或抑制相应基因的转录。历史上,研究最多的转录因子之一是大肠杆菌(Escherichia coli)的乳糖阻遏蛋白[6]。近年来,在解析微生物生理功能或某些功能成分生物合成研究中的转录因子不断被挖掘,转录因子与结合位点及其调控方式已经成为研究的热点。大量研究表明,找到这些特定的片段和位点对于研究基因的转录调控有重要的意义,有助于进一步揭开蛋白质与核酸相互作用的规律,了解原核生物的基因转录调控机制。随着分子生物学及相关技术研究不断发展与完善,一些新的方法应运而生,为阐明原核生物的基因调控提供了有力的工具。通过改造原核生物的转录调控网络,调整代谢流向构建高效的细胞工厂,更好发挥原核生物作为生产宿主的优势,对于突破传统代谢工程方法改变细胞表型及提高目标化合物产量等方面具有广阔的前景[7]。
本文主要综述了有关原核生物转录调控中核酸-蛋白质相互作用现有的一些研究技术,包括凝胶电泳迁移率实验(electrophoretic mobility shift assay,EMSA)、DNase I footprinting技术、染色质免疫共沉淀(chromatin immunoprecipitation,ChIP)技术、微量热泳动技术(MicroScale Thermophoresis,MST)、等温滴定量热法(isothermal titration calorimetry,ITC)及细菌单杂交系统(bacterial-one-hybrid system,B1H),分别从6种方法的原理、应用和优缺点等方面进行介绍(表1),并展望了未来的研究热点,为进一步阐明原核生物的基因调控提供借鉴。
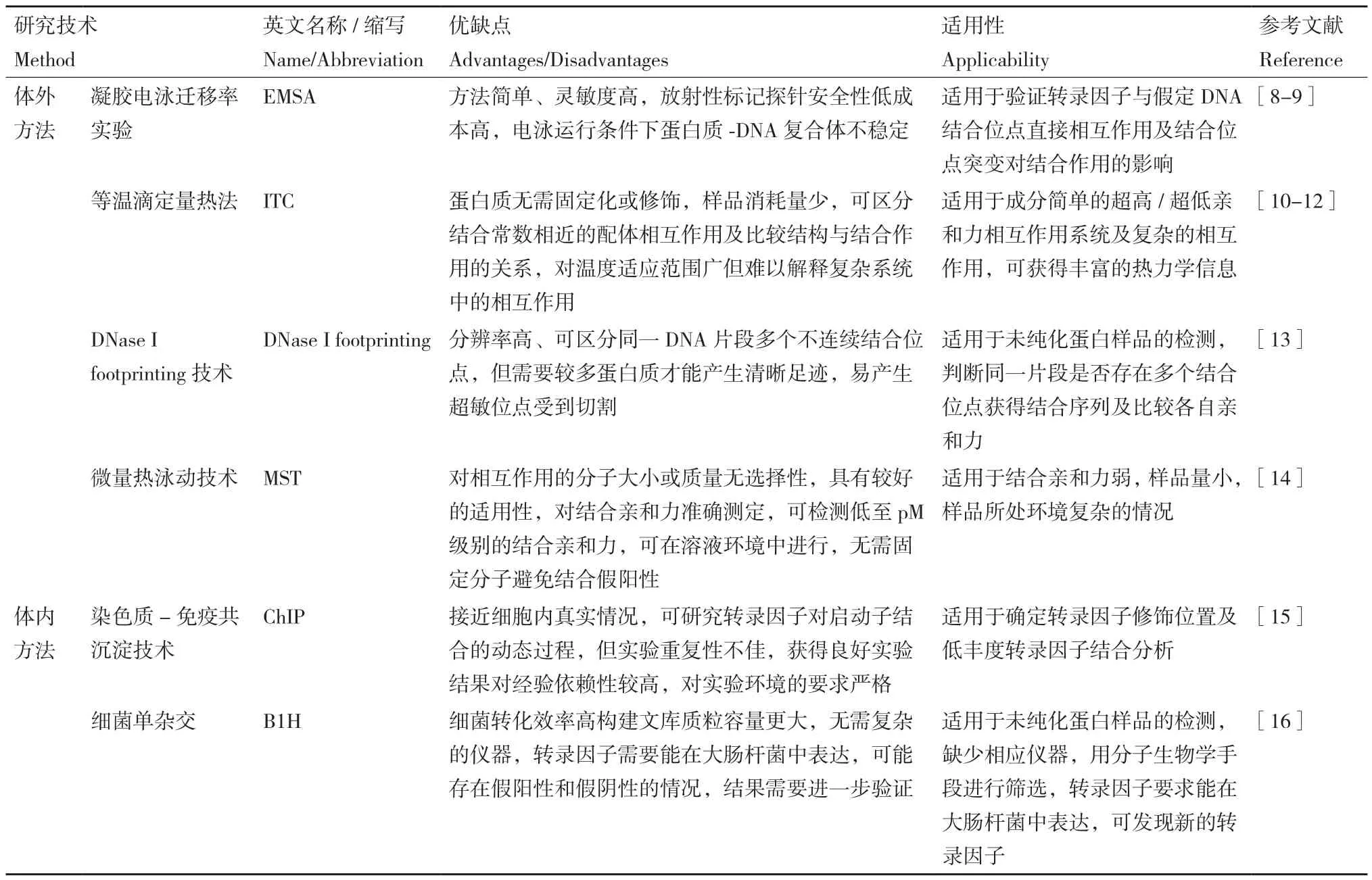
表1 原核生物转录调控研究技术总结Table 1 Summary of research techniques on transcriptional regulation in prokaryotes
1 凝胶电泳迁移率实验(EMSA)
1.1 EMSA基本原理
EMSA是一种在体外研究蛋白质与核酸相互作用的简单、廉价、快速而灵敏的技术,它可实现对目的蛋白与核酸作用的定性和定量分析[8,17],此外,还可用于热力学和动力学参数的定量测量以及构象变化的分析[18]。EMSA的作用原理为(图1):通常是将寡核苷酸标记的DNA或RNA探针与DNA特异结合蛋白或RNA一同孵育,随后通过非变性聚丙烯酰胺凝胶电泳(non denaturing polyacrylamide gel electrophoresis,Native-PAGE)分离蛋白质-核酸复合物与游离的探针。由于目的蛋白与核酸探针结合后分子量增大,故其电泳迁移率会降低,在凝胶中将会看到阻滞的条带[17]。其电泳迁移率是生物分子的固有物理性质,与生物大分子大小、电荷和分离介质的粘度有关[19]。根据所显示滞后带的有无和量的多少,来反映DNA结合蛋白与DNA探针的结合活性和特异性,并可以计算出两者的结合常数或解离常数。
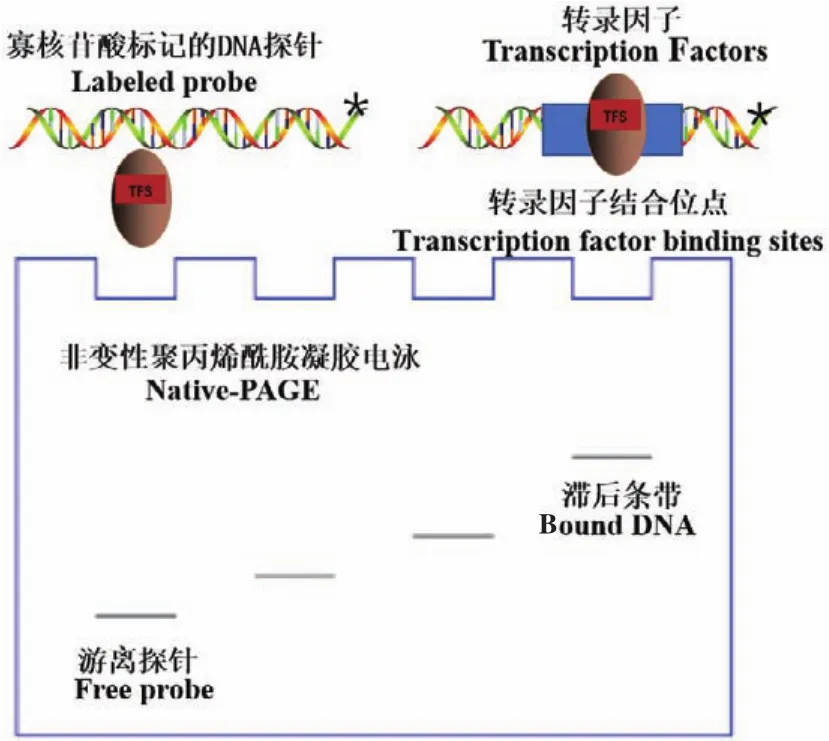
图1 EMSA实验的基本原理Fig.1 Basic principle of EMSA experiment
1.2 EMSA鉴定转录调控因子-结合位点直接相互作用及适用性分析
EMSA实验有许多优点,传统的EMSA实验一般采用放射性同位素(32P)标记核酸探针,其基本操作技术简单,灵敏度高,研究表明,即使待测样品的浓度≤0.1 nmol/L,样品的体积≤20 μL也可进行测定[8]。但是同位素的半衰期短,实验周期长,对人体及环境均会产生危害且处理成本高[20]。因此许多研究者对该方法进行了改进,出现了采用生物素或地高辛标记的核酸探针代替传统的放射性同位素标记的探针,利用碱性磷酸酶或辣根过氧化物酶化学发光检测系统检测[21]。例如葛兴枫等[22]利用经蛋白酶K消化步骤优化的生物素标记的EMSA方法,也可明显观察到核酸适配体与靶蛋白结合的阻滞条带,与同位素标记的EMSA实验无明显差异。此外这两种方法安全、成本低且检测时间短。然而Vavrova等[20]研究表明,网织红细胞系统体外翻译蛋白质不适用于非放射性EMSA,因为网织红细胞裂解物中存在干扰的生物素化蛋白质;Tokunaga等[23]也发现在使用地高辛标记的探针检测转录因子的过程中阴性对照实验存在相当大的非特异性变化带的问题,影响目标蛋白的检测。另一个限制因素是电泳过程中蛋白质与核酸的复合物处于非化学平衡状态,转录因子的结合作用可能被减弱,同时某些络合物在凝胶中的稳定性显著高于溶液环境,使得转录因子与DNA的亲和力检测结果偏低,或是EMSA实验对某些相互作用分子的研究效果不佳[8,24]。
EMSA实验的主要应用之一是鉴定转录因子与DNA结合蛋白的作用位点,Brown等[25]在研究德氏乳杆菌乳酸亚种CRL581(Lactobacillus delbrueckii subsp.lactis CRL 581)中与蛋白水解系统相关的基因时,通过EMSA实验发现YebC与蛋白水解酶系统相关基因prtL、oppA1和opts启动子区域结合,表明YebC是关键蛋白水解酶基因的转录调控因子。王丽滨[26]利用EMSA实验验证了碳代谢调控蛋白A(catabolite control protein A,CcpA)与肺炎链球菌(Streptococcus pneumoniae)荚膜多糖基因座转录调控区域的结合,为研究肺炎链球菌的致病机理及防治提供了理论依据。本课题组研究植物乳杆菌CcpA对碳分解代谢抑制作用(carbon catabolite repression,CCR)的调控作用时,通过EMSA证实CcpA与分解代谢反应元件(catabolite responsive element,cre)位点的结合[27]。
EMSA是一项较为成熟的研究DNA-蛋白质相互作用的技术,可获得蛋白质-核酸相互作用的亲和力、特异性、动力学特性及最小结合位点。EMSA的优点是可以分析与核酸结合的多个蛋白质或蛋白质复合体,但EMSA在体外进行实验不能精确揭示体内蛋白质-DNA的相互作用,转录因子需要通过硫酸铵分步沉淀、凝胶过滤层析、离子交换层析等技术进行一定程度的纯化,增加了实验操作的复杂性。此外有许多变量只能通过经验确定,要根据具体相互作用的物质调整反应条件和凝胶运行条件,核酸靶标、结合反应的条件以及凝胶电泳的条件是实验优化主要考虑的因素[28]。
2 DNase I footprinting技术
2.1 DNase I footprinting基本原理
DNase I footprinting技术是用于体外研究蛋白质与DNA结合位点的方法。它的原理是(图2):首先对待测双链DNA片段中一条链的一端选择性地进行标记,再加入适当浓度的脱氧核糖核酸酶I(DNase I),在DNA双链上形成缺口,经过变性后聚丙烯酰胺凝胶电泳分离,放射自显影,即形成以相差一个核苷酸为梯度的DNA条带[29]。但当DNA片段与相应的序列特异性DNA结合蛋白结合后,DNA结合蛋白可局部抑制DNase I对相应DNA片段的消化,因此在放射自显影图谱上,DNA梯度条带在结合区域中断,从而形成一空白区域,恰似蛋白质在DNA片段上留下的足迹,根据其放射自显影呈现的结果把这种研究DNA-蛋白质特异性结合的方法形象地称为足迹法。如果要得出与目的蛋白特异性结合的碱基序列,可以进一步对DNA进行测序。经过40余年的发展DNase I footprinting技术不断得到完善,包括:(1)对双链DNA片段其中一条链进行标记时采用生物素进行末端标记,避免放射性同位素的掺入,减少危害;(2)可利用固定相磁珠对传统DNase I footprinting技术进行改进,在获取特异性DNA结合蛋白时省略了硫酸铵分步沉淀、离子交换层析、凝胶过滤层析等蛋白纯化步骤,可适用于未经纯化的蛋白粗提物内特异性DNA结合蛋白的研究,与传统方法相比具有操作简便、效率高、使用范围更广泛等显著优势[30]。
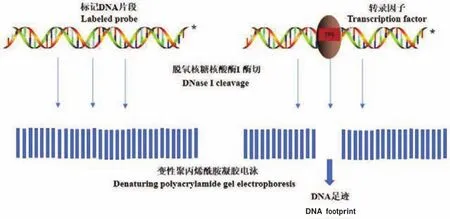
图2 DNase I footprinting技术基本原理Fig.2 Basic principle of DNase I footprint technology
2.2 DNase I footprinting获取结合位点及适用性分析
DNase I footprinting技术在研究生物体转录调控中有广泛的应用,常用来得到对某个确定基因具有调控作用的调控因子在基因组上的结合位点。Gong等[31]研究短链乳杆菌(Lactobacillus brevis)GlnR对谷氨酸依赖的耐受性的调节作用,为确定GadR基因和gadCB操纵子的GlnR结合位点,用6-羧基荧光素(6-FAM)标记的DNA片段进行DNase I footprinting分析发现了PgadR’和PgadCB’分别和GInR的结合序列。Ihara等[32]研究大肠杆菌在L-丙氨酸积累后,编码一种L-丙氨酸输出蛋白的alaE基因的表达受到氮代谢全局调控因子亮氨酸反应调节蛋白(LRP)的正向调控,为进一步识别LRP的结合位点采用了DNase I footprinting技术找出LRP与alaE上游区域-161- -83 bp的核苷酸序列结合。较多情况下转录因子与受到调控作用的基因存在多个结合位点,DNase I footprinting技术能找出位于同一片段不连续的结合位点并获得各自的结合序列。Yang等[33]研究植物乳杆菌(Lactobacillus pl-antarum)在高浓度乙醇环境下的强适应性发现调节因子AcrR与编码3-羟基酰基-ACP脱水酶(FabZ1)基因的启动子PFabZ1结合调控FabZ1的转录表达,利用DNase I footprinting技术获得PFabZ1上两个特异的AcrR结合位点。
与其他转录调控研究技术相比,DNase I footprinting技术不仅证实了DNA序列与目标蛋白质特异性结合,还可以得出调控因子与DNA调控元件特异性结合的DNA序列,为研究生物体转录调控网络提供了极大的便利。DNase I footprinting也可用于研究EMSA技术表现不佳的转录因子,例如一种相对低亲和力的DNA结合蛋白xUBF在DNase I footprinting分析中取得比EMSA更好的效果[13]。但在进行DNase I footprinting分析时要有足够多的蛋白质才能产生清晰的足迹,而蛋白质含量增加要求DNase I浓度也要相应升高,由于DNase I本身是一种蛋白,也可竞争结合在相应的位点干扰蛋白质结合靶基因出现假阳性影响实验结果准确性,所以实验前必须仔细滴定DNase I浓度[34]。
3 染色质-免疫共沉淀技术(ChIP)
3.1 ChIP基本原理
ChIP技术是研究蛋白质和基因组DNA特定区域相互作用一种有效的方法[35],是用于评估蛋白质与体内特定DNA区域结合的多用途技术。1993年,在果蝇的培养细胞中,Polycomb阻遏蛋白是第一个使用ChIP技术进行定位的蛋白质,为了解其生物学功能奠定基础[36]。ChIP技术是一个多步骤的过程,每个步骤都需要标准化以获得最佳结果。其作用原理为(图3)[37]:通常是在生理状态下将细胞内的DNA与蛋白质交联在一起[37],通过机械或酶消化将染色质碎片化后,用目的蛋白的特异性抗体免疫沉淀富集DNA-蛋白质交联复合体[38],只有与目的蛋白结合的DNA才能被沉淀下来,获得目的蛋白结合的DNA后,通过PCR、芯片和测序技术等,以定量检测特定的DNA靶标[15]。ChIP技术可用于分析转录因子、转录辅因子、DNA复制因子和DNA修复蛋白的结合[39],是研究细胞内蛋白质-DNA相互作用的重要技术。
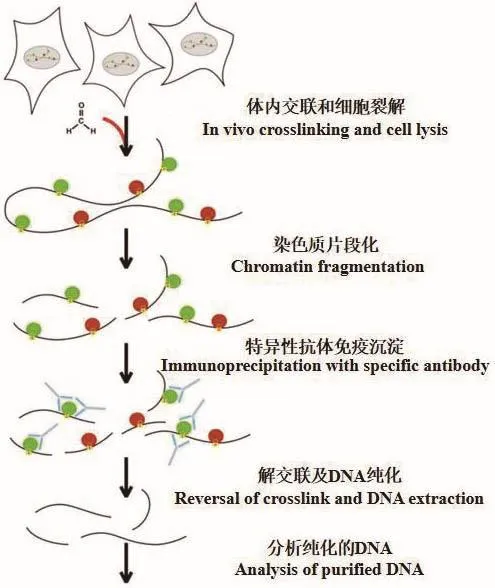
图3 ChIP实验的基本原理Fig.3 Basic principle of ChIP experiment
3.2 ChIP筛选结合位点及适用性分析
最初,ChIP技术用于研究高乙酰化组蛋白与特定DNA序列的结合,以进一步了解组蛋白乙酰化在转录中的作用[40]。近年来,为确定转录因子在基因组内的结合位点并监测其分布的动态变化,开发了与芯片杂交结合的染色质免疫沉淀技术(ChIP-chip),并且已经被广泛应用于鉴定真核生物基因组上DNA结合蛋白的位置[41],研究表明此方法也可用于原核生物。Ratib等[42]利用ChIP-chip技术先在体内交联DNA-蛋白质复合物再与基因芯片杂交检测到ChvI蛋白除EMSA实验获得的其他结合位点,通过ChIP-chip技术获得了较其他体外检测方法更多的与ChvI蛋白直接结合的靶基因。Yun等[43]利用ChIP-chip技术筛选恶臭假单胞菌KT2440(Pseudomonas putida KT2440)基因组中调控因子Pmr、TurA和TurB的结合区域。
ChIP与高通量测序相结合(ChIP-seq)是体内蛋白质-DNA相互作用的全基因组分析的有力工具。基本原理是先获得与转录调控因子特异性结合的DNA片段,再通过测序技术确定其基因序列。近年来,ChIP-seq逐渐超越了ChIP-chip,越来越多地应用于转录调控研究中。Shao等[44]在研究囊性纤维化患者的条件致病菌铜绿假单胞菌(Pseudomonas aeruginosa)内一种全局调控因子RpoN对铜绿假单胞菌一组毒力因子和群体感应基因的作用,利用ChIP-seq鉴定了RpoN的1 068个结合位点,并通过EMSA等技术证实它们之间存在特异性结合作用。2012年,Bard等[45]通过分析转录因子EVI1靶向序列的特征,发现在EVI1的ChIP-seq峰附近常常伴随有低聚果糖结合位点,并通过ChIP-seq和芯片技术证实FOS是EVI1的相互作用分子。本课题组通过ChIP-seq技术证实两个调控植物乳杆菌中FOS代谢的基因簇的预测启动子区域存在6个潜在的CcpA结合cre位点,揭示了依赖CcpA的CCR作用调节FOS代谢的调控网络[27]。
ChIP技术是研究DNA-蛋白质互作的常用技术,具有特异性较强且DNA富集效率高的特点。从研究目的来说,更多情况下研究者希望得到真实反映在相应生理条件下转录因子与DNA的结合情况,作为一种体内研究方法ChIP技术则能很好满足这一需求。但ChIP技术也相对考验操作经验,ChIP技术对染色质片段的长度以及实验中其他影响因素有较高要求,如以甲醛为交联剂时影响转录因子的分布情况,使用交联剂时需要注意交联剂浓度和交联时间、优化超声破碎参数使染色质的破碎片段长度在一定范围、需要选择合适的抗体等,结果分析时也需要格外注意,与目的蛋白结合的其他蛋白质也会与DNA结合影响实验结果[46]。
4 微量热泳动技术(MST)
4.1 MST基本原理
MST是一种基于荧光检测的优势,与热泳动相结合,能快速、精确量化微升溶液中生物分子相互作用的方法。这项技术的基础是一种称为热泳的物理效应,它描述了分子通过温度梯度的定向运动。分子的热泳特性取决于其大小、电荷和水化层[47]。其作用原理为(图4)[48]:运行MST实验时,红外激光瞬间加热生成一个微观温度梯度场,通过共价结合的荧光染料、荧光融合蛋白或蛋白自身色氨酸荧光来监测并定量分析分子的定向移动[49],以检测相互作用的生物分子间的亲和性。简而言之,其中一种分子用荧光染料标记并保持恒定的浓度,另一种分子被稀释至适宜的倍数,建立等浓度梯度稀释系列,以此产生大量浓度范围,随后将这两种分子混合并加载到用以扫描的毛细管中,样品经温度梯度,荧光探测器跟踪荧光标记分子的运动。分子在初始温度和新温度下的荧光差异产生未标记分子的结合曲线[50]。另外,MST还可以进行分子间竞争结合测定、多结合配体的测定、稳定性测定和筛选测定,获取化学计量和热力学参数等[51]。
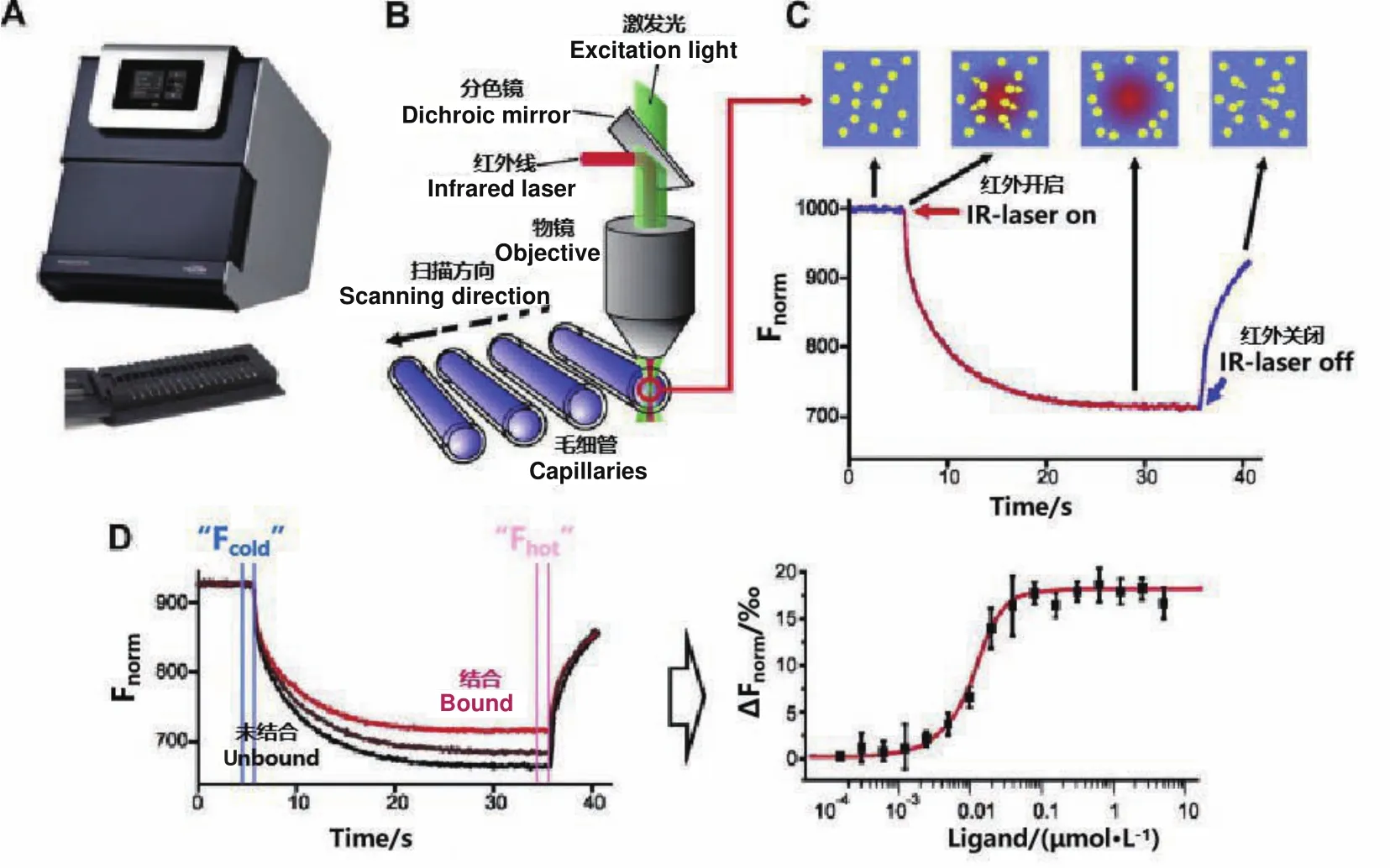
图4 MST实验的基本原理Fig.4 Basic principle of MST experiment
4.2 MST定量分析结合相互作用及适用性分析
近年来,MST成功地用于分析各种生物分子相互作用,如寡核苷酸相互作用[52]、蛋白质-DNA相互作用[53]、蛋白质-小分子蛋白质相互作用和蛋白质-脂质相互作用[54-55]。在蛋白质与DNA的相互作用研究中,Papageorgiou 等[56]用MST方法检测嗜热栖热菌(Thermus thermophilus)组蛋白类DNA结合蛋白HUTth 与pUC18质粒DNA的相互作用,测得了HUTth与pUC18结合的解离常数,并在相同实验条件下测定了另外两个HU蛋白与pUC18的亲和力得出了HU蛋白与DNA分子结合的化学计量比,发现MST方法重复性好。MST技术提供了方便的数据分析系统,根据实验的需要配置了多种曲线拟合的选项,在数据采集的动态过程中即可分析数据[47]。而且MST技术在几乎任何的溶液环境都适用,使某些复杂体系如细胞裂解液中相互作用分子的测量更加简便[57]。
MST技术的优势是快速、灵敏、所需样品量少、对研究对象并无特殊的选择性、无相互作用分子质量和大小以及相互作用后尺寸变化较大等要求[14],同时由于MST的检测过程是在溶液中进行所以该技术提供了接近自然状态的检测方法[58],并且利用检测仪器分析软件得出MST结合曲线并进行数据分析可以方便得出配体分子是否结合、结合位点数量及亲和力等信息[59]。因此MST技术已广泛用于生命科学研究的诸多方面。但是,MST也存在一定的局限性,比如仪器价格昂贵、不能直接确定具体的结合位点等。
5 等温滴定量热法(ITC)
5.1 ITC基本原理
ITC是一种广泛用于研究分子间结合相互作用的方法,ITC实验的主要数据是热分析图,图中记录了绝热的对照池和样品池之间保持恒定的温差所需的时间进程。其原理为(图5)[10]将已知浓度的反应物滴定到样品中,每次样品的注入都会引起样品池中溶液成分的变化,并且在其反应达到新的平衡期间,系统将记录反应热并形成相应的峰,每个峰的面积表示在样品与样品池中组分反应时产生或消耗的总热量。例如,在模拟整合加热的等温线之后,可以得到反应焓变、结合亲和力和结合化学计量等信息[60]。
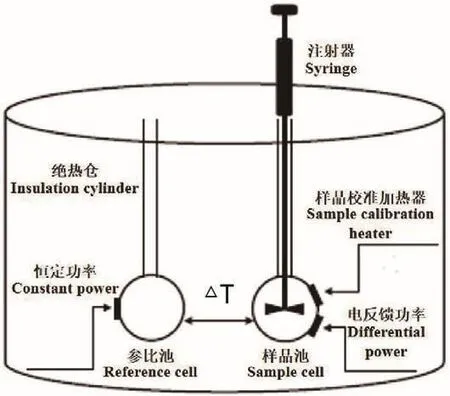
图5 ITC实验的基本原理Fig.5 Basic principle of ITC experiment
5.2 ITC研究结合作用模式及适用性分析
在过去的几十年里,ITC已经被应用到众多的系统中。这些研究大多集中于分子间的相互作用,如蛋白质之间、蛋白质与小配体之间、DNA与其他大分子系统之间以及酶动力学中[61]。在原核生物的转录调控研究中,Zhuo等[62]研究黄色粘球菌DK1622(Myxococcus xanthus DK1622)groELs差异性表达的转录调控网络,通过ITC技术用DNA滴定粘球菌DK1622的HrcA蛋白,分析HrcA蛋白和分子伴侣表达反向重复序列(controlling inverted repeat of chaperone expression,CIRCE)之间的相互作用,发现HrcA蛋白不能与CIRCE2groESL1元件结合,而倾向于与CIRCE1groESL1结合,其次是CIRCEgroEL2。2014年,Wang等[63]通过该方法对一些调控因子与启动子的结合参数进行了详细的表征,另外还对调控因子结合效应物小分子的相关参数进行了分析,展示了外源抗生素JdB 对天蓝色链霉菌ScbR2(Streptomyces coelicolor ScbR2)的结合情况,数据表明它们近似1∶1结合,取得了较好的研究效果。虽然ITC技术可应用于多种系统相互作用的研究,但传统的ITC技术直接滴定的方法难以用于超高(Ka>108M-1)/超低(Ka<104M-1)亲和力系统的测定,利用与目标分子具有较弱亲和作用的竞争性配体采取置换滴定的方式,可获得准确的热力学信息,提高该方法的适用性[11-12]。ITC技术还可更进一步研究转录调控因子的某一结构域与DNA序列的关系,得出结合亲和力的具体信息、反应焓变和熵等参数可帮助研究相互作用分子之间的结合模式,为更深入研究转录调控因子提供了丰富的信息[64]。
ITC在分子相互作用研究方面有两大优势。首先,ITC主要应用于天然蛋白,不需要对其进行修饰;其次,ITC提供了丰富的热力学信息,包括焓值、熵值和热容量等。因此,该技术提供的丰富的信息常用来推断分子间相互作用反应机制,例如结合位点的数目、不同构象反应物滴定的热力学差异等[65]。然而,传统的ITC也存在局限性,其分析受到随机热谱图噪声影响,且单个滴定实验信息量有限。因此,Brautigam 等[66]提出一个基于自动形状分析注射峰的无偏差热成像积分的方案,然后将来自不同量热滴定实验的等温线组合进行全局分析,并将结合参数的统计分析结果以图形直观地表示。
6 细菌单杂交系统(bacterial-one-hybrid system)
6.1 细菌单杂交系统基本原理
细菌单杂交系统是一种在体内研究转录因子和目的基因相互作用的研究技术,该技术是在酵母单杂交和细菌双杂交技术的基础上发展而来。细菌单杂交系统的原理是转录因子与RNA聚合酶的α亚基融合表达,识别随机文库载体上的DNA片段并与之结合,使RNA聚合酶聚集到报告基因上游弱启动子区域,激活报告基因his3和ura3表达(图6)[67]。该系统包括3个组成部分:转录因子表达载体、结合随机位点文库的报告载体和营养缺陷型宿主菌[68]。转录因子与启动子区域结合激活报告基因his3和ura3,有利于组氨酸和尿嘧啶合成,转录因子表达载体和随机文库报告载体转化到营养缺陷型宿主菌中使其在含有一定浓度3-氨基-1,2,4-三唑(3-AT)的缺乏组氨酸和尿嘧啶筛选平板上生长,筛选出与转录因子相互作用的DNA片段。
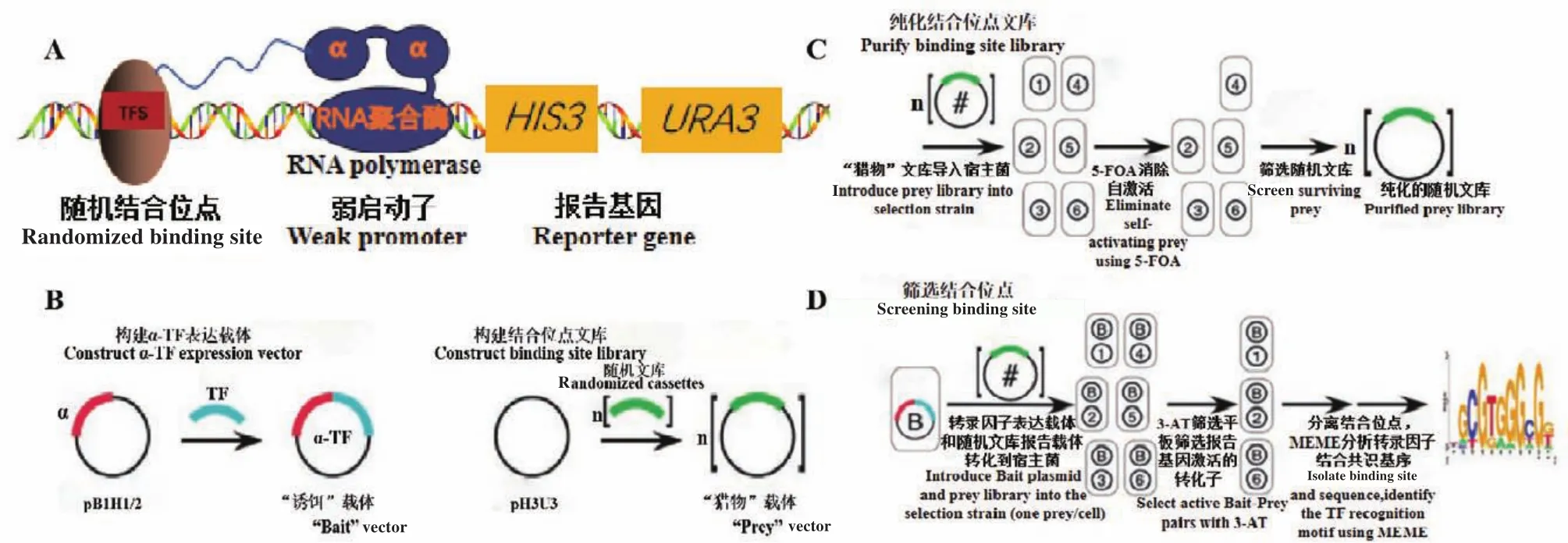
图6 细菌单杂交系统基本原理Fig.6 Basic principle of bacterial one hybrid system
6.2 细菌单杂交筛选转录调控因子及适用性分析
细菌单杂交系统在研究蛋白质-DNA相互作用方面得到了广泛的应用,如Hebdon等[69]利用细菌单杂交系统筛选了艰难梭状芽孢杆菌R20291(Clostridioides difficile R20291)反应调节因子RR_1586的可能调控靶点,并利用EMSA验证了RR_1586与该段基因序列的结合,证实了RR_1586结合位点位于可能的靶基因上游。Zhai等[70]发现保加利亚乳杆菌(Lactobacillus bulgaricus)在酸胁迫条件下预测的转录调控因子Ldb0677表达上调1.84倍,用细菌单杂交和EMSA测定了Ldb0677的DNA结合特异性,表明它可能是保加利亚乳杆菌酸胁迫反应的主要调节因子。对于基因转录调控研究,建立有关物种的转录调控因子数据库,包括已经证实的转录因子、全基因组结合位点、位置权重矩阵(position frequency matrix,PFM)等信息,对快速构建相关的转录调控网络有很大帮助,细菌单杂交系统因其较高的转化效率被用于建立转录因子特异性结合数据库[71]。
与EMSA相比,细菌单杂交系统不需要对转录因子进行蛋白纯化;此外,细菌生长速度较快,具有更高的转化效率,与酵母单杂交系统相比此技术可以搜索更大的随机文库。通过此方法有利于发现与相应DNA序列特异性结合的潜在转录因子。此方法具有操作简单,无需复杂仪器且转化效率高的特点,适用于转录因子结合位点鉴定并进行高通量分析,但有可能出现假阳性的结果,所以往往需要与其他技术联用来验证结果的准确性。
7 总结与展望
基因表达调控是生物遗传调控的一个重要方面,转录调控是原核生物关键调控步骤且是一个非常复杂的过程。本文总结了原核生物DNA-蛋白质相互作用的研究技术,主要从基本原理、实验方法、注意事项及其进展进行综述。通过其具体应用了解这些研究技术在研究原核生物基因转录调控网络的重要作用及其发展趋势。
尽管上述的每项研究技术都是一项独立的技术,但是目前看来某种研究技术都存在各自的优缺点和适用性,可以根据情况选择合适的技术而且往往需要相互补充才能确认DNA-蛋白质相互作用的特异性和亲和性,因此在研究中通常两项或两项以上技术结合使用。此外,对于转录调控网络的研究技术还有待于进一步地发展,通过各种技术手段建立转录因子与启动子之间的动态调控网络更为重要,以期能够重新解释原核生物的各种生理现象,为构建更加复杂、更准确地反映原核生物生命现象的动态网络提供更多的经验。随着后基因时代的发展,原核生物基因转录调控研究技术的应用会越来越广泛。对这些研究技术的改进或在原有技术基础上开发出新技术使其适用于更多的情况,为原核生物的基因改造提供新的靶点和思路,从而更好地发挥原核生物作为生产宿主的优势,助力创造绿色低碳、可持续发展的资源节约型生产方式,为人类造福。

