细粒棘球绦虫Eg95抗原表位的生物信息学预测*
李玉娇,王 晶,赵 慧,贾海英,李 博,马秀敏,温 浩,丁剑冰,
2.新疆医科大学附属中医医院,乌鲁木齐 830000;
3.新疆自治区人民医院,乌鲁木齐 830000;
4.新疆医科大学基础医学院,乌鲁木齐 830011
细粒棘球绦虫Eg95抗原表位的生物信息学预测*
李玉娇1,王 晶2,赵 慧1,贾海英3,李 博2,马秀敏4,温 浩1,丁剑冰1,4
目的 应用软件和在线网络分析Eg95的蛋白二级结构,预测B细胞表位和T细胞表位,为确定和筛选优势表位,研制安全、高效的表位疫苗奠定基础。方法 采用DNAStar软件和在线网站IEDB、SYFPEITHI等对Eg95的B细胞表位和 T细胞表位进行预测。结果 Eg95存在潜在的抗原表位,B细胞表位区域:16-38,41-48,50-67,68-90,92-100,103-113,120-132。分值高的 T细胞表位区域:6-14,14-22,48-56,4-12,98-106,142-150,146-154。结论 运用生物信息学方法确定Eg95存在7个抗原表位,对进一步研究Eg95的抗原性和研发优势表位疫苗具有重要的意义。
Eg95;抗原表位;生物信息学
2.新疆医科大学附属中医医院,乌鲁木齐 830000;
3.新疆自治区人民医院,乌鲁木齐 830000;
4.新疆医科大学基础医学院,乌鲁木齐 830011
细粒棘球蚴病(echinococcosis)又称囊型包虫病(cystic echinococcosis,CE),是由细粒棘球绦虫(Echinococcusgranulosus,Eg)幼虫引起的一种人兽共患慢性寄生虫病,呈全球性分布。我国是细粒棘球蚴病发病最高的国家之一,至今已在25个省、市、自治区发现有人、畜原发性棘球蚴病,主要流行在西部的新疆、青海、甘肃、宁夏等牧区和半牧区[1]。经过流行病学统计,2004-2008年总体上报告包虫病病例数呈增加趋势[2]。目前对于包虫病的治疗主要以手术治疗为主,药物治疗为辅,效果欠佳。采用给犬定期驱虫和宣传教育为主的综合性防治措施的效果也不理想,因此人们一直在研究更有效的防治措施。免疫预防是防止包虫病流行比较理想的途径。采用现代分子生物学技术对细粒棘球绦虫的有效免疫原成分进行筛选和克隆,制备基因工程疫苗,为包虫病的免疫预防和免疫诊断开辟了新途径[3]。Lightowlers等[4]发现Eg95重组蛋白疫苗免疫中间宿主(羊),可抵抗六钩蚴的感染获得95%~100%的免疫保护作用,其中86%得到完全保护,认为Eg95是较理想的保护性抗原。贾海英等[5]将Eg95与pET28a重组融合构建了pET28a-Eg95重组质粒,经过Western blot实验,其中11例阳性患者的血清中有8份都与pET28a-Eg95重组蛋白有阳性反应,其中5份正常人的血清不与其发生阳性反应,该结果表明Eg95抗原在CE病人体内有较高免疫原性。随着分子生物学技术的发展,表位疫苗[6](Epitope vaccine)成为分子疫苗的研究热点,它是根据抗原表位氨基酸序列制备而成的疫苗,包括合成肽疫苗[7](synthetic peptide vaccine)、重组表位疫苗[8](Recombinant epitope-based vaccine)及表位 核 酸 疫 苗[9](Epitope DNA vaccine,minigenes/epigenes)等形式,是目前研制抗感染性疾病、恶性肿瘤、寄生虫病疫苗的发展方向,研制表位疫苗面临的最大挑战就是如何找到抗原上免疫原性最强的区域,即表位所在的位置[10]。Eg95分子是一种分子量为24.5kDa的天然六钩蚴抗原,本研究运用生物信息学方法进行Eg95的B细胞和T细胞抗原表位预测,为包虫病诊断和表位疫苗的研制奠定基础。
1 材料与方法
1.1 材料
1.1.1Eg95氨基酸序列 根据GenBank的Eg95基因序列,经过多重序列比对,确定最终进行分析的Eg95基因序列(GenBank登录号:HM345607),长471bp,用DNAMAN软件推导出Eg95抗原氨基酸序列,它编码156个氨基酸残基:MAFQLCLILFATSVLAQEYKGVGKGQGQQETPLRNHFNL TPVGSQGIRLSWEVQHLSDLKGTDISLRAVN PSDPLVCKRQTAKFSDGQLAVGELKPSTLYK MTVEAVKAKKTILGFTVDIETPRAGKKEST VMTSGSALTSAIAGFVFSCIVVVLT
1.1.2 蛋白质二级结构预测软件TMHMM Sever2.0 (http://www.cbs.dtu.dk/services/ TMHMM)分析Eg95抗原的跨膜序列,SOPMA Sever(http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_sopma.html)预测Eg95的蛋白二级结构。
1.1.3 抗原表位预测软件 B细胞表位:DNAStar软件及在线预测软件IEDB (http://tools.immuneepitope.org/main/index.html)。T 细胞表位:(SYFPEITHI http://www.syfpeithi.de)和(BIMAS http://bimas.dcrt.nih.gov/ molbio/hla_bind/)。
1.2 方法
1.2.1 采用DNAStar软件的Protean程序以及在线预测网站IEDB (http://tools.immuneepitope.org/main/index.html)结合亲水性参数、可及性参数、抗原性指数、柔韧性参数及二级结构方案对Eg95抗原的B细胞表位进行综合预测。
1.2.2 通过在线网络资源的使用在以下两个网站:(SYFPEITHI http://www.syfpeithi.de)和(BIMAS http://bimas.dcrt.nih.gov/molbio/hla_bind/)通过其各种算法对Eg95抗原的T细胞表位进行预测。
2 结 果
2.1 B细胞表位预测结果
2.1.1 Eg95跨膜结构预测 用 TMHMM 对Eg95的跨膜序列进行分析,Eg95的跨膜区在133-155位区域,其中1-132位区域显示为膜外区,156为膜内区(见图1)。
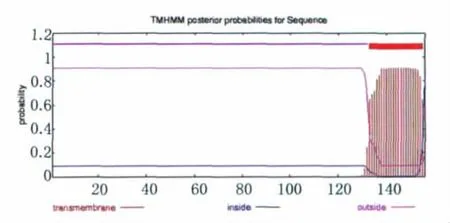
图1 TMHMM分析的Eg95跨膜结构Fig.1 Transmembrane struture of Eg95using TMHMM analysis
2.1.2Eg95抗原蛋白质二级结构预测 利用SOPMA Sever对Eg95的二级结构进行预测,发现Eg95基因中柔性结构占氨基酸总数的43.59%,其中无规则卷曲占40.38%,β转角为3.21%。α螺旋和β折叠分别占到32.05%和24.36%,各种结构在Eg95抗原中的分布情况见图2。

图2 SOPMA分析的Eg95二级结构Fig.2 Secondary structure of Eg95using SOPMA analysis
2.1.3 用DNAStar软件对Eg95B细胞表位预测DNAStar软件对Eg95的预测结合了以下有代表性的参数综合预测方案(见图3):1)以 Hopp和Woods方案预测的Eg95亲水性区域为:15-40,47-65,69-87,96-101,120-134。2)用Emini蛋白可及性方案分析,分值比较高的区域为:24-37,79-86,94-111,121-131。3)经过 Karplus-Sohulz柔性区域的分析,Eg95可塑性较强的区域为:18-35,40-48,57-65,70-75,78-88,93-99,108-112,120-139。4)采用Jameson-Wolf方法对Eg95的抗原性指数进行预测,抗原性指数较高的表位区域为:17-38,41-48,55-67,68-90,92-99,103-113,120-133,136-138。联合以上的参数和Eg95的二级结构进行综合分析,得出Eg95有7个潜在的的B细胞抗原表位,分别为:17-38,41-48,57-67,68-90,92-99,104-113,120-133。
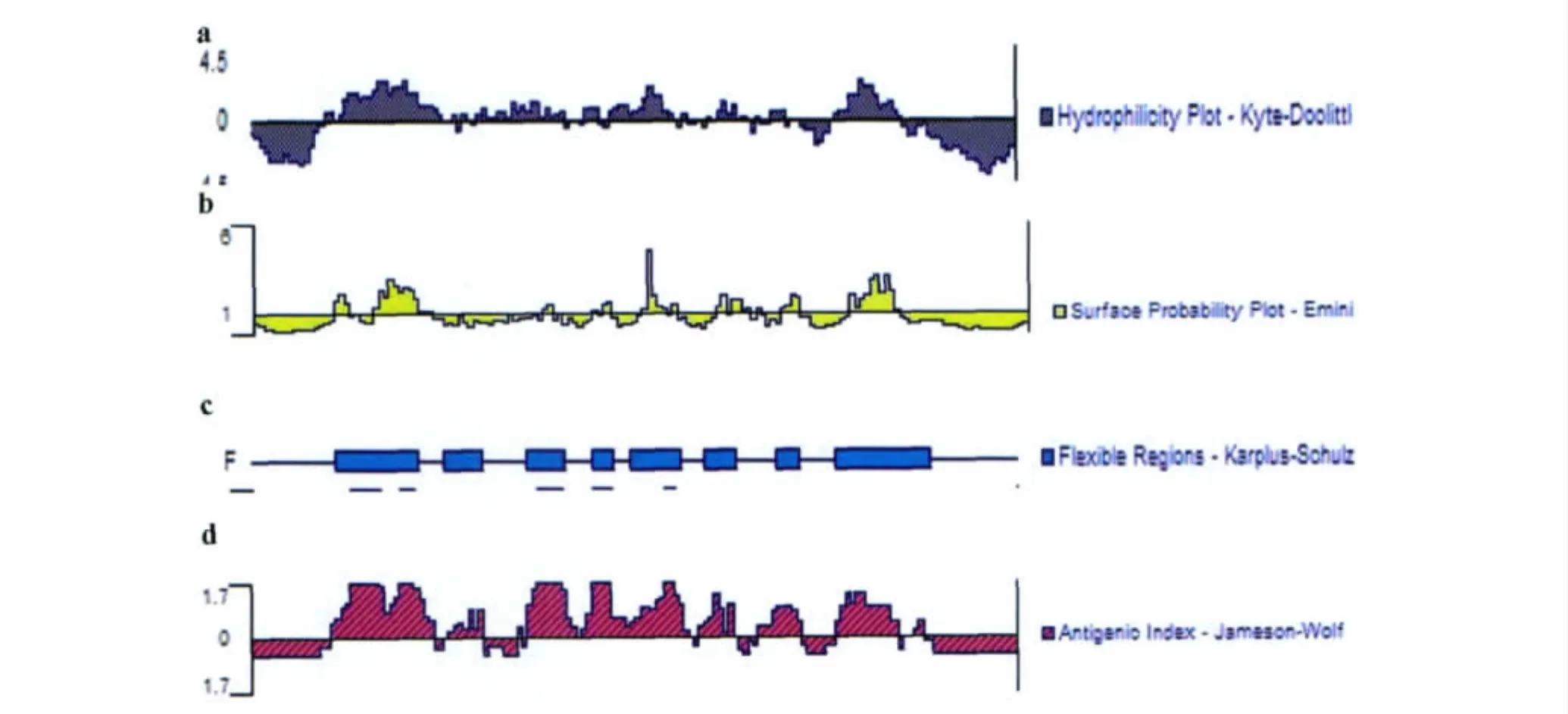
图3 DNAStar分析Eg95的各项参数Fig.3 The parameters of Eg95using DNAStar analysis
2.1.4 在线网络IEDB预测Eg95抗原B细胞表位 通过在线网络IEDB对Eg95进行表位预测(见图4):1)Chou& Fasmanβ转角的预测,分值较高的区域为:19-49,56-65,68-78,82-91,93-100,121-140。2)对Eg95进行Emini表面可及性的分析,分值较高的区域为:16-22,23-37,68-73,78-88,92-103,107-111,120-130。3)在Karplus &Schulz柔韧性区域的预测中,Eg95分值高的区域为:18-37,40-47,57-65,70-75,78-89,92-100,109-113,120-139。4)通过Kolaskar &Tongaonkar抗原性指数预测,可能存在的表位区域为:4-22,39-48,50-59,65-80,86-94,101-121。5)Parker的亲水性预测显示Eg95的高亲水性区域为:16-34,40-47,55-65,68-76,78-90,103-112,119-140。6)通过 Bepipred预测抗原表位,Eg95存在多个Bepipred线性抗原表位区域为:19-33,41-44,69-76,82-86,120-134。综合以上参数分析,得出Eg95的潜在抗原表位区域也为 7个,分别是:16-37,41-47,50-65,68-90,92-100,103-112,120-139。
通过综合以上两个分析软件的结果,将两个软件预测为表位的相同区域确定为最终Eg95的B细胞抗原表位区域,分别为:16-38,41-48,50-67,68-90,92-100,103-113,120-132。
2.2 T细胞表位预测结果
2.2.1 SYFPEITHI预测Eg95抗原T细胞表位
在进行T细胞表位的预测后,选取了分值高的13个T细胞抗原表位,预测的结果见表1。
2.2.2 在线网络预测Eg95抗原T细胞表位 为了提高预测的准确性,同时用在线预测工具(http://bimas.dcrt.nih.gov/molbio/hla_bind/)对Eg95的T细胞表位进行预测,结果见表2。
综合两个在线软件的预测,确定了在两种方法中分值都比较高的七个表位为Eg95的T细胞表位区域,分别是:6-14,14-22,48-56,4-12,98-106,142-150,146-154。
3 讨 论
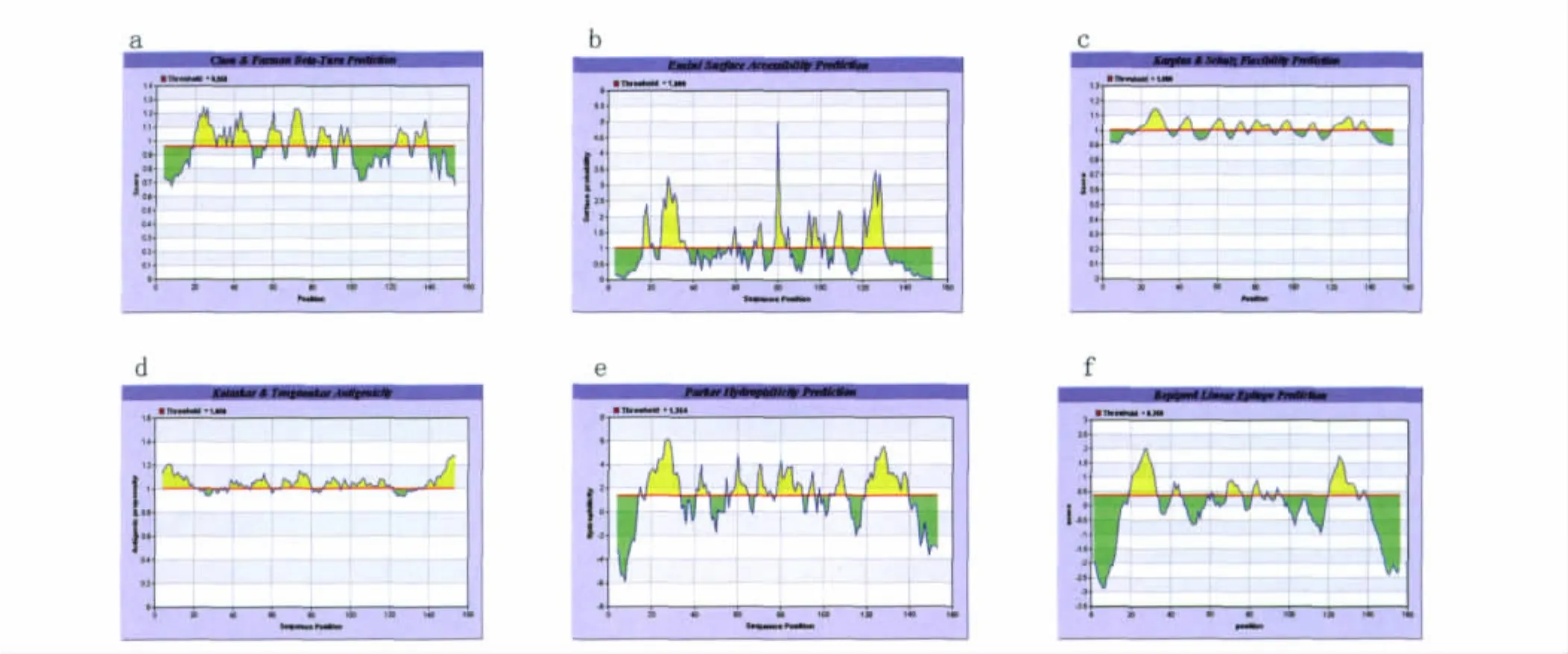
图4 IEDB分析的Eg95的B细胞抗原表位Fig.4 The parameters of Eg95using IEDB analysis
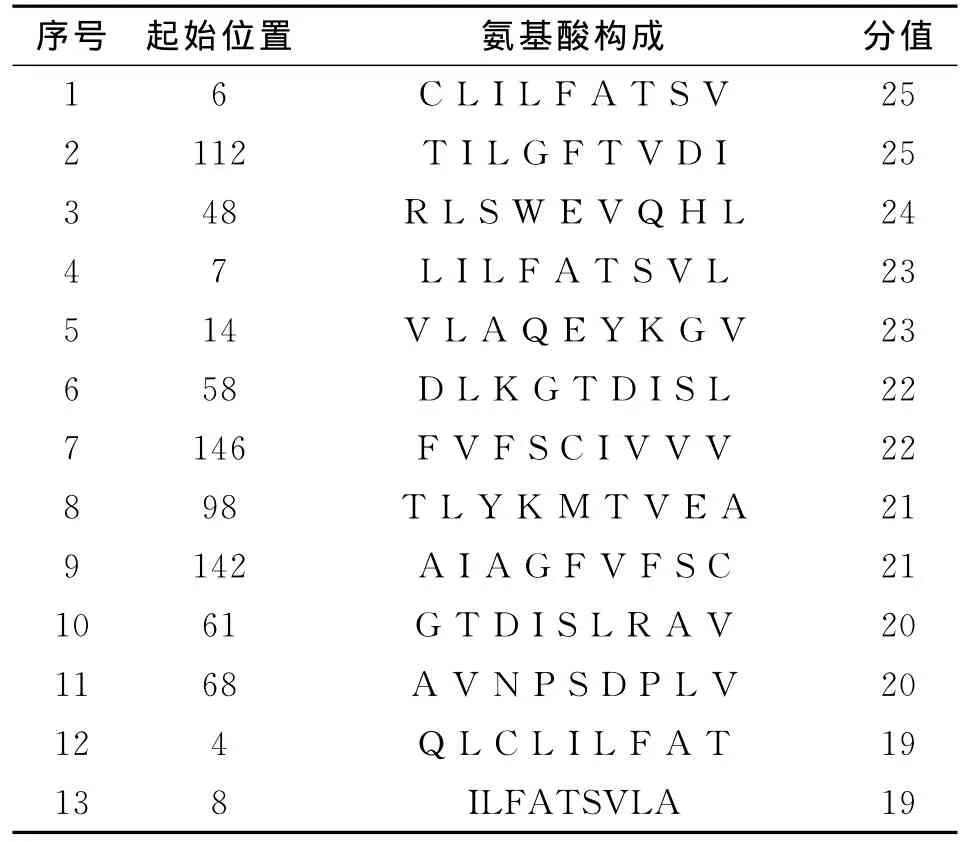
表1 SYFPEITHI预测的MHCⅠ类HLAA 0201限制性T细胞表位Table 1 HLA-A*0201nonamers T cell epitope using SYFPEITHI
寄生虫病仍是发展中国家亟待解决的公共卫生问题,研制抗寄生虫病疫苗是人们关注的热点和难点问题。包虫病严重影响我国西部牧区的经济发展和人民的身体健康,而且新的感染和发生区域也在不断扩大,因此通过免疫预防途径控制和消灭包虫病将是行之有效的方法。20世纪80年代Strohmaier[11]等发现口蹄疫病毒(FMDV)的 146-154及200-213氨基酸肽段含有免疫性位点,从而找到了一种新型的疫苗,即表位疫苗。表位(epitope)是抗原分子中决定抗原特异性的特殊化学基团,又称抗原决定簇(antigenic determinant),它是T细胞抗原受体(Tcell receptor,TCR)和B细胞抗原受体(B cell receptor,BCR)及抗体特异性结合的基本单位,分为T细胞表位和B细胞表位。
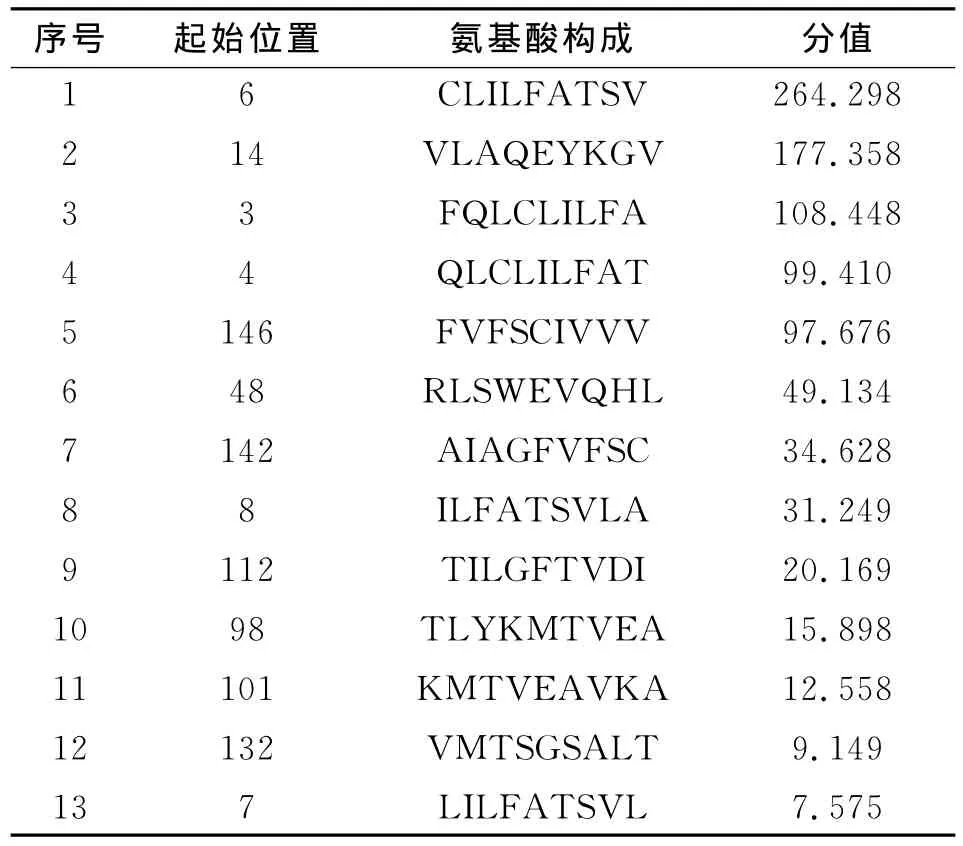
表2 在线网络预测的MHCⅠ类HLAA 0201限制性T细胞表位Table 2 HLA-A*0201nonamers T cell epitope using Internet
分子生物学和免疫学研究快速向前发展,如何利用细胞表位预测新方法,高效快速地获得表位的信息,已经成为抗包虫病疫苗研究中的重要内容。传统的筛选方法,包括噬菌体肽库,核磁共振技术,质谱技术,SPR等多种手段都需要做大量的工作,花费大量的经费,但是预测的准确性却有限,这让人们感到困扰。随着计算机知识的普及和网络技术的发展,预测表位的参数计算变得容易操作。以往表位预测多采用单参数预测,有其局限性,随着生物信息网络技术的发展和数据库的扩展,多参数多方法的综合预测已成为当前表位预测的主流,可显著提高预测的准确性[12]。李晋涛[13]运用多种方法对Eppin的二级结构和表面特性,如亲水性、理化性质、可及性、免疫原性和可塑性等方面进行分析,得出其存在多个潜在的抗原表位。经过体外实验证明,所预测抗原表位区域基本能与免疫抗血清发生抗原特异性反应。李章球[14]应用SYFPEITHI等7种软件对人、鼠源血小板糖蛋白GPⅡb/Ⅲa抗体蛋白进行 HLA-A*0201、HLA-A*1101和 HLA-A*2401限制性表位预测,发现了GPⅡb/Ⅲa抗体蛋白的T细胞表位。
本研究利用生物信息网络技术对Eg95抗原的B细胞表位预测,发现其跨膜结构中有一个跨膜区,一般比较稳定,不容易发生氨基酸的变异和表位形成。抗原的柔性区域主要集中在膜外区,本研究氨基酸序列也证实了这一结果。用TMHMM对Eg95抗原的跨膜序列进行分析,膜外区占了大部分的区域,说明这些区域产生抗原表位的可能性也比较大。用SOPMA Sever进行预测,发现Eg95氨基酸序列中柔性结构占氨基酸总数的43.59%,1-132区域是抗原表位富集的区域,表明Eg95的抗原表位也集中在该区域内。
在二级结构的预测中,蛋白质二级结构预测方案认为蛋白质二级结构分析与蛋白质表位的分布关系密切,无规则卷曲和β转角主要位于蛋白质的表面,结构突出,有利于与抗体嵌合,成为抗原表位的可能性也较大[15]。本研究用SOPMA Sever对Eg95进一步分析发现其无规则卷曲占40.38%,β转角为3.21%,表明在Eg95的这些区域中,抗原表位将容易产生并发挥特异性反应。
用抗原表位预测的经典方案对Eg95进行多参数的预测,(1)Hopp和 Woods为代表的蛋白质的亲水性方案(hydrophilicity),此方案认为蛋白质抗原各氨基酸残基可分为亲水残基和疏水残基两类。在机体内,疏水性残基一般埋在蛋白内部,而亲水性残基位于表面,因此蛋白的亲水部位与蛋白抗原表位有密切的联系[16]。(2)Emini蛋白可及性方案(accessibility),指蛋白质抗原中氨基酸残基被溶剂分子接触的可能性,它反映了蛋白质抗原内、外各层残基的分布情况[17]。(3)Karplus-Sohulz柔性区域预测方案(flexibility)认为蛋白抗原构象不是刚性不变的,其多肽链骨架有一定程度的活动性,也就是蛋白质的柔性区域。活动性强的氨基酸残基即可塑性大的位点,易形成抗原表位[18]。(4)Jameson-Wolf的抗原性指数方案(Antigenic index)通过对20个已研究得很透的蛋白质的69个连续位点的606个氨基酸统计分析,用各氨基酸残基在已知B细胞表位中出现的百分率与其通常在蛋白质中出现的百分率比值的对数建立了抗原性刻度,并以此计算蛋白中各亚序列的抗原性[19]。(5)β转角的预测方案认为β转角是蛋白质二级结构的基本类型之一,由4个氨基酸残基组成,其中第一个残基的CO基团和第四个残基的NH基团之间形成氢键,使多肽链的方向发生U形改变。正因为这样的改变,使表位容易在β转角存在的区域形成。这些方案的综合运用,将会提高B细胞表位预测的准确性。
在T细胞的预测中,MHCI类抗原表位预测的准确率较高,可达90%左右,MHCI类表位是由9个氨基酸残基构成的短肽,汉族人种最常见的是HLAA 0201限制的抗原表位[20]。因此本研究对Eg95进行了HLAA 0201MHCI类9肽限制性表位预测。研究者们发现根据蛋白质氨基酸的分布,运用各种软件对氨基酸的构成和分布进行计算得出一个T细胞表位分值,以表格的形式显示[21]。本研究将两种预测法分值排在前13位的表位进行综合分析,发现两种不同方法的结果有许多重复之处,说明综合两种方法的结果能够提高预测方法的准确性。联合各参数预测Eg95含有7个潜在的B细胞表位分别是16-38,41-48,50-67,68-90,92-100,103-113,120-132,7个分值较高的 T细胞表位是6-14,14-22,48-56,4-12,98-106,142-150,146-154。
本研究通过生物信息学预测的这些表位区域有利于下一步研究和筛选Eg95抗原B细胞和T细胞优势抗原表位,构建优势抗原表位疫苗,为获得高效的保护性疫苗和预防包虫病的流行奠定实验基础。
[1]刘春燕,马秀敏,丁剑冰,等.我国棘球绦虫感染的不同宿主状况[J].中国人兽共患病学报,2009,25(6):586-588.
[2]王立英,伍卫平,朱雪花.2004-2008年全国包虫病疫情分析[J].中国人兽共患病学报,2010,26(7):699-701.
[3]鲍佳春,袁凤来,陆伟国.包虫病治疗进展[J].中国血吸虫病防治杂志,2010,22(2):197-199.
[4]Ligtowlers MW,Lawrence SB,Gauci CG,et al.Vaccination against hydatidosis using a defined recombinant antigen[J].Parasite Immunol,1996,18(9):457-462.
[5]贾海英,马秀敏,丁剑冰,等.细粒棘球蚴Eg95重组蛋白表达及其血清学反应的研究[J].中国人兽共患病学报,2009,25(4):344-347.
[6]Ben YT,Arnon R.Towards an epitope based human vaccine for influenza[J].Hum Vaccin,2005,1(3):95-101.
[7]Corradin G,Swpertini F,Verdini A.Medicinal application of long synthetic peptide technology[J].Expert Opin Biol Ther,2004,4(10):1629-1639.
[8]骆利敏,李明,夏虎,等.乙型肝炎病毒多表位抗原基因的设计、合成及表达[J].中华微生物学和免疫学杂志,2004,24(6):426-432.
[9]Wang X,Yang Q,Wang P,et al.Derp2-mutant gene vaccine inhibits airway inflammation and up-regulates Toll-like receptor 9 in an allergic asthmatic mouse model[J].Asian Pac J Allergy Immunol,2010,28(4):287-293.
[10]You L,Brusic V,Gallagher M,et al.Using Gaussian process with test rejection to detect T-Cell epitopes in pathogen genomes[J].IEEE/ACM Trans Comput Biol Bioinform,2010,7(4):741-751.
[11]Schuler MM,Nastke MD,Stevanovikc S.SYFPEITHI:database for searching and T-cell epitope prediction[J].Methods Mol Biol,2007,409:75-93.
[12]Sompuram SR,Bastas G,Vani1K,et al.Accurate identification of paraprotein antigen targets by epitope reconstruction[J].Blood,2008,111(1):302-308.
[13]李晋涛,陈正琼,梁志清,等.Eppin抗原的二级结构分析B细胞表位预测[J].第三军医大学学报,2008,30(24):2254-2257.
[14]李章球,张梅霞,胡海燕,等.GPⅡb/Ⅲa抗体的二级结构及T、B细胞表位的预测[J].生物医学工程学杂志,2010,27(5):1146-1151.
[15]Sikic K,Tomic S,Carugo O.Systematic comparison of crystal and NMR protein structures deposited in the protein data bank[J].Open Biochem J,2010,3(4):83-95.
[16]Sy SM,Chen J,Huen MS.The 53BP1-EXPAND1connection in chromatin structure regulation[J].Nucleus,2010,1(6):472-474.
[17]Liliana F',Bjarni H,Oliver K,et al.Epitope prediction algorithms for peptide-based vaccine design [J].Proc Comput Sys Bioinform,2003,33:7690-7695.
[18]Casella G,Ferrante F,Saielli G.A.A DFT study of the Karplus-type dependence of vicinal(3)J(Sn-C-X-C),X=N,O,S,in organotin(iv)compounds:application to conformationally flexible systems[J].Org Biomol Chem,2010,8(12):2711-2718.
[19]Ie SI,Thedja MD,Roni M,et al.Prediction of conformational changes by single mutation in the hepatitis B virus surface antigen(HBsAg)identified in HBsAg-negative blood donors[J].Virol J,2010,7:326-334.
[20]Yan C,Wang R,Li J,et al.HLA-A gene polymorphism defined by high-resolution sequence-based typing in 161Northern Chinese Han people[J].Gen Prot Bioinform,2003,1(4):304-309.
[21]Tong JC,Tan TW,Ranganathan S.Methods and protocols for prediction of immunogenic epitopes[J].Briefings in Bioninformatics,2007,8(2):96-108.
Bioinformatics prediction onEg95antigen epitopes ofEchinococcusgranulosus
LI Yu-jiao,WANG Jing,ZHAO Hui,JIA Hai-ying,LI Bo,MA Xiu-min,WEN Hao,DING Jian-bing
(MedicalResearchCenterandXinjiangKeyFirst,FirstAffiliatedHospital ofXinjiangMedicalUniversity,Urumqi830011,China)
The protein secondary structure was analyzed by TMHMM Sever2.0and SOPMA Sever,and B cell and T cell epitopes ofEg95were predicted by DNAStar,IEDB and SYFPEITHI.Many distinct antigenic epitopes ofEg95were identified by computation,and the possible B cell epitopes of which were in the regions of 16-38,41-48,50-67,68-90,92-100,103-113,and 120-132.The high score of T cell epitopes were in the regions of 6-14,14-22,48-56,4-12,98-106,142-150,and 146-154.These results indicated that there are 7antigen epitopes inEg95antigen determined by bioinformatics methods,which are significant in the further research ofEg95antigenic and development of dominant epitope vaccine.
Eg95;antigen epitope;bioinformatics
R383.3
A
1002-2694(2011)10-0892-05
*国家自然科学基金项目 (30901374,81060135,30560146,30860263)资助
丁剑冰,Email:djbing002@163.com
1.新疆医科大学第一附属医院新疆重点实验室,乌鲁木齐 830011;
2010-11-17;
2011-03-23

