网络药理学:认识药物及发现药物的新理念
周文霞,程肖蕊,张永祥
(军事医学科学院毒物药物研究所,北京 100850)
过去十余年来,新药候选物转化成临床有效新药的速率显著下降,而在Ⅱ和Ⅲ期临床试验中因缺乏有效性和出现非预期的毒性所导致的损耗呈现令人担忧的增长趋势,约占研发失败原因的60%[1]。2007年,英国Dundee大学药理学家Hopkins[2-3]认为,造成上述失败的主要原因并不是技术和条件方面的因素,甚至也不是科学的因素,而是哲学(或理念)的因素(may not be technological,environmental or even scientific but philosophical),因此,他率先提出并系统地阐述了网络药理学(network pharmacology)的概念[3],为从新的角度认识药物作用机制、以新的策略发现新药提供了新的思想、理论及方法,在生物医药研究领域产生了深远影响。
与传统药理学的最大区别在于,网络药理学是从系统生物学(systems biology)和生物网络平衡的角度阐释疾病的发生发展过程、从改善或恢复生物网络平衡的整体观角度认识药物与机体的相互作用并指导新药发现。网络药理学紧紧围绕系统生物学、生物网络构建和分析、连接性、冗余性和多效性等进行药物有效性、毒性、代谢特性的揭示,是建立在高通量组学数据分析、计算机虚拟计算及网络数据库检索基础上的生物信息网络构建及网络拓扑结构分析策略和技术基础上的科学思想和研究策略[2-3],代表了现代生物医药研究的全新理念和模式,使以“一个药物,一个靶标,一种疾病(one drug,one target,one disease)”为主导的传统新药研发理念产生了革命性转变,对认识药物及发现药物的理念、策略和方法具有深刻影响。
1 网络药理学的理论基础
网络药理学的产生离不开基因组学、蛋白质组学、代谢组学等组学技术以及高通量、高内涵筛选等现代新药发现技术的发展,也离不开生物信息学、系统生物学、网络生物学及计算生物学等相关学科的基础理论和研究技术的发展,可以说,网络药理学是生命科学发展到系统生物学阶段的产物,是生物学、医学、计算科学、生物信息学等多学科基本理论和研究技术综合集成的结果。
1.1 系统生物学
美国科学院院士莱诺伊·胡德(Leory Hood)最早提出了系统生物学的概念和研究体系,按照他的定义,系统生物学就是一门研究生物系统中所有组成成分的构成,以及在特定条件下这些组分间相互关系的学科[4]。系统生物学认为生物体是一个包含多个个体和多个层次相互作用的复杂系统,系统生物学研究就是要在细胞、组织、器官和整体水平对结构和功能各异的所有组成成分(如基因、蛋白、mRNA、小分子代谢物等)的相互作用关系,以及在特定条件下(如遗传、环境因素变化时)这些组成成分间相互作用关系的变化进行研究,同时还要通过生物信息学来定量阐明和预测生物功能、表型和行为。生物系统具有涌现性(emergence)、复杂性(complexity)和鲁棒性(robustness)的特点[5-7]。
生物系统由若干个子系统构成,每个子系统中又涉及到不同种类的生物大分子,研究时需着眼于将这些所有的分子、子系统放入整个生物系统的大环境中去考察其所有的相互关系;系统生物学不是用单一的方法同时研究成千上万个基因或蛋白质的“水平”研究,而是要将“水平”研究和“垂直”研究统一起来,成为一种“高维”的研究,既要同时考虑多个层次、多种类型的生物信息,还要考虑时间因素;“部分之和大于整体”是系统科学的核心思想,系统特性是由于不同组成部分、不同层次间相互作用而“涌现”的新性质,如果只是针对组成部分或低层次的分析并不能真正准确地预测高层次的行为。
系统生物学的发展带给药理学工作者的启示是“分子到药物”的简单模式将逐渐被“生物学到药物(biology to drug)”的模式所取代,药物靶点将从单一分子扩展至分子组合、某个信号转导通路甚至几个通路的组合。对新药研发工作者来说,则认识到那些“dirty drug(不纯的药物)”尽管对单个靶点的亲和性和选择性可能都不高,但因为可作用于疾病网络的多个靶点,或对各靶点的作用可以产生协同效应,使其总效应大于各单效应之和,从而达到比单靶点药物更佳的治疗效果[8-9]。
1.2 网络生物学和生物网络
网络生物学(network biology)的概念是 Barabasi和Oltvai[10]于2004年提出的,他们提出可以利用网络学科的语言,将复杂生物系统的相互作用抽象表达为网络,建立网络模型,进而通过研究代表生物体的复杂网络的成分关系和特性来揭示生物体的原理和本质。
采用数学领域图论的研究手段,借助网络的概念、属性和复杂网络的研究方法,将生物体中各种生物分子及其相互作用加以抽象(将构成系统内的各个组分如生物分子抽象成节点,它们之间的联系或相互作用关系如分子间的相互作用描绘成边,由节点和边构成的图就是网络),组成一个包含多个体、多层次相互作用的复杂网络,称为生物网络(biological network)[11]。度 分 布 (degree distribution)、小 世 界 效 应(small-world effects)、聚集性(clusterness)和鲁棒性(robustness,也称稳健性或弹性)等是描述生物网络性质的基本参数(表 1)[12-13]。
从网络生物学角度来讲,当生物网络为稳态和平衡态时,机体处于健康状态。网络平衡被扰乱或破坏时即可导致病理或疾病状态。药物对疾病的治疗作用其本质在于重建生物网络的平衡或减轻平衡被破坏的程度[10,14]。
1.3 生物网络平衡理论
勒夏特列原理(Le Chatelier's principle)又称平衡移动原理,是一个定性预测化学平衡点的原理。该原理认为化学平衡是动态平衡,如果改变影响平衡的一个因素,平衡就向能够减弱这种改变的方向移动,以抗衡该改变。该原理体现在生物网络上就是生物网络具有鲁棒性,表现在拓扑结构的鲁棒性、功能的鲁棒性和动力学的鲁棒性,这是由于生物分子的功能冗余性和替代补偿性信号通路的存在造成的[15]。生物网络的鲁棒性反映了机体或网络对错误和攻击的容忍能力。
生物网络的勒夏列特原理,即如果一个系统(生物网络)的平衡(健康状态)经历了一个改变(疾病状态),那么有效药物的作用将使平衡向能够减弱这种改变的方向移动。网络药理学是基于网络生物学和生物网络平衡理论提出的,它为新药发现提供了新的思维体系(a new framework)和研究策略,同样也为研究和理解药物作用方式(作用机制)提供了新的思路和途径[1,3]。
生物网络保持平衡的能力或鲁棒性带给药理学和新药研发工作者深刻的启示就是要干扰“致病网络(disease-causing network)”,寻求“扰动(perturbations)”而不是寻找“致病基因(disease-causing gene)”,可能才是药物作用机制研究和新药发现的有效途径,这也正是网络药理学的核心[10,16]。
2 网络药理学在药理学研究和新药发现中的作用
建立在系统生物学和网络生物学之上的网络药理学能够在系统的分子水平上更好地理解细胞以及器官的行为对功能表型的影响,推动对药物作用机制的重新认识,为药物重定位(drug repositioning)提供理论依据和技术支撑,为临床合理用药及多药组合使用提供科学依据,并系统的预测和解释药物相互作用、优化药物的使用,预测及发现影响药物有效性和安全性的因素,加速药物靶点的确认以及发现生物标志物。
2.1 对认识药物作用机制及指导临床合理用药的意义
过去几十年间,药理学科将研究重点主要放在分子药理学研究方面,对体内大量单个分子靶点及其与许多疾病和药物的关系有了较为深入地认识,但从系统生物学角度深入研究药物作用及作用机制的研究当时还非常有限。
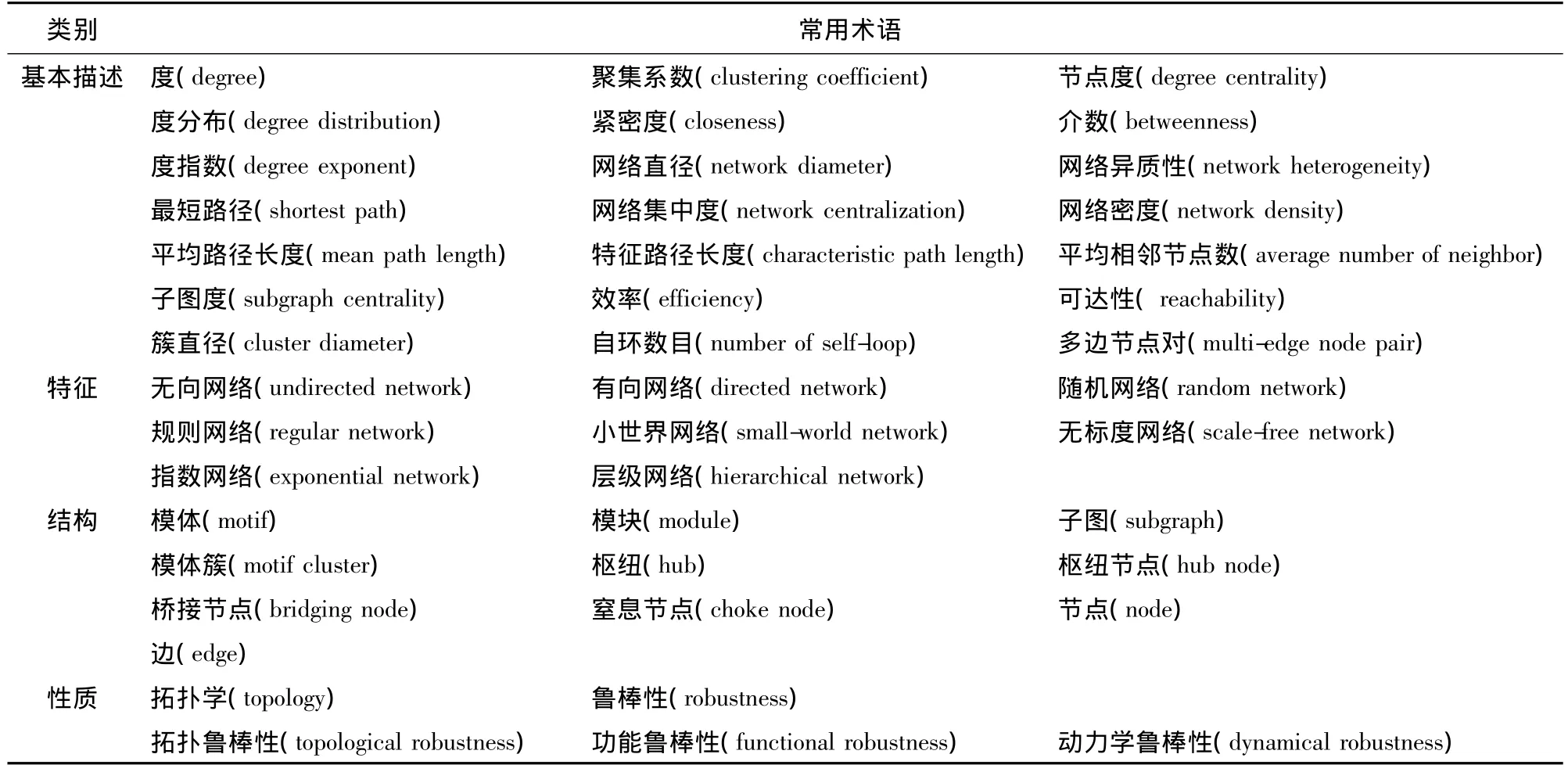
表1 网络生物学常用科学术语
网络药理学的重要任务之一就是从整体网络的角度认识药物的作用机制,指导临床合理用药。已如前述,从系统的角度来看,疾病的本质在于生物网络失平衡,药物治疗疾病的本质在于重建生物网络的平衡或减轻平衡被破坏的程度[14,17]。因此,从整体生物网络稳态的角度来讲,理解单个生物分子在生物体系中的生物学地位和动力学过程要比理解单个生物分子的具体生物功能更为重要。同样,对于药理学和新药研发工作者来说,理解药物在生物体系中的地位和动力学过程要比理解药物在个别靶点或组合靶点上的有效性更为重要。寻找那些作用于生物网络中多个构件(而不是网络中的单一构件)、恢复或改善生物网络平衡的药物才是突破目前新药尤其是治疗多因素复杂性疾病药物研发瓶颈的出路。
生物网络具有鲁棒性,即对于内外干扰的承受能力。鲁棒性关系到生物体的生存,是生物体承受内外环境变化的一种能力。然而,这种鲁棒性在多个扰动因子的作用下则变得非常脆弱。如对酵母的基因敲除与化学干预、环境影响的合成致死(synthetic lethality)实验结果表明,酵母中只有34%的单个基因敲除可引起死亡或疾病,但是单基因敲除叠加一个小分子化合物或一个环境因素干预,则可对63%的单基因敲除产生增强效应,如死亡或疾病;若单基因敲除、一个小分子化合物、一个环境因素三者同时干预,则可对97%的基因敲除产生预期的缺陷,如死亡或疾病。该结果表明,尽管大多数基因在任何一种环境下可能是冗余的,但是通过遗传扰动结合化学干预的各种条件谱实验,说明这些基因似乎很少是冗余的[18]。这充分体现了生物网络鲁棒性的容错和抗攻击能力,也从反面说明了具有多向药理学(或多靶标)性质的药物在临床上将更为有效。
在临床药物治疗学中,基于具有多向药理学性质的药物对多个靶点的低亲和力相互作用,或平衡调节与疾病相关且具有内在联系的多个靶点,或许将产生更好的疗效和更小的副作用,从而达到最佳治疗效果。而且药物对各靶点的作用可能产生协同(协同有效或协同毒性),使总效应大于各单效应之和[19]。在给定剂量(配伍比例)的药物在联用时,其联用的顺序可对网络产生不同的扰动,并可能对功效产生巨大的作用[20-21]。因此,在临床上,无论是多药联用,使用同一药物载体中含多组分配伍药物还是使用基于设计的可选择性作用于多靶点的单一药物,除了关注这些药物的协同作用所产生的显著疗效外,不可忽视其同样的对表型的改变作用(毒性作用)。
在目前临床药物治疗中,抗药性(如肿瘤药、抗菌药)的产生已然成为常态。这是因为目前所用药物多为基于单靶点设计的药物,而靶点蛋白的一个单独的氨基酸突变通常就足以导致药物抗性,因此,许多有效的抗生素通常是通过同时靶向多个蛋白而不是单个蛋白来发挥作用的[22]。如β-内酰胺类(β-lactams)的抗菌作用依赖于多青霉素结合蛋白(multiple penicillin-binding proteins,PBPs)中的至少2个来发挥抗菌作用的,这主要基于多PBPs缺失而对表型无变化的事实[23]。氟喹诺酮(fluoroquinolone)抗生素是蛋白ParC和GyrA蛋白的双靶标抑制剂[24]。而基于网络药理学的药物,可以同时调节疾病网络系统中的多个环节,不易产生抗药性。
2.2 对新药发现的影响
网络药理学与传统药理学一样,其重要任务之一就是新药的发现和新药作用评价,并开发新药。但网络药理学却为新药研发提供了全新的视角,首先,药物靶点并不是随机分布在网络中,而是具有靶点分布的特点和一定的规律,因此,可通过网络药理学的研究来寻找、优化或确认靶点;为多靶点药物设计和优化提供重要信息;预测和分析药物毒性作用产生的可能性。
2.2.1 加速治疗靶点的发现和确认
传统的药物靶点发现研究是一种“垂直式”的研究,即以个别分子为研究对象,采用多种手段研究其生物学性质。这种将分子靶点孤立起来的研究不但耗时长、投资大,而且很难对靶点的功能建立全面的认识,以该模式开发出的新药进入临床后通常会因药动学或毒性作用而终止。而以整体性和系统性为特征的网络药理学则具有强大的预测能力,而且着眼于靶点分子或靶点分子组合或子网组合在整体生物网络中的定位和生物动力学以及调节动力学,并注重研究其扰动后所“涌现”的表型。因此,对于药物治疗靶点的发现和确认会更加迅速,以其为模式进行新药的研发其临床成功率会更高。
网络药理学研究结果告诉我们,在靶点发现中需考虑以下基本事实:① 多靶标的成药能力在根本上优于单靶标。②对于生物体,靶标不必要是唯一的,或在宿主中是缺失的。尽管许多基本的看家酶在宿主和传染原之间是共同的,但是药物在宿主和传染原之间的选择性即在结合位点水平上是不相同的。③组合靶标(作用于多靶点)的作用是致死性,但在单个基因敲除研究中却被忽略为非必要的靶标(单靶点敲除对表型无作用)。④被预测为有可能和相同化合物结合的靶标群优于单个可药性靶标[2]。
在药物靶点发现中,各种不同的网络被构建和不同的模型算法被开发以用于建立连接、分析拓扑结构和定量优先位次,如使用多种药物-靶点数据库如 Drugbank,SuperTarget,TDR,TTD,Matador,Pdtd和STITCH等,利用多种靶点预测工 具 如 CellDesigner, COPASI, iPATH, SABIO-RK,SYCAMORE和Tide等,在构建均相的(homogenous)和异质的(heterogeneous)蛋白质相互作用网络、信号转导网络、代谢网络、基因调控网络、miRNA网络[25]等上进行靶点的预测和发现[26]。如使用与疾病相关的生理通路联合其已知的药物和药物靶点构建网络,预测生物药过程-药物相关性(biological process-drug relationships),所建立的模型预测的2078个相关性中有401(18.1%)个已经进入临床试验[27]。
聚腺苷二磷酸核糖聚合酶-1〔poly(ADPribose)-polymerase-1,PARP〕抑制剂对有BRAC1或BRAC2缺陷的细胞具有高致死性,因而PARP抑制剂作为BRAC1和BRAC2突变携带者肿瘤患者的有效治疗药物而进入临床。PARP抑制剂合成致死siRNA筛选结果表明,许多激酶的沉默(或敲低)对PARP抑制剂具有强烈的敏感性,包括 CDK5,MAPK12,PLK3,PNKP,STK22c和STK36。尤其是CDK5对于非神经元细胞的DNA损伤反应、细胞周期的内-S(intra-S)和G2/M期检查点(checkpoints)是必需的[28]。分析乳腺癌基因表达数据揭示了4个基因:COX2,MMP1,MMP2和表皮调节素(epiregulin)对于肺癌迁移是必需的[29-30]。在小鼠模型中,这4个基因的遗传和药理学抑制皆可导致肿瘤迁移进程的停止[29]。
2.2.2 指导新药研发
网络药理学的发展使得新药研发从指导思想上发生了根本的改变,这主要归结于以下4个事实:① 针对单一靶点的高选择性药物似乎呈现出更低的临床有效性和成功率[2];②多靶点药物在临床上已经成功,特别是是双重或多重激酶抑制剂[31],如2005年批准的索拉非尼(sorafenib)、2006年批准的达沙替尼(dasatinib)、2007年批准的舒尼替尼(sunitinib)和拉帕替尼(lapatinib);③ 许多被批准的药物其选择性比最初预想的要差[32],如肿瘤治疗药物(伊马替尼imatinib)和舒尼替尼可与多个激酶的结合[9];④生物网络的鲁棒性说明药物-靶标网络具有无标度性质[14,33],则意味着单个蛋白具有功能障碍的补偿机制,抑制单个通常在治疗上是无效的[2]。
在具体的新药研发实践中,已有研究表明,在基于多靶点设计合成具有多向药理学性质的药物时,片段或分子量相对小的化合物成药性更好[33]。药-靶相互作用呈现化合物分子结构和靶蛋白氨基酸序列成对碎片相互嵌入,因此,建立分子的子结构和序列片段对(subgraph-subsequence pairs)进行药物的研发就是可行的。而且已批准药物的75%药物-靶点相互作用符合显著的亚结构对,而且这些亚结构对的聚类具有高度排他性,提示着每个聚类有其对应的独特的多向药理学类型。同时,说明基于序列片段的药物设计方法在网络药理学指导下的新药研发实践中一样具有使用价值[34]。
传统的虚拟筛选方法关注药物分子与靶点结合的亲和力,而不是药物分子对疾病系统的表型数据,这远离了复杂性疾病的本质特性。基于网络药理学的药物评价则会给出不同靶点在生物途径中的权重,并能给出多个靶点部分抑制后的显著有效性证据。如将多靶点对接(multi-target docking)研究的亲和力预测与生物网络有效性分析进行整合,来评价化合物的抗凝血活性。人类凝血级联网络效率的计算结果表明,在人类凝血级联系统中,因子Ⅹa和凝血酶是两个最脆弱的酶,而复合体ⅨA∶ⅧA所介导的催化反应和复合体ⅧA∶ⅨA的形成是两个最脆弱的生物物质。实验数据和网络有效性降低的相关性(r=0.671)表明基于网络的多靶点药物评价方式是有效的[35]。
目前,基于网络药理学进行新药研发中,药物联合应用和多组分药物使用的策略需要研究药物彼此间发生相互作用而产生的不良反应,证明所用药物组分无论是在单独应用还是联合应用都是安全的。基于靶向设计合成的有选择性的非选择性化合物(selectively non-selective drugs)的策略需要找到选择性地作用于多个靶点的药效团且化合物具有平衡生物网络系统的活性和所能接受的药动学特征。
3 网络药理学的研究方法
网络药理学目前的研究思路一是根据公共数据和公开发表的已有数据,建立特定疾病及其防治药物靶点预测网络模型,预测所研究药物的作用靶点,进而构建所研究药物-靶点-疾病网络,解析所研究药物的网络药理学机制,并通过相应的实验进行机制的验证。二是利用组学技术以及高内涵高通量技术,观察药物对模型(细胞和动物)的作用或模型对药物的作用,针对所产生的大量数据,采用生物信息学的手段分析和构建药物-靶点-疾病网络,进而解析在研药物的网络药理学机制。当然,这两个思路也可并行发展、综合分析。其所涉及的主要研究技术如下述。
3.1 网络构建以及可视化方法
构建疾病网络、疾病-疾病网络、疾病-药物网络、药物-药物网络、药物-靶点网络、靶点-疾病网络、药物-靶点-疾病网络等,用于药物的快速重新定位和药物靶点或作用途径的确证以及药物新的临床适应证预测等。
目前常用的网络构建以及可视化工具主要有三类:使用直接编程语言或工具,例如Java,C,Perl等;使用半编程性质的脚本性软件,例如Matlab,R project等;使用专门用于构建网络的工具,主要有 Cytoscape,GUESS,Pajek,Osprey,MultiNet,UCINET,NetMiner,NetworkX 等。
3.2 网络分析方法
通过网络拓扑结构及网络平衡或鲁棒性分析,可以客观、准确地找出具有特定生物功能的关键节点、亚结构,明确药物干预的主要靶点、次要靶点和协同靶点,理解网络达到平衡状态涉及的亚网络间关系,为预测干预药物提供理论计算的参考。
网络分析通常包括:网络拓扑学信息计算(如Cytoscape中的Network Analysis)、随机网络生成和比较(如Cluster)、网络分层和聚类(如AllegroMCODE)。网络比对方法通常包括:基于图模型的启发式搜索方法(如MaWISH,Network-BLAST,Græmlin)、基于目标函数的约束优化方法(如IsoRank,MNAligner)、基于分治策略的模块化比对方法(如Match-and-Split,BiNA,DivAfull)。
3.3 预测网络模型建立方法
通过整合网络搜索算法、数据标准化算法和生物活性预测算法以及相关软件等,建立网络预测模型,能够迅速且较稳定地筛选出具有较强的结构相关性及功能相关性的靶点或靶点组合或子网,进行网络药理学的研究。
常见的有最小二乘法(least square)、偏最小二乘法(partial least square)、布尔网络(Boolean network)模型、线性组合模型(linear modeling)、加权矩阵模型(weighted matrix models)、贝叶斯网络(Bayesian network)模型、径向基神经网络(RBFNetwork)、随机森林法(RandomForest)、随机游走算法(Random walk)、PRINCE算法以及微分方程等。
3.4 网络药理学实验方法
网络的基本特性就是当网络被扰动时,蛋白就会“涌现”一定的功能。因此,网络药理学的具体实验方法通常通过调节网络的节点来进行,如缺失节点、调节节点的浓度。缺失是完全去除了一个网络节点的所有相互作用和功能。调节则为衰减或增强。衰减(如拮抗剂)仅仅是部分去除了一些相互作用(比如降低代谢产物的浓度)而不是移除所调节节点的整体的连接(比如蛋白-蛋白相互作用)。相反,增强(如激动剂)可加强网络中的特异连接。这样基于连接而不是节点的研究思路与药物作用的具体情况更为接近。但是研究表明,一个网络中仅仅大约有15%的蛋白节点可能是由小分子药物化学可控的[36],因此,除了工具药筛选方法之外,药物混合物、化学工具和RNAi/过表达等联合筛选方法非常重要。同时,基于具有系统性和整体性的系统生物学和网络生物学之上的网络药理学,其根本的要求就是要搞清楚节点和节点组合、通路和通路组合或子网和子网组合的可药性(druggability)和多向药理学相互作用在整体的生物学网络中的定位、作用动力学(dynamics)和生物动力学(kinetics),由此也要求网络药理学研究的方法应该是高通量、高内涵的技术。
网络药理学实验方法通常有组学(基因组学、蛋白质组学、代谢组学、相互作用组等)技术、生物芯片(核酸、蛋白和细胞芯片等)技术、高通量酵母双杂交技术、高通量/高内涵基因过表达技术、基于siRNA和miRNA文库的高通量反义核酸/RNAi技术、报告基因检测技术、转基因和基因敲除技术、高通量/高内涵细胞表型筛选技术,如进行基因过表达,进行信号转导通路筛选、多通路协同筛选、细胞表型变化筛选等,进行基因敲减和抗体封闭以及药物调节等。
4 网络药理学目前所面临的挑战
网络药理学作为药理学的一门新兴学科,是建立在基于海量组学数据的系统生物学和网络生物学之上的,其发展面临的挑战除了来自于基础医学理论知识、系统生物学和网络生物学理论知识的限制以外,主要还有以下几点。
(1)靶点或靶点组合的鉴定:一方面,生物网络分析和比对研究是生物学、数学、信息学和计算机科学等学科综合交叉领域的研究问题,另一方面,由于生物网络的无尺度特性,使得理想的网络模型算法、结果的评价与比较标准难以界定。而且还有来自于具体实验技术方面的限制,使得通过扰动其即可导致理想的治疗功效的节点或节点组合的鉴定困难重重。
(2)多向药理学药物的发现:尽管多靶标策略高于单靶标策略的生物学原理是不容置疑的,但优化多活性药效团的同时需要平衡成药性所具备的特性以及控制不想要的脱靶效应依然是一项困难的工作。药物分子均是以特定的立体结构在生物体内与立体的靶点蛋白产生相互作用,这种药物与靶点间基本的分子作用机制也使得基于多靶点抑或是子网的具有多向药理学性质的药物发现举步维艰。
(3)数据库数据的扩充:网络药理学依据现有数据库和已有实验数据,进行网络的建模,进行药物药理学性质的预测和评价。尽管目前的数据能够反映一些生物体内真实的细胞网络的特点,但其肯定不能反映全部,或者其中一些被反映出的特点可能并非是真实的,因为大部分原始实验数据、图谱信息来源于不同的实验条件,相关性差,数据的假阳性率和假阴性率都较高;已经评价的小分子化合物及其作用靶点数量均有限;公用数据库的信息有限,且存在不同程度的局限性。因此,供作网络分析的数据库数据的完整性,已成为制约这类网络分析与客观真实相接近的关键。对数据库加以完善,才能完成均一、无偏见的网络的构筑,这是确保网络分析可靠、客观的重要手段。
5 展望
作为药理学新兴分支学科的网络药理学,其揭示的是比现代的分子药理学更复杂的药理学规律,基于网络的药物研发在于创制新型药物-网络调节药,代表了现代生物医药研究的哲学理念与研究模式。但由于网络药理学严重受限于现有实验数据和数据库数据的完整性,基于现在已有知识的网络还不能提供网络对意外疾病(或未知疾病)和新(未知)干预方式的反应。因而,这些分析不能预测那些反直觉或反常的系统应答。而且尽管目前网络药理学研究还远没有达到系统地理解生命现象的要求,生物网络在深度、广度和生物学意义等方面随时在发生变化。到目前为止,还没有建立一种生物完全的蛋白质相互作用网络。但随着疾病和药物相关数据的不断积累,合成生物学(synthetic biology)的进展,以及网络计算方法和计算软件的发展与完善,网络药理学也必将得到快速发展,为新药研发提供更多更有意义的信息。这种基于网络药理学的药物研发新模式,必将对复杂疾病的诊治带来重大突破。
[1]Kola I,Landis J.Can the pharmaceutical industry reduce attrition rates[J]?Nat Rev Drug Discov,2004,3(8):711-715.
[2]Hopkins AL.Network pharmacology:the next paradigm in drug discovery[J].Nat Chem Biol,2008,4(11):682-690.
[3]Hopkins AL.Network pharmacology[J].Nat Biotechnol,2007,25(10):1110-1111.
[4]Hood L.A personal view of molecular technology and how it has changed biology[J].J Proteome Res,2002,1(5):399-409.
[5]Hood L.Systems biology:new opportunities arising from genomics,proteomics and beyond[J].Exp Hematol,1998,26:681.
[6]Ideker T,Galitski T,Hood L.A new approach to decoding life:systems biology[J].Annu Rev Genomics Hum Genet,2001,2:343-372.
[7]Hood L.Systems biology:integrating technology,biology,and computation[J].Mech Ageing Dev,2003,124(1):9-16.
[8]Schrattenholz A,Groebe K,Soskic V.Systems biology approaches and tools for analysis of interactomes and multi-target drugs[J].Methods Mol Biol,2010,662:29-58.
[9]Frantz S.Drug discovery:playing dirty[J].Nature,2005,437(7061):942-943.
[10]Barabasi AL,Oltvai ZN.Network biology:understanding the cell's functional organization[J].Nat Rev Genet,2004,5(2):101-113.
[11]Bader S,Kuhner S,Gavin AC.Interaction networks for systems biology[J].FEBSLett,2008,582(8):1220-1224.
[12]Barabasi AL,Gulbahce N,Loscalzo J.Network medicine:a network-based approach to human disease[J].Nat Rev Genet,2011,12(1):56-68.
[13]Arrell DK,Terzic A.Network systems biology for drug discovery[J].Clin Pharmacol Ther,2010,88(1):120-125.
[14]Yildirim MA,Goh KI,Cusick ME,Barabasi AL,Vidal M.Drugtarget network[J].Nat Biotechnol,2007,25(10):1119-1126.
[15]Kitano H.Towards a theory of biological robustness[J].Mol Syst Biol,2007,3:137.
[16]Chen Y,Zhu J,Lum PY,Yang X,Pinto S,MacNeil DJ,et al.Variations in DNA elucidate molecular networks that cause disease[J].Nature,2008,452(7186):429-435.
[17]Janga SC,Tzakos A.Structure and organization of drug-target networks:insights from genomic approaches for drug discovery[J].Mol Biosyst,2009,5(12):1536-1548.
[18]Hillenmeyer ME,Fung E,Wildenhain J,Pierce SE,Hoon S,Lee W,et al.The chemical genomic portrait of yeast:uncovering a phenotype for all genes[J].Science,2008,320(5874):362-365.
[19]Zimmermann GR,Lehar J,Keith CT.Multi-target therapeutics:when the whole is greater than the sum of the parts[J].Drug Discov Today,2007,12(1-2):34-42.
[20]Kitano H.A robustness-based approach to systems-oriented drug design[J].Nat Rev Drug Discov,2007,6(3):202-210.
[21]Dudekula N,Arora V,Callaerts-Vegh Z,Bond RA.The temporal hormesis of drug therapies[J].Dose Response,2005,3(3):414-424.
[22]Lange RP,Locher HH,Wyss PC,Then RL.The targets of currently used antibacterial agents:lessons for drug discovery[J].Curr Pharm Des,2007,13(30):3140-3154.
[23]Denome SA,Elf PK,Henderson TA,Nelson DE,Young KD.Escherichia coli mutants lacking all possible combinations of eight penicillin binding proteins:viability,characteristics,and implications for peptidoglycan synthesis[J].J Bacteriol,1999,181(13):3981-3993.
[24]Janoir C,Zeller V,Kitzis MD,Moreau NJ,Gutmann L.Highlevel fluoroquinolone resistance in Streptococcus pneumoniae requires mutations in parC and gyrA[J].Antimicrob Agents Chemother,1996,40(12):2760-2764.
[25]Satoh JI.Molecular network of microRNA targets in Alzheimer's disease brains[J].Exp Neurol,2011.DOI:10.1016/j.expneurol.2011.09.003
[26]Klipp E,Wade RC,Kummer U.Biochemical network-based drugtarget prediction[J].Curr Opin Biotechnol,2010,21(4):511-516.
[27]Mathur S,Dinakarpandian D.Drug repositioning using disease associated biological processes and net work analysis of drug targets[J].AMIA Annu Symp Proc,2011,2011:305-311.
[28]Turner NC,Lord CJ,Iorns E,Brough R,Swift S,Elliott R,et al.A synthetic lethal siRNA screen identifying genes mediating sensitivity to a PARP inhibitor[J].EMBO J,2008,27(9):1368-1377.
[29]Gupta GP,Nguyen DX,Chiang AC,Bos PD,Kim JY,Nadal C,et al.Mediators of vascular remodelling co-opted for sequential steps in lung metastasis[J].Nature,2007,446(7137):765-770.
[30]Eltarhouny SA,Elsawy WH,Radpour R,Hahn S,Holzgreve W,Zhong XY.Genes controlling spread of breast cancer to lung"gang of 4"[J].Exp Oncol,2008,30(2):91-95.
[31]Apsel B,Blair JA,Gonzalez B,Nazif TM,Feldman ME,Aizenstein B,et al.Targeted polypharmacology:discovery of dual inhibitors of tyrosine and phosphoinositide kinases[J].Nat Chem Biol,2008,4(11):691-699.
[32]Campillos M,Kuhn M,Gavin AC,Jensen LJ,Bork P.Drug target identification using side-effect similarity[J].Science,2008,321(5886):263-266.
[33]Morphy R,Rankovic Z.Fragments,network biology and designing multiple ligands[J].Drug Discov Today,2007,12(3-4):156-160.
[34]Takigawa I,Tsuda K,Mamitsuka H.Mining significant substructure pairs for interpreting polypharmacology in drug-target network[J].PLoSOne,2011,6(2):e16999.
[35]Li Q,Li X,Li C,Chen L,Song J,Tang Y,et al.A networkbased multi-target computational estimation scheme for anticoagulant activities of compounds[J].PLoS One,2011,6(3):e14774.
[36]Hopkins AL,Groom CR.The druggable genome[J].Nat Rev Drug Discov,2002,1(9):727-730.

