大菱鲆生长性状相关单核苷酸多态位点的多家系验证❋
田 涛, 王伟继, 陈再忠, 胡玉龙, 王陌桑, 吕 丁,4, 李之乡
(1.上海海洋大学水产与生命学院,上海 201306;2.中国水产科学研究院黄海水产研究所农业部海洋渔业可持续发展重点实验室,山东 青岛 266071;3.海洋国家实验室海洋渔业科学与食物产出过程功能实验室,山东 青岛 266071;4.南京农业大学无锡渔业学院,江苏 无锡 214081)
大菱鲆生长性状相关单核苷酸多态位点的多家系验证❋
田 涛1,2,3, 王伟继2,3❋❋, 陈再忠1, 胡玉龙2,3, 王陌桑1,2,3, 吕 丁2,3,4, 李之乡1,2,3
(1.上海海洋大学水产与生命学院,上海 201306;2.中国水产科学研究院黄海水产研究所农业部海洋渔业可持续发展重点实验室,山东 青岛 266071;3.海洋国家实验室海洋渔业科学与食物产出过程功能实验室,山东 青岛 266071;4.南京农业大学无锡渔业学院,江苏 无锡 214081)
为在多家系群体中进一步验证与大菱鲆生长性状紧密关联的SNP位点,本研究以QTL定位作图筛选获得的100个与大菱鲆(ScophthalmusmaximusL.)生长性状相关的候选单核苷酸多态(SNP)位点为基础,利用飞行质谱法,在4个大菱鲆家系群体中进行基因分型,并与体重、体长2个生长性状数据进行关联分析。对关联显著的位点,统计位点在家系群体中的多态性,并进行连锁不平衡分析和单倍型分析;根据显著位点所在基因序列信息,在NCBI数据库中进行比对分析,对匹配到的基因功能进行初步探讨。关联分析发现,有9个SNP位点与生长性状关联显著(P<0.05)。9个位点中,4个位点与体重关联显著(P<0.05),7个位点与体长关联显著(P<0.05),其中,SNP51和SNP111与体重和体长2个性状均关联显著(P<0.05)。SNP位点的多态性分析发现,观测杂合度在0.126~0.719之间(平均为0.387),期望杂合度在0.118~0.501之间(平均为0.338);9个位点的平均多态信息含量为0.267,属于中度多态性位点;哈迪温伯格平衡检验结果显示,9个位点中有5个位点偏离平衡。连锁不平衡和单倍型分析发现,标记间存在显著的连锁不平衡;单倍型CTTGT和CTTGA是2个主要的单倍型,单倍型频率分别为26.8%和25.9%。根据关联显著SNP位点所在的序列信息,在NCBI数据库比对分析后发现,COL11A1和Notch1基因可能是与大菱鲆生长性状相关的重要功能基因。本研究结果将推进大菱鲆生长相关分子标记的研究,为下一步基因定位和分子标记辅助育种提供更多的标记基础。
大菱鲆;生长性状;单核苷酸多态性(SNP);关联分析
大菱鲆(ScophthalmusmaximusL.)属于鲽形目(Pleuronectiformes)鲆科(Scophthalmidae)菱鲆属(Scophthalmus),俗称“多宝鱼”,是原产于欧洲沿海的一种名贵的比目鱼。中国于1992年首次从英国引进,因其生长快,易驯化,抗逆性强等特点,迅速成为北方海水养殖的主要经济鱼类[1]。然而,随着集约化养殖水平的不断提高,大菱鲆养殖面临诸多问题。由于苗种资源匮乏、累代养殖以及近亲交配,使得大菱鲆种质资源退化严重,加上大菱鲆疾病频发,生长周期已从原来的12个月增长到18个月,严重制约了大菱鲆养殖产业的可持续发展[2-4]。因此对大菱鲆进行遗传改良,选育具有优良生长性状的大菱鲆新品种成为发展大菱鲆养殖产业的当务之急。
生长性状是影响大菱鲆经济价值最重要的性状,在大菱鲆育种工作中也主要是针对生长性状进行遗传改良。传统的育种方式主要是通过杂交育种或者选择育种的方法,经过多代选育来改良水产动物的生长性状。随着生物技术的不断创新与发展,分子标记技术被引入到育种工作中,利用分子标记能够准确的标记遗传性状,并且在生物体的各个生长时期均能进行检测,显著缩短育种年限,弥补了传统育种中的不足。分子标记辅助育种(Marker assisted selection, MAS)逐渐成为一种新的育种模式,在水产动物育种领域也取得了快速的发展[5]。
单核苷酸多态性(Single nucleotide polymorphism, SNP)是基因组DNA中由于单个核苷酸变异所引起的序列多态性,包括单碱基的转换、颠换、插入及缺失等形式,最早由Lander于1996年提出[6]。因SNP标记在基因组中密度高,遗传稳定性好,并且易于自动化分析,被广泛应用于人类、动物和植物的遗传研究中。在动物遗传育种领域,SNP标记可用于遗传连锁图谱的构建、群体遗传多样性分析、亲缘关系鉴定以及性状的关联分析研究[7-9]。其中,SNP标记与动物经济性状的关联分析研究近几年逐渐兴起,它基于全基因组范围内高密度的SNP位点或者候选基因内部的SNP标记,通过关联分析,研究遗传标记和表型性状的关联性,进而寻找与重要遗传性状相关联的SNP标记[10]。生长性状是水产动物最重要的经济性状之一,国内外学者利用SNP标记与生长性状进行关联分析已经取得一定进展。Lv等[11]基于三疣梭子蟹转录组学数据,利用关联分析方法在三疣梭子蟹生长性状分离群体中发现一个与生长性状关联的SNP位点(comp58070-R31);Li等[12]研究大口黑鲈鱼(Micropterussalmoniodes)胰岛素样生长因子基因(IGF-1)5’侧翼区域,通过关联分析发现,该基因内部SNP位点的多样性与体重、体长显著关联。另外,SNP位点与性状的关联分析研究在栉孔扇贝(Chlamysfarreri)[13]和鲤鱼(Cyprinuscarpio)[14]等水产动物中也有报道,但在大菱鲆的研究中还鲜有报道。
水产动物的生长性状是典型的数量性状,具有连续遗传的特点,受多基因位点的控制,而这些位点被称为数量性状位点(Quantitative traits locus)。Wang等[8]以一个大菱鲆家系共149个父母本及子代个体为作图群体,构建了基于SNP标记的遗传连锁图谱,最终定位了220个与大菱鲆性别及生长性状相关的QTL位点。本研究以此为基础,选取其中100个与生长性状相关的候选SNP位点,利用飞行质谱法(Time of flight mass spectrometer,TOF-MS)在4个大菱鲆家系群体中进行SNP位点基因分型,并结合大菱鲆的体重和体长数据进行关联分析。接着,对于关联显著的SNP标记位点,统计各位点在大菱鲆家系群体中的观测杂合度、期望杂合度、基因型频率和等位基因频率等群体遗传参数,并进行连锁不平衡分析和单倍型分析;最后,根据SNP标记位点周围的序列信息,在NCBI数据库中进行序列比对分析,对相似性高的基因的功能进行讨论。本研究旨在大菱鲆多个家系群体中进一步验证与生长性状相关联的SNP标记位点,为加快大菱鲆分子标记辅助育种进程及生长性状相关基因研究提供理论基础。
1 材料与方法
1.1 实验材料
试验群体为2014年构建的大菱鲆家系,饲养于烟台海阳市黄海水产有限公司。随机挑选同一批次构建的4个大菱鲆家系,4个家系的父母本各不相同。受精卵孵化后100天左右在大菱鲆腹部注射荧光标记以区分不同的家系,每个家系随机挑选100条鱼,混养于同一水泥池中。12月龄时每个家系随机选取40尾鱼,测量体重和体长,并记录数据,同时剪取鳍条放入液氮中,之后转移至-80℃超低温冰箱中保存,用于后续基因组DNA的提取。
1.2 基因组DNA的提取
采用醋酸铵法提取大菱鲆鳍条组织基因组DNA,使用紫外光分光光度计测定基因组DNA的浓度和纯度,所有基因组DNA用双蒸水将浓度统一定量到100ng/μL,OD260/OD280值在1.8~2.0之间,符合后续实验要求。利用1%浓度的琼脂糖凝胶电泳检测基因组DNA的完整性,条带明亮且无拖尾现象可用于下一步实验。
1.3 SNP位点的筛选
根据Wang等[8]利用QTL定位作图筛选获得的与大菱鲆生长性状相关的QTL位点,选取其中的100个候选SNP位点((LOD>3),部分位点的详细信息见表1。同时,根据SNP位点所在的序列,选取SNP位点突变碱基前后各200个碱基长度的序列用于飞行质谱引物设计。
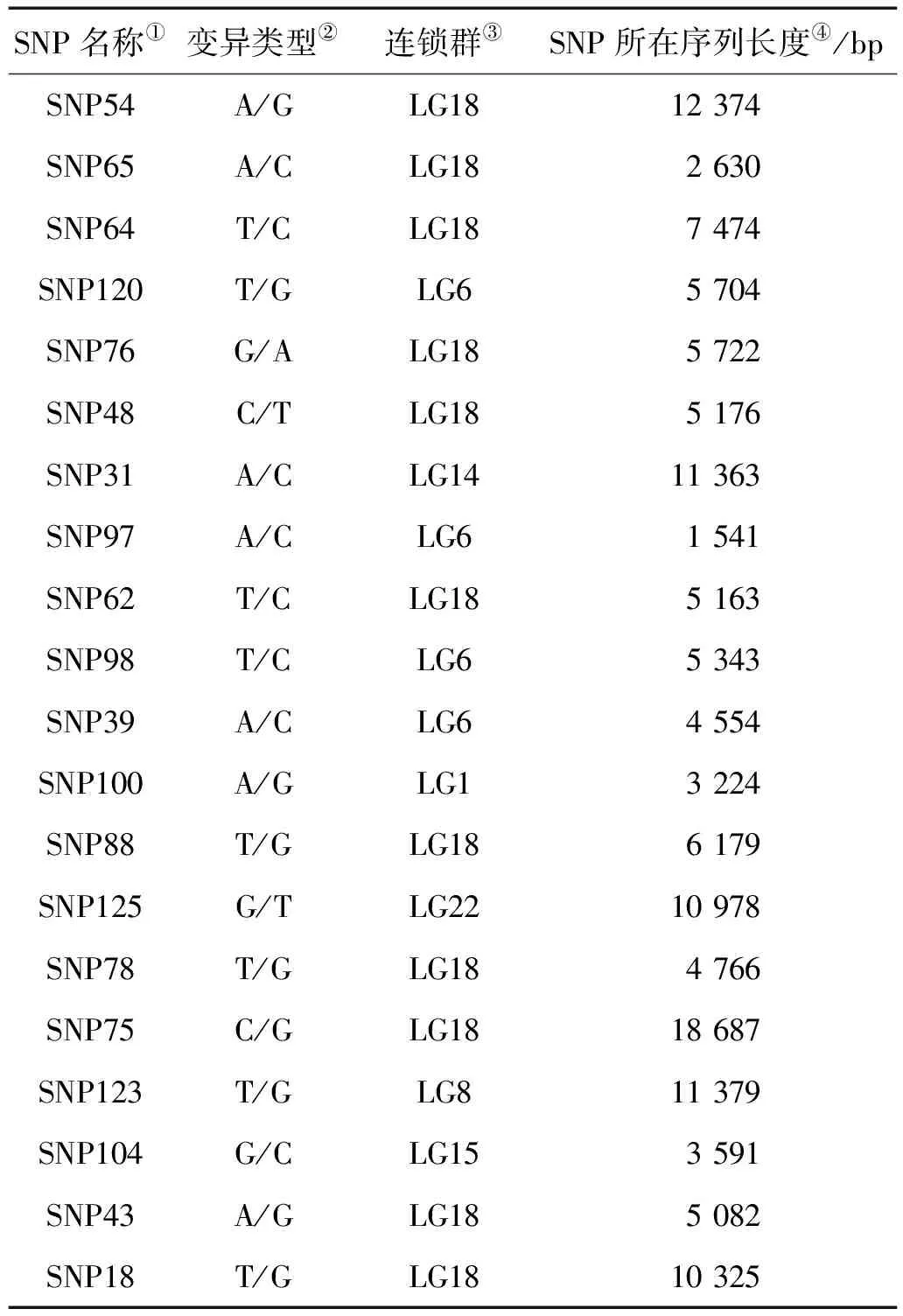
表1 部分SNP位点信息
Note:①SNP locus; ②Variants; ③Linkage group; ④Scaffold length
1.4 SNP位点的分型
SNP位点基因分型采用飞行质谱法(TOF-MS),结合SNP所在序列信息,共设计4个多重PCR反应,每个PCR反应分别包括26/26/26/22个SNP位点,飞行质谱分型由北京翔燕凯杰科技有限公司完成,质谱反应结束后根据质谱峰图判读各样本目标位点基因型。
1.5 统计分析
统计4个家系所有个体在每个SNP标记位点上的基因型,利用SPSS19.0软件的一般线性模型(General linear model, GLM),对基因型数据与生长性状(体重和体长)数据进行关联分析,模型为:
Y=μ+Gi+eij。
式中:Y为性状观察值(体重和体长);μ为总体平均值;Gi为SNP的效应值;eij为随机误差。利用POPGENE32软件[15]统计显著关联位点的观测杂合度(Observed heterozygosity,Ho)、期望杂合度(Expected heterozygosity,He)、有效等位基因数(Ne)以及进行哈迪-温伯格平衡(Hardy-Weinberg equilibrium, HWE)检验。利用PIC_CALC(0.6)[16]计算各SNP标记位点的多态信息含量(Polymorphism information content,PIC)。利用SHESIS软件[17]统计关联位点的基因型频率和等位基因频率,并进行连锁不平衡分析和单倍型分析。
1.6 基因功能预测
根据关联显著的SNP位点所在序列信息,在美国国家生物技术中心((National center for biotechnology information,NCBI)利用BLAST功能进行序列比对分析,查找相似度比较高的基因序列,通过基因注释信息以及查阅相关文献,研究基因的功能,为进一步研究提供参考。
2 实验结果
2.1 SNP位点分型结果统计
利用飞行质谱法对100个SNP位点进行分型,结果88个位点成功分型,分型成功率为88%。在成功分型的88个位点中,41个属于转换,47个属于颠换,具体变异类型分布见图1。
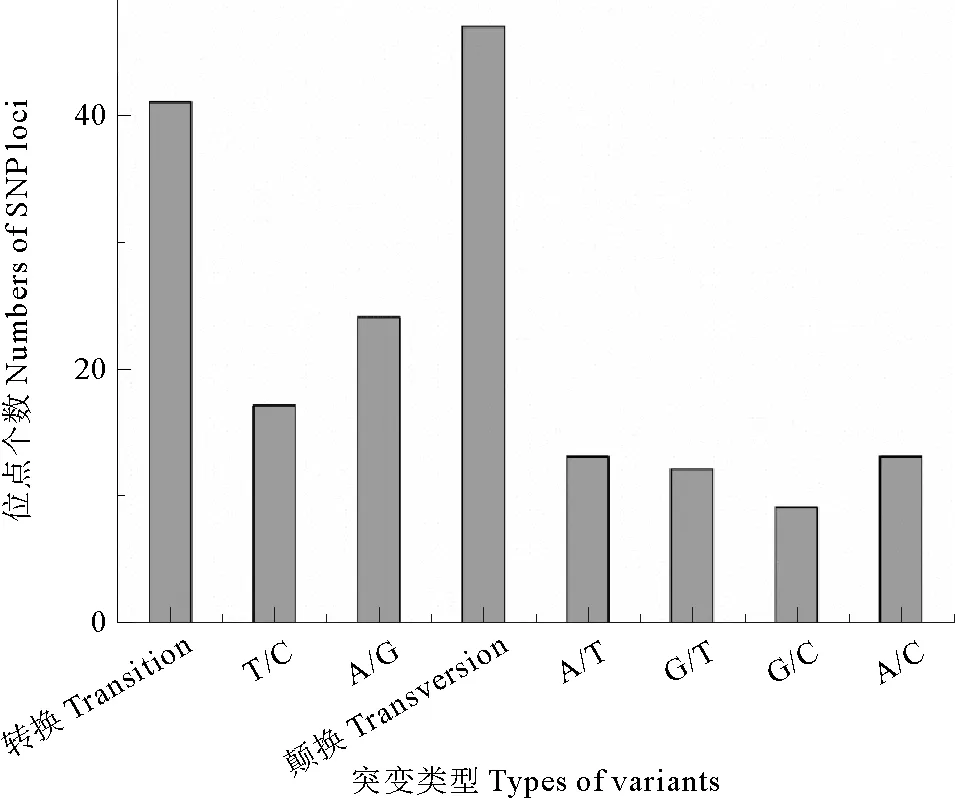
图1 SNP位点变异类型分布图
2.2 SNP位点与生长性状关联分析结果
大菱鲆4个家系生长性状(体重和体长)数据见表2。利用一般线性模型(GLM)对SNP位点的基因型和生长性状进行关联分析,结果见表3。从表3的结果中可见共有2个SNP标记(SNP51和SNP111)位点与体重和体长2个性状均显著关联。4个SNP标记位点(SNP51、SNP55、SNP101和SNP111)与体重显著关联(P<0.05),7个位点(SNP50、SNP51、SNP56、SNP86、SNP92、SNP105和SNP111)与体长显著关联(P<0.05),其中SNP56和SNP105与体长性状极显著关联(P<0.01)。
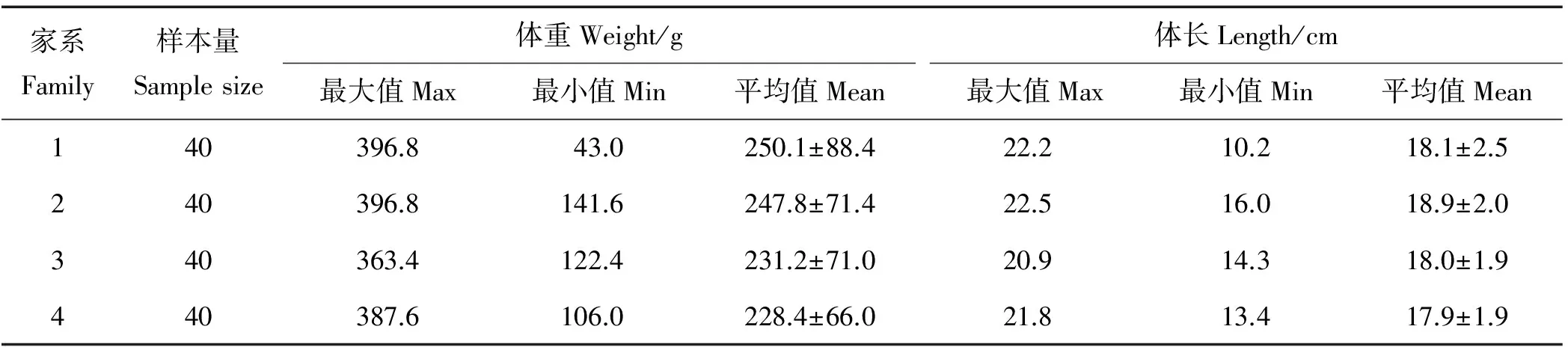
表2 生长性状(体重和体长)详细信息
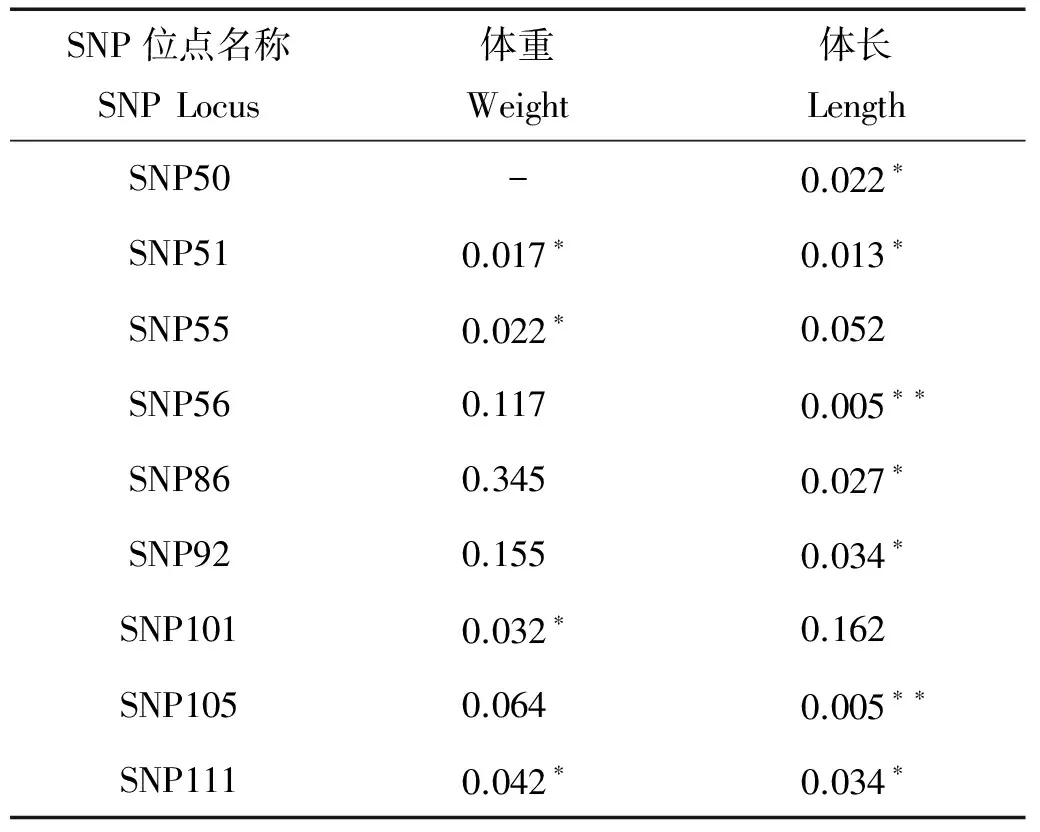
表3 SNP位点与生长性状的关联分析结果
注:*表示关联显著(P<0.05);**表示关联极显著(P<0.01)。
Note:* indicates significant association;** indicates very significant association.
2.3 关联显著位点的多态性分析
9个SNP位点在大菱鲆家系群体中的多态性信息见表4。9个位点中,4个位点(SNP50、SNP51、SNP56和SNP101)在群体中只检测到2种基因型,其它5个位点为3种基因型,平均有效等位基因数为1.600个。观测杂合度在0.126~0.719之间,平均观测杂合度为0.387;期望杂合度在0.118~0.501之间,平均期望杂合度为0.338。9个位点的平均多态信息含量为0.267,其中SNP50、SNP56和SNP111为低度多态位点(PIC<0.25),其它6个位点为中度多态位点(0.25 2.4 单倍型和连锁不平衡分析 关联显著的9个SNP位点中,有5个位点位于同一连锁群(LG18)(SNP50、SNP51、SNP55、SNP56和SNP92),连锁不平衡分析结果见表5。从表5的结果中看出有2对SNP位点之间属于完全连锁不平衡(D’=1),另外,6对位点之间存在紧密连锁(D’>0.9)。同时,对5个位点进行单倍型分析(见表6),结果表明,5个位点共组成9种单倍型,其中有6种单倍型的频率大于0.03,CTTGT的频率最高,为26.8%,其次是CTTGA,频率为25.9%。 表4 显著位点的多态性信息 注:HE为期望杂合度;HO为观测杂合度;PIC为多态信息含量;HWE为哈迪温伯格平衡;Ne为有效等位基因数;A表示腺嘌呤;T表示胸腺嘧啶;G表示鸟嘌呤;C代表胞嘧啶。 Note:HOandHEindicate observed and expected heterozygosity;PICis Polymorphism information content; HWE means Hardy-Weinberg equilibrium;Neindicates effective number of alleles; A, T, G, C is adenine, thymine, guanine and cytosine respectively. 2.5 关联显著SNP位点基因功能预测 根据关联显著SNP位点所在序列信息,在NCBI数据库中进行序列比对查找相似度高的基因(E值<10-10)(见表7),其中7个位点的序列能匹配到相关基因,共6种不同的基因,分别为补体成分9基因(Complement compoment 9 gene)、细胞珠蛋白-2(Cytoglobin-2)、干扰素4a1基因(Requlatory factor-4a1 gene)、Collagen alpha-1(Ⅺ)chain-like (COL11A1)基因、StrainHd-rR基因以及Notch1基因。 表5 连锁不平衡检验D’值 表6 单倍型分析结果 注:*代表单倍型频率大于0.03(LFT>0.03); A表示腺嘌呤;T表示胸腺嘧啶;G表示鸟嘌呤;C代表胞嘧啶。 Note: *indicate the haplotype frequency>0.03(LFT>0.03)A, T, G, C is adenine, thymine, guanine and cytosine respectively. 本研究利用飞行质谱法对100个与生长性状相关的候选SNP位点进行分型,其中成功分型88个,成功率为88%。飞行质谱法是基于单碱基延伸技术和质谱检测技术,通过检测延伸产物和未延伸产物的分子量大小来确定SNP位点的碱基信息。作为一种中通量的SNP分型方法,具有准确率高、检出速度快等特点[18]。另外,此技术还可以同时对多个位点进行检测,提高分型的效率,节省基因分型的成本,比如,如美国Sequenom公司的Sequenom MassArray可设计多达40重的基因型检测[19]。本研究在分型过程中设计了4个多重PCR反应,每个反应里面包括20多个位点,节省了实验的成本。统计位点的突变类型(见图1),A/G类型最多(24个),其次是T/C突变(17个),这类突变属于转换突变。理论上,发生转换突变和颠换突变的比例应该相同的,但是实际情况下,发生转换突变的概率要大一些,比例约2∶1,这种现象称为“转换偏差”。WANG等[20]统计分析人类基因组SNP突变类型的结果也证明了这一点。原因可能是因为CpG二核苷酸上的胞嘧啶残基通常会自发地脱去氨基,进而形成胸腺嘧啶,在一定程度上增加了转换的比例。 关联分析在复杂性状关联标记的发掘上发挥了重要的作用,尤其在全基因组序列已知的高等动物中,利用高密度的SNP基因芯片,在全基因组水平上筛选与高等动物经济性状相关联的SNP位点,如羊的肉质性状、牛的生长性状及京海黄鸡生长性状等[21-23]。但在大菱鲆的研究中,由于基因组数据的缺乏,无法完成全基因组水平的关联分析。对于像大菱鲆这样缺乏基因组数据的非模式生物来说,两阶段研究正被更多的研究者所采用[24]。2个阶段研究是指第一阶段通过小样本的QTL定位得到与数量性状相关联的SNP位点,第二阶段在更大的样本中对第一阶段得到的阳性结果进行验证,结合2个阶段结果进行分析。本研究以QTL定位作图初步筛选获得的100个与大菱鲆生长性状相关联的候选SNP位点为基础,利用关联分析的方法,在多个大菱鲆家系群体中进一步验证。许多研究证实,连锁图谱与关联分析在数量性状的定位中都具有不可替代的作用,两者在定位的精度和广度、提供的信息量、统计分析方法等方面具有良好的互补性[25-26]。通过关联分析(见表3),共有9个位点与体重或体长性状显著关联,其中,SNP51和SNP111与2个性状均关联显著。9个位点共分布于3个连锁群,其中18号连锁群(LG18)有5个显著位点。结合第一阶段通过QTL作图得到的结果,2/3的位点都位于18号连锁群上,而且SNP51也在此连锁群上,因此可以推测18号连锁群可能是与大菱鲆生长性状紧密关联的,下一阶段可以针对此连锁群做进一步的研究。 表7 9个SNP位点所在序列基因功能预测 对9个关联显著的SNP位点在家系群体中的多态性进行分析,其中,3个位点存在基因型缺失(见表4),其他6个位点基因型分布比较均匀;群体的平均观测杂合度为0.387,稍大于期望杂合度(0.338)。9个位点的多态信息含量(PIC)都小于0.5,属于低度或中度多态性位点。哈迪温伯格平衡(HWD)检验结果显示9个位点中5个位点偏离了平衡(P<0.05)。本研究实验群体是人工选育的大菱鲆家系群体,在选育过程中因多代选育使得部分等位基因的频率发生了改变。张德宁等[27]对“黄选1号”三疣梭子蟹生长性状SNP的鉴定中,也有部分位点偏离了哈迪温伯格平衡,这表明与生长性状相关联SNP位点可能容易受到选种选配的影响。 连锁不平衡(Linkage disequilibrium,LD)是指同一条染色体上等位基因间的非随机相关,包括2个标记间、2个基因或QTL间的非随机关联。SNP标记之间的连锁不平衡在群体遗传学、基因精确定位以及关联分析中有重要应用[28]。尤其在关联分析研究中,从根本上讲,关联分析就是检测的遗传标记和表型性状的连锁不平衡[29]。LD一般通过D’来进行度量。D’的取值从0~1,当D’=1时,意味着2个标记之间完全LD。本研究中,有2对SNP位点之间完全LD(D’=1),6对位点之间紧密LD(D’>0.9),在18号连锁群上(LG18)80%位点间存在连锁不平衡,因此该连锁群是与生长性状关联紧密的区域,具有很高的研究价值。生长性状是复杂的数量性状,受多基因和多通路的调控。针对于复杂的性状,基于单倍型的关联分析在很多研究中被证明要比单个的位点分析更有统计效力[30-31]。因此,单倍型的研究更有利于发掘基因与表型性状的相关性。在本研究中,对位于同一连锁群的5个显著关联SNP进行了单倍型分析(见表6),5个SNP位点共组成9种单倍型,其中,6种单倍型的频率大于3%。CTTGA和CTTGT的单倍型频率都大于25%,是2种主要的单倍型。通过连锁不平衡分析和单倍型分析得知,18号连锁群的某段区域和生长性状密切关联,是连锁不平衡区域,这与关联分析得到的结果是一致的。 在对9个SNP位点序列进行比对分析后发现,共匹配到6个功能基因。其中补体9基因(Complement compoment 9 gene)和干扰素因子4a1基因(Requlatory factor-4a1 gene)是与免疫相关的基因,与生长相关的功能还没有报道。细胞珠蛋白(Cytoglobin-2)是血红素蛋白家族中的一员,具有携氧、氧感受器的功能,在脊椎动物中普遍存在,有研究表明,它可能与胶原的形成有关[32]。COL11A1基因功能报道目前只出现在人类疾病研究中,它编码Ⅺ型胶原α1亚基,与软骨胶原纤维的组装有关。COL11A1基因突变会导致人类身高发育异常,有研究表明,这可能与骨骼中胶原纤维的错配有关[33]。周伏圣等[34]利用全基因组关联分析的方法,发现COL11A1基因启动子2个SNP位点与汉族人的身高显著有关。另外,小鼠的Cho基因与人类COL11A1基因是同源基因,有研究发现[35],Cho纯合子的缺失导致小鼠软骨发育不全,体长明显变短。Notch1基因编码一类高度保守的细胞表面受体,它主要功能是调节生物细胞的发育,影响细胞正常形态发生的多个过程,包括细胞分化、细胞凋亡、细胞增殖以及细胞边界的形成。目前,Notch1基因在鱼类的研究中已有相关报道,在斑马鱼中,Notch1基因主要参与鱼类中枢神经系统的分化,特别是与体节中胚层的形成密切相关;而在虹鳟鱼中,Notch1基因的功能与生殖细胞的发育有关[36-37]。COL11A1基因和Notch1基因在大菱鲆的研究中还未见报道,但相关研究结果表明此类基因与细胞生长以及胚胎发育息息相关,某些基因的突变会对生长性状造成影响,因此后续的研究中可以将这些基因作为影响大菱鲆生长性状的候选基因,进一步研究基因的功能。 本研究根据QTL作图筛选获得的100个与大菱鲆生长性状相关的候选SNP位点,在多个大菱鲆家系中利用关联分析的方法进一步验证,结果发现9个位点与大菱鲆体重或体长性状显著关联。9个位点在大菱鲆家系群体中的多态性显著,属于中度多态性位点。连锁不平衡和单倍型分析结果表明,位点间存在显著的连锁不平衡,其中,CTTGA和CTTGT是2种主要的单倍型。最后,通过分析显著位点的序列信息探讨了相关基因的功能,推测COL11A1和Notch1基因可能是与大菱鲆生长相关的重要功能基因。这些结果将有利于推进大菱鲆生长相关分子标记的研究,为下一步基因定位以及分子标记辅助育种提供更多的标记基础。 [1] 雷霁霖, 刘新富. 大菱鲆ScophthalmusmaximusL. 引进养殖的初步研究[J]. 现代渔业信息, 1995, 10(11): 1-3. Lei J L, Liu X F. An primary study on culture of turbot,ScophthalmusmaximusL [J]. Modern Fisheries Information, 1995, 10(11): 1-3. [2] 雷霁霖, 马爱军, 陈超, 等. 大菱鲆(ScophthalmusmaximusL.)养殖现状与可持续发展[J]. 中国工程科学, 2005, 7(5): 30-34. Lei J L, Ma A J, Chen C, et al. The present status and sustainable development of turbot (ScophthalmusmaximusL. ) culture in China [J]. Engineering Science, 2005, 7(5): 30-34. [3] 申雪艳, 宫庆礼, 雷霁霖, 等. 进口大菱鲆ScophthalmusmaximusL. 苗种的遗传结构分析[J]. 海洋与湖沼, 2004, 35(4): 332-341. Shen X Y, Gong Q L, Lei J L, et al. Population genetic structure analysis of the imported turbot seedlingsScophthalmusmaximusL. using RAPD and microsatellite technique [J]. Oceanologia et Limnologia Sinica, 2004, 35(4): 332-341. [4] 许可, 马爱军, 王新安, 等. 大菱鲆(Scophthalmusmaximus)生长性状相关的微卫星标记筛选[J]. 海洋与湖沼, 2009, 40(5): 577-583. Xu K, Ma A J, Wang X A, et al. microsatellites molecular markers and the correlation to growth trait ofScophthalmusmaximus[J]. Oceanologia et Limnologia Sinica, 2009, 40(5): 577-583. [5] 孙效文, 鲁翠云, 智英, 等. 水产动物分子育种研究进展[J]. 中国水产科学, 2009, 16(6): 981-990. Sun X W, Lu C Y, Jia Z Y, et al. The progress of molecular marker-based breeding for aquatic species [J]. Journal of Fishery Sciences of China, 2009, 16(6): 981-990. [6] Vignal A, Milan D, SanCristobal M, et al. A review on SNP and other types of molecular markers and their use in animal genetics[J]. Genetics Selection Evolution, 2002, 34(3): 275-306. [7] Creelman E K, Hauser L, Simmons R K, et al. Temporal and geographic genetic divergence: characterizing sockeye salmon populations in the Chignik Watershed, Alaska, using single-nucleotide polymorphisms[J]. Transactions of the American Fisheries Society, 2011, 140(3): 749-762. [8] Wang W, Hu Y, Ma Y, et al. High-density genetic linkage mapping in Turbot (ScophthalmusmaximusL. ) based on SNP markers and major sex and growth-related regions detection[J]. Plos One, 2015, 10(3): 120410. [9] Buzanskas M E, Grossi D A, Ventura R V, et al. Genome-wide association for growth traits in Canchim beef cattle[J]. Plos One, 2014, 9(4): 94802. [10] McCarthy M I, Abecasis G R, Cardon L R, et al. Genome-wide association studies for complex traits: Consensus, uncertainty and challenges[J]. Nature Reviews Genetics, 2008, 9(5): 356-369. [11] Lv J, Liu P, Gao B, et al. Transcriptome analysis of the Portunus trituberculatus: de novo assembly, growth-related gene identification and marker discovery[J]. Plos One, 2014, 9(4): 94055. [12] Li XH, Bai JJ, Ye X, et al. Polymorphisms in the 5′flanking region of the insulin-like growth factor I gene are associated with growth traits in largemouth bassMicropterussalmoides[J]. Fisheries Science, 2009, 75(2): 351-358. [13] Wang X, Meng X, Song B, et al. SNPs in the myostatin gene of the molluskChlamysfarreri: association with growth traits[J]. Comparative Biochemistry and Physiology Part B: Biochemistry and Molecular Biology, 2010, 155(3): 327-330. [14] Sun Y, Yu X, Tong J. Polymorphisms in myostatin gene and associations with growth traits in the common carp (CyprinuscarpioL.)[J]. International Journal of Molecular Sciences, 2012, 13(11): 14956-14961. [15] Yeh F, Yang R, Boyle T, et al. Pop Gene32, Microsoft Windows-based freeware for population genetic analysis, version 1. 32[CP]. Edmonton, Alberta, Canada: Molecular Biology and Biotechnology Centre, University of Alberta, 2000. [16] Nagy S, Poczai P, Cernák I, et al. PICcalc: an online program to calculate polymorphic information content for molecular genetic studies[J]. Biochemical Genetics, 2012, 50(9-10): 670-672. [17] Yong Y, Lin H. SHEsis, a powerful software platform for analyses of linkage disequilibrium, haplotype construction, and genetic association at polymorphism loci[J]. Cell Research, 2005, 15(2): 97-98. [18] 赵琼一, 李信, 周德贵, 等. 后基因组时代下作物的 SNP 分型方法[J]. 分子植物育种, 2010, 8(1): 125-133. Zhao Q Y, Li X, Zhou D G, et al. SNP genotyping methods for crops in post-genomic Era [J]. Molecular Plant Breeding, 2010, 8(1): 125-133. [19] Tang K, Opalsky D, Abel K, et al. Single nucleotide polymorphism analyses by MALDI-TOF MS[J]. International Journal of Mass Spectrometry, 2003, 226(1): 37-54. [20] Wang D G, Fan J B, Siao C J, et al. Large-scale identification, mapping, and genotyping of single-nucleotide polymorphisms in the human genome[J]. Science, 1998, 280(5366): 1077-1082. [21] Li Z, Jiasen L, Fuping Z, et al. Genome-wide association studies for growth and meat production traits in sheep[J]. Plos One, 2013, 8(6): 66569. [22] 樊庆灿, 王金玉, 张跟喜, 等. 京海黄鸡生长性状与15个单核苷酸多态(SNP)位点的关联分析[J]. 农业生物技术学报, 2014, 22(8): 1009-1017. Fan Q C, Wang J Y, Zhang G X, et al. Association analysis of fifteen single nucleotide polymorphism(SNP) loci with growth traits in Jinghai yellow chicken(Gallusgallus) [J]. Journal of Agricultural Biotechnology, 2014, 22(8): 1009-1017. [23] Bolormaa S, Hayes B J, Savin K, et al. Genome-wide association studies for feedlot and growth traits in cattle[J]. Journal of Animal Science, 2011, 89(6): 1684-1697. [24] 胡艳玲. 复杂性状与基因组多位点的关联分析方法研究[D]. 上海: 上海交通大学, 2009: 4-6. Hu Y L. Study on the Methods of Genome Multi-loci Association Analysis for Complex Traits[D]. Shanghai: Shanghai Jiao Tong University School of Agriculture and Biology, 2009: 4-6. [25] Gupta P K, Rustgi S, Kulwal P L. Linkage disequilibrium and association studies in higher plants: Present status and future prospects[J]. Journal of Microbiological Methods, 2005, 57(4): 461-485. [26] 于海霞, 肖静, 田纪春, 等. 关联分析及其在植物中的应用[J]. 基因组学与应用生物学, 2009, 28(1): 187-194. Yu H X, Xiao J, Tian J C, et al. Association analysis and its application in plants genetics[J]. Genomics and Applied Biology, 2009, 28(1): 187-194. [27] 张德宁, 吕建建, 刘萍, 等. 三疣梭子蟹生长相关 SNP 位点的鉴定[J]. 中国水产科学, 2015, 22(3): 393-401. Zhang D J, Lv J J, Liu P, et al. Identifying SNP markers correlated with growth of swimming crab (Portunustrituberculatus) based on a comparative transcriptome[J]. Journal of Fishery Sciences of China, 2015, 22(3): 393-401. [28] Marty A, Amigues Y, Servin B, et al. Genetic variability and linkage disequilibrium patterns in the bovine DNAJA1 gene[J]. Molecular Biotechnology, 2010, 44(3): 190-197. [29] 何云刚, 金力, 黄薇. 单核苷酸多态性与连锁不平衡研究进展[J]. 基础医学与临床, 2004, 24(5): 78-81. He Y G, Jin L, Huang W. Advance in the research of single nucleotide polymorphism and linkage disequilibrium [J]. Basic Medical Sciences and Clinics, 2004, 24(5): 78-81. [30] BaderJ S. The relative power of SNPs and haplotype as genetic markers for association tests[J]. Pharmacogenomics, 2001, 2(1): 11-24. [31] Zhang K, Calabrese P, Nordborg M, et al. Haplotype block structure and its applications to association studies: power and study designs[J]. The American Journal of Human Genetics, 2002, 71(6): 1386-1394. [32] Hankeln T, Ebner B, Fuchs C, et al. Neuroglobin and cytoglobin in search of their role in the vertebrate globin family[J]. Journal of Inorganic Biochemistry, 2005, 99(1): 110-119. [33] Majava M, Hoornaert K P, Bartholdi D, et al. A report on 10 new patients with heterozygous mutations in the COL11A1 gene and a review of genotype-phenotype correlations in type XI collagenopathies[J]. American Journal of Medical Genetics Part A, 2007, 143(3): 258-264. [34] 周伏圣, 孙良丹, 高敏, 等. COL11A1 基因启动子多态与汉族人身高的关联分析[J]. 安徽医科大学学报, 2009, 44(3): 331-334. Zhou F S, Sun L D, Gao M, et al. Association between polymorphisms of COLl 1A1 gene promoter and adult height in Chinese Han population[J]. Acta Universitatis Medicinalis Anhui, 2009, 44(3): 331-334. [35] Li Y, Lacerda D A, Warman M L, et al. A fibrillar collagen gene, Col11a1, is essential for skeletal morphogenesis[J]. Cell, 1995, 80(3): 423-430. [36] Yano A, Von Schalburg K, Cooper G, et al. Identification of a molecular marker for type a spermatogonia by microarray analysis using gonadal cells from pvasa-GFP transgenic rainbow trout (Oncorhynchusmykiss)[J]. Molecular Reproduction & Development, 2009, 76(3): 246-254. [37] Westin J, Lardelli M. Three novel Notch genes in zebrafish: implications for vertebrate Notch gene evolution and function[J]. Development Genes & Evolution, 1997, 207(1): 51-63. 责任编辑 朱宝象 Validation of Single Nucleotide Polymorphism (SNP) Loci Related to Growth Traits in Turbot (Scophthalmus maximus L.) Families TIAN Tao1,2,3, WANG Wei-Ji2,3, CHEN Zai-Zhong1, HU Yu-Long2,3, WANG Mo-Sang1,2,3,LV Ding2,3,4, LI Zhi-Xiang1,2,3 (1.College of Fisheries and Life Science, Shanghai Ocean University, Shanghai 201306, China; 2.The Key Laboratory of Sustainable Development of Marine Fisheries, Ministry of Agriculture, Yellow Sea Fisheries Research Institute, Chinese Academy of Fisheries Science, Qingdao 266071, China; 3.Function Laboratory for Marine Fisheries Science and Food Production Processes, National Laboratory for Ocean Science and Technology, Qingdao 266071, China; 4.Wuxi Fisheries College, Nanjing Agriculture University, Wuxi 214081, China) In order to further identify single nucleotide polymorphism (SNP) associated with the growth traits of turbot (ScophthalmusmaximusL.), the time of flight mass spectrometer(TOF-MS)was used to genotyping 100 SNP loci of four turbot families, 160 individuals in total, and the association of SNPs with growth traits (body weight and body length)was analyzed based on the high-density linkage map of turbot. For the significant SNP loci, polymorphism of loci each was detected in the population, meanwhile, linkage disequilibrium (LD) and haplotype analysis were performed for the significant SNPs located in the same linkage group (LG). At last, the sequence around the site was aligned in NCBI database and then detected the function of the matched gene. The results showed that 9 SNPs were identified to be associated with the growth traits (P<0.05). Among these SNPs, four loci were associated with body weight and seven were associated with body length significantly (P<0.05). SNP51 and SNP111 were identified to be associated with both body weight and body length (P<0.05). The genetic parameters showed that the observed heterozygosity (Ho) ranged from 0.126 to 0.719 (Mean=0.387) and expected heterozygosity (He) estimates from 0.118 to 0.501 (Mean=0.338). The average polymorphism information content (PIC) was 0.267, indicating that the 9 loci were reasonably informative. Five of the 9 SNP loci deviated significantly from Hardy-Weinberg equilibrium (HWE). Strong linkage disequilibrium occurred among the five SNPs in the LG18 according to the LD analysis results. Moreover, haplotype CTTGT and CTTGA were two important types as they accounted for 26.8% and 25.9% of total haplotypes, respectively. Finally, the alignment results showed thatCOL11A1 gene andNOTCH1 gene may be related to the growth traits. However, further studies are needed to explore their function in the turbot development. These findings will boost molecular marker relating to growth traits screening and provide theoretical basis to the marker assisted selection (MAS) and gene location. ScophthalmusmaximusL.; growth trait; single nucleotide polymorphism(SNP); association analysis 国家十二五“863”重要鲆鲽鱼类良种培育子课题大菱鲆良种培育项目(2012AA10A408-7)资助 Supported by the National High Technology Research and Development Program of China (2012AA10A408-7) 2016-03-03; 2016-05-04 田 涛(1991-)男,硕士生,主要从事水产动物遗传育种方向研究。 ❋❋ 通讯作者:E-mail:wangwj@ysfri.ac.cn Q754; S917.4 A 1672-5174(2016)12-032-09 10.16441/j.cnki.hdxb.20160055 田涛, 王伟继, 陈再忠, 等. 大菱鲆生长性状相关单核苷酸多态位点的多家系验证[J]. 中国海洋大学学报(自然科学版), 2016, 46(12): 32-40. TIAN Tao, WANG Wei-Ji, CHEN Zai-Zhong, et al. Validation of single nucleotide polymorphism (SNP) loci related to growth traits in turbot (ScophthalmusmaximusL.) families[J]. Periodical of Ocean University of China, 2016, 46(12): 32-40.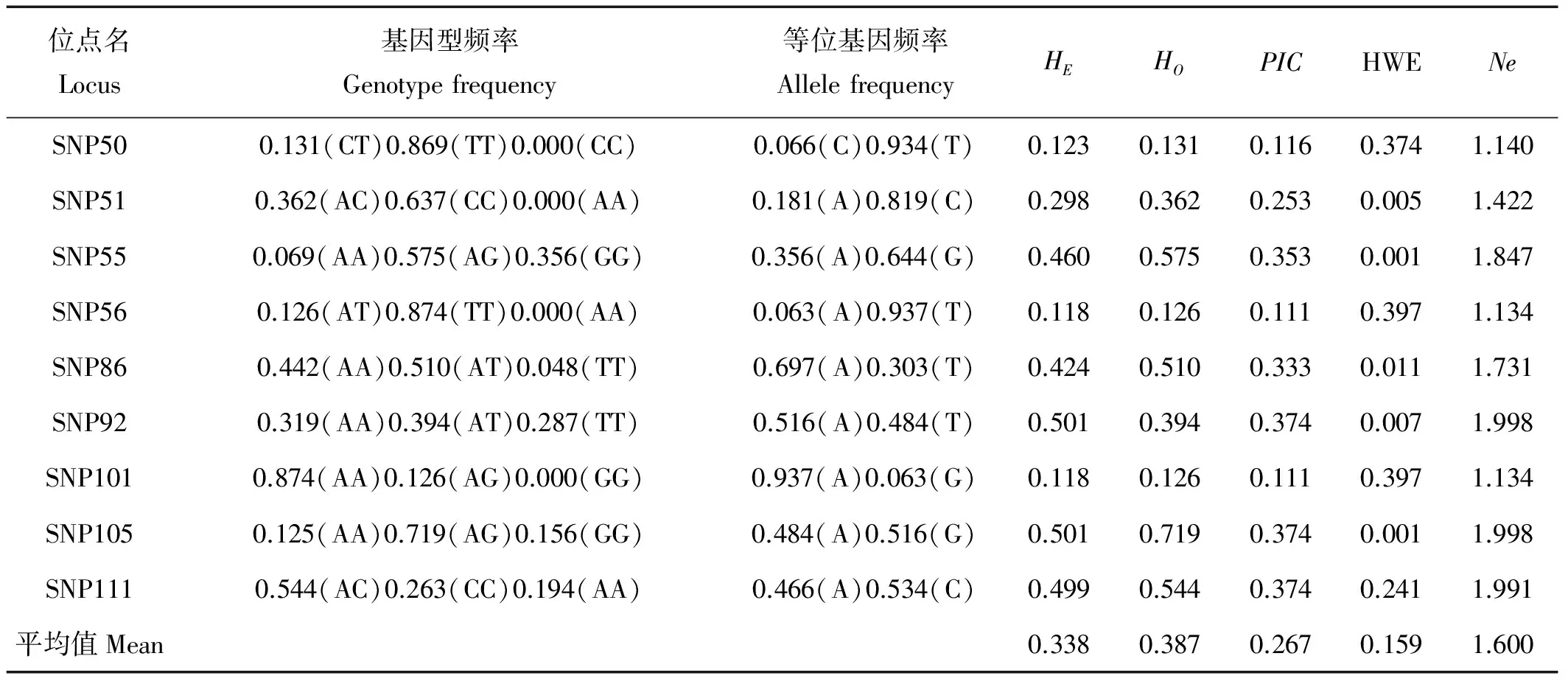
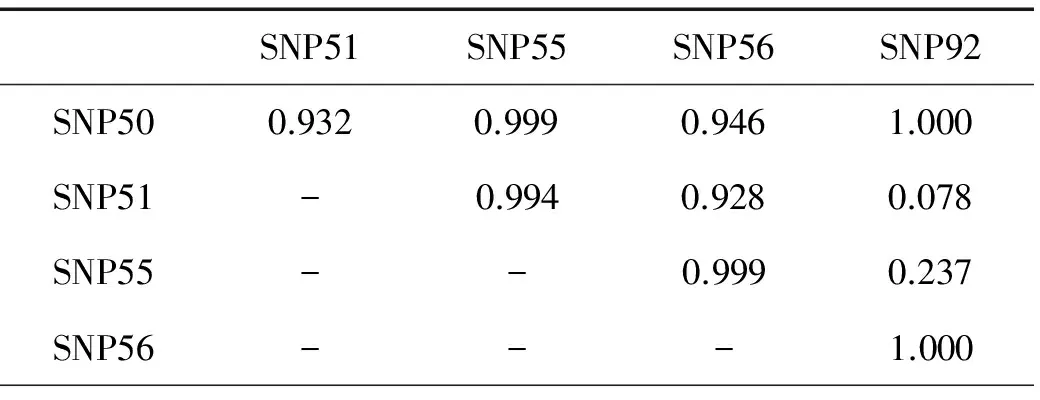
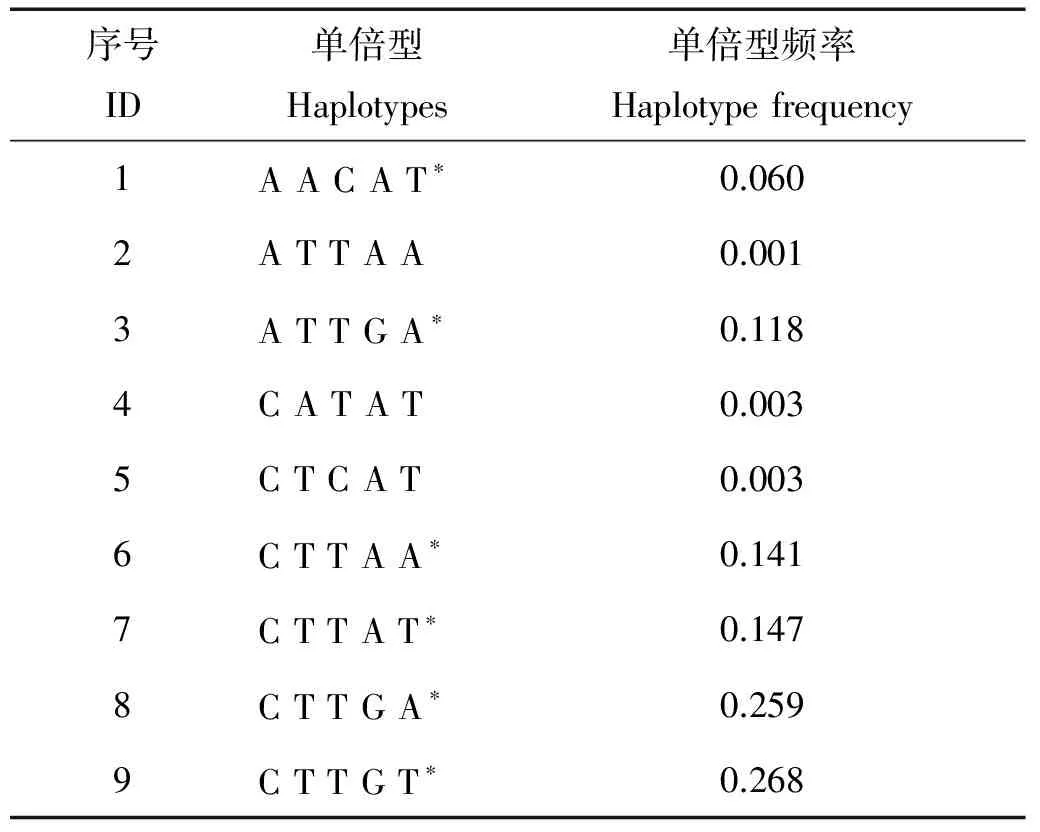
3 讨论

4 结语

