耐碱性蛋白A色谱配基的构建及性能评价
管志龙,白姝,孙彦,史清洪
耐碱性蛋白A色谱配基的构建及性能评价
管志龙,白姝,孙彦,史清洪
(系统生物工程教育部重点实验室,天津大学化工学院,天津 300072)
提出了优化分子内作用力提高蛋白A(SpA)的结构域Z分子稳定性的方法,构建了突变结构域Zʹ和Zʺ。圆二色光谱分析结果表明,突变结构域具有与结构域Z一致的特征峰,但突变结构域Zʹ中α-螺旋含量明显高于突变结构域Zʺ。在pH 6.0下,突变结构域Zʹ的热转变温度(m)较结构域Z提高了3.9℃,而突变结构域Zʺ的T值仅提高了1.6℃。这表明,突变结构域Zʹ中第12位引入疏水性更大的Ile残基提高了结构域分子的稳定性。色谱介质耐碱性实验结果表明,突变结构域Zʹ具有更好的耐碱性,其耐碱性在突变结构域Zʹ四串联体融合蛋白中得到了进一步提升。研究工作为SpA色谱配基的分子改造提供了新思路。
抗体;吸附;热力学;蛋白A色谱;示差扫描量热;圆二色光谱;耐碱性
引 言
蛋白A(protein A,SpA)是一种来自金黄色葡萄球菌表面的膜蛋白,包含5个可与抗体特异性结合且高度同源串联结构域(依次为E、D、A、B和C)。SpA色谱是当前抗体药物制备的核心技术,一直被业界视为单克隆抗体提取单元操作的“金标准”[1-3]。这一技术成功的基础在于持续提升的SpA色谱介质性能[4-5]。自首个商品化SpA色谱介质1978年问世后的若干年中[6],SpA色谱介质的化学稳定性曾一度饱受诟病。这种状况直到Nilsson等[7]构建了突变结构域B(G29A)后才得以缓解。这种突变结构域被命名为结构域Z,其通过消除Asn-Gly结构的去酰胺化效应使SpA的化学稳定性得以明显改善。此后,一大批以MabSelect SuRe和ProSep vA Ultra等为代表基于结构域Z的新型SpA色谱介质不断地涌现[8-10]。
当前除MabSelect SuRe以外的绝大多数商品化SpA色谱介质仍无法耐受苛刻的标准原位清洗(CIP)溶液(0.5 mol·L-1NaOH溶液)。因此,需要采用折中(0.05~0.1 mol·L-1NaOH溶液)或替代(6 mol·L-1盐酸胍溶液,pH约4.0)方法实现CIP操作[10]。针对上述问题,Linhult等[11]通过基因突变逐一替代结构域Z中Asn残基的方法研究了其对结构域碱液耐受性的影响。结果显示,N23T突变显著提高了结构域的碱液耐受性。以突变结构域Z(F30A,N23T)为配基的SpA色谱介质在标准CIP溶液中连续浸泡7.5 h后仍保持了70%以上的抗体结合能力;而同样处理后的结构域Z(F30A)的抗体结合能力仅剩25%。N23T突变对结构域Z化学稳定性的影响也得到了Xia等[12]的验证。Asn残基对化学稳定性影响也见于蛋白G(Streptococcal protein G,SpG)结构域C2的报道中[13]。研究发现,N7A和N36A双突变的结构域C2在碱液中最为稳定。另外,Minakuchi等[14]发现,结构域C的G29W和G29Y两种突变体具有比G29A突变体更好的化学稳定性。Palmer等[15]通过自由能随pH变化的比较发现,Lys、Arg和Tyr侧链基团的去质子化导致了SpG在碱性溶液中稳定性降低。这种SpG蛋白中表面电荷影响稳定性的讨论为改善SpA配基的稳定性提供了另一思路。
在前人工作基础上[16],本文提出了通过优化分子内作用力提高结构域Z分子整体稳定性的方法,其是通过对结构域Z非关键氨基酸残基中去酰胺化效应的消除(N11T和N23T)和疏水性相互作用的强化(A12I和Q32T)两种途径实现的。基于上述思路,本文构建了两种突变结构域,分别命名为Zʹ和Zʺ(如图1所示)。借助于圆二色(CD)光谱、示差扫描量热(DSC)分析和抗体吸附实验,系统地比较了结构域Z、Zʹ和Zʺ的分子结构、稳定性及抗体结合能力;进而构建了结构域Z和Zʹ四串联体融合蛋白Z4和Zʹ4,实现了SpA色谱配基碱液耐受性的进一步提升。本研究工作为SpA色谱配基的分子改造提供了新思路。
1 实验材料和方法
1.1 实验材料
结构域Z及突变结构域Zʹ和Zʺ由北京华大蛋白质研发中心有限公司合成。含有融合蛋白Z4和Zʹ4的表达质粒pET-30a(+)-Z4和pET-30a(+)- Zʹ4由生工生物工程(上海)有限公司构建并转入感受态细胞BL21(DE3)大肠杆菌中表达。Q Sepharose FF和Sepharose FF凝胶购自通用电气医疗系统(中国)有限公司。实验用的抗体、1,4-丁二醇二缩水甘油醚(BDDE)和缩水甘油醚三甲基氯化铵(GTMAC)为Sigma-Aldrich公司产品,购自天津博瑞星科技有限公司。蛋白质分子标准品Blue Plus Protein Maker (14×103~100×103)购于北京全式金生物技术有限公司。胰蛋白胨、酵母提取物为英国Oxoid公司产品,其和其他化学试剂均购自当地,化学试剂除特殊说明外均为分析纯试剂。
1.2 圆二色光谱分析
结构域Z、Zʹ和Zʺ的CD光谱分析采用JASCO J-180圆二色光谱仪在0.15 mg·ml-1浓度和1 mm光程条件下完成。样品缓冲液为含100 mmol·L-1NaCl的20 mmol·L-1PBS溶液(pH 6.0)。CD光谱采用连续扫描模式,扫描速度为50 nm·min-1,扫描波长范围为250~190 nm,每个样品重复扫描3次。分析中以样品缓冲液为参照扣除背景干扰。
1.3 示差扫描量热分析
结构域Z、Zʹ和Zʺ的DSC分析采用MicroCal VP-DSC示差扫描量热仪(GE Healthcare, PA, USA)在1.0 mg·ml-1浓度下完成。样品分别溶解于含100 mmol·L-1NaCl的20 mmol·L-1PBS溶液(pH 6.0)和0.1 mol·L-1NaOH溶液中。DSC的温度扫描范围为25~110℃,升温速率为1℃·min-1。实验数据采用MicroCal Origin软件的MN2State模型进行拟合,得到热变性温度m。
1.4 蛋白表达与纯化
含表达质粒pET-30a(+)-Z4和pET-30a(+)-Zʹ4的BL21(DE3)大肠杆菌在37℃下于LB培养基平板培养后,挑选单菌落置于10 ml含30 μg·ml-1卡那霉素的LB液体培养基中培养过夜作为种子液。种子液按照1:100(体积比)接种于200 ml LB液体培养基(卡那霉素的终浓度为30 μg·ml-1)。上述菌液在37℃培养后加入终浓度为0.5 mmol·L-1IPTG;含pET-30a(+)-Z4的BL21(DE3)大肠杆菌在37℃下诱导6 h后收集菌体;pET-30a(+)-Zʹ4的BL21(DE3)大肠杆菌在20℃下诱导过夜后收集菌体。收集的菌体复悬于20 mmol·L-1Tris-HCl缓冲液(pH 7.5)后,于冰浴中超声破碎。细胞破碎液于12000 r·min-1下离心收集上清液。
上清液经0.45 μm针头滤器过滤后采用Q Sepharose FF离子交换色谱柱纯化得到目标蛋白,其步骤如下:填充于XK 16/26色谱柱中的Q Sepharose FF经20 mmol·L-1Tris-HCl缓冲液(pH 7.5)平衡至少5CV后,连续输入上清液直至穿透。目标蛋白收集以含1 mol·L-1NaCl的20 mmol·L-1Tris-HCl缓冲液(pH 7.5)为洗脱液,通过1%洗脱液/min的线性梯度洗脱分步回收。目标蛋白经透析和冻干后保存于-20℃备用。
1.5 SpA色谱介质合成
通过结构域Z、Zʹ和Zʺ以及融合蛋白Z4和Zʹ4分别偶联于Sepharose FF合成5种SpA色谱介质,步骤简述如下:将经去离子水反复清洗并抽干的凝胶Sepharose FF(10 g)加入到装有75%(质量)GTMAC(5 ml)和0.645 mol·L-1NaOH(5 ml)混合溶液的锥形瓶中,悬浮液在170 r·min-1和25℃下反应过夜。收集的凝胶经去离子水反复冲洗后,称取2 g抽干后凝胶重悬于 4.6 mol·L-1NaOH溶液(1.2 ml)中并加入4 ml BDDE。上述悬浮液于170 r·min-1和25℃下反应2 h以引入环氧基团活化Sepharose FF。活化凝胶经去离子水反复冲洗并抽干后用于SpA色谱介质合成。
活化Sepharose FF在10 mmol·L-1NaHCO3溶液中平衡过夜后,转移至含有配基的10 mmol·L-1NaHCO3溶液中。上述悬浮液于170 r·min-1和37℃下反应2 h。反应产物经去离子水反复清洗后转移至巯基乙醇(1 ml)和含0.5 mol·L-1NaCl的0.2 mol·L-1NaHCO3(9 ml)的混合溶液中于37℃下反应2 h封闭凝胶表面残留的环氧基团。SpA色谱配基的修饰密度通过Water C18反相色谱柱测定反应前后溶液中配基浓度变化后计算得到。本文中合成的5种SpA色谱介质依据配基的种类分别命名为Z SepFF、Zʹ SepFF、Zʺ SepFF、Z4SepFF和Zʹ4SepFF。
1.6 SpA色谱介质耐碱性实验
SpA色谱介质的耐碱性实验分别在浓度为0.1和0.5 mol·L-1的NaOH溶液中进行。简要步骤如下:SpA色谱介质在NaOH溶液中浸泡2、6和12 h;继而经去离子水冲洗后,SpA色谱介质于含100 mmol·L-1NaCl的20 mmol·L-1PBS缓冲液(pH 7.5)中平衡过夜;称取10 mg 平衡后的介质依次转入装有10 ml浓度为0.2~3.0 mg·ml-1抗体溶液的锥形瓶中。上述悬浮液在25℃和120 r·min-1条件下反应过夜达到吸附平衡。最后,在4000 r·min-1下离心收集上清液,在280 nm下测定上清液中抗体浓度,根据质量守恒计算出抗体吸附量。根据式(1)的Langmuir吸附等温线可计算出抗体饱和吸附容量m(mg·g-1)和解离常数d(mg·ml-1)

2 结果与讨论
2.1 结构域Z、Zʹ和Zʺ的圆二色光谱
本文利用CD光谱分析了结构域Z、Zʹ和Zʺ在含100 mmol·L-1NaCl的20 mmol·L-1PBS溶液(pH 6.0)和0.1 mol·L-1NaOH溶液中的二级结构及其变化规律。3种结构域的CD光谱结果如图2所示。
图2(a)中结构域Z的CD光谱显示,其在190~200 nm间有一正特征峰,而在208和222 nm附近各有一负特征峰。这与典型的α-螺旋结构CD特征峰相吻合[17-18]。此外,Jendeberg等[19]在200~250 nm的CD光谱扫描结果也显示,结构域Z在208和222 nm附近各有一个负特征峰。迄今结构域Z及其与抗体复合物分子结构的研究均表明,结构域Z分子由空间上3条反向平行α-螺旋构成[19-21]。由图2(a)结果可知,本文中结构域Z不仅与文献报道的结构域Z一级序列相同而且均有典型的α-螺旋结构,由此证明两种分子具有相同的空间结构。
(a) 100 mmol·L-1NaCl in 20 mmol·L-1PBS buffer (pH 6.0); (b) 0.1 mol·L-1NaOH solution
突变结构域Zʹ和Zʺ在pH 6.0下的CD光谱结果亦绘于图2(a)中。从中可知,突变结构域Zʹ和Zʺ具有与结构域Z一致的特征峰,即分别为190~200 nm间的正特征峰和208与222 nm附近两个负特征峰。由此可知,结构域Z中氨基酸突变(如图1所示)并未引起突变结构域分子二级结构的改变,突变结构域Zʹ和Zʺ保持了与结构域Z相同的二级结构。而在0.1 mol·L-1NaOH溶液中,结构域Z、Zʹ和Zʺ中仅有222 nm附近的负特征峰仍然可见[图2(b)]。图2(a)结果进一步显示,在222 nm下突变结构域Zʹ比突变结构域Zʺ和结构域Z具有更强的负特征峰,表明突变结构域Zʹ的α-螺旋含量更高。对比图1中结构域序列发现,其变化趋势与结构域中第12位上Trp(-0.9)、Ala(1.8)和Ile(4.5)残基的Kyte-Doolittle疏水性指数增加相一致[22-23]。这说明在结构域Z的12位引入疏水性更大的氨基酸残基有利于提高α-螺旋含量。
2.2 结构域Z、Zʹ和Zʺ的示差扫描量热分析
为了进一步考察其对结构域分子稳定性的影响,本文采用DSC测量结构域Z、Zʹ和Zʺ的热转变温度m。不同条件下,结构域Z、Zʹ和Zʺ的偏摩尔热容(ΔC)随温度变化结果如图3所示。从中可以看出,在含100 mmol·L-1NaCl的20 mmol·L-1PBS溶液(pH 6.0)中结构域Z的m为75.3℃。这个结果与Dincbas-Renqvist等[24]通过CD光谱[]222变化测定的结构域Z在pH 5.7下热转变温度(m=78.5℃)非常接近。相同条件下,突变结构域Zʺ的m为76.9℃,其较结构域Z提高了1.6℃;突变结构域Zʹ的m为79.2℃,其相较于结构域Z和Zʺ分别提高了3.9℃和2.3℃。同样的趋势也出现在0.1 mol·L-1NaOH溶液中。如图3(b)所示,0.1 mol·L-1NaOH溶液中结构域Z、Zʹ和Zʺ的m分别降至61.6、64.8和62.8℃。对比突变结构域Zʹ和Zʺ的氨基酸序列(如图1所示)可以发现,两者之间唯一差别在于突变结构域Zʺ第12位上的Trp替换为突变结构域Zʹ中疏水性更大的Ile。由此,突变结构域Zʹ分子内的疏水性相互作用得到了强化,从而显著提高突变结构域的分子稳定性。而当Kyte-Doolittle疏水性指数较小的Trp存在时,突变结构域Zʺ的m值较突变结构域Zʹ下降。突变结构域Zʹ不仅在3种结构域中具有最高的m值而且其在0.1 mol·L-1NaOH溶液中的m值接近Minakuchi等[14]报道的中性条件下结构域B的热转变温度(m= 67.5℃)。这说明突变结构域Zʹ具有更好的分子稳定性。
(a) 100 mmol·L-1NaCl in 20 mmol·L-1PBS buffer (pH 6.0); (b) 0.1 mol·L-1NaOH solution
2.3 融合蛋白Z4与Zʹ4表达与纯化
采用Q Sepharose FF离子交换色谱柱对BL21(DE3)大肠杆菌表达的融合蛋白Z4和Zʹ4进行纯化。离子交换色谱平衡后,连续输入细胞破碎后的上清液直至穿透;上样后清洗除去未吸附的蛋白样品,然后引入洗脱液通过线性梯度洗脱吸附组分并收集电导率为6~10 mS·cm-1的洗脱峰。实验结果如图4所示。图4(a)为融合蛋白Z4的色谱图及电泳照片。从中可以看出,阴离子交换色谱Q-Sepharose FF收集的洗脱蛋白质分子量介于26×103~27×103,其与融合蛋白Z4的理论分子量相当。该目标蛋白的纯度达到95%以上。相同的结果也出现在图4(b)中。经离子交换色谱Q Sepharose FF纯化收集的融合蛋白Zʹ4同样具有95%以上的纯度。两种融合蛋白直接用于SpA亲和色谱介质的合成。
2.4 SpA色谱介质的耐碱性分析
本文中突变结构域Zʹ和Zʺ与结构域Z分别偶联于琼脂糖凝胶Sepharose FF表面合成Z SepFF、Zʹ SepFF和Zʺ SepFF 3种SpA色谱介质。3种色谱介质的SpA配基密度依次为9.7、9.7和9.5 mg·g-1介质。上述3种介质分别浸于0.1 mol·L-1NaOH溶液2、6和12 h后,测定其抗体吸附能力。抗体在Z SepFF和Zʹ SepFF介质上的静态吸附结果如图5所示。抗体吸附的Langmuir参数列于表1中。结果显示,Z SepFF和Zʹ SepFF介质在含100 mmol·L-1NaCl的20 mmol·L-1PBS缓冲液(pH 7.5)中的抗体饱和吸附容量相当,分别为22.1和21.4 mg·ml-1。但是,抗体在Zʺ SepFF介质上的吸附量非常低(本文未列出)。对比图1中突变结构域Zʹ和Zʺ的序列可知,两者间唯一差别在于突变结构域Zʹ中Ile替换为突变结构域Zʺ中Trp。这种差别也导致突变结构域Zʺ中α-螺旋含量及m值的降低。本文将专注于Z SepFF和Zʹ SepFF介质的耐碱性分析。浸泡于0.1 mol·L-1NaOH溶液12 h后,Z SepFF和Zʹ SepFF介质的m值也仅略微降低不到10%(如表1所示)。
本文进一步合成了Z4SepFF和Zʹ4SepFF介质。Z4SepFF和Zʹ4SepFF介质的SpA配基密度分别为9.5和9.3 mg·g-1介质。在此基础上,进一步比较了上述4种SpA色谱介质对标准CIP溶液(0.5 mol·L-1NaOH溶液)的耐受性。实验结果如图6所示。从中可以看出,Z SepFF介质的抗体吸附容量随着浸泡时间的延长下降得最为显著,在标准CIP溶液中浸泡12 h后仅保留了50%的抗体吸附能力。这种抗体吸附容量的下降程度与Linhult等[11]报道相近,其发现结构域Z在0.5 mol·L-1NaOH溶液浸泡12 h后的抗体吸附量相当于浸泡1 h后的65%。图6结果进一步显示,Zʹ SepFF对标准CIP溶液的耐受性更好,其在标准CIP溶液中浸泡12 h后仍保留了初始抗体吸附容量的62%。本文中融合蛋白Z4和Zʹ4的引入不仅提高了SpA色谱介质的抗体吸附容量而且进一步提高了其对碱液的耐受性。在含100 mmol·L-1NaCl的20 mmol·L-1PBS缓冲液(pH 7.5)中,抗体在Z4SepFF和Zʹ4SepFF介质上的m分别达到55.4和57.9 mg·ml-1。这不仅比von Roman等[25]报道的以结构域B四串联体融合蛋白为配基SpA色谱介质的m高出25%,也接近商品化MabSelect Xtra和MabSelect SuRe介质的m值[26]。
(a) chromatography of SpA-Z4and native PAGE image; (b) chromatography of SpA-Zʹ4and native PAGE image; F and P stands for feedstock and product, respectively
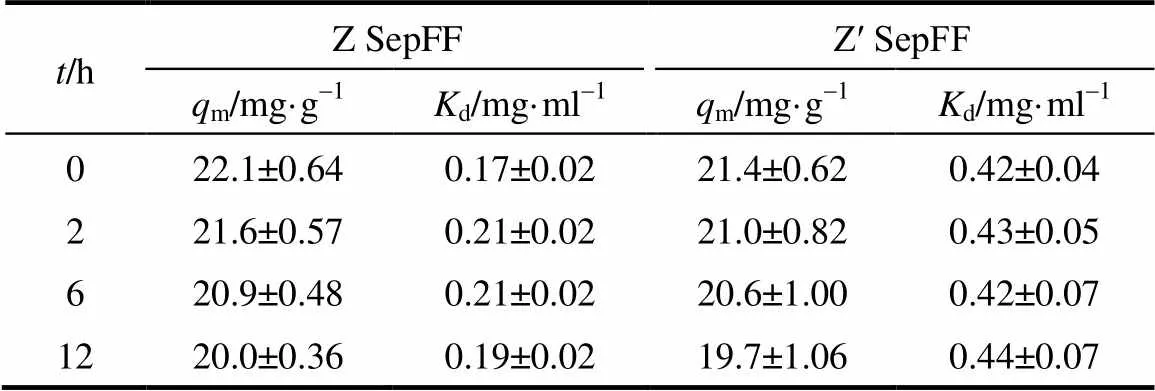
表1 不同碱液浸泡时间t后Z SepFF和Zʹ SepFF介质的Langmuir参数
在标准CIP溶液中浸泡12后,Z4SepFF和Zʹ4SepFF的m分别降至60%和68%。这表明SpA色谱介质的碱液耐受性得到进一步改善。耐碱性实验结果不仅证明了突变结构域Zʹ具有更好的碱液耐受性,而且这种碱液耐受性在突变结构域Zʹ四串联体融合蛋白为配基的SpA色谱介质中得到了提高。
3 结 论
本文通过对结构域Z非关键氨基酸残基中去酰胺化效应的消除(N11T和N23T)和疏水性相互作用的增强(Q31T和A12I)提高SpA蛋白结构域Z分子整体稳定性。突变结构域Zʹ和Zʺ的CD光谱结果显示,两者的特征峰与结构域Z相一致。这表明突变结构域中氨基酸的替换并未引起结构域分子二级结构的改变。突变结构域Zʹ在222 nm下具有最强的负特征峰,暗示着其α-螺旋含量要明显高于其他分子。在pH 6.0下,突变结构域Zʹ的m较突变结构域Zʺ提高了2.3℃,达到了79.2℃。这一结果表明,突变结构域Zʹ中第12位替换为疏水性更大的Ile显著提高了结构域分子的稳定性。SpA色谱介质耐碱性实验结果表明,突变结构域Zʹ具有更好的碱液耐受性,这种碱液耐受性在以其四串联体融合蛋白为配基的SpA色谱介质中得到了进一步提高。
References
[1] HOBER S, NORD K, LINHULT M. Protein A chromatography for antibody purification[J]. Journal of Chromatography B-Analytical Technologies in the Biomedical and Life Sciences, 2007, 848(1): 40-47.
[2] LOW D, O'LEARY R, PUJAR N S. Future of antibody purification[J]. Journal of Chromatography B-Analytical Technologies in the Biomedical and Life Sciences, 2007, 848(1): 48-63.
[3] GHOSE S, ALLEN M, HUBBARD B,. Antibody variable region interactions with protein A: implications for the development of generic purification processes[J]. Biotechnology and Bioengineering, 2005, 92(6): 665-673.
[4] BOLTON G R, MEHTA K K. The role of more than 40 years of improvement in protein A chromatography in the growth of the therapeutic antibody industry[J]. Biotechnology Progress, 2016, 32(5): 1193-1202.
[5] TAJIMA N, TAKAI M, ISHIHARA K. Significance of antibody orientation unraveled: well-oriented antibodies recorded high binding affinity[J]. Analytical Chemistry, 2011, 83(6): 1969-1976.
[6] EY P L, PROWSE S J, JENKIN C R. Isolation of pure IgG1, IgG2a and IgG2b immunoglobulins from mouse serum using protein A-sepharose[J]. Immunochemistry, 1978, 15(7): 429-436.
[7] NILSSON B, MOKS T, JANSSON B,. A synthetic IgG-binding domain based on staphylococcal protein A[J]. Protein Engineering, 1987, 1(2): 107-113.
[8] HAHN R, SCHLEGEL R, JUNGBAUER A. Comparison of protein A affinity sorbents[J]. Journal of Chromatography B-Analytical Technologies in the Biomedical and Life Sciences, 2003, 790(1/2): 35-51.
[9] GHOSE S, HUBBARD B, CRAMER S M. Binding capacity differences for antibodies and Fc-fusion proteins on protein A chromatographic materials[J]. Biotechnology and Bioengineering, 2007, 96(4): 768-779.
[10] HAHN R, SHIMAHARA K, STEINDL F,. Comparison of protein A affinity sorbents(Ⅲ): Life time study[J]. Journal of Chromatography A, 2006, 1102(1/2): 224-231.
[11] LINHULT M, GULICH S, GRASLUND T,. Improving the tolerance of a protein A analogue to repeated alkaline exposures using a bypass mutagenesis approach[J]. Proteins-Structure Function and Bioinformatics, 2004, 55(2): 407-416.
[12] XIA H F, LIANG Z D, WANG S L,. Molecular modification of protein A to improve the elution pH and alkali resistance in affinity chromatography[J]. Applied Biochemistry and Biotechnology, 2014, 172(8): 4002-4012.
[13] GULICH S, LINHULT M, STAHL S,. Engineering streptococcal protein G for increased alkaline stability[J]. Protein Engineering, 2002, 15(10): 835-842.
[14] MINAKUCHI K, MURATA D, OKUBO Y,. Remarkable alkaline stability of an engineered protein A as immunoglobulin affinity ligand: C domain having only one amino acid substitution[J]. Protein Science, 2013, 22(9): 1230-1238.
[15] PALMER B, ANGUS K, TAYLOR L,. Design of stability at extreme alkaline pH in streptococcal protein G[J]. Journal of Biotechnology, 2008, 134(3/4): 222-230.
[16] HUANG B, LIU F F, DONG X Y,. Molecular mechanism of the effects of salt and pH on the affinity between protein A and human immunoglobulin G1 revealed by molecular simulations[J]. Journal of Physical Chemistry B, 2012, 116(1): 424-433.
[17] JOHNSON W C JR. Protein secondary structure and circular dichroism: a practical guide[J]. Proteins, 1990, 7(3): 205-214.
[18] KELLY S M, PRICE N C. The use of circular dichroism in the investigation of protein structure and function[J]. Current Protein & Peptide Science, 2000, 1(4): 349-384.
[19] JENDEBERG L, TASHIRO M, TEJERO R,. The mechanism of binding staphylococcal protein A to immunoglobin G does not involve helix unwinding[J]. Biochemistry, 1996, 35(1): 22-31.
[20] ZHENG D Y, ARAMINI J M, MONTELIONE G T. Validation of helical tilt angles in the solution NMR structure of the Z domain ofprotein A by combined analysis of residual dipolar coupling and NOE data[J]. Protein Science, 2004, 13(2): 549-554.
[21] TASHIRO M, TEJERO R, ZIMMERMAN D E,. High-resolution solution NMR structure of the Z domain of staphylococcal protein A[J]. Journal of Molecular Biology, 1997, 272(4): 573-590.
[22] KYTE J, DOOLITTLE R F. A simple method for displaying the hydrophatic character of a protein[J]. Journal of Molecular Biology, 1982, 157(1): 105-132
[23] 阎隆飞, 孙之荣. 蛋白质分子结构[M]. 北京: 清华大学出版社, 1999: 1-21.
YAN L F, SUN Z R. Molecular Structure of Protein[M]. Beijing: Tsinghua University Press, 1999: 1-26.
[24] DINCBAS-RENQVIST V, LENDEL C, DOGAN J,. Thermodynamics of folding, stabilization, and binding in an engineered protein-protein complex[J]. Journal of the American Chemical Society, 2004, 126(36): 11220-11230.
[25] VON ROMAN M F, BERENSMEIER S. Improving the binding capacities of protein A chromatographic materials by means of ligand polymerization[J]. Journal of Chromatography A, 2014, 1347: 80-86.
[26] HAHN R, BAUERHANSL P, SHIMAHARA K,. Comparison of protein A affinity sorbents(Ⅱ): Mass transfer properties[J]. Journal of Chromatography A, 2005, 1093(1/2): 98-110.
Construction and characteristics of alkali-tolerance mutants of Z domain for protein A chromatography
GUAN Zhilong, BAI Shu, SUN Yan, SHI Qinghong
(Key Laboratory of Systems Bioengineering of Ministry of Education, School of Chemical Engineering and Technology, Tianjin University, Tianjin 300072, China)
A novel strategy to optimize intramolecular interaction of Z domain was proposed to improve alkali-tolerance of the domain ofprotein A (SpA) and two mutants of Z domain, named as Zʹ domain and Zʺ domain, were prepared to evaluate their alkali tolerance as the ligand for protein A chromatography. The result of circular dichroism showed that both the mutants had the identical characteristic peaks to Z domain whilst Zʹ domain had higher α-helix content than Zʺ domain. At pH 6.0, heat transition temperature of Zʹ domain increased by 3.9℃ compared with domain Z whilst heat transition temperature of Zʺ domain increased merely by 1.6℃. It indicated that the introduction of more hydrophobic Ile at position 12 in Zʹ domain improved the stability of the Zʹ domain. Using Z, Zʹ and their tetrameric domains as the ligand for protein A adsorbents, adsorption performance of SpA adsorbents was evaluated after their exposure in alkaline solutions. The results demonstrated that domain Zʹ had a higher alkali tolerance among three domains and the tolerance was further improved by applying a tetrameric Zʹ domain. The research provided a new clue for the development of alkali-tolerance protein A ligand.
antibody; adsorption; thermodynamics; protein A chromatography; differential scanning calorimetry; circular dichroism; alkali tolerance
10.11949/j.issn.0438-1157.20170366
TQ 028.8
A
0438—1157(2017)09—3459—07
2017-04-10收到初稿,2017-05-10收到修改稿。
史清洪。
管志龙(1989—),男,硕士研究生。
国家自然科学基金项目(21276189, 21476166);天津市应用基础研究计划项目(15JCYBJC48500);生化工程国家重点实验室开放基金(2014KF-03)。
Receiving date: 2017-04-10.
Prof. SHI Qinghong, qhshi@tju.edu.cn
supported by the National Natural Science Foundation of China (21276189, 21476166), the Tianjin Natural Science Foundation (15JCYBJC48500) and the Open Funding Project of the State Key Laboratory of Biochemical Engineering (2014KF-03).

