云南省登革4型病毒全基因组序列特征研究
胡挺松,张海林,刘永华,邓 波,尹小雄,徐松淼,李 萍,范泉水,张富强
云南省登革4型病毒全基因组序列特征研究
胡挺松1,张海林1,刘永华2,邓 波1,尹小雄2,徐松淼1,李 萍2,范泉水1,张富强1
目的阐明云南省2015年5株登革4型病毒(DENV-4)流行株的全基因组序列特征及分子流行病学特点。方法采用C6/36 细胞培养法分离病毒,用RT-PCR法扩增新分离DENV-4的全基因组序列,采用ClastalX1.83和MEGA6等生物信息学软件进行核苷酸和推导氨基酸序列同源性及系统进化分析。结果从2015年云南省瑞丽市登革热患者血清中分离到5株DENV-4(本地病例2株,来自缅甸腊戍和南坎市输入性病例3株)。经RT-PCR和序列测定,获得这5株DENV-4的全基因组序列(10 661 nt),其开放读码框(103-10 264)编码3 386个氨基酸。全基因组或结构蛋白和非结构蛋白基因序列的系统进化和同源性分析表明,云南分离株间高度同源并聚集为一个进化支,并与泰国不同年代DENV-4基因I型(G-I)流行株具有较近的进化关系和较高的同源性,同属G-I。云南株和泰国株均与同基因型的DENV-4原型株(H241,1956年菲律宾)和中国广州1990年B5株亲缘性和同源性都较低。云南株与H241株在结构蛋白或非结构蛋白中分别存在21和45个氨基酸位点差异。结论首次获得云南省DENV-4分离株的全基因组序列并发现它们与近期东南亚DENV-4 G-I流行株亲缘关系较近。首次证实云南省存在DENV-4 本地流行,传播来源为相邻缅甸北部边境地区。云南分离株某些氨基酸位点的改变是否与其抗原性和毒力有关尚需进一步研究。
登革4型病毒;全基因组;系统进化分析;同源性分析;氨基酸位点分析
登革病毒(dengue virus, DENV)分为4种血清型(DENV-1、2、3和4),每一种血清型又可分为多个基因型(genotype)[1-4]。DENV基因组全长约10~11 kb,编码3种结构蛋白(C、prM/M 和E)和7种非结构蛋白(NS1、NS2a、NS2b、NS3、NS4a、NS4b 和NS5)[2]。登革热(dengue fever, DF)广泛分布于全球热带和亚热带地区,尤以东南亚地区流行较为严重[1]。2000年以来,云南省每年均有来自相邻东南亚国家的DF输入性病例[5-12]。2013-2015年云南省德宏州、西双版纳州和临沧市相继发生DENV-1、DENV-2和DENV-3引起的本地DF暴发疫情[8-12],其中德宏州瑞丽市连续3年发生DENV-1和DENV-2本地流行[8-9,13]。冯云等[13]曾从2014年瑞丽市来自缅甸的一例输入性病例中检测到DENV-4,本研究从2015年瑞丽市输入性病例和本地病例中均分离到DENV-4,首次证实瑞丽市存在该血清型病毒的本地流行,并对新分离病毒株进行全基因组序列测定以及同源性、遗传进化和氨基酸位点分析。
1 材料与方法
1.1病毒样本 2015年瑞丽市DF患者急性期血清标本,经DENV C/prM基因检测和血清型鉴定[2]为DENV-4阳性的血清标本。
1.2病毒分离 采用白纹伊蚊C6/36 细胞(军事医学科学院流行病和微生物研究所提供)培养法分离病毒。取100 μL阳性患者血清,用细胞培养液1∶10稀释,接种于已长成单层的C6/36细胞,置4% CO2恒温培养箱28 ℃培养。每日于倒置显微镜下观察细胞病变(CPE),待75% 以上细胞出现CPE,收取细胞上清液,低温冰箱保存待鉴定。
1.3RT-PCR和序列测定 用Axygen公司的AxyPrep Body Fluid Viral DNA/RNA Miniprep Kit 提取病毒分离物中的病毒RNA,用美国Invitrogen公司的SuperScript○RIII One-Step RT-PCR System with Platinum○RTaq Kit 试剂盒一步法RT-PCR扩增DENV全基因序列,将上述PCR产物进行1% 琼脂糖凝胶电泳并割胶回收, 直接测序。引物为自行设计,根据GenBank中的DENV-4基因序列,利用MEGA6软件进行比对,找出相对保守区域,利用Primer Premier软件设计DENV-4全基因序列的扩增引物(表1)。引物合成和扩增产物的测序均由昆明硕挚生物科技有限公司完成。
表1 DENV-4全基因序列引物信息
Tab.1 Specific primers for DENV-4 used in this study

引物名Primer引物序列Sequencedata(5′-3′)DEV4⁃1FAGTTGTTAGTCTGTGTGGACCGDEV4⁃1013RTAGCACCAGATCGACCCATGDEV4⁃662FAACACCACATTCAGGAATGGGDEV4⁃2622RCAGTGAGGTCATGTCCTCCTTCDEV4⁃2280FTTTGGAGGAGTCTCATGGATGDEV4⁃4337RTGTTATGTCTGCCATCTCATCCDEV4⁃3946FCAACAATGCCATTGGTCATGDEV4⁃6301RGGCCTGAGTTTTTTCTTCTCTCDEV4⁃6085FGAGAGTTCCGCCTCAGAGGDEV4⁃7771RATCCCTCTTTCAACAATCCATCDEV4⁃7362FCTCTTGATGAGAACAACATGGGDEV4⁃9088RCCTTCCACTCCACTCCATGAGDEV4⁃8804FGGAAGAACAGGGATGGACATCDEV4⁃10650RAGAACCTGTTGGATCAACAACA
1.4序列分析 采用ClastalX1.83软件进行核苷酸序列比对,采用MEGA6软件邻接法(neighbor-joining)进行病毒基因核苷酸序列系统发生树分析[14]。病毒核苷酸和氨基酸序列同源性分析、氨基酸位点分析及系统进化分析均引用GenBank中14株DENV-4(表2),其中基因I型(GenotypeI, G-I)8株,基因II型(G-II)、基因III型(G-III)和sylyatic型各2株。H241株为DENV-4原型株(prototype strain),除与其它13株参考株同时用于同源性及进化树分析外,并作为氨基酸位点差异比对分析所参照的标准株。
表2 本研究引用的登革4型病毒参考株的背景信息
Tab.2 Background information of 14 strains of DENV-4 from GenBank
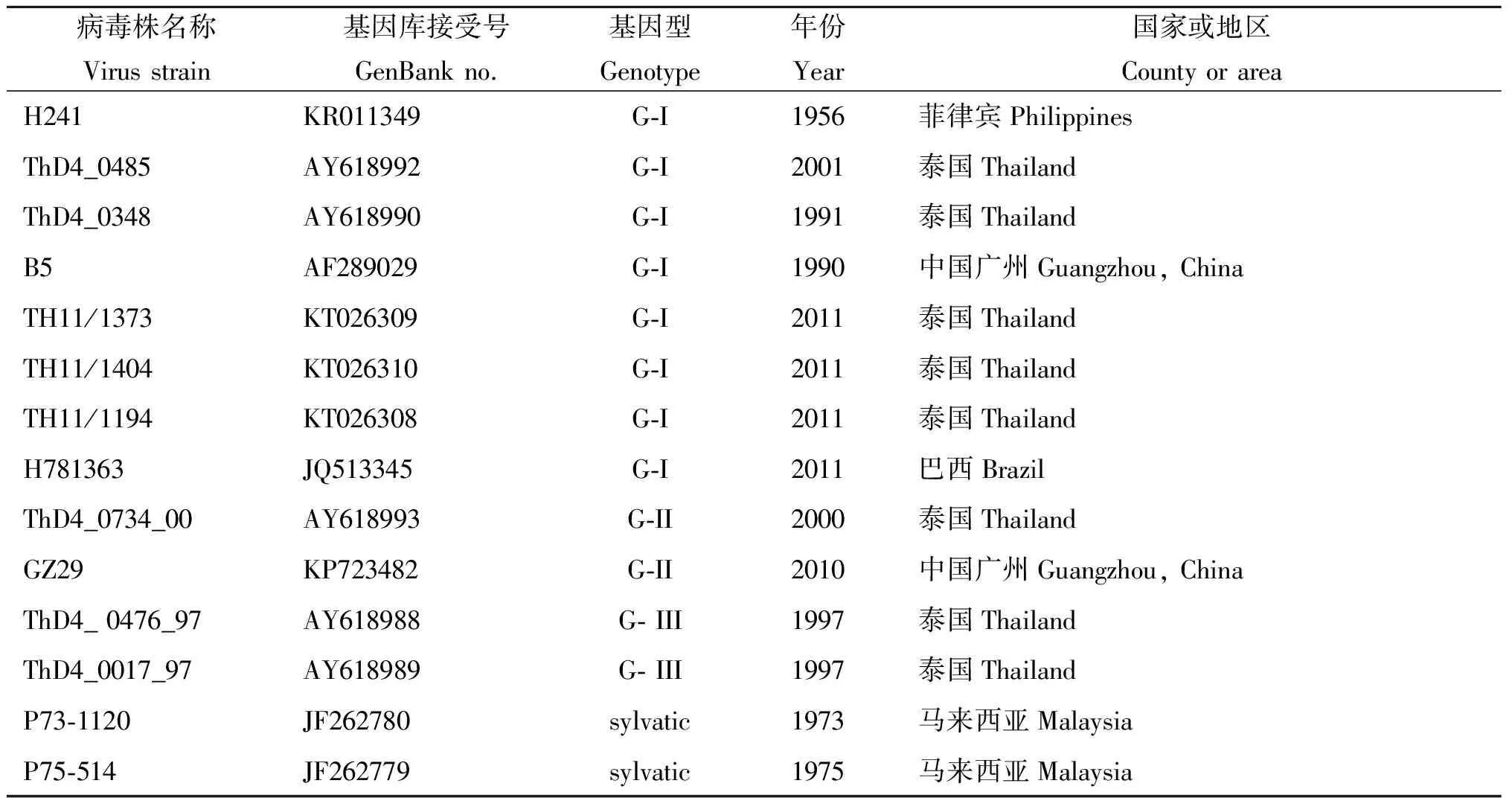
病毒株名称Virusstrain基因库接受号GenBankno.基因型Genotype年份Year国家或地区CountyorareaH241KR011349G⁃I1956菲律宾PhilippinesThD4_0485AY618992G⁃I2001泰国ThailandThD4_0348AY618990G⁃I1991泰国ThailandB5AF289029G⁃I1990中国广州Guangzhou,ChinaTH11/1373KT026309G⁃I2011泰国ThailandTH11/1404KT026310G⁃I2011泰国ThailandTH11/1194KT026308G⁃I2011泰国ThailandH781363JQ513345G⁃I2011巴西BrazilThD4_0734_00AY618993G⁃II2000泰国ThailandGZ29KP723482G⁃II2010中国广州Guangzhou,ChinaThD4_0476_97AY618988G⁃III1997泰国ThailandThD4_0017_97AY618989G⁃III1997泰国ThailandP73⁃1120JF262780sylvatic1973马来西亚MalaysiaP75⁃514JF262779sylvatic1975马来西亚Malaysia
2 结 果
2.1病毒分离 将DENV-4核酸阳性的DF患者急性期血清接种C6/36 细胞,共分离到5株DENV-4,它们均能引起C6/36 细胞典型CPE。其中2株分离自本地感染病例,3株分离自缅甸输入性病例(表3)。
表3 云南省5株登革4型病毒的背景信息
Tab.3 Background information of 5 strains of DENV-4 isolated from Yunnan Province, China
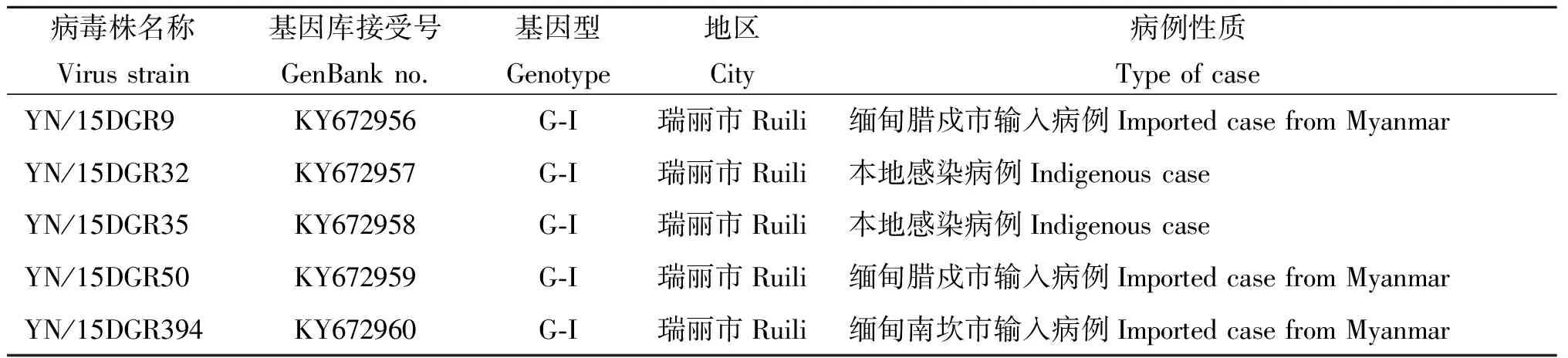
病毒株名称Virusstrain基因库接受号GenBankno.基因型Genotype地区City病例性质TypeofcaseYN/15DGR9KY672956G⁃I瑞丽市Ruili缅甸腊戍市输入病例ImportedcasefromMyanmarYN/15DGR32KY672957G⁃I瑞丽市Ruili本地感染病例IndigenouscaseYN/15DGR35KY672958G⁃I瑞丽市Ruili本地感染病例IndigenouscaseYN/15DGR50KY672959G⁃I瑞丽市Ruili缅甸腊戍市输入病例ImportedcasefromMyanmarYN/15DGR394KY672960G⁃I瑞丽市Ruili缅甸南坎市输入病例ImportedcasefromMyanmar
2.2序列测定 对本次分得的5株DENV-4进行全基因组序列测定,获得这5株病毒的全基因组核苷酸序列,基因组全长为10 661 nt,其中非编码区5′UTR和3′UTR分别为102 nt和397 nt。开放读码框(103-10 264)编码 3 386个氨基酸,包括3个结构蛋白(C、prM/M、E)和7个非结构蛋白(NS1、NS2a、NS2b、NS3、NS4a、NS4b和NS5)。云南株DENV-4全基因核苷酸序列均已提交GenBank(表3)。
2.3进化分析 云南5株DENV-4与来自GenBank中14株不同基因型DENV-4参考株的核苷酸序列构建系统进化树,图1、2和3显示,云南分离株无论在全基因组(comeplete genome)还是在C/prM/M、E、NS1、NS2a、NS2b、NS3、NS4a、NS4b和NS5基因核苷酸序列均高度聚集在一个进化支,并与泰国不同年代的DENV-4 G-I流行株(ThD4_0485、ThD4_0348、TH11/1373、TH11/1404和TH11/1194)位于同一进化群,具有较近的亲缘关系,它们的基因型同属G-I。所有云南株均与DENV-4原型株(H241)和中国广州1990年B5株同处一个大分支,但亲缘关系较远。此外,云南株均与DENV-4的其它基因型(G-II、G-III和sylvatic)病毒株的进化关系较远(图1和2)。本次云南株非编码区3′UTR的进化分析结果与编码区结构和非结构蛋白分析结果基本相似(图2)。
2.4同源性分析 云南5株DENV-4与14株DENV-4参考株的E基因进行核苷酸和氨基酸序列同源性分析。表4显示,云南5株DENV-4的E基因核苷酸(氨基酸)同源性为98.84%~100.0%(99.18%~100.0%),它们间不仅高度同源并与泰国流行株(TH11_1194、TH11_1373、TH11_1404、ThD4_0348和ThD4_0485)E基因核苷酸(氨基酸)同源性为95.95%~98.08%(98.57%~99.59%),同属G-I。云南株与同基因型的DENV-4原型H241株E基因核苷酸(氨基酸)同源性仅为92.14%~94.84%(96.48%~96.90%),与广州1990分离的B5株也仅为91.24%~91.96%(96.48%~96.90%)。云南株与G-II(ThD4_0734和GZ29株)E基因核苷酸(氨基酸)同源性为90.23%~90.65%(96.27%~96.90%),与G-III
(ThD4_0476_97和ThD4_0017_97株) E基因核苷酸(氨基酸)同源性为88.84%~89.27%(96.27%~96.90%),与sylvatic基因型(P73-1120和P75-514株)E基因核苷酸(氨基酸)同源性为84.14%~84.42%(94.52%~95.00%)。
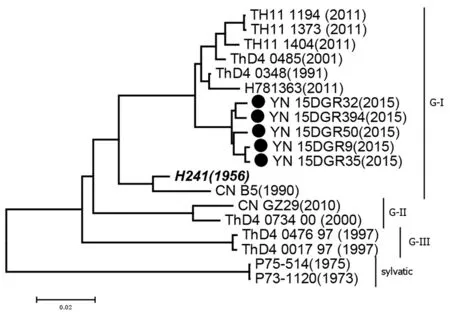
注:黑色圆形为分离自云南省的DENV-4Black circles represent the strains isolated from Yunnan Province, China图1 云南省5株DENV-4 全基因组序列进化分析Fig.1 Phylogenetic tree based on the comeplete genome of 5 DENV-4 strains isolated from Yunnan Province, China
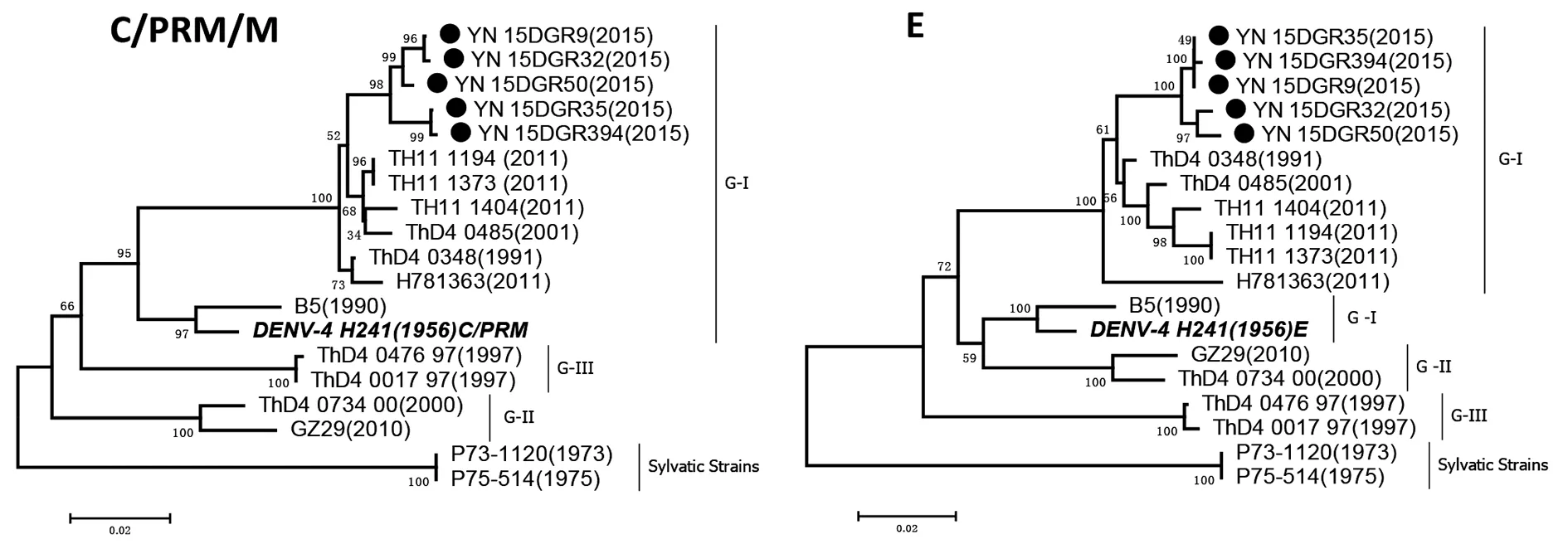
注:黑色圆形为分离自云南省的DENV-4Black circles represent the strains isolated from Yunnan Province, China图2 云南省5株DENV-4 C/PrM/M和E基因序列进化分析Fig.2 Phylogenetic tree based on the C/PrM/M and E genes of 5 DENV-4 strains isolated from Yunnan Province, China
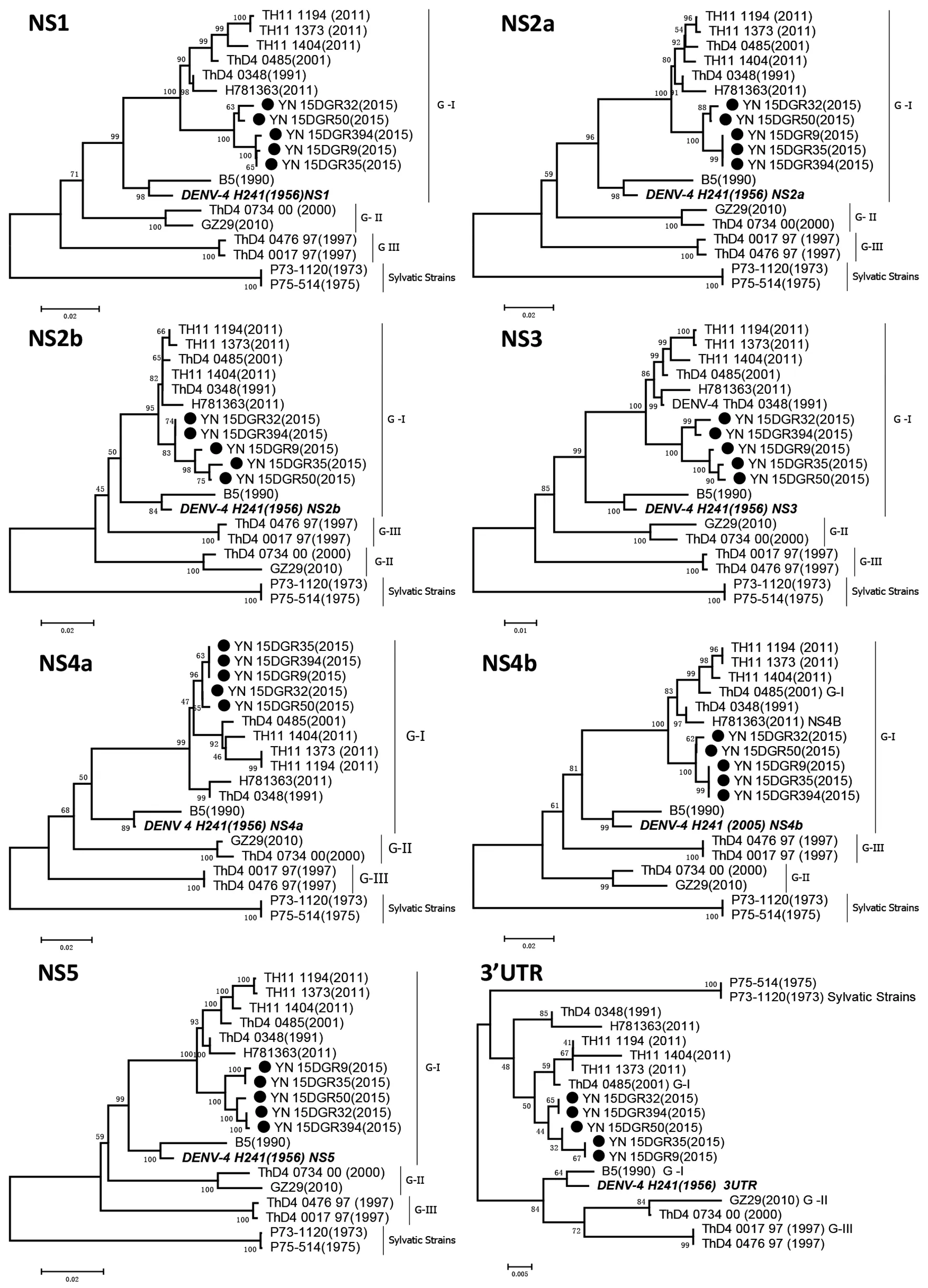
注:黑色圆形为分离自云南省的DENV-4Black circles represent the strains isolated from Yunnan Province, China图3 云南省5株DENV-4 NS1、NS2a、NS2b、NS3、NS4a、NS4b、NS5和3′UTR基因序列进化分析Fig.3 Phylogenetic tree based on the NS1, NS2a, NS2b, NS3,NS4a, NS4b, NS5 and 3′UTR genes of 5 DENV-4 strains isolated from Yunnan Province, China
表4 云南DENV-4分离株与DENV-4参考株E基因核苷酸和氨基酸同源性比较
Tab.4 Homology comparisons of the E gene sequences of dengue serotype 4 viruses and Yunnan isolates in Yunnan Province, China
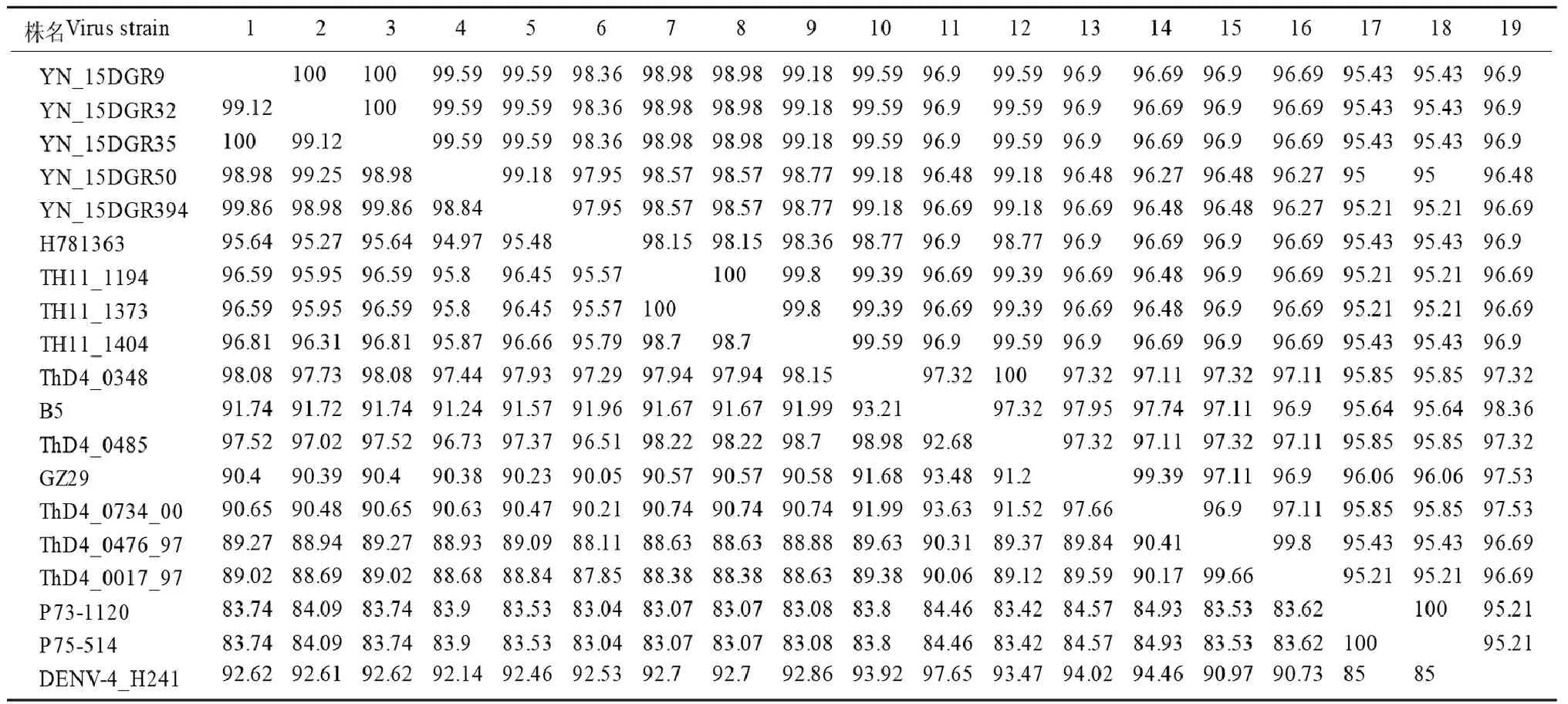
注:左下三角数据为E基因核苷酸序列同源性百分率,右上三角数据为E基因氨基酸序列同源性百分率。
Note: The percentages of nucleotide sequence identities of the genome are presented on the lower left side; the percentages of amino-acid sequence identities of the genome are presented on the upper right side.
此外,还对云南5株DENV-4与14株参考株的NS5基因进行同源性比较分析,结果表明,云南株间的NS5基因核苷酸(氨基酸)同源性为98.45%~99.85%(99.09%~99.89%),并与前述5株泰国流行株NS5基因核苷酸(氨基酸)同源性为96.30%~98.07%(98.86%~99.32%),同样均为G-I。云南株与同基因型的DENV-4原型H241株和广州1990分离的B5株的NS5基因核苷酸(氨基酸)同源性分别仅为94.67%~94.84%(98.05%~98.51%)和92.83%~93.04%(97.70%~98.17%)。云南株与前述G-II、G-III和sylvatic基因型NS5基因核苷酸(氨基酸)同源性较低,依次仅为90.60%~91.14%(96.54%~97.24%)、90.00%~90.35%(97.12%~97.59%)和84.14%~84.42%(94.52%~95.00%)。
2.5氨基酸差异位点分析 云南5株DENV-4与13株DENV-4参考株进行全编码区氨基酸位点差异分析,并与原型株(H241)进行氨基酸位点比对。结果发现,无论结构蛋白或非结构蛋白的氨基酸残基均存在不同程度的差异。云南株结构蛋白共有21个氨基酸位点发生改变,其中C、prM/M、E分别有2、4和15个位点改变(表5)。如所有云南株和各基因型参考株均在prM/M-16、prM/M-52、E-155、E-156、E-221、E-329、E-360、E-402和E-461位点分别出现R-K、E-K、I-T、P-S、A-T、A-T、Y-N、L-F 和F-L的改变;所有云南株和东南亚G-I参考株均在C-102、prM-156、E-96、E-203、E-227、E-233、E-265和E-478位点依次出现M-I、V-I、V-M、K-T、S-L、Y-H、T-A 和T-S的改变;仅有云南5株在E-21和E-384分别发生W-G和D-E改变;仅云南2株在C-99出现R-K改变。此外,表5空白处为该氨基酸位点与H241株相同,未发生改变。
云南株与H241株在非结构蛋白中共发现45个氨基酸位点存在差异,其中NS1、NS2a、NS2b、NS3、NS4a、NS4b和NS5依次有11、2、3、13、1、3和12个位点改变。在NS1中所有云南株和所有基因型参考株均在NS1-2(T-M)、NS1-51(E-K)、NS1-66(I-V)和NS1-129(K-R)有改变;所有云南株和东南亚G-I参考株在NS1-128(A-T)、NS1-135(I-V)有改变;仅云南株在NS1-153(L-F)、NS1-214(K-R)、NS1-251(F-V)和NS1-265(V-L)发生改变。云南株和东南亚G-I参考株在NS3共同存在6个表位的改变;仅云南株NS3有8个表位的改变,其中株间存在差异。NS2a、NS2b、NS4a、NS4b位点仅有较少改变。所有云南株和东南亚G-I参考株在NS5中的NS5-53(H-Y)、NS5-59(S-T)、NS5- 200(K-R)、NS5-256(V-A)、NS5-335(I-V)、NS5-631(Q-R)和NS5-826(T-I)均发生改变,仅5株云南株在NS5-51(I-T)、3株云南株在NS5-269(T-I)和2株云南株在NS5-628(V-A)、NS5-651(C-Y)出现改变。
表5 DENV-4云南株与H241株结构蛋白(C、prM/M和E)氨基酸差异位点比较
Tab.5 Different sites of amino acid in structural protein (C,prM/M和E ) sequences in Yunnan strains and H241 strain of DENV-4
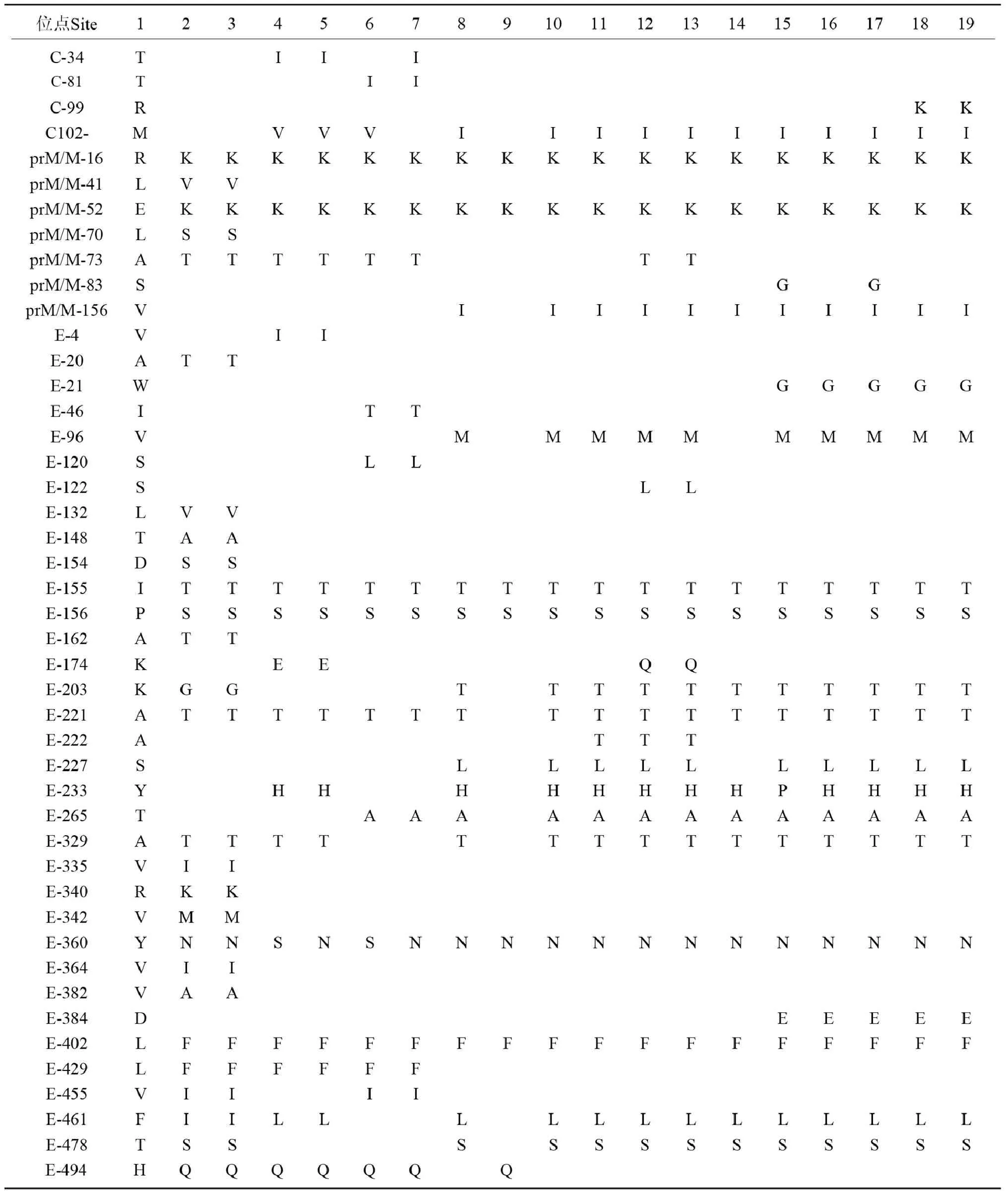
注:阿拉伯数字表示本研究中的病毒株名称。
Note:Arabic numeral represent the virus strain names in the study: 1: H241; 2: P75-514; 3: P73-1120; 4: ThD4_0017_97; 5: ThD4_ 0476_97; 6: ThD4_0734_00; 7: GZ29; 8: ThD4_0485; 9: B5; 10: ThD4_0348; 11: TH11/1404; 12: TH11/1373; 13: TH11/1194; 14: H781363; 15: YN/15DGR394; 16: YN/15DGR50; 17: YN/15DGR35; 18: YN/15DGR32; 19: YN/15DGR9.
3 讨 论
东南亚地区广泛存在登革4种血清型病毒的流行[1,15],对云南省构成威胁。云南省西南边境地区自2013年以来相继发生过来自相邻东南亚国家的DENV-1、DENV-2和DENV-3感染的输入性病例引起的本地DF流行[8-13],其中瑞丽市于2013-2016年连续4年发生DENV-1和DENV-2流行[8,9,13]。20世纪80年代,曾从云南省西南边境地区白纹伊蚊中分离到DENV-4[16-17],表明云南省局部边境地区曾发生过该血清型病毒的流行。近几年云南省曾经由来自缅甸的输入性病例中检测到DENV-4(2013年昆明和2014年瑞丽)[13,18],本研究首次从中缅边境重要口岸城市瑞丽市的本地感染病例和缅甸输入性病例中同时分离到DENV-4,证实2015年该市存在DENV-4引起的本地DF流行,此为首次证实云南省存在DENV-4本地流行。DENV-4同源性和进化分析表明,瑞丽市无论来自缅甸的输入性病例或本地感染病例的病毒株,它们间均具有较高的同源性和较近的进化关系,且基因型均为G-I,并发现这些流行株均与东南亚G-I流行株亲缘关系最为接近,同属一个进化群。由此认为,导致2015年瑞丽DENV-4本地流行的传播来源为缅甸输入性病例,同时还间接证实,与中国瑞丽紧邻的缅甸北部边境地区的南坎市和腊戍市2015年均存在DENV-4 G-I流行。至此,云南省已发生过登革4种血清型病毒的DF本地流行,其中瑞丽除DENV-3外,其它3种血清型病毒均有本地流行,今后应加强DF跨境传播及本地流行的综合防控工作。瑞丽多种血清型病毒的共同流行很有可能形成DF地方性流行区,当地具有引发重症DF或登革出血热(dengue haemorrhagic fever)的风险,疾控和医疗部门对此应予以高度重视。
近几年,我国南方多以DENV-1和DENV-2流行。新中国成立后我国首次于1978年在广东佛山市暴发DF疫情,其病原体为DENV-4,基因型为G-II(78-42和78-56株)[19],随后广州2010年也发生了该血清型的G-II流行[20]。期间1990年广州株(B5)和2005年深圳株(SZ0524)DENV-4则为G-I,并发现SZ0524株与H241株和B5株的E基因核苷酸(氨基酸)同源性则分别高达99.7%(99.2%)和97.7%(98.8%),相似度较高[21]。本次进化分析发现,云南株与同为G-I的1956年分离自菲律宾的DENV-4原型株(H241)和1990年广州的B5株亲缘关系较远,核苷酸和氨基酸同源性也较低。本次氨基酸差异位点分析还发现,与H241株相比,云南株和东南亚G-I参考株间在某些位点的改变既有共性也有不同,且云南株与H241和B5株间也有较多差异位点。这些结果表明,不同时期和地域的DENV-4 G-I流行株间存在明显差异,并随着流行时间的推移该基因型病毒变异更为明显,此可能与DENV在自然界传播中与宿主共进化有关。由此认为,开展不同年代和不同地区的DENV监测对掌握自然界流行株的遗传进化和变异均有重要意义。
目前认为E 蛋白中的结构域III区(氨基酸303~395位点)、II区(52~136 和190~284)及II区和III区分界面之间的区域(294~305)与DENV的抗原性和毒力有关[22-24]。本次云南株E基因中有15个氨基酸位点的改变,其中属于III区(E-329、E-360、E-384)和II区(E-96、E221、E-203、E-227、E-233、E265)的位点有9个,但它们可能不属于关键位点。刘志文等[25]对DENV-4自然分离株和实验室减毒株的比较发现,E-155位氨基酸由T(苏氨酸)转变为 I(异亮氨酸)后,可能会使病毒毒力增强,反之毒力减弱,认为该位点与毒力相关。本次云南株中的E-155位与毒力较强的原型株(H241)相比则由I变为T,似乎意味着当前云南流行株相较于原型株在毒力上有减弱现象。对于上述位点的变异与抗原性或毒力的关系以及非结构蛋白某些氨基酸位点改变的意义尚需进一步研究。
[1] Bhatt S, Gething PW, Brady OJ, et al. The global distribution and burden of dengue[J]. Nature, 2013, 496(7446): 504-507. DOI:10.1038/nature12060
[2] Bureau of Disease Control and Prevention, Health Department of the People’s Republic of China. Handbook of prevention and control for dengue fever[M]. 2nd ed. Beijing: People’s Health Press, 2008: 13-23. (in Chinese)
卫生部疾病预防控制局. 登革热防治手册[M]. 2版. 北京: 人民卫生出版社,2008:13-23.
[3] Rico-Hesse R. Molecular evolution and distribution of dengue viruses type 1 and 2 in nature[J]. Virology, 1990, 174(2): 479-493.
[4] Zhang YJ, Zhou CH, Huang M, et al. Genomic analyses of dengue viruses and origins of dengue strains from China[J]. Chin J Zoonoses, 2012, 28(6): 515-520. DOI:10.3969/j.issn.1002-2694.2012.06.001 (in Chinese)
张拥军,周朝晖,黄萌,等. 登革病毒的基因组分析及中国登革毒株的来源[J]. 中国人兽共患病学报,2012,28(6):515-520.
[5] Zhang HL, Fu SH, Deng Z, et al. An outbreak of imported dengue fever from Myanmar to the border of China, with its viral molecular epidemiological features[J]. Chin J Epidemiol, 2013, 34(5):428-432.DOI: 10.3760/cma.j.issn.0254-6450.2013.05.004(in Chinese)
张海林,付士红,邓掌,等. 云南中缅边境一起输入性登革热暴发的分子流行病学研究[J]. 中华流行病学杂志,2013, 34(5):428-432.
[6] Guo X, Zhao Q, Chao WC, et al. First isolation of dengue virus from Lao PDR in a Chinese traveler[J]. Virol J, 2013, 10:70. DOI:10.1186/1743-422X-10-70
[7] Yang WH, Huang Y, Feng Y, et al. Investigation on dengue fever imported cases of Kunming City, Yunnan Province in China and sequence analysis of C/PreM gene of dengue virus[J]. Intl JVirol, 2015, 22(1):11-16.DOI:10.3760/cma.j.issn.1673-4092.2015.01.003 (in Chinese)
杨卫红,黄瑛,冯云,等. 昆明市登革热输入性病例的调查及C/PreM基因序列分析[J]. 国际病毒学杂志,2015, 22(1):11-16.
[8] Feng Y, Liu YH, Yin ZL, et al. Molecular epidemiologic analysis of an outbreak of dengue fever in 2013 at Ruili city, Yunnan province of China[J]. Chin J Viral Dis, 2014, 4(4):306-311. (in Chinese)
冯云, 刘永华, 尹正留, 等. 云南省瑞丽市2013年登革热暴发的分子流行病学研究[J]. 中国病毒病杂志, 2014, 4(4):306-311.
[9] Liu YH, Feng Y, Yin XX, et al. Molecular epidemiology of dengue virus types 1 and 2 in Ruili, Yunnan, 2014[J]. Dis Surveill, 2016, 31(1):8-13.DOI:10.3784/j.issn.1003-9961.2016.01.004 (in Chinese)
刘永华,冯云,尹小雄,等. 2014年云南省瑞丽市登革1和2型病毒流行的分子流行病学研究[J]. 疾病监测,2016, 31(1):8-13.
[10] Feng Y, Fan JH, Zhu J, et al. Molecular epidemiology of an outbreak of dengue fever in Jinghong City, Yunnan Province, China, 2013[J]. Chin J Epidemiol, 2014, 35(12):1409-1411.DOI:10.3760/cma.j.issn.0254-6450.2014.12.0023 (in Chinese)
冯云,范建华,朱进,等. 云南省景洪市2013年登革热暴发的分子流行病学研究[J]. 中华流行病学杂志, 2014, 35(12):1409-1411.
[11] Li YY, Feng Y, Fan JH, et al.Investigation on an outbreak of dengue serotype 2 virus in Xishuangbanna prefecture of Yunnan province in 2015[J]. Chin J Exp Clin Virol, 2016, 30(4):340-345. DOI:10.3760/cma.j.issn.1003-9279.2016.04.002(in Chinese)
李园园,冯云,范建华,等. 2015年云南省西双版纳州登革2型病毒暴发疫情的调查研究[J]. 中华实验和临床病毒学杂志,2016, 30(4):340-345.
[12] Li HC, Pan H, Feng Y, et al.Epidemiological survey of an outbreak of dengue fever in Lincang, Yunnan, 2015[J]. Dis Surveill, 2016, 31(7):561-565.DOI:10.3784/j.issn.1003-9961.2016.07.007 (in Chinese)
李华昌,潘虹,冯云,等.2015年云南省临沧市登革热暴发的流行病学调查[J]. 疾病监测,2016, 31(7):561-565.
[13] Feng Y, Zhang HL, Pan H, et al. Diversity of serotypes and genotypes and molecular tracing of dengue viruses isolated from dengue fever cases in Yunnan[J]. Chin J Exp Clin Virol,2017, 31(1):24-30. DOI:10.3760/cma.j.issn.1003-9279.2017.01.005 (in Chinese)
冯云,张海林,潘虹,等.云南省登革病毒血清型和基因型多样性及溯源研究[J]. 中华实验和临床病毒学杂志,2017,31(1):24-30.
[14] Tamura K, Stecher G, Peterson D, et al. MEGA6: Molecular evolutionary genetics analysis version 6.0[J]. Mol Biol Evolut,2013, 30(12):2725-2729.DOI:10.1093/molbev/mst197
[15] Thu HM, Lowry K, Myint TT, et al. Myanmar dengue outbreak associated with displacement of serotypes 2, 3, and 4 by dengue 1[J]. Emerg Infect Dis, 2004,10(4):593-597.
[16] Zhang HL, Zi DY, Gong ZD. Epidemiological survey of dengue fever in Yunnan Province China[J]. Endem Dis Bull, 1999, 14(3):50-54. (in Chinese)
张海林,自登云,龚正达. 云南省登革热流行病学调查分析[J]. 地方病通报,1999, 14(3):50-54.
[17] Wang JL, Zhang HL, Sun XH, et al. Identification and sequence analysis of NS1 and NS2a of two strains of dengue type-4 virus from mosquitoes in Yunnan Province[J]. Chin J Zoonoses, 2008, 24(7):636-640. (in Chinese)
王静林,张海林,孙肖红,等. 云南登革4型病毒的鉴定及NS1和NS2a基因序列分析[J]. 中国人兽共患病杂志,2008, 24(7):636-640.
[18] Huang Y, Fu SH, Shi L, et al. Detection of mosquito-borne arbovirus infectious in sixty two feverish patients of Yunnan[J]. Chin J Exper Clin Virol, 2016, 30(4):351-354. DOI: 10.3760/cma.j.issn.1003-9279.2016.04.004 (in Chinese)
黄瑛,付士红,史蕾,等.云南省62例发热患者中蚊传虫媒病毒感染的病原学检测[J].中华实验和临床病毒学杂志,2016, 30(4):351-354.
[19] Liu ZW, Yu YX, Jia LL, et al. Sequence determination and phylogenetic analysis of two dengue virus type 4 strains isolated in the first outbreak of dengue fever after the foundation of the People’s Republic of China[J]. Chin J Zoonoses, 2007, 23(7):678-682.(in Chinese)
刘志文,俞永新,贾丽丽,等. 建国后首次登革热暴发分离的2株登革4型病毒prM和E基因序列测定及其系统发生分析[J]. 中国人兽共患病学报,2007,23(7):678-682.
[20] Zhao H, Yu XD, Zhang XY, et al. Complete genome sequence of a dengue virus serotype 4 strain isolated in Guangdong, China[J]. J Virol, 2012, 86(12):7021-7022. DOI:10.1128/JVI.00858-12
[21] Yang F, He JF, Xian HX, et al. Sequence determination and phylogenetic tree analysis of the E gene of dengue virus type 4 isolated from a patient in Shenzhen[J]. Chin J Zoonoses, 2010, 26(1): 17-20. DOI:10.3969/j.issn.1002-2694.2010.01.005 (in Chinese)
阳帆,何建凡,冼慧霞,等. 登革4型病毒深圳分离株E基因序列测定及其系统发生分析[J]. 中国人兽共患病学报, 2010, 26(1):17-20.
[22] Bray M, Men R, Tokimatsu I, et al. Genetic determinants responsible for acquisition of dengue type 2 virus mouse neurovirulence[J]. J Virol, 1998, 72(2):1647-1651.
[23] Rey FA, Heinz FX,Mandl C, et al. The envelope glycoprotein from tick-borne encephalitis virus at 2 A resolution[J]. Nature, 1995, 375(6529):291-298.
[24] Gromowski GD, Barrett ADT. Characterization of an antigenic site that contains a dominant, type-specific neutralization determinant on the envelope protein domain III (ED3) of dengue 2 virus[J]. Virology, 2007, 366:349-360.
[25] Liu ZW, Yu YX, Jia LL, et al. Study on the molecular characteristics of two dengue type 4 virus strains, Ban18 strain and its attenuated strain Ban18-30[J]. J Pathog Biol, 2008, 3(5): 343-347. (in Chinese)
刘志文, 俞永新, 贾丽丽, 等. 登革4 型病毒 Ban18 原株及其减毒株的分子特征分析[J]. 中国病原生物学杂志, 2008, 3(5): 343-347.
Molecularcharacteristicsofthefull-lengthgenomeofdengueserotype4virusstrainsisolatedfromdenguefevercasesinYunnanProvince,China
HU Ting-song1, ZHANG Hai-lin1, LIU Yong-hua2, DENG Bo1, YIN Xiao-xiong2, XU Song-miao1,LI Ping2, FAN Quan-shui1, ZHANG Fu-qiang1
(1.CenterforDiseaseControlandPrevention,ChengduMilitaryRegion,Kunming650118,China;2.RuiliCenterforDiseaseControlandPrevention,Ruili678600,China)
We investigated the molecular characteristics of the full-length genome of 5 dengue serotype 4 virus (DENV-4) strains isolated in Yunnan Province, China, 2015 and their molecular epidemiological feature.Isolation of dengue virus was using C6/36 cell culture method. Viral RNA was extracted from virus isolates, then the full-length genome was amplified by RT-PCR. The homology and phylogenetic analysis was made on the nucleotide and deduced amino acid sequences by bioinformatics softwares including ClastalX1.83 and MEGA6 etc. Results showed that five strains of DENV-4 isolated from dengue fever cases in Ruili City of Yunnan Province in 2015, of these, 2 strains from indigenous cases, 3 from imported cases from Lashio and Nam Can cities of Myanmar to Ruili of China. RT-PCR and sequencing indicated that the full-length genes (10 661 nt) of 5 DENV-4 strains were obtained, and their open reading frame (103-10 264) were coded 3 386 amino acid residues. Phylogenetic tree and homology analysis based on the comeplete genome or structural and non-structural protein genes showed that the 5 DENV-4 isolates were highly homologous and gathered in an evolution as well as they have a closer genetic relationship with the DENV-4 genotype I (G-I) strains isolated from Thailand. Results indicated that the Yunnan strains belonged to G-I.Yunnan strains and Thailand strains compared with DENV-4 prototype strain (H241, Philippines 1956) and Guangzhou strain (B5, 1990) of China and showed low homology and evolutionary relationship.When Yunnan strains compared with H241 strain, there were 21 and 45 different sites in amino acid of structural and non-structural proteins, respectively.This is the first time in Yunnan to obtain full-length genomes sequence of DENV-4 and they have closer evolutionary relationship with DENV-4G-I strains from Southeast Asia region in recent years. The autochthonous DENV-4 epidemic in Yunnan was detected for the first time, and the virus transmission sources were from neighboring northern Myanmar. It is necessary to further study that change of the amino acid sites of Yunnan strains of DENV-4 is related to its antigenicity and virulence.
dengue serotype 1 virus; full-length genome; phylogenetic analysis; homology analysis; amino acid site analysis
s: Zhang Fu-qiang, Email: zfq1968@aliyun.com; Fan Quan-shui, Email:fqs168@126.com
10.3969/j.issn.1002-2694.2017.10.002
国家十三五重大专项(No.2016YFC1200100),全军青年培育项目(No.15QNP035)和中国博士后基金项目(No.2015M582907和2016T91009)联合资助
张富强,Email: zfq1968@aliyun.com
范泉水,Email:fqs168@126.com
1.成都军区疾病预防控制中心,昆明 650118;2.瑞丽市疾病预防控制中心,瑞丽 678600
R373.3
A
1002-2694(2017)10-0859-09
Supported by the National Key Basic Research Program of China (No. 2016YFC1200100), the Science and Technology Programs of Military (No. 15QNP035), and the General Financial Grant from the China Postdoctoral Science Foundation(No. 2015M582907 and 2016T91009)
2017-04-17编辑李友松

