牛支原体与无乳支原体的相似性比对分析
韩杨 罗海霞 马春骥 金华 李敏 郝秀静

摘要:运用生物信息学方法对牛支原体(Mycoplasma bovis)和无乳支原体(Mycoplasma agalactiae)的相似性进行多重比对分析。使用Mega 7、Clustal a、Sibeilia等生物信息学软件对M.bovis与M.agalactiae的全基因组、16S rRNA、脂蛋白家族进行相似性比对分析。通过多重比对发现,M.bovis与M.agalactiae在全基因、16S rRNA、以及脂蛋白家族方面均表现出较高的相似性。
关键词:牛支原体;无乳支原体;全基因组比对分析
中图分类号:S858.236.3文献标识码:A文章编号:1000-4440(2020)02-0398-06
Abstract: This study aims to conduct multiple comparative analysis on the similarity of Mycoplasma bovis and Mycoplasma agalactiae with the method of bioinformatics. M.bovis and M. agalactiae were compared for similarity in their whole genome, 16S rRNA, and lipoprotein families using bioinformatics softwares such as Mega 7, Clustal a and Sibeilia. Through the multiple alignment, M.bovis and M.agalactiae showed higher similarity in terms of whole gene, 16S rRNA and lipoprotein family.
Key words:Mycoplasma bovis;Mycoplasma agalactiae;whole genome alignment analysis
支原体为自然界中独立生存的最小原核微生物,擁有能够进行自我复制的最小基因组[1]。支原体能攻击宿主细胞,引起宿主细胞的免疫反应并损害宿主细胞,以便在宿主细胞内生长繁殖[2]。牛支原体(Mycoplasma bovis)是一种主要感染牛呼吸道的病原体[3],能够持续感染宿主[4],并引发包括牛肺炎在内的多种慢性疾病[5],如乳腺炎、中耳炎、生殖障碍、关节炎[6]、脑膜炎以及角膜结膜炎等[7]。M.bovis与无乳支原体(Mycoplasma agalactiae)在16S rRNA序列上有较高的相似性[8-11]。M.agalactiae也可以感染牛,并引发乳腺炎、角膜结膜炎和关节炎等疾病[12],但主要会引起小型反刍动物的“传染性无乳症”(CA)综合征[13]。所以在特异性检测病原[14]甚至是根据特定的病原研制疫苗[15]预防疫病时就需要对二者进行精准区分[16]。为此我们拟通过对M.bovis与M.agalactiae的全基因组序列比对,探究二者在基因组层面的相似性,寻找两者独特的基因,为M.bovis快速检测方法研究奠定基础,为鉴定物种特异性提供更有效的证据。此外,我们还想探究二者分离株在16S rRNA上的亲缘关系,以及主要毒力因子膜表面脂蛋白家族的相似性。
1材料与方法
1.1材料来源
从美国国立生物技术信息中心(National Center for Biotechnology Information, NCBI)中找到牛支原体(Mycoplasma bovis)与无乳支原体(Mycoplasma agalactiae)的全基因组序列。
1.2统计分析
运用生物信息学软件Mega 7构建M.bovis与M.agalactiae的16S rRNA、膜表面脂蛋白家族的系统进化树,用Clustal w进行多重序列比对,用OrthoMCL v2.0.3对M.bovis(GenBank:CP002188.1)与M.agalactiae(GenBank:CU179680.1)的全基因组编码的氨基酸序列进行比对(由北京基华生物技术服务有限公司进行)。使用OAT-ANI软件对M.bovis与M.agalactiae进行全基因组共线性分析。
2结果
2.1M.bovis与M.agalactiae的基因组共线性
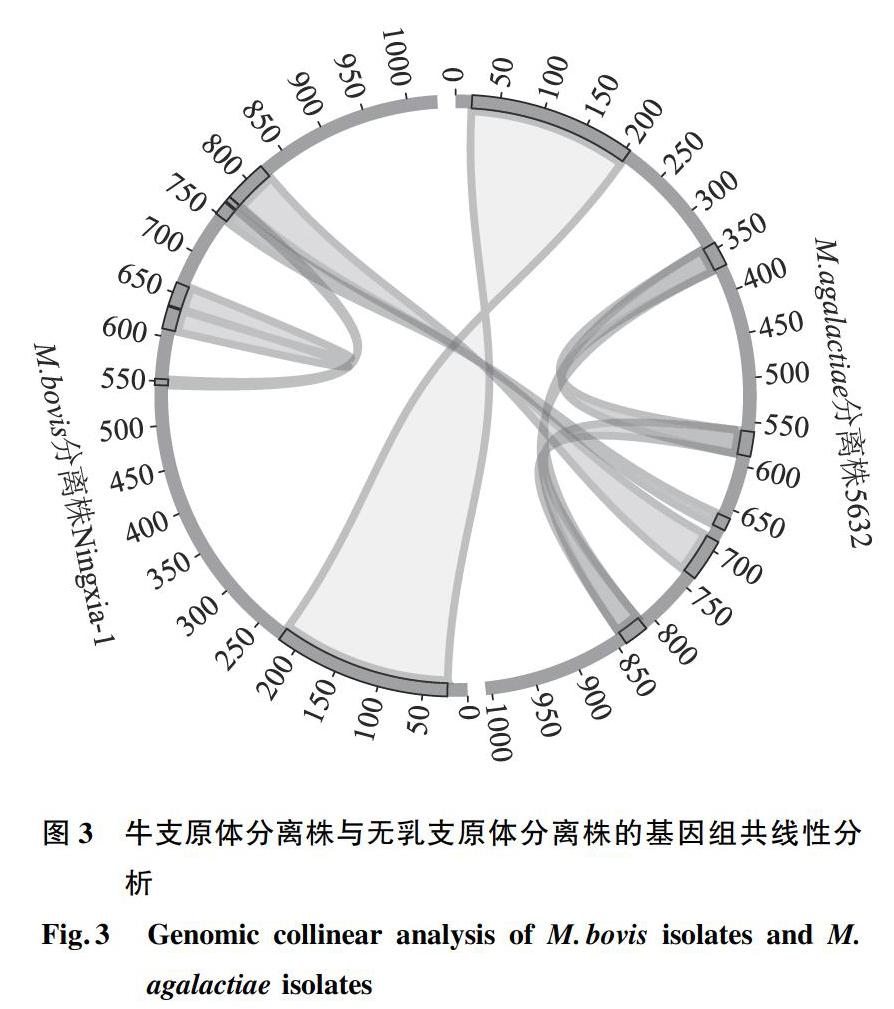
运用生物信息学软件Sibelia对M.bovis的标准株PG45与M.agalactiae的标准株PG2进行全基因组共线性分析。由共线性圆形图(图1)可以发现,PG45与PG2之间有4个位置的基因具有共线性关系,说明M.bovis与M.agalactiae的标准株具有一定的相似性。
在M.bovis与M.agalactiae标准株表现出共线性的前提下,对M.bovis标准株PG45与M.agalactiae分离株5632进行全基因组共线性分析(图2),对M.bovis分离株Ningxia-1与M.agalactiae分离株5632也进行了全基因组共线性分析(图3)。结果表明M.bovis与M.agalactiae不仅在标准株中具有共线性关系,在分离株中同样存在共线性关系,说明二者在基因组共线性层面上具有相似性。
2.2M.bovis与M.agalactiae的全基因组比对分析
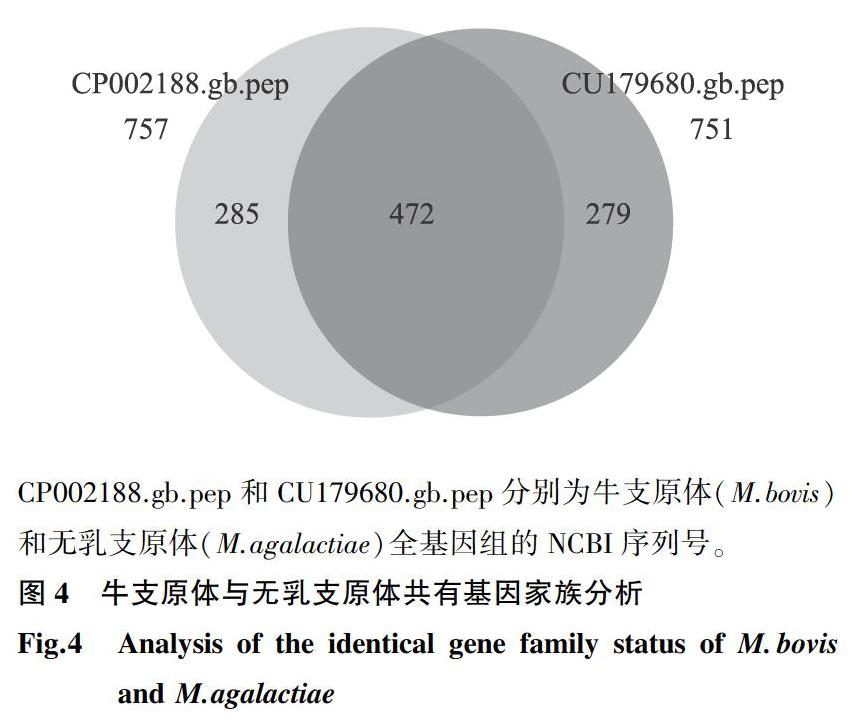
采用OrthoMCL v2.0.3软件对M.bovis(GenBank:CP002188.1)与M.agalactiae(GenBank:CU179680.1)的全基因组编码的氨基酸序列进行比对,选取阈值(BLASTP E值不大于1×10-5,MCL_INFLATION值=1.5)进行相似性聚类,获得同源基因的列表,以确定M.bovis与M.agalactiae的同源性。
比对结果显示, M.bovis与M.agalactiae之间共有基因家族472个。使用MUSCLE v3.7软件(http://www.ebi.ac.uk/Tools/msa/muscle/)将每个单拷贝基因家族中各成员进行全序列比对。结果(图4)表明,牛支原体(M.bovis)特有基因数量为285,无乳支原体(M.agalactiae)特有基因数量为279,牛支原体与无乳支原体共有的基因家族数量为472。M.bovis与M.agalactiae共有的基因家族高达472个,说明二者具有较高的相似性和较近的亲缘关系。
为了进一步探究M.bovis与M.agalactiae在基因组层面的相似性,使用生物信息学软件Jspecies对M.bovis与M.agalactiae进行全基因组平均核苷酸序列同源(ANI)分析。平均核苷酸序列同源性是通过比较物种全基因组同源基序列判定物种间遗传关联性的重要参数。对M.bovis及M.agalactiae全基因组序列比对分析后得到的ANI值高达83.08%,再次说明M.bovis与M.agalactiae具有较高的相似性。
2.3支原体16S rRNA系统进化树
在NCBI中找到M.bovis标准株PG45和M.agalactiae标准株PG2的16S rRNA碱基序列,并运用生物信息学软件Clustal w对二者进行多重序列比对(图5),发现相似性高达97%。由于二者16S rRNA碱基序列相似度过高,为了探究M.bovis与M.agalactiae间的亲缘关系,在NCBI中找到较常见的11种支原体16S rRNA碱基序列,并找到NCBI中M.bovis与M.agalactiae其余分离株的16S rRNA碱基序列,使用生物信息学软件MAGA 7构建系统进化树(图6),由进化树可以看到M.bovis与M.agalactiae无论标准株还是分离株,均位于同一分枝上,且亲缘关系较近。
2.4M.bovis与M.agalactiae的16S rRNA相似性
对16S rRNA进行生物信息学分析后,使用引物设计软件Primer Premier 5随机对M.bovis的16S rRNA碱基序列设计4对引物,同时以M.bovis与M.agalactiae基因組DNA为模板进行PCR扩增。结果(图7)显示,M.bovis与M.agalactiae均扩增出明亮的目的条带,再次证明二者在16S rRNA层面的相似性。
2.5M.bovis与M.agalactiae表面可变脂蛋白家族进化树
在NCBI中找到M.bovis与M.agalactiae的表面可变脂蛋白家族的基因序列,运用生物信息学软件MAGA 7构建M.bovis与M.agalactiae表面可变脂蛋白家族的进化树。结果(图8)显示,M.bovis的Vsps家族可细分为3个亚类,M.agalactiae的Vpma家族与其中一类的亲缘距离较近,表面可变脂蛋白家族进化的层面上说明M.bovis与M.agalactiae的亲缘关系较近。运用生物信息学软件Clustal w,在M.bovis与M.agalactiae的表面可变脂蛋白家族中相似性较高的多条序列中,各选择2条进行多重序列比对(图9),发现M.bovis与M.agalactiae也显示出相似性。说明M.bovis与M.agalactiae不仅在16S rRNA碱基序列上相似,在与感染致病相关的高度变异的表面可变脂蛋白方面上也表现出相似性。
3讨论
支原体会感染人类[17-19]和动物[20-22],而且往往难以根除[23]。虽然治疗支原体病具有较大的社会经济意义[24],但由于缺乏对支原体致病机制[25]的认识,目前正确预防并控制支原体病疫情还未成系统。牛支原体病的高传染性和低治愈率[26]对中国牛养殖业的健康发展造成了巨大威胁,作为宁夏回族自治区农业特色优势产业之一的牛养殖业[27],同样受到该病的潜在威胁[28]。本课题组在建立M.bovis的快速检测方法时,筛查特异性基因的过程中发现M.agalactiae和M.bovis具有极高的相似性。所以本研究运用生物信息学软件分析了M.bovis和M.agalactiae的相似性,这对M.bovis快速检测方法的建立具有实际意义。
全基因组序列比较结果表明M.bovis与M.agalactiae共有的基因家族高达472个,占全基因组的2/3。M.bovis与M.agalactiae的ANI值高达83.08%,二者分属于不同种的支原体,但相似性之高显而易见。16S rRNA碱基序列比对以及进化树分析结果也揭示了M.agalactiae和M.bovis在16S rRNA碱基序列上的相似性。表面可变脂蛋白家族为支原体主要的毒力因子[29]。氨基酸序列比对结果显示, M. agalactiae的Vpma与M.bovis的Vsp具有高度的氨基酸序列相似性。表面可变脂蛋白家族进化树显示,Vpma家族与Vsp家族中的一类亲缘距离较近。对Vsp家族和Vpma家族的相似性分析不仅可以在表面可变脂蛋白家族的层面上验证M.bovis与M.agalactiae的相似性,也可以探究M.agalactiae的致病机制。M.bovis的Vsp蛋白已被证明参与宿主细胞粘附[30],但是关于M.agalactiae Vpma蛋白的确切功能未见报道。通过Vsp家族和Vpma家族的相似性分析,或许可以推测M.agalactiae的Vpma蛋白也参与宿主细胞粘附[31]。多方面比对结果都表明M.bovis与M.agalactiae具有较高的同源性,所以在建立检测方法时要将二者进行区分[32]。
参考文献:
[1]HEGDE S, ZIMMERMANN M, ROSENGARTEN R, et al. Novel role of Vpmas as major adhesins of Mycoplasma agalactiae mediating differential cell adhesion and invasion of Vpma expression variants[J]. International Journal of Medical Microbiology Ijmm, 2018, 308(2): 263.
[2]ERIC B, DOMINIQUE B, EVELINE S, et al. Experimental infections with Mycoplasma agalactiae identify key factors involved in host-colonization[J]. PLoS One, 2014, 9(4): e93970.
[3]JI W H, WU Y Q. Advances on the research of vaccine against Mycoplasma bovis[J]. China Animal Husbandry & Veterinary Medicine, 2018,45(3):763-769.
[4]NICHOLAS R A J, AYLING R D. Mycoplasma bovis : disease, diagnosis, and control[J]. Research in Veterinary Science, 2003, 74(2): 105-112.
[5]BRAS A L, BARKEMA H W, WOODBURY M R, et al. Clinical presentation, prevalence, and risk factors associated with Mycoplasma bovis-associated disease in farmed bison (Bison bison) herds in western Canada[J]. Javma Journal of the American Veterinary Medical Association, 2017, 250(10): 1167-1167.
[6]LIMIYED B P G. Mycoplasma bovis arthritis and pneumonia in calves[J]. Veterinary Record, 2017, 180(11): 272.
[7]CALCUTT M J, LYSNYANSKY I, SACHSE K, et al. Gap analysis of Mycoplasma bovis disease, diagnosis and control: An aid to identify future development requirements[J]. Transboundary & Emerging Diseases, 2018, 65 (1): 91-109.
[8]李媛,董惠,張美晶,等. 牛支原体套式PCR检测方法的建立[J]. 中国预防兽医学报, 2009, 31(9): 709-711.
[9]SULYOK K M, BEK K, KREIZINGER Z, et al. Development of molecular methods for the rapid detection of antibiotic susceptibility of Mycoplasma bovis[J]. Veterinary Microbiology, 2018, 213: 47-57.
[10]HEGDGE S, ZIMMERMANN M, ROSENGARTEN R, et al. Novel role of Vpmas as major adhesins of Mycoplasma agalactiae mediating differential cell adhesion and invasion of Vpma expression variants[J]. International Journal of Medical Microbiology, 2018, 308(2):263.
[11]TIMONEN A A E, KATHOLM J, PETERSEN A, et al. Within-herd prevalence of intramammary infection caused by Mycoplasma bovis and associations between cow udder health, milk yield, and composition[J]. Journal of Dairy Science, 2017, 100(8): 6554-6561.
[12]CUBEDDU T, CACCIOTTO C, PISANU S, et al. Cathelicidin production and release by mammary epithelial cells during infectious mastitis[J]. Vet Immunol Immunopathol, 2017, 189: 66-70.
[13]TATAY-DUALDE J, HAM P V D, FE C D L, et al. Mutations in the quinolone resistance determining region conferring resistance to fluoroquinolones in Mycoplasma agalactiae[J]. Veterinary Microbiology, 2017, 207: 63-68.
[14]PARKERR A M, SHEEHY P A, HAZELTON M S, et al. A review of mycoplasma diagnostics in cattle[J]. Journal of Veterinary Internal Medicine, 2018,32(3):1241-1252.
[15]季文恒,吴娅琴,翟肖辉,等. 牛支原体疫苗的研究进展[J]. 中国畜牧兽医, 2018, 45(3): 763-769.
[16]MANSO-SILVN L, DUPUY V, LYSNYANSKY I, et al. Phylogeny and molecular typing of Mycoplasma agalactiae and Mycoplasma bovis by multilocus sequencing[J]. Veterinary Microbiology, 2012, 161(1/2): 104-112.
[17]KUMAR S. Mycoplasma pneumoniae: A significant but underrated pathogen in paediatric community-acquired lower respiratory tract infections[J]. Indian Journal of Medical Research, 2018, 147(1):23-31.
[18]MOYNIHAN K M, BARLOW A, NOURSE C, et al. Severe Mycoplasma pneumoniae infection in children admitted to pediatric intensive care[J]. Pediatric Infectious Disease Journal, 2018, 37: 1.
[19]SNDERGAARD M J, FRIIS M B, HANSEN D S, et al. Clinical manifestations in infants and children with Mycoplasma pneumoniae infection[J]. PLoS One, 2018, 13(4): e0195288.
[20]GILLE L, CALLENS J, SUPR K, et al. Use of a breeding bull and absence of a calving pen as risk factors for the presence of Mycoplasma bovis in dairy herds[J]. Journal of Dairy Science, 2018,101(9):8289-8290.
[21]管禮麟,马媛,杨丹茹,等. 绵羊肺炎支原体P208蛋白质的分子特征分析及免疫原性[J]. 江苏农业学报, 2018,34(1):81-86.
[22]徐引弟,焦文强,王治方,等. 鸡毒支原体河南株的分离鉴定及生物学特性研究[J]. 山东农业科学,2018, 50(11):124-128.
[23]KHAN F A, RASHEED M A, FAISAL M, et al. Proteomics analysis and its role in elucidation of functionally significant proteins in Mycoplasma bovis[J]. Microbial Pathogenesis, 2017, 111: 50.
[24]ROSENGARTEN R, CITTI C, GLEW M, et al. Host-pathogen interactions in mycoplasma pathogenesis: Virulence and survival strategies of minimalist prokaryotes[J]. International Journal of Medical Microbiology Ijmm, 2000, 290(1): 15-25.
[25]BRKI S, FREY J, PILO P. Virulence, persistence and dissemination of Mycoplasma bovis[J]. Veterinary Microbiology, 2015, 179(1/2): 15-22.
[26]张文劲,张文皓,唐鑫,等. 牛支原体免疫学及免疫学诊断研究进展[J].中国兽医学报,2019,39(9):1868-1872.
[27]邵倩. 宁夏地区永宁县牛支原体病流行病学调查及防治技术研究[D].兰州:甘肃农业大学,2017.
[28]郭亚男,曹思婷,何生虎,等. 2013-2015年宁夏地区牛支原体感染的血清学调查分析[J]. 动物医学进展, 2017, 38(4): 129-133.
[29]CITTI C, BLANCHARD A. Mycoplasmas and their host: emerging and re-emerging minimal pathogens[J]. Trends in Microbiology, 2013, 21(4): 196-203.
[30]ZHAO G, ZHANG H, CHEN X, et al. Mycoplasma bovis NADH oxidase functions as both a NADH oxidizing and O2 reducing enzyme and an adhesin[J]. Scientific Reports, 2017, 7(1): 44.
[31]CHOPRA-DEWASTHALY R, SPERGSER J, ZIMMERMANN M, et al. Vpma phase variation is important for survival and persistence of Mycoplasma agalactiae in the immunocompetent host[J]. PLoS Pathogens, 2017, 13(9): e1006656.
[32]WAWEGAMA N K, BROWNING G F. Improvements in diagnosis of disease caused by Mycoplasma bovis in cattle[J]. Animal Production Science, 2017, 57(7): 1482.
(责任编辑:张震林)

