山西省酸枣遗传多样性及遗传结构分析
胡晓艳,杜淑辉,王兆山,韩有志*
(1. 山西农业大学林学院,北方功能油料树种培育与研发山西省重点实验室,山西 太谷 030800;2. 中国林业科学研究院林业研究所,北京 100091)
野生种质资源具有丰富的遗传变异,能抵御各类生物或非生物胁迫的影响,其蕴含的有益基因对于栽培植物遗传改良至关重要。然而,由于生境破坏以及全球气候变化等对野生居群存续和自然进化的影响,其保护甚为迫切[1]。开展野生种质资源遗传多样性研究有助于制定其保护策略。酸枣(Ziziphus jujuba var. spinosa (Bunge) Hu ex H.F. Chow)是栽培枣的近缘野生种质,在我国分布范围广泛,华北、西北、东北和华东等地区均有分布,集中分布在山西、陕西、河北、山东等地[2]。酸枣适应能力强,抗旱耐寒、耐贫瘠,树势强健,根系深广发达,具有很强的水土保持作用。由于人为活动的影响和资源的不合理开发利用,酸枣的分布正日益缩减[3]。开展酸枣种质资源遗传多样性研究,将为其有效保护和可持续利用提供科学依据,同时推动枣相关育种研究进程[4]。山西省是我国红枣主要产区之一,也是酸枣主要分布区之一,但是目前酸枣资源面积逐年减少,开发利用严重不足[3]。
单拷贝核基因标记(single-copy nuclear gene markers)具有易于扩增测序、含有大量SNPs、不存在谱系分选等优点,目前在植物遗传多样性及谱系地理研究中得到广泛应用[5-6]。栽培枣基因组序列的公布[7-8],使得利用生物信息学方法挖掘枣属物种中通用的单拷贝核基因标记成为可能。本研究筛选酸枣中的单拷贝核基因标记并分析山西省酸枣居群的遗传多样性和遗传结构,为后续酸枣资源的保护及可持续利用奠定基础。
1 材料和方法

图1 采样点分布图Fig. 1 Distribution of sample sites in Shanxi
1.1 样品采集
本研究采集了山西省14 个野生酸枣居群213 个单株的叶样。采样地点、居群编号、样品个数等具体信息见图1、表1。选取居群内生长势良好、无病虫害的单株,且相邻单株间距至少100 m,采集新鲜叶片置于变色硅胶中带回实验室用于总DNA 的提取。
1.2 标记筛选与扩增测序
使用改良的CTAB 法提取总DNA。所提DNA经1.0% 琼脂糖凝胶电泳检测后于-20℃储存备用。采用以下方法筛选酸枣中的单拷贝核基因标记:(1)利用Duarte 等[9]提供的单拷贝核基因标签在GENE(http://www.ncbi.nlm.nih.gov/gene)中检索,获得相应的核苷酸序列;(2)将核苷酸序列在枣基因组中进行BLAST 检索(https://blast.ncbi.nlm.nih.gov/Blast.cgi?PAGE_TYPE=BlastSearch&PR OG_DEF=blastn&BLAST_PROG_DEF=megaBlast&BLAST_SPEC=OGP__326968__182558),以确定枣基因组中与其同源的序列,将此同源序列确定为参考序列;(3)利用参考序列进行核苷酸BLAST(http://blast.ncbi.nlm.nih.gov/Blast.cgi) 以确定所选基因在枣基因组中为单拷贝;(4)根据选定的参考序列,利用Primer Premier 5.0 软件设计引物。通过检索分析初步筛选了50 个单拷贝核基因标记并设计了相应引物。选取3 个居群总共15 个样品(DTYG、JZTG、YCRC 各5 个样品)对筛选出的单拷贝核基因标记的变异情况进行检测。PCR 扩增采用25 μL 体系:50 ng DNA,dNTP 0.24 mmol·L-1,Mg2+2.0 mmol·L-1, Taq 酶1.5 U, 引 物0.24 μmol·L-1(所有试剂均为北京擎科生物科技有限公司生产)。PCR 扩增程序为:94℃预变性5 min;94℃变性30 s,退火90 s(退火温度随引物而定),72℃延伸90 s,30 个循环;72℃延伸5 min;10℃保存。扩增产物用1.5% 琼脂糖凝胶电泳检测后采用DNA 纯化试剂盒( Amersham Pharmacia Biotech,Piscataway, USA) 纯化后, 利用ABI3730 DNA analyzer (Applied Biosystems, Foster City, Califonia,USA) 直接测序。扩增和测序采用相同引物。在标记筛选时未选用含有poly-结构的参考序列,因此,不存在需要使用克隆测序的情况。当所选标记在15 个样品中扩增或测序结果差,或不包含变异信息时,弃用该标记。最终确定6 个单拷贝核基因标记对所有样品进行扩增测序(表2),PCR 扩增和测序步骤与标记筛选时一致。经过扩增测序得到了所有酸枣样品在6 个单拷贝核基因标记的序列数据。

表1 酸枣居群采样信息 Table 1 Sampled populations of Z. jujuba var. spinosa in this study
1.3 数据统计与分析
测序结果处理时遵循简并碱基的原则。所有序列数据采用软件ClustalX[10]比对,并在BioEdit[11]中进行校正后输出用于后续分析的数据文件。利用DnaSp5.10[12]对每个单拷贝核基因标记计算如下参数:多态核苷酸位点数目(S)、单倍型多样性(Hd)[13]、π(一对序列间每碱基平均差异值)[14]、基于多态核苷酸位点数目S的Watterson’s 参数θw[15]、Tajima’D中性检验(显著性检验由999 次联合模拟计算得到)[16]、重组事件(Rm) 和居群间基因流Nm。利用DnaSp5.10 计算居群水平的遗传多样性参数S、Hd、π和θw。同时利用DnaSp5.10中的失配分布分析(Mismatch distribution analysis)[17]检验居群历史动态,当居群大小保持相对稳定时,失配分布曲线会呈多峰分布;当居群经历扩张或持续增长时,失配分布曲线一般会呈现单峰。利用Arlequin 3.5.1.3[18]中的分子方差分析(AMOVA) 计算在每个单拷贝核基因标记物种内居群间以及居群内差异对总变异的贡献率(显著性检验利用999次重复模拟得到)和居群间遗传分化指数Fst[19](显著性检验利用999 次重复模拟得到),同时计算两两居群间遗传距离。使用GenAlEx 6.5[19]中的Mantel test检验遗传距离和地理距离之间的相关性。采用STRUCTURE 2.3.4[20]进行群体水平的遗传结构分析,参数设置如下:选用混合模型(admixture model) 和群体间相关联的等位基因频率模型( correlated alleles frequencies), length of burnin 长度设置为500 000,马尔科夫链蒙特卡洛迭代次数(MCMC replicates after burn-in)设置为2 000 000,分类数目(K)值范围为1~10,每个K值重复计算10 次。利用最大值ΔK对应的K值确定为最佳K值[21]。
2 结果
2.1 山西酸枣遗传多样性
6 个单拷贝核基因标记的长度在199~846 bp 之间,分离位点数目和单倍型多样性的变化区间为7~64 和0.663~0.979;核苷酸多样性参数π和θw的平均值分别为0.007 20 和0.009 25;Rm的平均值为8,表明酸枣基因组内重组事件发生频率较高(表3)。在居群水平上,单倍型多样性平均值为0.986,π和θw的平均值分别为0.006 50 和0.005 43,多样性较高的居群如YQYX(π=0.007 89,θw=0.007 19),较低的如XZDX(π=0.005 02,θw=0.003 48)(表4)。

表3 6 个单拷贝核基因标记多样性Table 3 Nucleotide variation of single-copy nuclear gene markers

表4 14 个山西省酸枣居群遗传多样性水平Table 4 Nucleotide variation of 14 Z. jujuba var. spinosa populations in Shanxi
2.2 山西酸枣遗传结构
AMOVA 分析结果(表5)表明:居群内变异占总变异的85.48%,居群分化指数Fst平均值为0.145,表明山西省酸枣居群间基因交流平均水平较低,这与Nm分析结果基本一致(Nm平均值为1.33)。居群间遗传距离结果(表6)表明:XZDX和DTYG 间遗传距离最大(0.360),YCRC 和CZQX 间最低(0.025)。Mantel test 检验表明:居群间地理距离与遗传距离没有相关性(r=0.177 5,P=0.216)。STRUCTU 分析显示:当K=2 时,山西省酸枣居群有最佳聚类结果,并没有形成明显的遗传结构,部分居群间个体遗传组成差异较大(如DTYG 和JZTG)(图2)。

表5 山西酸枣居群遗传结构分析Table 5 AMOVA and Fst analysis of Z. jujuba var. spinosa populations in Shanxi

表6 山西酸枣居群间遗传距离Table 6 Genetic differentiation between Z. jujuba var. spinosa populations in Shanxi
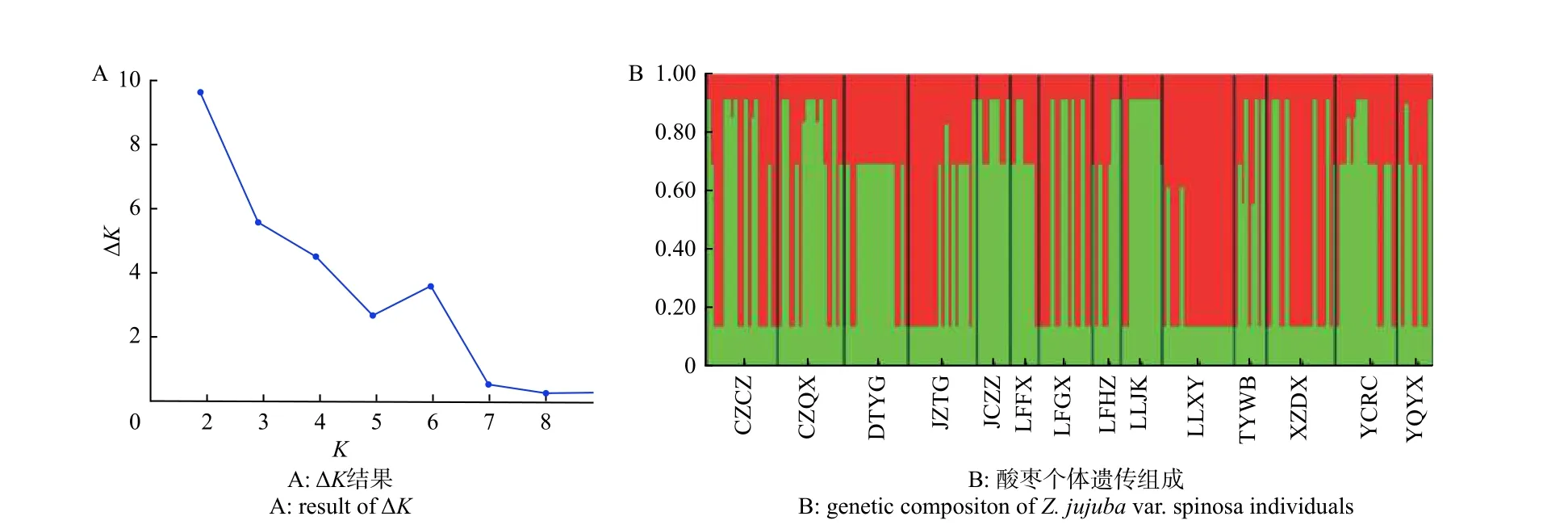
图2 STRUCTURE 运行结果图Fig. 2 Result of STRUCTURE
2.3 山西酸枣居群动态
Tajima’D检验结果均不显著且大部分为负值,表明所选用的单拷贝核基因标记符合中性进化假设,且山西酸枣居群在进化历史上可能经历过居群扩张(表3)。失配分析结果呈现明显的单峰,表明山西省酸枣自然野生居群历史上经历过扩张,这与Tajima’D 检测结果一致(图3)。

图3 失配分布分析结果Fig. 3 Result of mismatch distribution analysis
3 讨论
3.1 酸枣单拷贝核基因标记的筛选
基因组复制事件和高水平的序列变异是导致单拷贝核基因标记筛选困难的主要原因[22]。例如在566 个中国白栎组(section Quercus)单拷贝核基因标记中,仅有19 个标记能够在本组不同物种间通用[5]。虽然枣近期没有发生过全基因组复制事件,但是枣染色体融合与片段复制事件发生频率很高[7],这在一定程度上导致单拷贝核基因标记筛选的困难。标记筛选过程中,在枣基因组中成功检索到50 个单拷贝核基因序列并设计了特异引物,但仅有6 个单拷贝核基因能够在所有样品中成功扩增并测序。据估计,在植物基因组中大约有10%的基因为单拷贝[9]。在枣基因组注释到的32 808 个功能基因中[7],单拷贝基因的个数在3 000 个左右,因此,未来能够挖掘更多的单拷贝基因标记应用于酸枣和枣居群遗传学和谱系地理学等相关研究中。
3.2 山西酸枣的遗传多样性
在位点水平上,分布于山西的酸枣表现出较高的遗传多样性水平(π=0.007 20,θw=0.009 25),比在我国分布的其他物种如中国山杨(Populus davidiana Dode)(π=0.004 40,θw=0.007 50)[23]、甘蒙柽柳(Tamarix austromongolica Nakai)(π=0.002 59)[24]、沙芥[Pugionium cornntum (L.) Gaertn](π=0.005 32)[25]的遗传多样性水平高。使用其他类型分子标记技术的研究(如SSR、RAPD 等)也表明自然分布的酸枣具有较高的遗传多样性水平,积累了丰富的遗传变异[26-27]。物种遗传多样性水平受取样代表性、自然选择、繁育系统及居群历史等因素的影响[23]。本研究的取样范围覆盖山西省,因此,可以排除取样代表性对本研究结果的影响。Tajima’D 中性检验结果表明,6 个单拷贝核基因标记均符合中性进化假设,因此,可以排除自然选择的影响。酸枣为异交植物,异交可以增加物种的有效群体大小和有效重组率,从而提高遗传多样性水平[28],并且自然状态下酸枣居群内很少发生近交繁殖[27]。第四纪冰期循环对北半球物种的多样性与分布产生了重大的影响[29-30],但在我国黄土高原地区冰期循环对物种的影响非常小,冰盖也仅仅在黄土高原南部部分地区形成,持续时间非常短[31]。因此,分布于山西省的酸枣受第四纪冰期循环的影响非常小,可以保留大量的遗传变异。同时,本研究中失配分布分析表明,山西省酸枣居群经历过居群扩张,也支持了上述推论。
3.3 山西酸枣的遗传结构
本研究发现,分布于山西省的酸枣居群间遗传距离差异较大(0.025~0.360)(表6),表明酸枣居群之间的基因交流存在较大的变化,由于Mantel test 表明地理距离与遗传距离之间没有相关性,因此,这可能与居群之间是否存在明显的地理隔离(如山脉、河流)或人类活动等因素相关。地理隔离可以阻碍居群之间的基因交流,而人类活动有时可以打破这种阻碍[32],因此,山西省酸枣居群间遗传距离差异是上述因素共同作用的结果。整体上居群间表现出较高水平的遗传分化(Fst=0.145)且基因流较小(Nm=1.33),但居群内的变异是总体变异的主要来源(表3、5)。张春梅等[27]研究了黄河流域3 个酸枣居群的遗传分化水平,发现3 个居群间分化水平非常低(Fst=0.017~0.039)且基因流较高,推测黄河水流携带种子传播是导致黄河流域3 个酸枣居群出现低分化水平的主要原因。Zhang等[33]进一步对全国范围内酸枣居群的遗传分化水平进行研究,发现酸枣居群间遗传分化指数Fst为0.12。Zhang 等[33]指出,河流在酸枣居群间基因交流中(种子传播)发挥着重要的作用;但本研究中,采样居群间无河流连接,因此,居群间种子传播的可能性非常小,同时酸枣花粉活力弱,扩散能力不强[34]。上述因素是导致山西省酸枣居群间表现出较高水平遗传分化的主要原因。随着人类活动的加剧,同时由于酸枣在枣培育过程中的使用及其种仁蕴含的药用价值,酸枣资源不合理开发利用的情况日益严重,使酸枣这一宝贵的野生果树资源得不到有效的保护与开发利用。本研究发现,分布于山西中部及南部的酸枣居群普遍表现出较高的遗传多样性水平,因此,在未来一方面应该加强保护,防止多样性丢失;另一方面应加强人工驯化栽培、就地抚育改造等工作,利用好这一宝贵的种质资源[3]。
4 结论
本研究利用生物信息学方法筛选了酸枣中的6 个单拷贝核基因标记并将其应用于山西酸枣居群遗传学相关研究,这为未来我国酸枣种质资源遗传学及谱系地理学等研究提供了重要的技术支持。通过对山西省酸枣居群遗传多样性、遗传结构及居群历史动态的初步分析,发现山西省酸枣表现出较高的遗传多样性水平,繁育系统及居群历史动态是影响遗传多样性的主要原因。山西省酸枣居群之间表现出较高水平的遗传分化,未来应对分布于山西中部及南部地区的酸枣居群加强保护与开发利用。本研究的开展为深入进行山西省及我国酸枣种质资源保护、可持续开发利用等奠定了基础。

