基于SRAP 标记的海雀稗遗传多样性及群体结构分析
洪佳琦,孙宗玖, ,杨志民
(1. 新疆农业大学草业与环境科学学院,新疆 乌鲁木齐 830052;2. 西部干旱荒漠区草地资源与生态重点实验室,新疆 乌鲁木齐 830052;3. 南京农业大学草业学院,江苏 南京 210095)
海雀稗(Paspalum vaginatum),隶属于禾本科(Gramineae)雀稗属,广泛分布于南北半球的沿海地区,在美国南部、非洲、澳大利亚及太平洋沿岸国家最为丰富[1],在中国主要分布于台湾、云南、广东和海南岛等地[2-3]。海雀稗是一种极具开发价值的盐生种质资源,其耐盐性优于其他暖季型草种;并且可在大部分植物生长受到抑制的恶劣环境中正常生长,具有较强的抗逆性和适应性,对土壤有较强的固着力,可防止水土流失[4-5],适宜于土壤改良、滩涂绿化等环境修复[6-8],故海雀稗是滨海滩涂发展草牧业和改良盐碱地的优良草种。国外对海雀稗的育种非常重视,美国的乔治亚州立大学是全球培育海雀稗新品种最多的单位[9]。我国虽有海雀稗分布,但野生种质资源收集不足40 份[10-11],迫切需要开发或引进种质,满足育种需求。
目前,我国对于海雀稗的研究主要集中在国外种质的引种栽培、抗性评价及育种方面[12-13]。在遗传多样性方面,解新明等[4]对广东3 个居群的海雀稗进行RAPD 分析,发现居群间基因组多态性比率高达64.77%,认为我国的海雀稗存在丰富的遗传变异;刘燕等[14]对国内野生与国外引进的42 份海雀稗种质进行SRAP 分析,发现国内与国外种质间遗传相似性很低。虽然国内针对海雀稗种质引种及开发已经开展了部分工作,但总体还较少,特别是对育种而言,缺乏亲本选择可用的遗传信息基础。因此本研究以国内外较广范围内收集到的49 份海雀稗种质为对象,利用SRAP 标记对其进行遗传多样性分析,明确不同种质间的遗传差异程度,为海雀稗育种提供基础遗传信息,也为海雀稗种质核心种质库的建立提供科学依据。
1 材料与方法
1.1 材料
本研究所用49 份海雀稗材料由南京农业大学草业学院提供,其中43 份为2016 年从美国引进(源自12 个国家),5 份为国内收集的海雀稗育成品种,1 份为自主选育新材料。所有供试材料种植于南京农业大学白马教学基地育草圃,株距1.5 m,行距2.0 m,并保持完全自然生长的状态,只定期进行人工除草,不施肥,不修剪,不灌溉,不防治病虫害,具体材料信息如表1 所列。
1.2 研究方法
1.2.1 样品采集
2017 年5 月,于南京农业大学白马教学基地育草圃分别剪取49 份海雀稗的幼嫩叶片1 g,置于2.0 mL EP 管中,用液氮速冻后置于便携式车载冰箱带回实验室,置于–80 ℃冰箱保存,每份材料4 个样,均为单株取样。
1.2.2 DNA 提取、SRAP 反应体系及PCR 扩增产物检测
(1) DNA 提取:采用改良的CTAB 法[15]对供试材料进行基因组DNA 提取。2017 年5 月采集每份材料4 个重复幼嫩叶片,将幼嫩叶均匀混合提取DNA。0.8%琼脂糖凝胶电泳检测DNA 质量,酶标仪检测DNA 原液浓度和纯度,工作液浓度稀释到20 ng·μL−1,−20 ℃保存备用。
(2) SRAP-PCR 反应体系:采用20 μL 体系,包括2 ×PCR mix 10 μL,引物(10 μmol·L−1) 1 μL,DNA(20 ng·μL−1)2 μL,ddH2O 6 μL。
(3) SRAP-PCR 反应程序:94 ℃预变性4 min;94 ℃变性30 s,37 ℃退火45 s,72 ℃延伸45 s (运行5 个循环);94 ℃变性30 s,56 ℃退火45 s,72 ℃延伸2 min(运行30 个循环) 72 ℃延伸7 min,最后4 ℃保存。
(4) PCR 产物检测:采用8%非变性聚丙烯酰胺凝胶电泳,上样量为2.5 μL,电泳上槽缓冲液为1 ×TBE,100 V 电压下电泳2 h,使用10%硝酸银快速银染显色。
1.3 数据分析与处理
运用Gel-pro 软件结合人工方法读带,进行条带统计,建立原始“1、0”原始矩阵,“1”表示存在条带,“0”表示同一位置无条带或不易分辨的弱带。根据试验统计的“1、0”原始矩阵,运用PopGenVer l.3.2 计算多态性位点的百分率(PP)、有效等位基因数(ne*)、Nei 遗 传 距 离(h*)、Shannon 信 息 指 数(I*)。运 用NTSYS-pc 软件计算供试材料的Jaccard 遗传相似系数,采用UPGMA 进行聚类分析,绘制系统发育树状图、PCA 散点分布图。使用Structure 2.3.4 软件对49 份海雀稗材料进行群体遗传结构分析,选取群体遗传结构的最优群体数目。
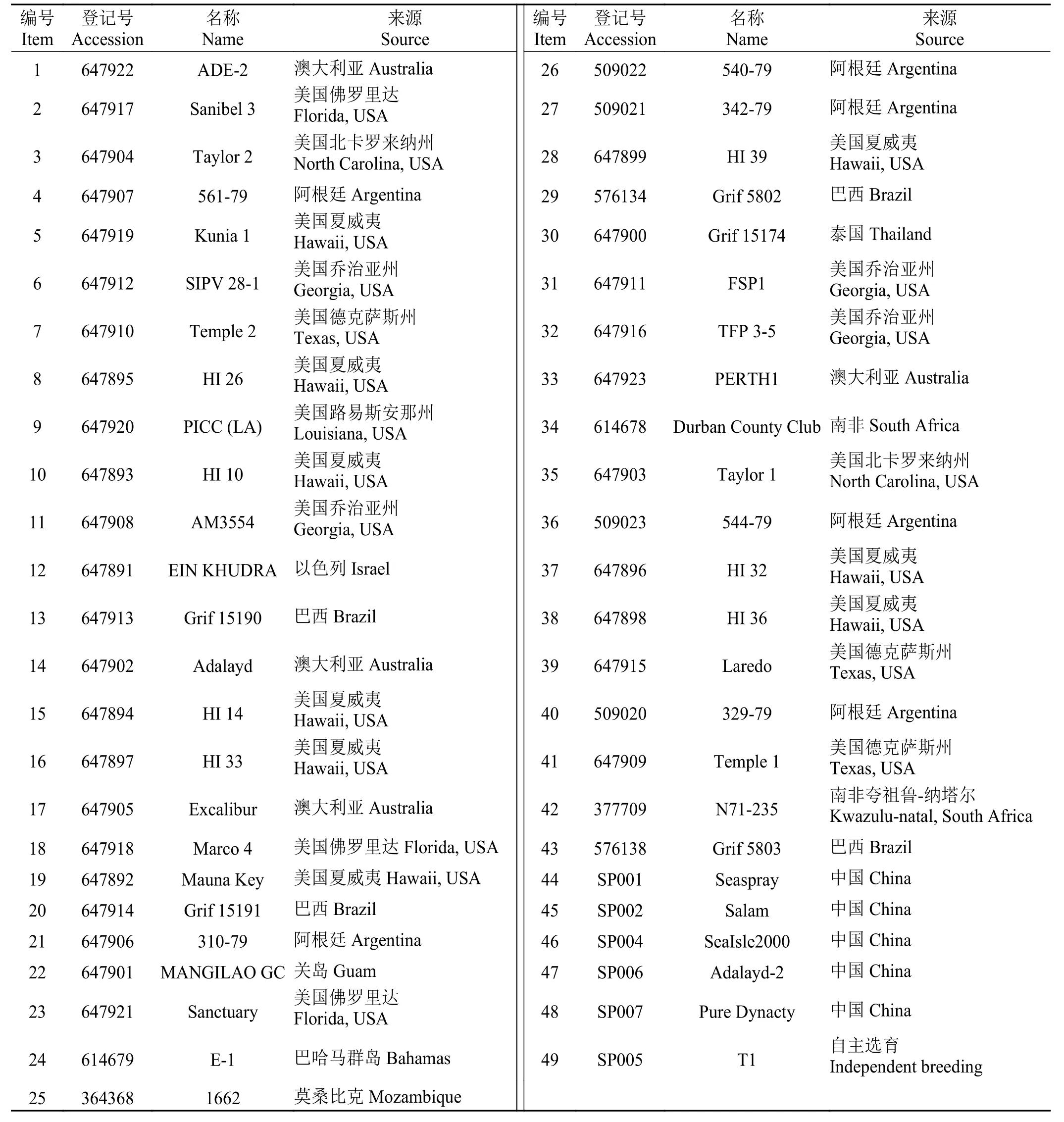
表1 材料编号及来源Table 1 Materials and sources
2 结果与分析
2.1 SRAP 引物多态分析
运用100 对SRAP 引物(表2)对49 份海雀稗种质资源的基因组DNA 扩增,20 对引物可扩增出条带清晰且重复性好的条带,80 对SRAP 不能扩出条带或扩增出不一致的条带。利用筛选出的20 对引物组合对49 份海雀稗种质进行PCR 扩增(图1、表3),共获得169 个条带,产物大小介于100~500 bp,各引物扩增条带为3~12 条,每条引物的平均扩增条带为8.5 条。在169 条DNA 扩增片段中,有118 条多态性条带,每条引物平均扩增多态性条带数为5.9,多态性位点百分率为69.80%。
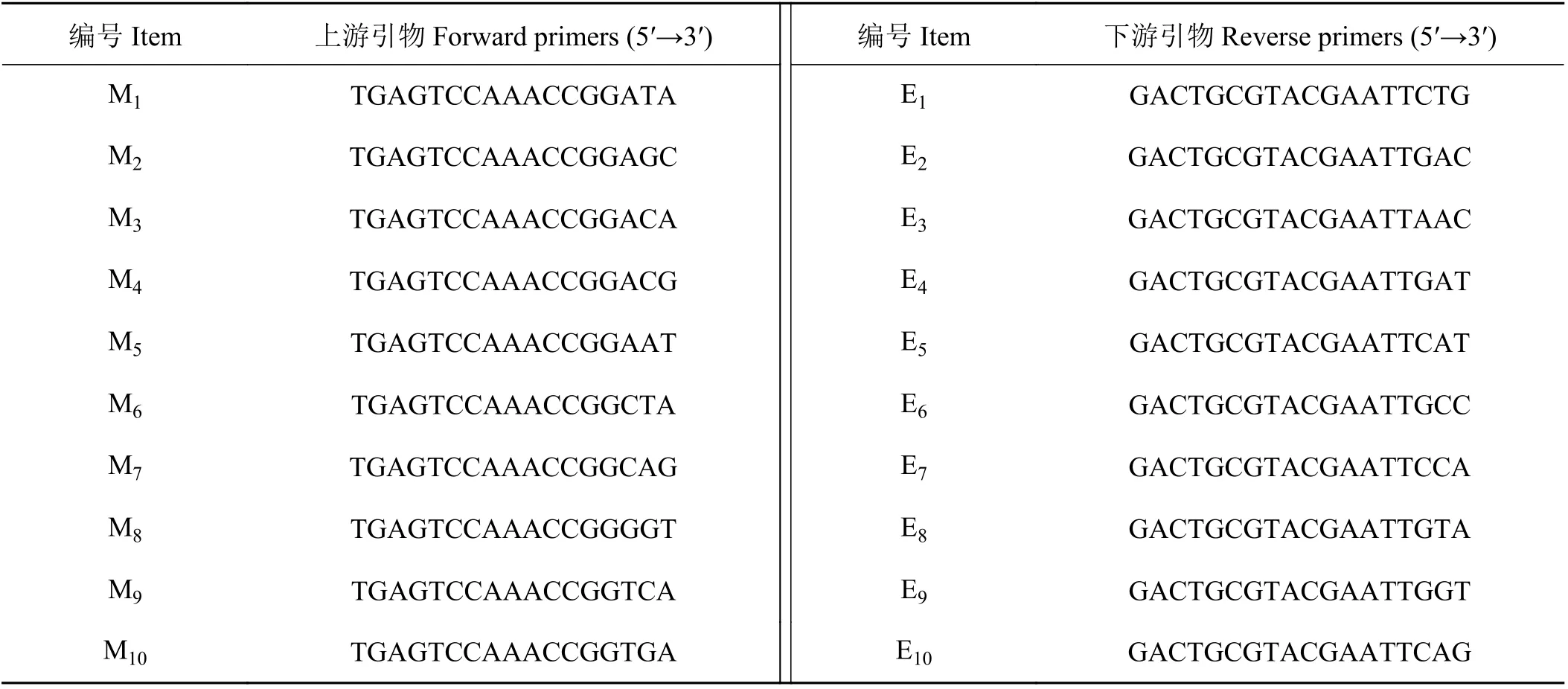
表2 SRAP 引物序列Table 2 Primer sequences used in SRAP analysis

图1 SRAP 引物筛选聚丙烯酰胺凝胶电泳Figure 1 SRAP primer screening using polyacrylamide gel electrophoresis
供试种质有效等位基因数为1.59~1.97,平均1.83;Nei 遗传距离为0.36~0.49,平均0.45;Shannon信息指数的范围在0.55~0.69,平均0.64 (表3),说明供试海雀稗种质资源间存在较丰富的遗传变异。
2.2 聚类分析
利用NTSYS-pc 计算的Jaccard 遗传相似系数,49份海雀稗种质资源的遗传相似系数介于0.52~0.88,平均值为0.67,说明海雀稗种质间存在一定的遗传差异。其中来自巴西的647913 和来自美国夏威夷的647897 相似系数最大(0.88),而来自美国乔治亚州的647908 和美国夏威夷647896 两份材料遗传相似系数最小(0.53),表明它们存在较大遗传差异。
UPGMA 聚类分析可知(图2),在遗传相似系数为0.69 时,可将供试海雀稗材料分为2 大类(Ⅰ、Ⅱ),在遗传相似系数为0.71 时将材料分为四类(A、B、C、D),其中A 类包含4 份材料,B 类包含12 份材料,C 类包含20 份材料,D 类包含13 份材料。
2.3 主成分分析
材料间的亲缘关系可以通过主成分分析(PCA)直观呈现,种质间位置越近表明它们的相似性越高,越远则表明种质间的遗传差异越大。根据统计“1、0”矩阵,用NTsys 软件进行主成分分析,第1、第2 和第3主成分的贡献率分别为16.63%、9.06%和5.70%,累计贡献率31.39%。从图3 可知,A 类与B 类、C 类海雀稗材料间有交叠,表明他们亲缘关系比较近;B 类和D 类有交叠,说明它们存在一定遗传相似性。
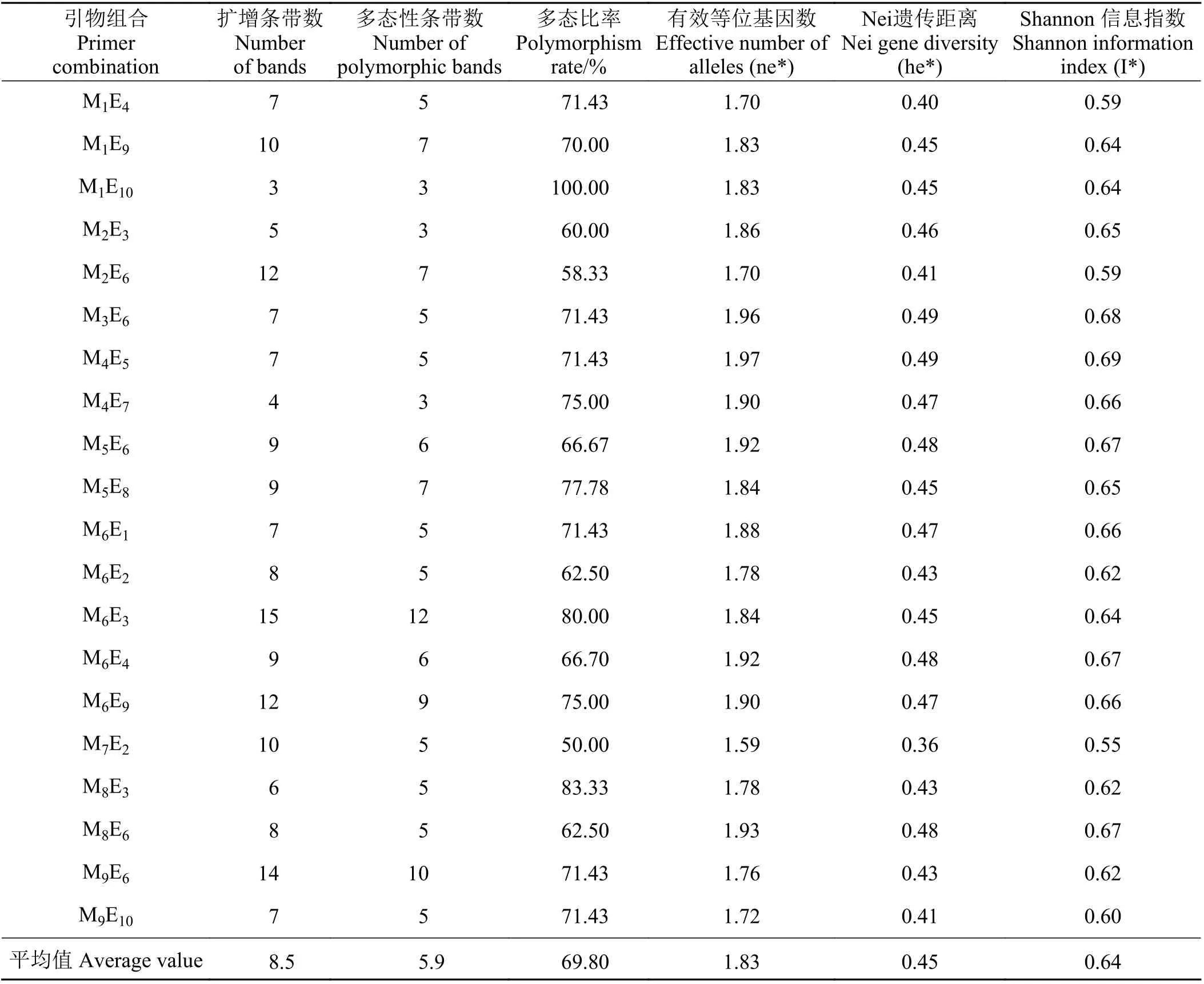
表3 20 对引物组合的多态性扩增结果Table 3 The results of polymorphic amplification of 20 pairs of primer combinations
2.4 群体结构分析
对49 份海雀稗种质进行群体遗传结构分析表明,当样本的等位变异频率特征类型数K = 2 时,其模型的后验概率最大。根据K 值将49 份海雀稗种质划分为PopⅠ、PopⅡ两个类群,分别以红色、绿色表示(图4),且每个亚类群中主要颜色所占的比例情况(Q 值)可以反映种质的来源情况。刘丽华等[16]认为,Q > 0.6 视为血缘单一,Q < 0.6 视为具有混合来源。49 份海雀稗材料的Q 值划分结果表明,PopⅠ亚群中各份材料只有1 份材料Q = 0.6,说明PopⅠ亚群的种质遗传背景较单一,与其他亚群间的基因交流不多;PopⅡ亚群部分种质的Q 值 < 0.6,说明种质资源的基因渗透较高,表明其具有混合来源,遗传背景较复杂。2 个类群中PopⅠ类群包含的海雀稗材料较多(34 份),占供试种质总数71.42%,其典型代表材料是647895 和647892,而其余15 份材料划分为另一类,典型代表材料是647908 和647899。
3 讨论

图2 海雀稗种质UPGMA 聚类Figure 2 UPGMA cluster map of seashore Paspalum
种质资源是开展育种工作的基础,种质资源利用的前提是要对其进行多样性分析和遗传背景研究[17]。本研究发现供试海雀稗引物的多态比率为69.80%,与解新明等[4]多态比率64.77%的结果相比,本研究海雀稗种质资源遗传背景相对较丰富。聚类分析与群体结构分析是进行种质资源遗传多样性分析的有效手段[18],49 份海雀稗种质资源的遗传相似系数平均值为0.67,表明49 份海雀稗种质资源亲缘关系相对较远。对供试材料按UPGMA 进行聚类分析,绘制亲缘关系树状图(图2),在遗传相似系数为0.69 时将材料分为2 大类。第Ⅰ类包含16 份材料,第Ⅱ类包含33 份材料,按UPGMA 进行聚类分析时,其中A 类材料和B 类材料则是按UPGMA 进行聚类分为2 大类中第Ⅰ类材料,C 类材料和D 类材料则是按UPGMA 进行聚类分为2 大类中第Ⅱ类材料,其拟合共相关系数 r = 0.669 表明聚类结果很好的体现了种质之间的遗传关系。
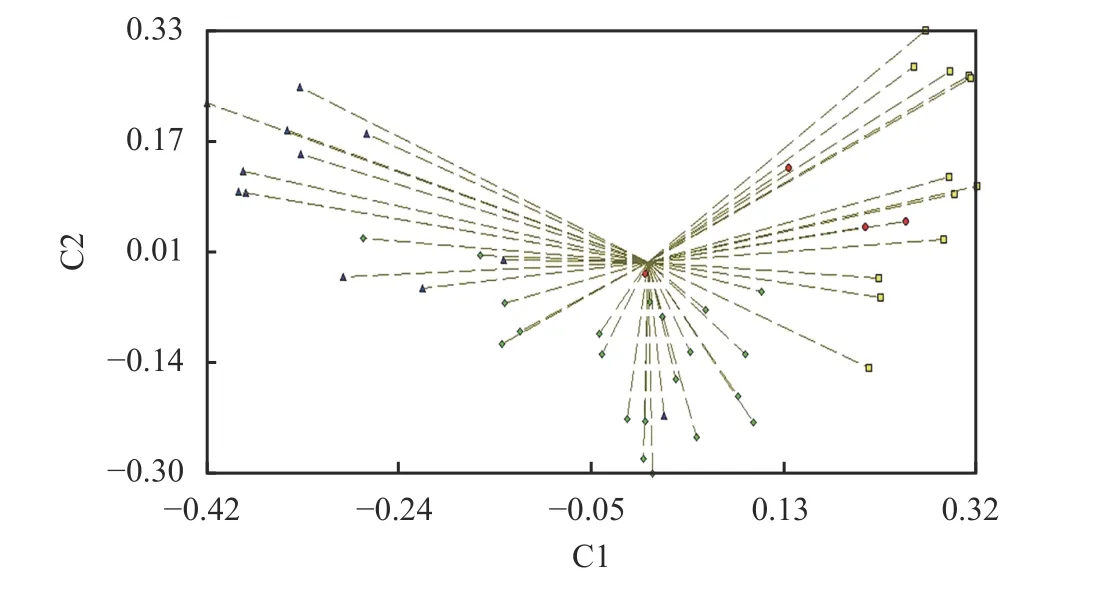
图3 海雀稗主成分分析Figure 3 Principal coordinate analysis of seashore Paspalum
用Structure2.3.4 软件对49 份海雀稗材料进行分析,K = 2 时,各群体内海雀稗的相似值最大,群体遗传结构分析结果将49 份海雀稗份材料划分为2 个类群。PopⅠ类群包含34 份材料;PopⅡ类群包括15 份材料。当K = 3、4、5 时,海雀稗的边缘群体出现分化(图4)。按UPGMA 进行聚类中第Ⅰ类包含的16 份材料都包含在Structure 分析时划分的PopⅠ类群里。
在Structure 分析时划分的PopⅡ类群里的15 份材料全部包含于按UPGMA 进行聚类中的第Ⅱ类中。但另有18 份材料的分类在按UPGMA 进行聚类和Structure 分析时出现不一致现象,这18 份材料在按UPGMA 进行聚类全部划分在第Ⅱ类中,而Structure 分析时则全部都划分在PopⅠ类群中。这个现象与主成分分析时A 类与B 类材料、C 类材料有交叠的部分,B 类和D 类中的海雀稗材料有交叠部分相呼应。只有部分主成分聚类结果与采用UPGMA进行聚类得出的结果一致,可能是因为主成分分析和系统聚类分析都能对材料进行聚类分析,但它们反映的信息却不同[19]。
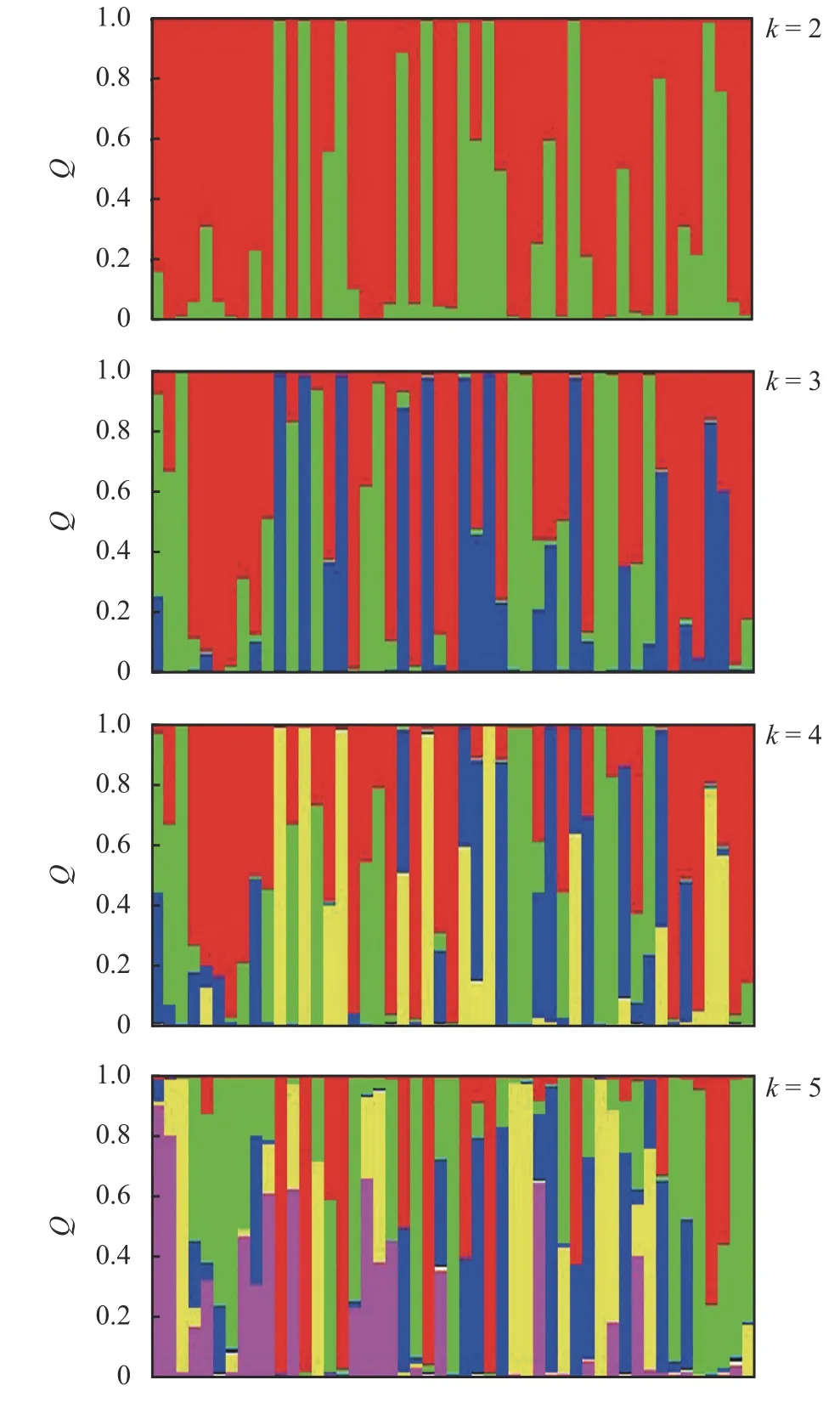
图4 海雀稗的群体遗传结构Figure 4 Population genetic structure of seashore Paspalum
综合分析49 份种质资源在3 种划分方法中的结果,其中4 份材料(647916、647922、647918、647917)、10 份材料(509022、647903、647906、647921、647899、647897、647913、509021、sp005、sp004)、12 份 材 料(647904、 647895、 647893、 647891、 647902、 647892、647900、 647911、 614678、 647896、 647898、 647909)、18 份材料(647907、647919、647912、647910、647920、647914、 647901、 614679、 364368、 576134、 647923、509023、509020、576138、sp001、sp002、sp006、sp007)在3 种划分方法时都能各自聚为一类,以上4 种类型的典型代表材料分别是647918、647899、647895、647910,这与前面群体结构分析的结果较为一致。另有5 份材料(647908、647894、647905、647916、377709)在3 种划分方法聚类时,在大体分类形同的情况下各自分类略有差异,这可能跟划分的方法与关。比较聚类分析和群体结构分析,群体结构分析能够反映出育种材料间的基因渗入和交流情况, 能够更好地揭示材料内在的遗传结构,对于育种工作者更准确掌握种质间的遗传关系有很大的帮助。
来自巴西的647913 和来自美国夏威夷的647897相似系数最大(0.88),而来自美国乔治亚州的647908和美国夏威夷647896 两份材料遗传相似系数最小(0.53),来自不同国家的两个材料遗传相似性最高,而来自同一国家的两个材料遗传相似性最小,目前尚缺乏明确的解释。
4 结论
本研究利用100 对SRAP 标记对49 份种质资源进行遗传多样性分析,筛选获得20 对扩增稳定、多态性好的SRAP 引物;对49 份海雀稗种质资源进行PCR 扩增,共获得169 个条带,多态性位点百分率为69.80%;平均每条引物扩增多态性条带数为5.9,有效等位基因数平均值为1.83,Shannon 信息指数平均值为0.64,Nei 遗传距离平均值为0.45。
49 份海雀稗种质资源的遗传相似系数介于0.52~0.88,平均值为0.67,进一步采用UPGMA 聚类分析、主成分分析、群体结构分析对其进行分类,44 份材料在3 种方法下均表现为较一致的分类,5 份材料表现出一定程度的变异,表明此研究结果反映的种质亲缘关系基本稳定,可用于未来育种工作的亲本选择。

