甘蓝BobHLH121基因克隆、亚细胞定位及表达分析
秦文斌 山溪 张振超 姚悦梅 戴忠良 秦岭

摘要:【目的】克隆甘藍bHLH转录因子(BobHLH121)基因,分析其在不同组织及低温胁迫下的表达模式,为深入研究bHLH转录因子在甘蓝低温响应中的作用机理提供理论参考。【方法】克隆BobHLH121基因编码区(CDS)序列,利用生物信息学软件进行预测分析,并构建pCAMBIA1300-FaFKF1-GFP融合表达载体,通过农杆菌介导转染烟草表皮细胞,观察荧光信号以确定蛋白亚细胞定位情况。采用实时荧光定量PCR(qRT-PCR)检测BobHLH121基因在低温胁迫下的相对表达量。【结果】BobHLH121基因CDS长度为1367 bp,定位于C06染色体上,其编码311个氨基酸,蛋白分子量为34.89 kD,理论等电点(pI)为5.42,不稳定系数为52.29,疏水性平均系数为-0.733,说明该蛋白为不稳定的亲水性蛋白,且呈酸性,定位于细胞核。BobHLH121蛋白的二级结构主要由α-螺旋(占30.87%)、延伸链(占9.32%)、β-转角(占2.89%)和无规则卷曲(占56.91%)组成,具有保守的HLH结构域。BobHLH121蛋白与拟南芥(NP_191768.2)、琴叶拟南芥(EFH52923.1)、亚麻荠(XP_01909381.1)、荠菜(XP_023638997.1)、油菜(CDY65446.1)、白菜(XP_009104362.1)和萝卜(XP_018442860.1)等多种植物bHLH蛋白的氨基酸相似性分别为55.6%、54.8%、52.0%、50.3%、76.4%、73.9%和61.1%。系统发育进化树分析结果显示,BobHLH121与拟南芥(NP_191768.2)和琴叶拟南芥(EFH52923.1)bHLH蛋白在同一小分支上。BobHLH121基因启动子区域存在植物生长发育、非生物胁迫、激素应答和光响应大量的顺式作用元件。BobHLH121基因在叶片的相对表达量显著高于其他组织(P<0.05,下同),在其他组织中的相对表达量均较低。低温胁迫6~12 h时BobHLH121基因的相对表达量较低温处理0 h(对照)显著降低,在低温处理24 h时急剧升高,显著高于其他处理时间,低温处理48 h时又显著降低,表明该基因可能在低温胁迫下延迟表达。【结论】BobHLH121基因含有bHLH家族典型的HLH保守结构域序列,具有明显的组织表达特异性,可能参与甘蓝叶片的生长发育调控,且响应低温胁迫,可能在甘蓝耐寒性中发挥调控作用。
关键词: 甘蓝;BobHLH121基因;亚细胞定位;低温胁迫;基因表达
中图分类号: S635.036 文献标志码: A 文章编号:2095-1191(2021)12-3320-10
Cloning,subcellular localization and expression analysis of BobHLH121 gene from Brassica oleracea L.
QIN Wen-bin, SHAN Xi, ZHANG Zhen-chao, YAO Yue-mei,
DAI Zhong-liang*, QIN Ling
(Zhenjiang Institute of Agricultural Sciences in Hilly Area, Jurong, Jiangsu 212400, China)
Abstract:【Objective】The whole length of Brassica oleracea L. bHLH transcription (BobHLH121 gene) was cloned by homologous cloning method,and its expression pattern under organ/tissue and low temperature stress was analyzed,which provided theoretical reference for studying the mechanism of bHLH transcription factor in low temperature response. 【Method】The sequence of the coding region(CDS) of BobHLH121 gene was cloned, the prediction analysis was performed using bioinformatics software, and the pCAMBIA1300-BobHLH121-GFP fusion expression vector was construc-ted through the transfection of tobacco epidermal cells, and the fluorescent signal was observed to determine the subcellular localization of the protein. The relative expression of the BobHLH121 gene under low temperature stress was measured using real-time fluorescence quantitative PCR (qRT-PCR). 【Result】 The CDS length of BobHLH121 gene was 1367 bp, localized on chromosome C06, encoding 311 amino acids, protein molecular weight of 34.89 kD, theoretical isoelectric point (pI) was 5.42, the instability coefficient was 52.29, and the average hydrophobicity was -0.733, indicating that the protein was an unstable hydrophilic protein, which was acidic and localized in the nucleus. The secondary structure of BobHLH121 protein mainly consisted of α-helix (30.87%), extended chain (9.32%),β-turn (2.89%) and random coil (56.91%), with a conserved HLH domain. BobHLH121 protein was closely related to bHLH proteins in Arabidopsis thaliana (NP_191768.2), A. lyrata (EFH52923.1),Camelina sativa (XP_01909381.1),Capsella brusa-pastoris (XP_023638997.1),B. napus(CDY65446.1),Chinese cabbage (XP_009104362.1) and radish (XP_018442860.1), and had amino acid similarities of 55.6%,54.8%,52.0%,50.3%,76.4%,73.9% and 61.1%,respectively. Phylogenetic tree analysis showed that BobHLH121 was on the same small clade as the A. thaliana (NP_191768.2) and A. lyrata (EFH52923.1) bHLH protein. A large number of cis-acting elements for plant growth and development, abiotic stress, hormone response, and light response existed in the promoter region of the BobHLH121 gene. The relative expression of the Bo-bHLH121 gene was significantly higher in leaves than in other tissues (P<0.05, the same below) and lower in all other tissues. The relative expression of BobHLH121 gene was significantly decreased under low temperature treatment for 6-12 h under low temperature treatment at 0 h (control), and increased sharply at 24 h under low temperature treatment, which was significantly higher than that of other treatments, and decreased significantly at 48 h under low temperature treatment. It indicated that the gene might be delayed in expression under low temperature stress. 【Conclusion】BobHLH121 has a conserved HLH domain typical of the bHLH family, has obvious tissue expression specificity, may participate in the re-gulation of growth and development of cabbage leaves, and respond to low temperature stress, may play a regulatory role in cabbage cold tolerance.
Key words: Brassica oleracea L.; BobHLH121 gene; subcellular localization; low temperature stress; gene expression
Foundation item:Jiangsu Science and Technology Plan Project(BE2020403); Zhenjiang Science and Technology Plan Project(NY2020001)
0 引言
【研究意义】碱性螺旋-环-螺旋(Basic helix-loop-helix,bHLH)转录因子是一类广泛存在于真核生物中的调节蛋白超家族,其家族成员具有高度保守的HLH结构域(Toledo-Ortiz et al.,2003;Li et al.,2006),通过转录调控或核定位等相关功能激活或抑制相关的下游基因,从而调节植物的生长发育过程以及生物和非生物胁迫响应(Agarwal et al.,2006a;Feller et al.,2011;Gong et al.,2015)。甘蓝(Brassica oleracea L.)为十字花科芸薹属植物,在我国广泛种植,年种植面积达到90万ha左右,但多为露地栽培,极易遭受冷害、日灼、病虫害等多种非生物和生物胁迫。其中,低温胁迫可改变植物细胞内蛋白结构和活性,从而影响光合作用等一系列酶促反应的速率,最终导致植株叶片萎蔫黄化等现象(Miura and Furumoto,2013)。因此,开展甘蓝bHLH转录因子基因克隆及低温胁迫下表达模式研究,以期挖掘其抗逆基因,对改良甘蓝品种抗性具有重要意义。【前人研究进展】随着不同植物基因组测序的完成,拟南芥(Carretero-paulet et al.,2010)、大白菜(Song et al.,2014)、番茄(Sun et al.,2015;Wang et al.,2015)、苹果(Mao et al.,2017)等植物中bHLH转录因子家族成员已被鉴定。研究发现,bHLH转录因子参与花青素的生物合成(Toledo-Ortiz et al.,2003;Gonzalez et al.,2008;Pires and Dolan,2010)、生物与非生物胁迫(Sorensen et al.,2003;Hichri et al.,2011)、植物器官发育(Goossens et al.,2017;Takahashi et al.,2017)等。bHLH转录因子家族蛋白具有高度保守的bHLH结构域,其长度约为50~60个氨基酸,分为2个不同的区域,即碱性区域和螺旋-环-螺旋区域(HLH),其中,N-端的碱性区域包含13~17个亲水性的碱性氨基酸,具有与DNA识别和结合的位点,参与DNA结合(Mao et al.,2017)。植物感知低温后,会将冷信号通过低温信号转导途径传递下去,最终诱导冷胁迫相关基因的表达,增加植株适应性,而CBF依赖的冷信号途径普遍存在于高等植物中,现已在拟南芥中发现(刘双等,2017)。拟南芥的ICE(Inducer of CBF expression)是CBF低溫响应通道上游的调控基因,编码一个类似MYC bHLH结构的转录因子(Kurbidaeva et al.,2015)。拟南芥中有2个ICE基因(AtICE1和AtICE2),其中AtICE1基因正调控CBF3信号途径,通过与MYB15蛋白互作,抑制MYB15对CBF的负调控作用(Kurbidaeva et al.,2015);AtICE2基因则通过调节CBF1基因表达来正调控植物的低温响应(Agarwal et al.,2006b)。此外,Li等(2010)研究发现,水稻OrbHLH001基因在拟南芥中过量表达时,显著提高了拟南芥的抗寒能力。Chen等(2012)研究发现,菊花bHLH家族基因CdICE1在低温、干旱和盐胁迫中均具有调控作用。Guo和Wang(2017)研究发现,小麦WbHLH046基因能被低温诱导,在低温胁迫下表达量显著升高。Man等(2017)研究发现,水稻ICE1基因受低温诱导上调表达。【本研究切入点】关于植物bHLH转录因子的结构特点和功能验证等研究已有很大进展,但bHLH家族成员众多,且不同植物bHLH转录因子的结构和调控功能变化多样。目前,甘蓝bHLH转录因子的相关研究鲜少,甘蓝bHLH转录因子参与非生物胁迫响应的研究在甘蓝中未见报道。【拟解决的关键问题】基于甘蓝bHLH转录因子家族的生物信息学分析结果,利用同源克隆的方法克隆BobHLH121基因编码区(CDS)序列,并通过构建pCAMBIA1300-FaFKF1-GFP融合表达载体确定蛋白亚细胞定位情况,采用实时荧光定量PCR(qRT-PCR)检测Bo-bHLH121基因在不同组织及低温胁迫下的相对表达量,为深入研究bHLH转录因子在甘蓝生长发育及低温胁迫的作用机制提供理论参考,对提高甘蓝产量和抗性具有重要意义。
1 材料与方法
1. 1 试验材料
供试的甘蓝材料由江苏丘陵地区镇江市农业科学院提供。总RNA提取试剂盒购自天根生化科技(北京)有限公司;PrimeScript 1st Strand cDNA Synthesis Kit、T4 DNA连接酶和pMD18-T质粒载体等均购自TaKaRa公司;DNA回收纯化试剂盒购自诺维赞生物科技有限公司。主要仪器设备:光照培养箱(南京恒裕仪器设备制造有限公司)、PCR仪(苏州东胜兴业科学仪器有限公司)、凝胶成像系统(上海培清科技有限公司)和实时荧光定量PCR仪(QuantStudio3,美国)。
1. 2 试验方法
1. 2. 1 样品采集 种子发芽后播种于基质中,在人工气候室中进行生长(白天25 ℃/夜晚18 ℃,光照14 h/黑暗10 h),待植株长至五叶一心时进行4 ℃低温胁迫处理(每个处理至少选取18株)。将幼苗移至春化室中低温处理0(对照)、6、12和24 h后取样,取每株顶端第三片完全展开的叶片,使用液氮迅速冷冻,保存于-80 ℃冰箱。
1. 2. 2 BobHLH121基因克隆 称取2份新鲜叶片0.1 g,分别用于RNA和DNA提取。以RNA为模板,使用PrimeScript 1st Strand cDNA Synthesis Kit反转录合成cDNA第一条链。采用CTAB法提取叶片总DNA。
从甘蓝基因组数据库(Bolbase,http://brassicadb.org/brad/index.php)下载BobHLH121(Bol010854)基因编码区(CDS)序列,使用Primer Premier 5.1设计引物(表1),委托金斯瑞生物科技有限公司合成引物。以甘蓝叶片DNA为模板进行BobHLH121基因的扩增。反应体系:10×PCR Buffer 2.0 μL,40 ng/μL DNA模板1.0 μL,10 μmol/L正、反向引物(BobHLH121-1-F/BobHLH121-1-R)各1.0 μL,2 mmol/L dNTP 2.0 μL,25 mmol/L MgCl2 2.0 μL,Taq聚合酶0.25 μL,ddH2O补足至20.0 μL。扩增程序:94 ℃预变性5 min;94 ℃ 30 s,60 ℃ 30 s,72 ℃ 60 s,进行35个循环;72 ℃延伸7 min。PCR产物用1%琼脂糖凝胶电泳检测,并使用凝胶成像仪拍照。利用PCR产物回收试剂盒回收纯化目的片段,将其连接至pMD18-T后转化大肠杆菌DH5α感受态细胞。使用LB培养基(含氨苄青霉素)进行菌落筛选,挑选阳性克隆送往南京斯普金生物科技有限公司进行测序。
1. 2. 3 生物信息学分析 在甘蓝基因组数据库中搜索BobHLH121基因的CDS、DNA和蛋白序列,利用NCBI数据库BLAST搜索同源序列。使用MEGA 6.0的邻接法(Neighbor-joining,NJ)构建系统发育进化树,Bootstrap值设置为1000,其他参数为默认值。使用ExPASy(https://web.expasy.org/protparam/)预测蛋白的理化性质。使用Mapchart绘制BobHLH121基因在染色体上的位置。利用New PLACE(https://sogo.dna.affrc.go.jp/)分析BobHLH121起始密码子上游2000 bp序列中顺式作用元件;使用SOPMA(http://npsa-pbi.libcp.fr/cgi-bin/secpred-sopma.pl)预测蛋白二级结构;使用Plant-mPLoc(http://www.csbio.sjtu.edu.cn/bioinf/plant-multi/)、BacelLo(http://gpcr.biocomp.unibo.it/bacello/pred.htm)和PredictProtein(https://www.predictprotein.org/)进行Bo-bHLH121亚细胞定位预测。
1. 2. 4 BobHLH121基因在不同组织及低温胁迫下的表达分析 BobHLH121基因在甘蓝不同组织中的表达数据均从NCBI的GEO数据库下载(登录号:GSE42891)。根据BobHLH121基因序列,设计实时荧光定量PCR的特异引物(BobHLH121-2-F/Bo-bHLH121-2-R)(表1),选用甘蓝GAPDH为内参基因(Brulle et al.,2014),并委托金斯瑞生物科技有限公司合成引物。以甘蓝叶片的cDNA第一条链为模板,使用荧光定量PCR试剂盒SYBR Premix Ex TaqTM II,检测BobHLH121基因在不同低温处理时间下的表达量。反应体系:40 ng/μL cDNA模板1.0 μL、0.2 μmol/L正、反向引物(BobHLH121-2-F/BobHLH121-2-R)各1.0 μL,SYBR Premix Ex Taq 10.0 μL,ddH2O补足20.0 μL。PCR扩增采用两步法:95 ℃ 5 s,60 ℃ 20.0 s,进行40个循環;熔解曲线分析:95 ℃ 15 s,60 ℃ 1 min,95 ℃ 1 s。
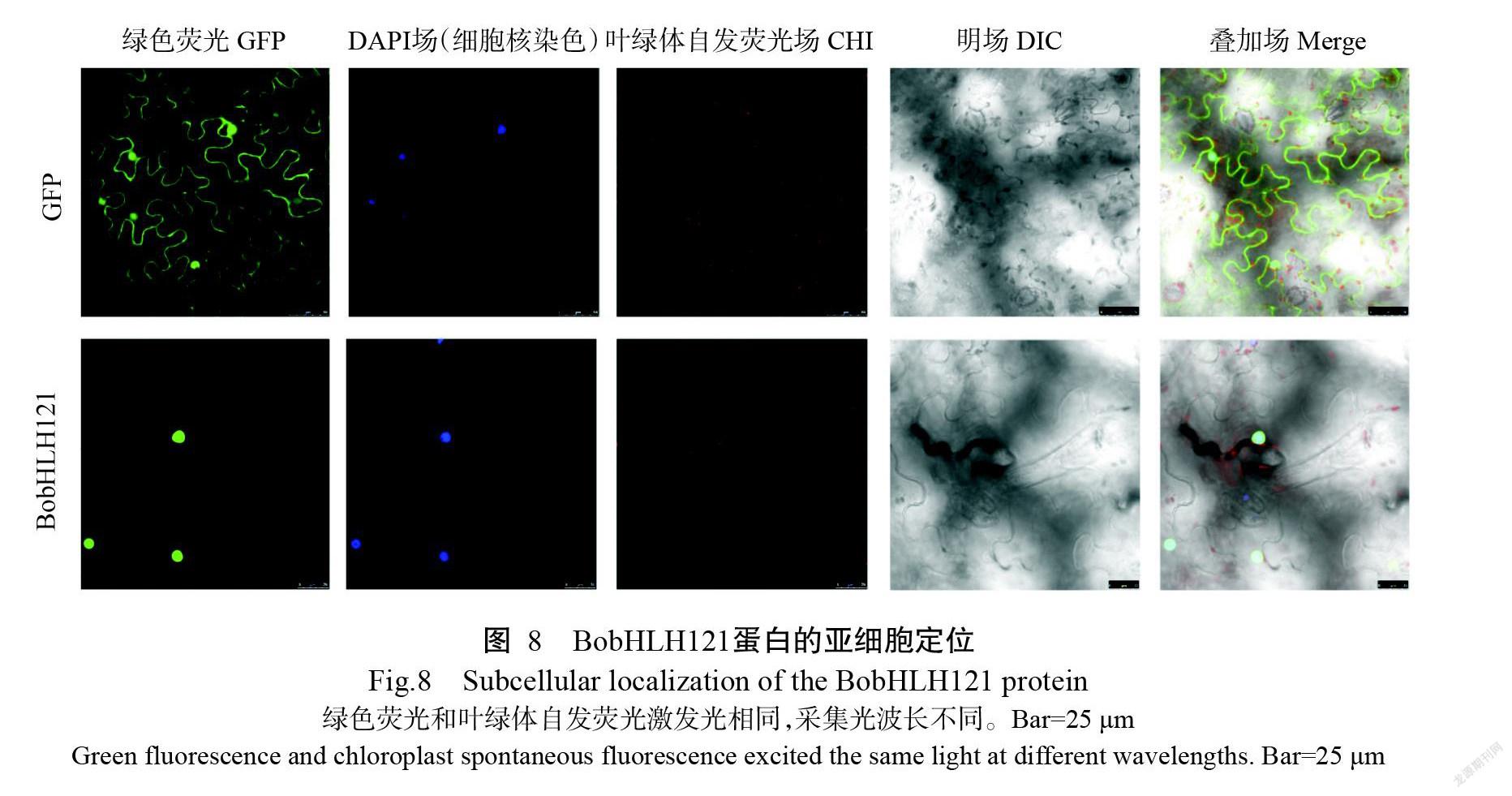
1. 2. 5 亚细胞定位 根据BobHLH121基因序列设计带有酶切位点的引物(BobHLH121-3-F/Bo-bHLH121-3-R)(表1),以携带目的基因序列的克隆载体pMD18-T为模板进行PCR扩增,回收纯化目的片段。分别使用Sac I和Xba I酶切带有荧光蛋白(GFP)基因的质粒载体PCAMBIA2300-GFP和目的片段,然后用T4连接酶连接,构建融合表达载体PCAMBIA2300-GFP-BobHLH121(贺丹等,2021),将其转化大肠杆菌DH5α感受态细胞,挑取阳性克隆进行测序,测序结果正确的质粒转化根瘤农杆菌GV3101。将农杆菌菌液注射烟草叶片,暗培养72 h后在激光共聚焦显微镜下观察叶片中荧光信号。
1. 3 统计分析
根据2-ΔΔCt法计算BobHLH121基因的相对表达量。采用GraphPad Prism 5.0整理分析数据及绘图。
2 结果与分析
2. 1 BobHLH121基因克隆结果
以甘蓝叶片DNA为模板进行PCR扩增,结果如图1所示。BobHLH121基因CDS长度为1367 bp,编码311个氨基酸残基。回收目的条带的测序结果与甘蓝基因组数据库中Bol010854基因CDS的序列完全一致,表明克隆片段为目的基因。
2. 2 染色体定位结果
在甘蓝基因组数据库中搜索BobHLH121基因信息,结果发现该基因定位于C06染色体上(图2),基因位置信息为C06:16463282-16464648(-),属于MF2亚组上的反义链基因。BobHLH121是个双拷贝基因,与白菜和拟南芥的同源基因之间具有相同的共线性区块(图3)。
2. 3 启动子顺式作用元件分析结果
利用PlantCARE数据库预测BobHLH121基因的上游2000 bp启动子区域的顺式作用元件,结果(表2)显示,BobHLH121基因的启动子区域存在大量的顺式作用元件,主要分为植物生长发育、非生物胁迫、激素应答和光响应四种类型。
2. 4 理化性质分析和二级结构预测结果
根据ExPASy在线工具推测BobHLH121蛋白的分子式为C1510H2389N421O494S17,分子量为34.89 kD,理论等电点(pI)为5.42,不稳定系数为52.29,疏水性平均系数为-0.733,说明该蛋白为不稳定的亲水性蛋白,且呈酸性。使用SOPMA在线工具预测Bo-bHLH121蛋白的二级结构,结果如图4所示。Bo-bHLH121蛋白的二级结构比较丰富,主要由α-螺旋(占30.87%)、延伸链(占9.32%)、β-转角(占2.89%)和无规则卷曲(占56.91%)组成,说明α-螺旋和无规则卷曲是该蛋白多肽链中主要的结构元件。
2. 5 序列比对及同源分析结果
SMART分析结果显示,BobHLH121蛋白具有保守的HLH结构域(图5)。通过NCBI数据库BLAST搜索BobHLH121蛋白的同源序列,结果发现Bo-bHLH121蛋白与拟南芥(NP_191768.2)、琴叶拟南芥(EFH52923.1)、亚麻荠(XP_01909381.1)、荠菜(XP_023638997.1)、油菜(CDY65446.1)、白菜(XP_009104362.1)和萝卜(XP_018442860.1)等多种植物bHLH蛋白的氨基酸相似性分别为55.6%、54.8%、52.0%、50.3%、76.4%、73.9%和61.1%(图6)。由图7可知,BobHLH121与拟南芥(NP_191768.2)和琴叶拟南芥(EFH52923.1) bHLH蛋白在同一小分支上。
2. 6 亚细胞定位分析结果
Plant-mPLoc、BacelLo和PredictProtein预测结果均显示,BobHLH121蛋白定位于细胞核中。将PCAMBIA2300-GFP-BobHLH121融合表达载体通过农杆菌介导转染烟草叶片表皮细胞,从而获得烟草瞬時表达体系,通过观察荧光信号发现对照组细胞核和细胞质可见明显的绿色荧光,处理组仅在烟草表皮细胞的细胞核检测到绿色荧光信号,即BobHLH121蛋白定位于细胞核中(图8),表明Bo-bHLH121具有核蛋白的功能。
2. 7 BobHLH121基因组织表达分析结果
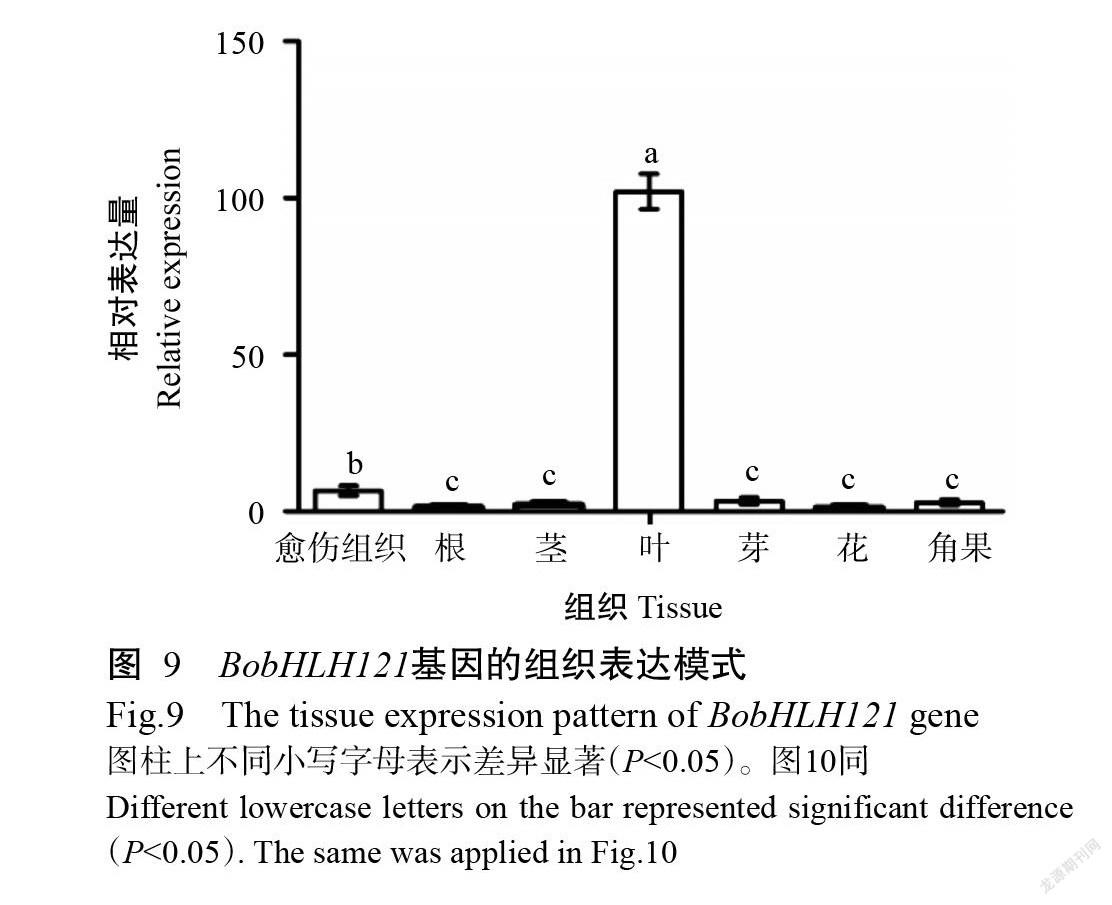
BobHLH121基因在甘蓝愈伤组织、根、茎、叶、芽、花和角果中的表达模式如图9所示。BobHLH121基因在不同组织中的相对表达量存在明显差异,其中在叶片的相对表达量显著高于其他组织(P<0.05,下同),在其他组织中的相对表达量均较低,尤其是在花中的相对表达量最低,表明BobHLH121基因可能参与甘蓝叶片的生长发育调控。
2. 8 BobHLH121基因在低温胁迫下的表达分析结果
对BobHLH121基因在低温胁迫下甘蓝幼叶中的表达模式进行分析,结果如图10所示。低温胁迫6~12 h时BobHLH121基因的相对表达量较低温处理0 h(对照)显著降低,低温处理24 h时相对表达量急剧升高,显著高于其他处理时间,低温处理48 h时又显著降低,表明BobHLH121基因可能在低温胁迫下延迟表达,响应24 h才被激活表达。
3 讨论
bHLH转录因子家族是植物中的第二大转录因子家族,在光合作用、光信号转导、色素生物合成、种子发育和胁迫响应等过程中具有重要的调节作用。有关bHLH转录因子的非生物胁迫响应研究在拟南芥(Xiang et al.,2008)、水稻(Liu and Zhou,2018)等模式植物中已广泛展开,但甘蓝中鲜见报道。本研究从甘蓝克隆获得BobHLH121基因CDS全长为1367 bp,其编码的氨基酸序列中具有HLH保守结构域,是bHLH转录因子家族典型的结构特征。HLH保守结构域主要参与结合下游基因中启动子区域的E-box/G-box顺式作用元件。BobHLH121蛋白与拟南芥bHLH家族Ia组蛋白的氨基酸序列相似性较高(Shan et al.,2019)。将BobHLH121与其他植物的bHLH蛋白进行比对及系统进化分析,结果发现Bo-bHLH121与拟南芥和琴叶拟南芥bHLH蛋白聚在一个分支上。BobHLH121蛋白与拟南芥bHLH蛋白(NP_191768.2)的氨基酸相似性分别为55.6%,推测甘蓝与拟南芥发生基因分化之后,出现了基因复制和基因丢失的现象,导致基因的多样化和功能分化。亚细胞定位结果显示,BobHLH121蛋白定位在细胞核,表明BobHLH121具有核蛋白功能,推测其主要功能是在细胞核中调控下游基因的转录水平,进而调控甘蓝在低温胁迫下的响应(刘海霞等,2010;豆玉娟等,2014)。因此,今后深入研究BobHLH121蛋白的生物学功能。
启动子顺式作用元件可调控基因的表达模式(Saeed et al.,2006),且与多种胁迫响应的基因密切相关(Walther et al.,2007;Le et al.,2012)。启动子位于转录起始位点上游的2000 bp片段,对结合靶基因具有重要影响(Fang et al.,2008;Wu et al.,2012)。本研究发现BobHLH121基因的启动子区域有许多激素应答相关的元件,表明BobHLH121基因可能在脱落酸(ABA)、赤霉素(GA)和茉莉酸甲酯(MeJA)信号通路中发挥积极作用。拟南芥AtICE2基因的启动子区域存在许多与非生物胁迫相关的顺式作用元件,且在低温响应中发挥重要的作用(Sorensen et al.,2003)。而BobHLH121基因的启动子区域也包含LTR等低温响应元件。目前已有大量研究证明,在胁迫条件下,部分bHLH转录因子被激活并结合到参与各种信号通路的关键基因启动子上,进而通过调控目的基因的转录水平来调节植物的抗逆性(Lu et al.,2017)。例如,拟南芥AtICE1(AtbHLH116)能识别CBF3基因启动子序列中的MYC位点(CANNTG),诱导CBF3基因的表达,提高拟南芥对低温胁迫的抗性(Badawi et al.,2008)。Feng等(2012)从苹果中获得AtICE1的同源蛋白(MdCIbHLH1),其与DREB家族基因MdCBF2启动子区域的MYC识别位点结合,导致MdCBF2基因表达上调,从而提高了苹果的低温耐性。Nakamura等(2011)研究发现在低温处理下水稻OsICE1和OsICE2蛋白可诱导转录因子基因OsDREB1B和OsHSFA3的表达上调,可调控植株的低温耐受性。李宇伟等(2012)在蒺藜苜蓿中发现1个低温响应基因MtbHLH1,在4 ℃低温胁迫下,不同处理时间的相对表达量具有明显的差异性。本研究通过荧光定量PCR检测发现,BobHLH121基因在低温处理24 h的相对表达量显著上调,说明BobHLH121基因参与调控低温胁迫响应,今后应深入研究揭示其低温响应机制。
4 結论
BobHLH121基因含有bHLH家族典型的HLH保守结构域序列,具有明显的组织表达特异性,可能参与甘蓝叶片的生长发育调控,且响应低温胁迫,可能在甘蓝耐寒性中发挥调控作用。
参考文献:
豆玉娟,曹飞,马跃,李贺,刘月学,张志宏. 2014. 栽培草莓果实中特异表达的bHLH78基因的克隆及过量表达载体构建[J]. 分子植物育种,12(3):456-465. [Dou Y J,Cao F,Ma Y,Li H,Liu Y X,Zhang Z H. 2014. Cloning of bHLH78 gene expressed specifically in fruits of cultiva-ted strawberry and construction of overexpression vector[J]. Molecular Plant Breeding,12(3):456-465.] doi:10. 13271/j.mpb.012.000456.
李宇伟,连瑞丽,王新民. 2012. 蒺藜苜蓿低温胁迫响应基因MtbHLH-1的克隆及功能分析[J]. 中国农业科学,45(16):3430-3436. [Li Y W,Lian R L,Wang X M. 2012. Isolation and functional analysis of the low-temperature-responsive gene MtbHLH-1 in Medicago truncatula[J]. Scientia Agricultura Sinica,45(16):3430-3436.] doi:10. 3864/j.issn.0578-1752.2012.16.024.
刘海霞,康俊根,颉建明,简元才,丁云花. 2010. 甘蓝OguCMS相关的花药优势表达转录因子BoBHLH1的克隆与表达分析[J]. 园艺学报,37(12):1953-1960. [Liu H X,Kang J G,Xie J M,Jian Y C,Ding Y H. 2010. Cloning and expression of an OguCMS-related anther-preferential transcription factor in Brassica oleracea[J]. Acta Horticulturae Sinica,37(12):1953-1960.] doi:10.16420/j.issn. 0513-353x.2010.12.023.
刘双,陈沼汀,董昭旭,孙国旭,李晖,关淑艳. 2017. 拟南芥抗寒基因ICE1的克隆及功能分析[J]. 西北农林科技大学学报,45(5):175-182. [Liu S,Chen Z T,Dong Z X,Sun G X,Li H,Guan S Y. 2017. Cloning and functional ana-lysis of Arabidopsis thaliana cold resistance gene ICE1[J]. Journal of Northwest A & F University,45(5):175-182.] doi:10.13207/j.cnki.jnwafu.2017.05.024.
Agarwal M,Hao Y J,Kapoor A,Dong C H,Fujii H,Zheng X W,Zhu J K. 2006a. A R2R3 type MYB transcription factor is involved in the cold regulation of CBF genes and in acquired freezing tolerance[J]. Journal of Biological Chemistry,281(49):37636-37645. doi:10.1074/jbc.M605 895200.
Agarwal P K,Agarwal P,Reddy M K,Sopory S K. 2006b. Role of DREB transcription factors in abiotic and biotic stress tolerance in plants[J]. Plant Cell Reports,25(12):1263-1274. doi:10.1007/s00299-006-0204-8.
Badawi M,Reddy Y V,Agharbaoui Z,Tominaga Y,Danyluk J,Sarhan F,Houde M. 2008. Structure and functional analysis of wheat ICE(inducer of CBF expression) genes[J]. Plant and Cell Physiology,49(8):1237-1249. doi:10.1093/pcp/pcn100.
Brulle F,Bernard F,Vandenbulcke F,Cuny D,Dumez S. 2014. Identification of suitable qPCR reference genes in leaves of Brassica oleracea under abiotic stresses[J]. Eco-toxicology,23(3):459-471. doi:10.1007/s10646-014-1209-7.
Carretero-Paulet L,Galstyan A,Roig-villanova I,Martinez-garcia J F,Bilbao-castro J R,Robertson D L. 2010. Genome-Wide classification and evolutionary analysis of the bHLH family of transcription factors in Arabidopsis,poplar,rice,moss,and algae[J]. Plant Physiology,153(3):1398-1412. doi:10.1104/pp.110.153593.
Chen L,Chen Y,Jiang J F,Chen S M,Chen F D,Guan Z Y,Fang W M. 2012. The constitutive expression of Chrysanthemum dichrum ICE1 in Chrysanthemum grandiflorum improves the level of low temperature,salinity and drought tolerance[J]. Plant Cell Reports,31(9):1747-1758. doi:10.1007/s00299-012-1288-y.
Fang Y J,You J,Xie K B,Xie W B,Xiong L Z. 2008. Systematic sequence analysis and identification of tissue-specific or stress-responsive genes of NAC transcription factor family in rice[J]. Molecular Genetics and Genomics,280(6):547-563. doi:10.1007/s00438-008-0386-6.
Feller A,Machemer K,Braun E L,Grotewold E. 2011. Evolutionary and comparative analysis of MYB and bHLH plant transcription factors[J]. The Plant Journal,66(1):94-116. doi:10.1111/j.1365-313X.2010.04459.x.
Feng X M,Zhao Q,Zhao L L,Qiao Y,Xie X B,Li H F,Yao Y X,You C X,Hao Y J. 2012. The cold induced basic helix-loop-helix transcription factor gene MdCIbHLH1 encodes an ICE-like protein in apple[J]. BMC Plant Bio-logy,12:22. doi:10.1186/1471-2229-12-22.
Gong H X,Rehman J,Tang H Y,Wary K,Mittal M,Chaturvedi P,Zhao Y Y,Komarova Y A,Vogel S M,Malik A B. 2015. HIF2α signaling inhibits adherens junctional disruption in acute lung injury[J]. The Journal of Clinical Investigation,125(3):1364. doi:10.1172/JCI81377.
Gonzalez A,Zhao M Z,Leavitt J M,Lloyd A M. 2008. Regulation of the anthocyanin biosynthetic pathway by the TTG1/bHLH/Myb transcriptional complex in Arabidopsis seedlings[J]. The Plant Journal,53(5):814-827. doi:10. 1111/j.1365-313X.2007.03373.x.
Goossens J,Mertens J,Goossens A. 2017. Role and functioning of bHLH transcription factors in jasmonate signalling[J]. Journal of Experimental Botany,68(6):1333-1347. doi:10.1093/jxb/erw440.
Guo X J,Wang J R. 2017. Global identification,structural analysis and expression characterization of Bhlh transcription factors in wheat[J]. BMC Plant Biology,17:90.
Hichri I,Barrieu F,Bogs J,Kappel C,Delrot S,Lauvergeat V. 2011. Recent advances in the transcriptional regulation of the flavonoid biosynthetic pathway[J]. Journal of Experimental Botany,62(8):2465-2483. doi:10.1093/jxb/erq442.
Kurbidaeva A,Novokreshchenova M,Ezhova T. 2015. ICE genes in Arabidopsis thaliana:Clinal variation in DNA polymorphism and sequence diversification[J]. Biologia Plantarum,59(2):245-252. doi:10.1007/s10535-015-0497-y.
Le D T,Nishiyama R,Watanabe Y,Vankova R,Tanaka M,Seki M,Harm L H,Yamaguchi-Shinozaki K,Shinozaki K,Tran L S P. 2012. Identification and expression analysis of cytokinin metabolic genes in soybean under normal and drought conditions in relation to cytokinin levels[J]. PLoS One,7(8):e42411. doi:10.1371/journal.pone.0042411.
Li F,Guo S Y,Zhao Y,Chen D Z,Chong K,Xu Y Y. 2010. Overexpression of a homopeptide repeat-containing bHLH protein gene(OrbHLH001) from Dongxiang wild rice confers freezing and salt tolerance in transgenic Arabidopsis[J]. Plant Cell Reports,29(9):977-986. doi:10. 1007/s00299-010-0883-z.
Li X X,Duan X P,Jiang H X,Sun Y J,Tang Y P,Yuan Z,Guo J K,Liang W Q,Chen L,Yin J Y,Ma H,Wang J,Zhang D B. 2006. Genome-wide analysis of basic/helix-loop-helix transcription factor family in rice and Arabidopsis[J]. Plant Physiology,141(4):1167-1184. doi:10. 1104/pp.106.080580.
Liu Y K,Zhou J. 2018. Mapping kinase regulation of ICE1 in freezing tolerance[J]. Trends Plant Science,23(2):91-93. doi:10.1016/j.tplants.2017.12.002.
Lu Y,Yang L,Yu M Y,Lai J B,Wang C,McNeil D,Zhou M X,Yang C W. 2017. A novel Zea mays ssp. mexicana L. MYC-type ICE-like transcription factor gene ZmmICE1 enhances freezing tolerance in transgenic Arabidopsis tha-liana[J]. Plant Physiology and Biochemistry,113:78-88. doi:10.1016/j.plaphy.2017.02.002.
Man L L,Xiang D J,Wang L N,Zhang W W,Wang X D,Qi G C. 2017. Stress-responsive gene RsICE1 from Raphanus sativus increases cold tolerance in rice[J]. Protoplasma,254(2):945-956. doi:10.1007/s00709-016-1004-9.
Mao K,Dong Q L,Li C,Liu C H,Ma F W. 2017. Genome-wide identification and characterization of apple bHLH transcription factors and expression analysis in response to drought and salt stress[J]. Frontiers in Plant Science,8:480. doi:10.3389/fpls.2017.00480.
Miura K,Furumoto T. 2013. Cold signaling and cold response in plants[J]. International Journal of Molecular Sciences,14(3):5312-5337. doi:10.3390/ijms14035312.
Nakamura J,Yuasa T,Huong T T,Harano K,Tanaka S,Iwata T,Phan T,Iwaya M. 2011. Rice homologs of inducer of CBF expression(OsICE) are involved in cold acclimation[J]. Plant Biotechnology,28(3):303-309. doi:10.5511/plantbiotechnology.11.0421a.
Pires N,Dolan L. 2010. Origin and diversification of basic-helix-loop-helix proteins in plants[J]. Molecular Biology and Evolution,27(4):862-874. doi:10.1093/molbev/msp288.
Saeed A I,Bhagabati N K,Braisted J C,Liang W,Sharov V,Howe E A,Li J W,Thiagarajan M,White J A,Quackenbush J. 2006. TM4 microarray software suite[J]. Methods in Enzymology,411:134-193. doi:10.1016/S0076-6879(06)11009-5.
Shan X,Zhang W,Yu F W,Wang S Y,Li J B,Tang J,Dai Z L. 2019. Genome-wide analysis of basic helix-loop-helix superfamily members reveals organization and chilling-responsive patterns in Cabbage(Brassica oleracea var. ca-pitata L.)[J]. Genes,10(11):914. doi:10.3390/genes1011 0914.
Song X M,Huang Z N,Duan W K,Ren J,Liu T K,Li Y,Hou X L. 2014. Genome-wide analysis of the bHLH transcription factor family in Chinese cabbage(Brassica rapa ssp. pekinensis)[J]. Molecular Genetics and Geno-mics,289(1):77-91. doi:10.1007/s00438-013-0791-3.
Sorensen A M,Kr?ber S,Unte U S,Huijser P,Dekker K,Saedler H. 2003. The Arabidopsis ABORTED MICROSPORES(AMS) gene encodes a MYC class transcription factor[J]. The Plant Journal,33(2):413-423. doi:10.1046/ j.1365-313X.2003.01644.x.
Sun H,Fan H J,Ling H Q. 2015. Genome-wide identification and characterization of the bHLH gene family in tomato[J]. BMC Genomics,16(1):9. doi:10.1186/s12864-014-1209-2.
Takahashi Y,Ebisu Y,Shimazaki K. 2017. Reconstitution of abscisic acid signaling from the receptor to DNA via bHLH transcription factors[J]. Plant Physiology,174(2):815-822. doi:10.1104/pp.16.01825.
Toledo-Ortiz G,Huq E,Quail P H. 2003. The Arabidopsis basic/helix-loop-helix transcription factor family[J]. The Plant Cell,15(8):1749-1770. doi:10.1105/tpc.013839.
Walther D,Brunnemann R,Selbig J. 2007. The regulatory code for transcriptional response diversity and its relation to genome structural properties in A. thaliana[J]. PLoS Genetics,3(2):e11. doi:10.1371/journal.pgen.0030011.
Wang J Y,Hu Z Z,Zhao T M,Yang Y M,Chen T Z,Yang M L,Yu W G,Zhang B L. 2015. Genome-wide analysis of bHLH transcription factor and involvement in the infection by yellow leaf curl virus in tomato(Solanum lycopersicum)[J]. BMC Genomics,16(1):39. doi:10.1186/s12864-015-1249-2.
Wu J,Peng Z,Liu S Y,He Y J,Cheng L,Kong F L,Wang J,Lu G. 2012. Genome-wide analysis of Aux/IAA gene fami-ly in Solanaceae species using tomato as a model[J]. Molecular Genetics and Genomics,287:295-311. doi:10. 1007/s00438-012-0675-y.
Xiang D J,Hu X Y,Zhang Y,Yin K D. 2008. Over-expression of ICE1gene in transgenic rice improves cold toleran-ce[J]. Rice Science,15(3):173-178. doi:10.1016/S1672-6308(08)60039-6.
(責任编辑 陈 燕)

