丝兰属6种植物叶绿体基因组特征分析
王 飞 赵文植 董章宏 马路遥 李卫英 李宗艳 辛培尧*
(1. 西南林业大学,国家林业和草原局西南风景园林工程技术研究中心,昆明 650224;2. 西南林业大学,西南地区生物多样性保育国家林业和草原局重点实验室,昆明 650224)
叶绿体在高等植物中广泛存在,大部分物种有着母系遗传特征的独立基因组,即叶绿体基因组[1]。与核基因组相比,叶绿体基因组具有全长序列短、易测序获得、基因结构稳定、保守性较高和进化速率适中等优势,在植物系统发育分析、物种分类鉴定及分子标记开发等研究中广泛应用[2-3]。例如对沙冬青属(Ammopiptanthus)[4]、鹅耳枥属(Carpinus)[5]、苜蓿属(Medicago)[6]、藜属(Chenopodium)[7]等属内多个物种的叶绿体基因组特征进行比对分析,并构建系统发育树,明确了各物种间亲缘关系和系统发育位置,为相关植物的分类鉴定及物种演化奠定了基础。基于叶绿体基因组还可进行分子标记开发,Alexander 等[8]利用焦磷酸测序(Pyrosequencing)技术在红槲栎(Quercus rubra)叶绿体基因组中发掘出多个高质量SNP位点,可用于该种质资源的开发及鉴定。总之,叶绿体基因组在植物系统发育、物种演化等研究中意义重大。
丝兰属(Yucca)隶属于天门冬科(Asparagaceae)龙舌兰亚科(Agavoideae),约有40多种,主要分布在中美至北美的部分地区。丝兰属植物形态优美,叶、茎纤维强韧,在园林观赏和缆绳制作中应用广泛[9-10]。近年来,对丝兰属植物的研究主要集中在组培快繁[11]、理化指标测定[12]、提取物在动物生产中的应用[13]以及该属植物与丝兰蛾(Tegeticula alba)互利共生关系[14]等方面,而有关叶绿体基因组特征及系统发育的研究鲜见报道。相关报道仅见McKain 等[15]从丝兰蛾传粉时间和起源入手,完成了多种丝兰属及近缘属植物的叶绿体基因组测序,通过构建系统发育树首次从分子角度明确了龙舌兰亚科各属间的亲缘关系。然而,关于丝兰属叶绿体基因组的种间变异情况、进化机制及其各物种间的系统发育关系鲜见报道,相关研究亟待进一步探究。
随着第二代测序技术的发展,多个物种的叶绿体基因组被测序并相继公布。截至2022 年1月,NCBI 数据库中已确定发表叶绿体全基因组的丝兰属植物有5种,分别是克雷塔罗丝兰(Y.queretaroensis)、西地格丝兰(Y. schidigera)、柔软丝兰(Y.filamentosa)、短叶丝兰(Y.brevifolia)和Y.jaegeriana。为进一步进行系统发育研究,探索丝兰属内种间关系,利用高通量测序技术获得无刺龙舌兰(Y. treculeana)叶绿体基因组序列,并与属内其他5个种的叶绿体基因组进行比较,通过重复序列检测、边界收缩与扩张分析、序列差异分析及核苷酸多态性分析等揭示丝兰属叶绿体基因组的结构特征和种间序列变异情况。基于叶绿体基因组构建系统发育树阐明6个种间的亲缘关系,以期为丝兰属种间变异情况、进化机制及系统发育等研究提供理论参考。
1 材料与方法
1.1 材料采集与数据收集
无刺龙舌兰新鲜叶片采集于西双版纳热带植物园,放置于超低温冰箱中-80 ℃保存,用于叶绿体基因组测序。在NCBI 数据库中检索丝兰属现已发表叶绿体全基因组的物种信息,下载各物种叶绿体基因组序列,记录物种名及对应的基因组登录号。
1.2 DNA提取及质量检测
选用无刺龙舌兰新鲜嫩叶,利用改良CTAB法[16]提取基因组DNA。提取时在传统CTAB 法的过程中,添加还原剂如β-巯基乙醇来避免褐化物质对DNA 的影响,通过使用氯仿和水饱和酚反复抽提以去除蛋白质、多糖和次生代谢物等杂质,进而加强DNA 的沉淀效果。提取的DNA 分别使用紫外分光光度计、琼脂糖凝胶电泳对其浓度及纯度进行检测。
1.3 叶绿体基因组测序、组装与注释
利用Illumina HiSeq 2500 平台[17]进行高通量DNA 测序,测序数据通过GetOrganelle v1.7.5 软件[18]组装无刺龙舌兰叶绿体基因组,以近缘种克雷塔罗斯兰(GenBank 号:KX931468)叶绿体基因组序列为参考,利用Geneous v8.1.3[19]和ORF Finder(https://www.ncbi.nlm.nih.gov/orffinder/)对叶绿体基因组进行注释和修正。使用在线工具OGDRAW(https://chlorobox. mpimp-golm.mpg.de/OGDraw.html)绘制叶绿体基因组结构图。无刺龙舌兰叶绿体基因组序列数据已上传NCBI 数据库(GenBank号:OL912952)。
1.4 重复序列与SSR分析
利 用 在 线 软 件REPuter(https://bibiserv.cebitec.uni-bielefeld.de/reputer)预测丝兰属叶绿体基因组内散在重复,包括正向重复(F)、反向重复(R)、回文重复(P)、互补重复(C),具体参数设置如下:最短重复单元设为30 bp,汉明距离(Hamming distance)为3。串联重复通过在线工具Tandem Repeats Finder(https://tandem.bu.edu/trf/trf.html)进行分析。丝兰属植物叶绿体基因组的SSR位点采用MISA 软件(https://webblast.ipk-gatersleben.de/misa/)进行检测,相关参数参考马孟莉等[20]进行设置,单核苷酸至六核苷酸单元重复数分别设为:≥10、≥5、≥4、≥3、≥3、≥3。
1.5 丝兰属植物叶绿体比较基因组分析
利 用 在 线 工 具IRscope(https://irscope.shinyapps.io/irapp/)[21]绘制丝兰属植物叶绿体基因组四分体的边界视图,根据4个边界上的基因差异分析其收缩与扩张情况。以无刺龙舌兰叶绿体基因组序列为参照,在mVISTA(https://genome.lbl.gov/vista/index.shtml)[22]分析程序下,用shuffle-LAGAN模式检测丝兰属植物叶绿体基因组的序列变异情况。并基于叶绿体基因组序列(去掉1 个IR 区)比对结果,利用DnaSP 6[23]分析丝兰属植物叶绿体基因组的核酸变异情况,根据核苷酸多样性指数(π)筛选高变异性位点,滑动窗口长度设为600 bp,步长设为200 bp。
1.6 丝兰属植物系统发育分析
为了明确6 种丝兰属植物的系统发育位置及种间亲缘关系,以龙舌兰属植物作为外类群,分别基于全叶绿体基因组和LSC+SSC 区,利用RAxML v8.2.12软件[24]下的HPC2 on XSEDE 模型构建系统发育树,自展值(Bootstrap value)设置为1 000。
2 结果与分析
2.1 丝兰属叶绿体基因组结构与基因注释
6 种丝兰属植物叶绿体基因组均包括LSC、SSC、IRa 和IRb 4 个独立的区域(见图1)。丝兰属植物叶绿体基因组大小为156 185 bp(Y. schidigera)~158 020 bp(Y.jaegeriana),相差1 835 bp。除sghg 叶丝兰的总GC 含量为37.9%之外,其余物种总GC 含量均为37.8%。从编码基因数目来看,无刺龙舌兰、柔软丝兰、克雷塔罗丝兰、短叶丝兰和Y.jaegeriana的基因总数相同,包括蛋白编码基因(85)、tRNA(38)和rRNA(8)的数目完全一致(见表1)。与前5种植物相比,西地格丝兰的叶绿体基因组缺失了1个蛋白编码基因rpl32;6种丝兰属植物叶绿体基因组的tRNA 和rRNA 数目和类型均一致。丝兰属植物叶绿体基因组编码基因主要由自我复制相关基因、光合作用相关基因、其他基因和ycf 类基因组成。统计发现,丝兰属植物叶绿体编码基因中有19 个基因存在双拷贝,包括所有的rRNA,tRNA 基因trnN-GUU、trnR-ACG、trnA-UGC、trnI-GAU、trnV-GAC、trnL-CAA、trnI-CAU、trnH-GUG和 蛋 白 编 码 基 因rps12、rps19、rps7、rpl2、rpl23、ndhB、ycf2。另外,在丝兰属植物叶绿体基因组中还存在trnK-UUU、trnG-GCC、trnL-UAA、trnV-UAC、trnA-UGC、trnI-GAU、rps16、rpl16、rpl2、rpoC1、ndhB、ndhA、atpF、petB、petD15 种含有2 个外显子的编码基因以及rps12、clpP、ycf33种含有3个外显子的编码基因(见表2)。
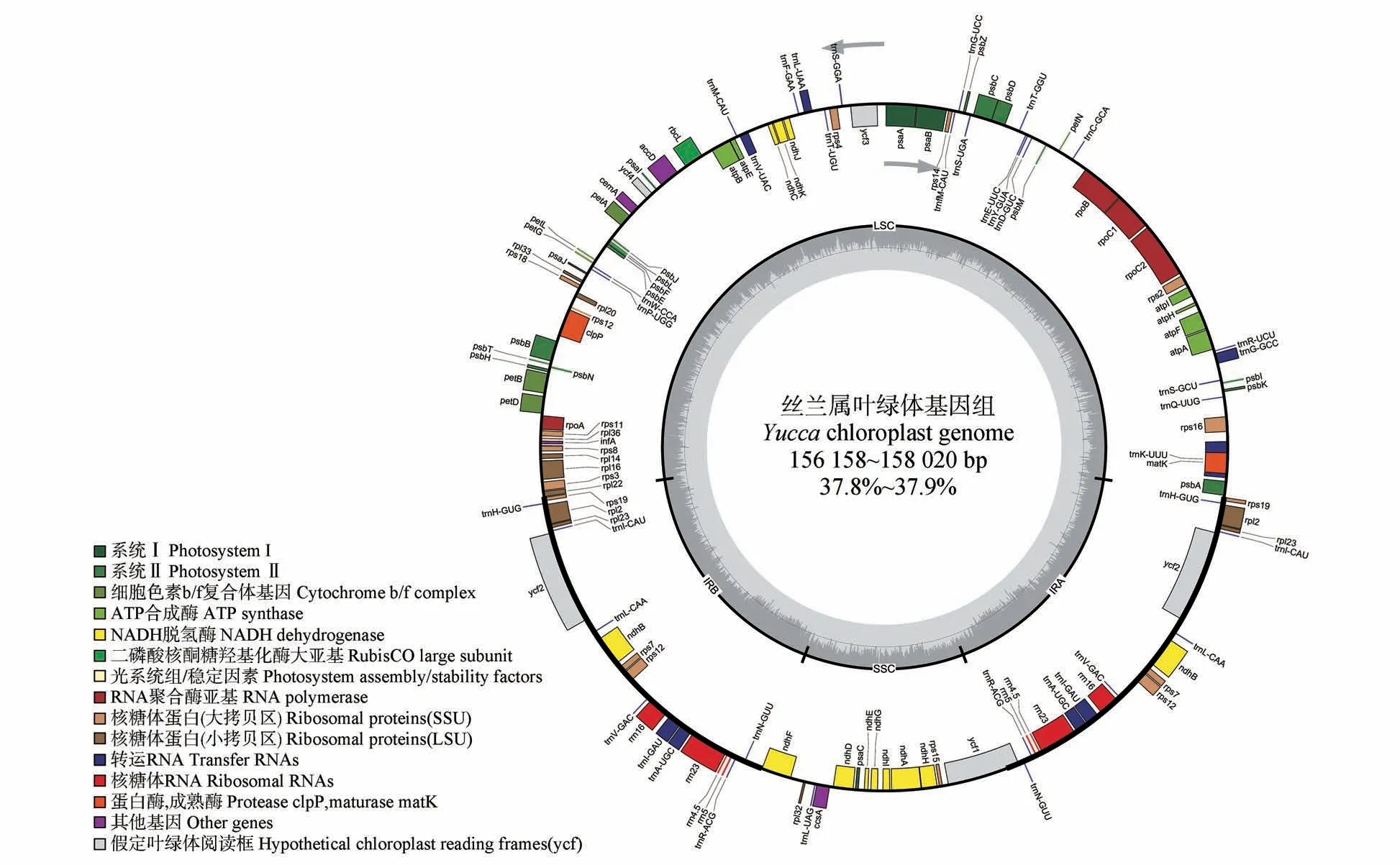
图1 丝兰属叶绿体基因组图谱Fig.1 Chloroplast genome map of Yucca

表1 6种丝兰属植物叶绿体基因组基本结构特征Table 1 Basic structural characteristics of the chloroplast genome of six Yucca species

表2 6种丝兰属植物叶绿体基因组基因类型Table 2 Chloroplast genome gene types of six Yucca species
2.2 重复序列与SSR分析
在6 种丝兰属植物叶绿体基因组序列中分别检测到不等数量的散在重复和串联重复,其中各物种的散在重复总数皆为49 个,除了短叶丝兰无互补重复之外,其余5个丝兰属植物叶绿体基因组序列中均存在正向、回文、互补和反向4 种重复类型,且同类型重复数在各物种之间无明显差异;而利用Tandem Repeats Finder 预测到的串联重复在6 种丝兰属植物中均存在,数目为23~29 个(见图2)。图3显示的是检测到的SSR类型及序列信息,可以看出在丝兰属植物中SSR 类型主要以单核苷酸为主,其次是双核苷酸和四核苷酸(见图3A),统计丝兰属植物各核苷酸重复类型,发现单核苷酸的重复单元基本都是A/T,双核苷酸的重复单元主要是AT,而四核苷酸的重复单元则是以AAAT/ATTT为主(见图3B)。

图2 重复序列类型及数目Fig.2 Type and number of repeats

图3 不同SSR单元及核苷酸重复序列Fig.3 Different SSR units and the nucleotide repeats
2.3 丝兰属叶绿体基因组边界分析
叶绿体基因组进化过程中,边界会发生收缩与扩张现象。6种丝兰属植物叶绿体基因组均为环形四分体结构,存在LSC-IRb、IRb-SSC、SSC-IRa 和IRa-LSC 4个边界。可通过比较边界区域基因的类型和分布状况来推断物种在进化过程中边界的收缩与扩张现象。丝兰属6 种植物叶绿体基因组边界情况如图4 所示,在LSC-IRb 边界区域的基因为rpl22和rps19,除了Y. jaegeriana的边界位于rpl22基因内,其他物种的边界均是在这2个基因的间隔区;丝兰属植物的IRb-SSC边界均位于ndhF基因左侧,但Y.jaegeriana边界距ndhF基因间隔42 bp,相差较大;6种丝兰属植物的SSC-IRa边界均位于ycf1基因编码区,但相比其他物种Y.jaegeriana的ycf1基因向IRa 区多扩张了30 bp;Y.jaegeriana的IRa-LSC边界在rps19基因编码区内,而其他丝兰属植物的IRa-LSC 边界则位于rps19基因和psbA基因的间隔区。由此可以看出,丝兰属植物叶绿体基因组边界在基因类型、收缩及扩张的序列长度等方面较为保守,但仍存在多样性。Y. jaegeriana的4 个边界相比其他物种均存在较大差异,可推测该物种的进化速率较快或是较早的分化物种。

图4 六种丝兰属植物叶绿体基因组四分体边界比较LSC/IRb、IRb/SSC、SSC/IRa和IRa/LSC 4个边界分别由JLB、JSB、JSA和JLA对应标注Fig.4 Comparisons of four regions boundary of chloroplast genomes in six Yucca species The four boundaries of LSC/IRb,IRb/SSC,SSC/IRa and IRa/LSC are marked by JLB,JSB,JSA and JLA,respectively
2.4 丝兰属叶绿体基因组差异
以无刺龙舌兰叶绿体基因组序列为参照,通过在线软件mVISTA 对6 种丝兰属叶绿体基因组全长序列差异进行可视化分析,结果见图5。6 种丝兰属植物叶绿体基因组的编码区(exon)均具有较高的保守性,而非基因编码区(CNS)差异较明显,序列变异程度较高,这主要体现在LSC 区和SSC 区,而IRa 区基本不存在变异。西地格丝兰在116 kb 处,即SSC 区的rpl32、truL-UAG、ccsA、ndhD上及基因间隔区存在较大差异,且在113 kb 处的ndhF基因上也存在较大的序列变异,这可能主要与该物种的基因缺失有关。除此之外,各物种的序列变异位点排列基本一致。变异程度较高的区域基本都位于2 个相邻基因的间隔区内,如psbKtrnS-GCU、rps4-trnF-GAA、rpl32-ccsA-ndhD等,这些高变异位点为丝兰属物种鉴定提供了新的分子标记资源。

图5 六种丝兰属植物叶绿体基因组全序列Fig.5 Full sequences alignment of chloroplast genomes of six Yucca species
结合上述序列差异分析,进一步对6种丝兰属植物叶绿体基因组的高变异区进行探究,发现丝兰属植物叶绿体基因组中π≥0.008 的核酸高变异区域有3 个,其中2 个位于LSC 区,1 个位于SSC区,而IR 区序列核酸变异程度均处于较低水平。结合vMISTA 图谱发现两者分析结果基本一致,进而根据核酸多态性π值确定了变异程度较高的3个基因间隔区位置,分别是psbK-psbl-trnS-GCU(7 861~9 771 bp)、rpl20-rps12(70 855~72 467 bp)和ccsA-ndhD(117 525~119 331 bp)(见图6),这有助于后续丝兰属植物的分子标记开发及分类鉴定。

图6 六种丝兰属植物叶绿体基因组核酸多态性Fig.6 Nucleic acid polymorphisms in the chloroplast genome of six Yucca species
2.5 系统发育分析
通过在线软件MAFFT 7 比对6 种丝兰属植物叶绿体基因组序列,以龙舌兰属植物龙舌兰(Agave americana)和剑麻(Agave sisalana)作为外类群,用最大似然法(ML)构建系统发育树。结果表明,6 种丝兰属植物以100%的检验值划分为两大组:Group Ⅰ和Group Ⅱ(见图7)。无刺龙舌兰、克雷塔罗丝兰、西地格丝兰和柔软丝兰为Group Ⅰ,其中无刺龙舌兰和克雷塔罗丝兰互为姊妹关系,其亲缘关系最近,检验值达86%;Group Ⅱ仅有短叶丝兰和Y.jaegeriana。而Group Ⅰ又可进一步将柔软丝兰单独划分为1 个小支,与其他3 个丝兰属物种亲缘关系较远。
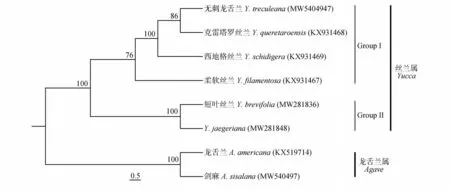
图7 基于全叶绿体基因组构建的丝兰属植物系统发育树Fig.7 Phylogenetic tree of Yucca constructed based on the whole chloroplast genome
3 讨论
植物叶绿体基因组因其结构稳定、进化速率适中、易测序获得等独特优势对植物生长发育、分类鉴定及系统进化等方面具有重大研究意义[25]。通过分析6 种丝兰属植物叶绿体基因组的结构特征,发现各物种间基因结构和序列大小相对保守,不存在大程度的变异,6种丝兰属植物叶绿体基因组均为典型的四分体结构(1 个LSC 区、1 个SSC 区和2 个IR 区),且序列差异多集中于LSC 区和SSC区。各物种所注释到的编码基因种类和数目也基本一致,尤其是tRNA 和rRNA 完全一致,这符合多数近缘种间植物叶绿体基因组具有遗传保守性及稳定性的基本特征[26]。一般来讲,陆生植物叶绿体基因组通常具有较高的保守性。因此,若某个物种叶绿体基因组序列中出现大片段连续碱基的插入或缺失而导致某一基因的获得或丢失,这可能意味着该物种在某一时期发生过进化事件[27]。在6种丝兰属植物中,西地格丝兰叶绿体基因组的SSC区大小仅为16 635 bp,相比其他5种丝兰属植物存在较大差异,最大差异为1 767 bp。通过序列比对发现,造成SSC 区大小差异的原因主要是宽叶丝兰叶绿体基因组的SSC 区出现了2 个大片段的碱基序列缺失(缺失碱基序列大小分别为1 534、116 bp),进而导致rpl32基因的丢失和ndhF基因的注释不完全。这一现象的出现可能与第四纪晚期该物种的历史扩张有关。De La 等[28]在西地格丝兰的遗传数据分析和环境生态位模型(ENMs)研究中就提出该物种具有较高的遗传多样性,且遗传变异随纬度的增加而显著减少,其研究结果支持西地格丝兰在第四纪晚期向加利福尼亚西北部扩张。植物在整个历史扩张过程中长期受自然选择压力的影响,进而可导致基因上的差异(插入、缺失及突变)。Ueda 等[29]在研究杨树(Populus)叶绿体基因组时就发现了rpl32基因丢失事件,但随后该丢失的基因又在核基因组中被鉴定出来,此现象被称为基因转移,是真核细胞进化的重要过程。因此,rpl32和ndhF基因有望作为一种标记资源用于研究西地格丝兰乃至丝兰属的进化与演化。
叶绿体基因组遗传稳定,进化路线独立,由此开发出来的SSR 标记在植物遗传多样性分析、物种分类鉴定等研究中具有明显的优势[30-31]。在6种丝兰属植物中检测到数目最多的SSR 类型是以A/T碱基为主的单核苷酸重复,其次是双核苷酸和四核苷酸,且其序列的碱基组成也均是A 和T,说明丝兰属植物叶绿体SSR 偏好使用A 和T 碱基。重复序列在植物叶绿体基因组中普遍存在,可导致叶绿体基因组某片段重复、缺失及重排,进而影响物种的进化以及种内基因的遗传变异[32]。在6种丝兰属植物中发现的散在重复总数一致,且重复序列类型主要为正向重复和回文重复,但各物种间包括串联重复在内的各类型重复数目却不一致,这说明6种丝兰属植物的突变频率存在一定差异。这一现象在梧桐属(Firmiana)叶绿体基因组中同样存在,从2种梧桐属植物叶绿体基因组中鉴定出来的重复序列总数均为49 个,且正向重复和回文重复占85%以上,但各类型重复序列的数量却不尽相同[33]。
IR 区在维持叶绿体基因组稳定中发挥着重要的作用。相关研究表明,植物在进化过程中IR 区常发生收缩或向单拷贝区扩张的现象,进而导致叶绿体基因组结构变异[34]。6 种丝兰属植物叶绿体基因组边界(LSC/IRb、IRb/SSC、SSC/IRa 和IRa/LSC)几乎不存在变异,只有Y.jaegeriana的边界相比其他物种向后扩张了约30 bp,表明其在进化过程中基因组结构发生了变异,推测原因可能是该物种进化速率较快或是较早分化。通过比对全叶绿体基因组发现,6种丝兰属植物基因编码区均具有较高的保守性,而非基因编码区差异较明显,序列变异程度较高,这主要表现在LSC 区和SSC 区。蒋礼玲等[7]在藜属植物叶绿体基因组核苷酸变异研究中也证明了这一点,说明对大多数被子植物而言,LSC 和SSC 区相比IRs 区具有更高的变异性,而丰富的变异区更有助于物种的系统发育研究。
通过分析叶绿体基因组序列多样性,进而检测基因变异情况,可初步确定各物种间的高度变异区或筛选出核苷酸多样性较高的基因,用于开发新的DNA 条形码,这对物种鉴定分类意义重大[2,35]。Cui 等[36]对比分析了6 种鼠尾草属(Salvia)植物叶绿体基因组序列,发现了6 个基因和4个基因间隔区可能是鼠尾草属植物的特异条形码。本研究在6 种丝兰属植物叶绿体基因组中根据核酸多态性π≥0.008 确定的3 个高变区psbK-psbl-trnS-GCU(7 861~9 771 bp)、rpl20-rps12(70 855~72 467 bp)和ccsA-ndhD(117 525~119 331 bp)可作为候选分子标记用于开发特异DNA 条形码,以帮助丝兰属植物的分类鉴定。现已有研究发现,在丝兰属植物叶绿体基因组中发现的非基因编码片段psbl-trnS和ccsA-ndhD因较高的序列变异性现已分别应用于石豆兰属(Bulbophyllum)[37]和蝴蝶兰属(Phalaenopsis)[38]的物种鉴别及DNA 条形码设计。
同属植物叶绿体基因组虽表现出高度的保守性,但各物种间仍存在一定程度的变异,这些变异位点非常适用于研究近缘同属植物的系统发育关系[7]。通过比对分析6 种丝兰属植物叶绿体基因组的序列变异程度,发现其高变异位点主要集中在叶绿体基因组的LSC 区和SSC 区。分别将这2个区域序列单独提取出来构建的系统发育树图各分支结果与利用全基因组序列构建的发育树图分类完全一致,且均具有较高的支持率。因此,在丝兰属植物中完全可以利用LSC+SSC 区丰富的核苷酸变异位点对该属植物进行精确分类。

