提取鼠肠道内微生物基因组DNA 的方法研究
李丽婷 许文涛, 郭 星 姚业成 黄昆仑,
(中国农业大学食品科学与营养工程学院1,北京 100083)
(农业部转基因产品检验监督测试中心2,北京 100083)
提取鼠肠道内微生物基因组DNA 的方法研究
李丽婷1许文涛1,2郭 星2姚业成1黄昆仑1,2
(中国农业大学食品科学与营养工程学院1,北京 100083)
(农业部转基因产品检验监督测试中心2,北京 100083)
通过改进鼠肠道微生物基因组DNA提取方法以期更全面地反映鼠肠道菌群的真实情况。采用CTAB和 SDS结合的方法裂解细胞,同时改进了传统的酚/氯仿抽提方法,提取全过程控制在 2 h以内。通过紫外分光光度计,细菌通用引物 PCR,总菌群实时定量 PCR,变性梯度凝胶电泳 (DGGE)对改进方法及两种试剂盒法提取 DNA的结果进行比较,评价了所建立的高效提取方法。与试剂盒相比,改进的方法 DNA得率较高,简便快速,成本低廉。同时后续的 PCR鉴定及DGGE菌群多样性分析显示,改进的方法可以更好地揭示鼠肠道菌群分布和特性。
鼠肠道微生物 DNA提取方法 CTAB/SDS实时定量 PCR 变性梯度凝胶电泳
鼠肠道内是一个复杂的生态体系,含有 500~1 000种细菌,总数高达 1013~1014,其包含的基因数目是自身基因组的 100倍[1]。肠道微生物群落与宿主个体的健康和疾病状况息息相关,它不仅能降解食物中不可消化的营养成分、提供宿主维生素等营养物质,还能促进肠上皮细胞的分化与成熟、激活肠道免疫系统以及调节宿主能量存储与代谢[2]。因此,越来越多的研究人员已经认识到肠道微生态研究的重要意义。
从肠道样品中提取的 DNA被广泛的用于遗传和生态学研究中。近年来,现代分子生物学方法如聚合酶链式反应 (PCR),实时定量 PCR(real-time PCR),变性梯度凝胶电泳 (DGGE),限制性片段长度多态性 (RFLP),随机扩增的多态性DNA(RAPD)等技术广泛用于分析肠道微生物多样性[3-6]。传统培养方法很难全面地反映肠道菌群的结构特征,而现代分子生物学技术不依赖于微生物培养,可以从肠道样品中分出更多具体的微生物属种,从而进一步揭示了肠道中微生物菌群结构的分布。然而,前面所提到的以 PCR分子技术为核心的试验结果很大程度上依赖于肠道微生物中 DNA模板的质量和数量,也就是说提取好的 DNA是分子生物学研究的关键一步。因此,很多研究者都报告了不同样品的传统DNA提取法的改良方法[7-9],各种DNA提取试剂盒的应用也很广泛。然而大多数传统方法复杂又耗时,还用到了有毒试剂和价格高的试剂,如β-巯基乙醇,溶菌酶等;很多 DNA提取试剂盒的价格偏高,不适合处理大批量的样品,而且有些试剂盒的 DNA产量相对较低[10],不适合做后续分子生物学的研究。目前市面上还没有专门用于从动物肠道中提取微生物基因组 DNA的试剂盒。因此,寻找一种适宜的方法是进一步研究鼠肠道菌群的当务之急。
本试验采用一种改进的高效快速的 DNA提取方法,并与两种常用 DNA提取试剂盒做比较,通过实时定量 PCR和DGGE考察不同提取方法的优劣和对后续分子生物学研究的影响。
1 材料和方法
1.1 主要材料及试剂
鼠肠道内容物样品:新鲜的肠道内容物从乙醚处理过的 SD大鼠盲肠中得到。样品收集在盛满冰的无菌袋中,立即被送到实验室,真空冷冻干燥后,-80℃冷冻保藏。
Qiagen DNA StoolMini Kit:德国 Qiagen公司;TI2 ANamp细菌基因组DNA提取试剂盒:天根生化科技有限公司;PCR扩增试剂盒 (dNTPs、Buffer、Taq酶 ),引物:大连宝生物公司;SYBR Green MasterMix:日本 TOYOBO公司;Tris-饱和酚、氯仿、异戊醇、醋酸钠、无水乙醇 (分析纯):科昊达生物技术公司;琼脂糖 (分析纯):西班牙 biowest公司;丙烯酰胺、甲叉双丙烯酰胺、尿素、去离子甲酰胺 (超级纯):Amresco公司。
1.2 主要仪器及设备
AB I-2720 PCR扩增仪:美国 Applied Biosystems公司;AB I7000实时定量 PCR扩增仪:美国 Applied Biosystems公司;CLASSⅡ TYPE B2 PCR生物安全柜:哈东连仪器设备厂;5804R型离心机:德国 Ep2 pendorf公司;CHEM I-S MART 3000WL/LC荧光凝胶成像系统:法国 VilberLour mat公司;UV IKON系列紫外可见分光光度计:法国 Secomam公司;电泳仪,DCode通用突变检测系统:美国 Bio-Rad公司。
1.3 方法
1.3.1 改良的酚 /氯仿抽提法
取约 50 mg肠道内容物样品各 3个,用液氮研磨5 min,分别置于 2 mL小离心管,加入 lmL TE缓冲液(pH 8.0,10 mmol/L Tris-HCl,1 mmol/L EDTA-Na2),振荡均质后 12 000 r/min离心 5 min,去上清液,沉淀中再加入 1 mL TE缓冲液,重复操作一遍。在离心得到的沉淀中加入 0.1 mL质量分数为10%SDS混匀,37℃保温 20 min。加入 5 mol/L NaC1,质量分数为 10%的 CTAB和质量分数为 1%的抗坏血酸各 0.2 mL,65℃保温 20 min。用等体积Tris-饱和酚 ∶氯仿 ∶异戊醇 (25∶24∶1)抽提,12 000 r/min离心5 min。取上清,重复操作一次。取上清;再加入等体积氯仿 ∶异戊醇 (24∶1),12 000 r/min离心 5 min。加入 1/10体积 3 mol/L NaAC,及 1倍体积异丙醇,液氮中放置 10 min立即取出。12 000 r/min离心 5 min取沉淀,用 70%乙醇洗涤一次,于室温晾干。沉淀用 100μL TE缓冲液溶解,加RNA酶 (5μL/100μL)37℃保温 10 min,-20℃保存备用。整个过程在 2 h左右。
1.3.2 TI ANamp细菌基因组 DNA提取试剂盒法
取约 50 mg肠道内容物样品各 3个,按照试剂盒的使用说明提取肠道菌群 DNA,溶于 100μL TE缓冲液中。
1.3.3 Qiagen DNA StoolMini Kit提取法
取约 50 mg肠道内容物样品各 3个,按照试剂盒的使用说明提取肠道菌群 DNA,溶于 100μL TE缓冲液中。
1.3.4 DNA质量检测
1%琼脂糖凝胶电泳、Gold Veiw染色、凝胶成像仪观察分析;紫外分光光度计分别在波长 260 nm和280 nm处检测样品总 DNA浓度和纯度,最后计算DNA得率。
1.3.5 总细菌通用引物 PCR
扩增通用引物为:上游引物 338f(5’-ACTC2 CTACGGGAGGCAGCAG-3’)和下游引物 518r(5’-ATTACCGCGGCTGCTGG-3’),其中上游引物带一个 GC串 (CGCCCGCCGCGCGCGGCGGGCGGGGC GGGGGCACGGGGGG),扩增菌群的 16S r DNA V3区,由大连宝生物公司合成。扩增反应体系为25μL,组成为:1×PCR缓冲液 (含 3%MgCl2),0.2 mmol/L dNTPs,0.5μmol/L双引物,1μL模板DNA和 0.025 U/μLTaq酶。扩增程序为预变性94℃5 min、35个循环 (94℃变性 30 s、58℃退火30 s、72℃延伸 30 s),72℃延伸 5 min。PCR产物用2%琼脂糖凝胶电泳,Gold Vei w染色后用凝胶成像仪观察分析。
1.3.6 实时荧光定量 PCR(SYBR Green I)
检测样品基因组 DNA引物如下,扩增片段大小为 466 bp:F:5’-TCCTACGGGAGGCAGCAGT-3’;R:5’-GGACTACCAGGGTATCTAATCCTGTT-3’。
标准曲线的构建:用紫外分光光度计测定大肠杆菌纯菌株 (Escherichia coli)DNA的 OD值及浓度,换算为标准菌株的拷贝数 (1.33×108),分别做10倍系列稀释的大肠杆菌纯菌株 DNA(10-1~10-5),用上述引物扩增,构建标准曲线。
实时定量 PCR反应体系及条件:总体积 30μL,包含 15μL 2×SYBRGreen Master Mix,引物(0.6μmol/L),DNA模板 1μL。PCR扩增反应条件:首先 95 ℃ 5 min;然后 95 ℃ 15 s、58 ℃ 30 s、72℃30 s进行 30个循环;熔点曲线分析 95℃15 s,60 ℃20 s,95 ℃15 s。
样品的检测:每种提取方法分别取 3个样,每个样做一个重复,反应体系及条件如上所述,结果 Ct值取平均,对照标准曲线计算菌群数量,用 log genome/g样品表示。
1.3.7 DGGE(变性梯度凝胶电泳)
DGGE在 DCode通用突变检测系统上运行,聚丙烯酰胺凝胶浓度 8%~12%(37.5∶1,丙烯酰胺 /甲叉双丙烯酰胺),电泳缓冲液 0.5×TAE(20 mmol/L Tris-acetate,pH 7.4,10 mmol/L乙酸钠,0.5 mmol/L Na2EDTA),变性梯度 35%~60%(100%指的是 7 mol/L尿素和体积分数为 40%去离子甲酰胺),电泳温度 59°C,电压 60 V,10 min然后跑160 V,4 h。SYBR Green I染色 30 min,用荧光凝胶成像仪照相。
2 结果与分析
2.1 琼脂糖凝胶电泳观察不同方法提取的鼠肠道微生物基因组 DNA效果
TI ANamp细菌基因组 DNA提取试剂盒是专门从各种革兰氏阴性、阳性细菌中快速提取到高质量的基因组DNA,试验用这种试剂盒提取鼠肠道内总菌群基因组 DNA,如图 1所示,在琼脂糖凝胶电泳图上几乎不可见,说明其用于肠道内总菌群的提取效率较低,DNA模板数较少。Qiagen DNA Stool Mini Kit使用硅胶膜技术,从新鲜或冷冻粪便或其他带有高浓度 PCR抑制剂样品中纯化提取基因组 DNA。在本试验中,Q I Aamp DNA Stool Mini Kit和改进的酚/氯仿抽提方法提取的基因组 DNA在琼脂糖凝胶电泳图上的条带都很亮,弥散较少,体现了改进酚/氯仿抽提法的优越性。
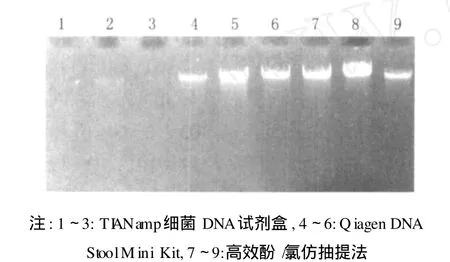
图 1 三种不同方法提取的肠道基因组DNA琼脂糖凝胶电泳图
2.2 紫外分光光度计检测鼠肠道菌群总 DNA质量
如表 1统计结果所示,三种不同提取方法均能达到 DNA纯度要求 (1.8~2.0之间)。就浓度和得率来看,高效酚/氯仿抽提法和 Qiagen DNA Stool Mini Kit法的 DNA产量均较高,其中高效酚 -氯仿抽提法比 Qiagen DNA StoolMini Kit法稍好一些,而TI ANamp细菌DNA试剂盒的浓度和得率只有上述两种方法的 1/3左右。
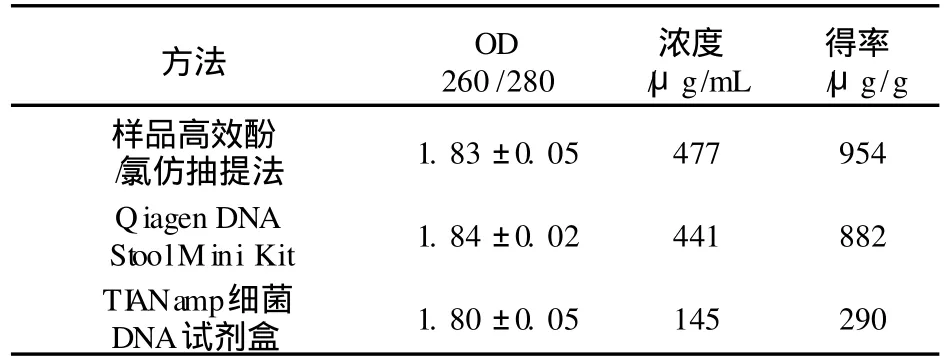
表 1 紫外分光光度法检测 3种方法提取的样品菌群总DNA浓度、纯度及得率
2.3 三种不同方法提取的肠道菌群DNA的应用考察
2.3.1 细菌通用引物 PCR
从图 2可见,使用细菌通用引物扩增,扩增片断根据肠道内细菌的种类分布在 200 bp左右。T IANa2 mp试剂盒法提取的菌群基因组 PCR产物亮度低,说明其起始模板拷贝数低或含有某些 PCR抑制因子。高效酚 -氯仿抽提法提取的菌群基因组 DNA经过扩增后的产物主带清晰明亮,无明显弥散带,无 RNA残留,说明其模板拷贝数最高,PCR抑制因子相对较少。
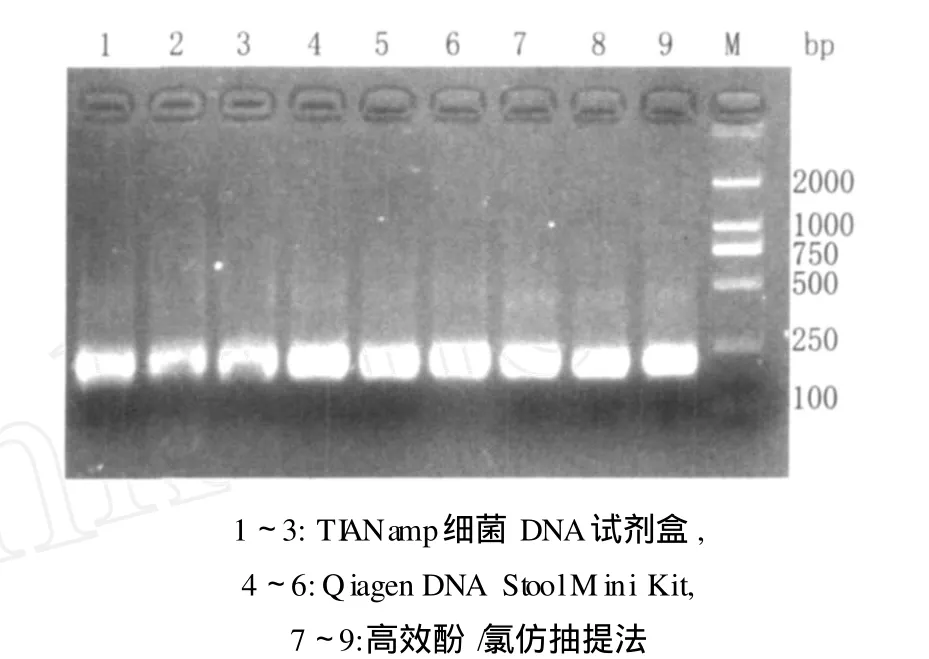
图 2 鼠肠道菌群基因组DNA的 16S r DNA扩增电泳图
2.3.2 SYBR GreenⅠ荧光染料实时定量 PCR
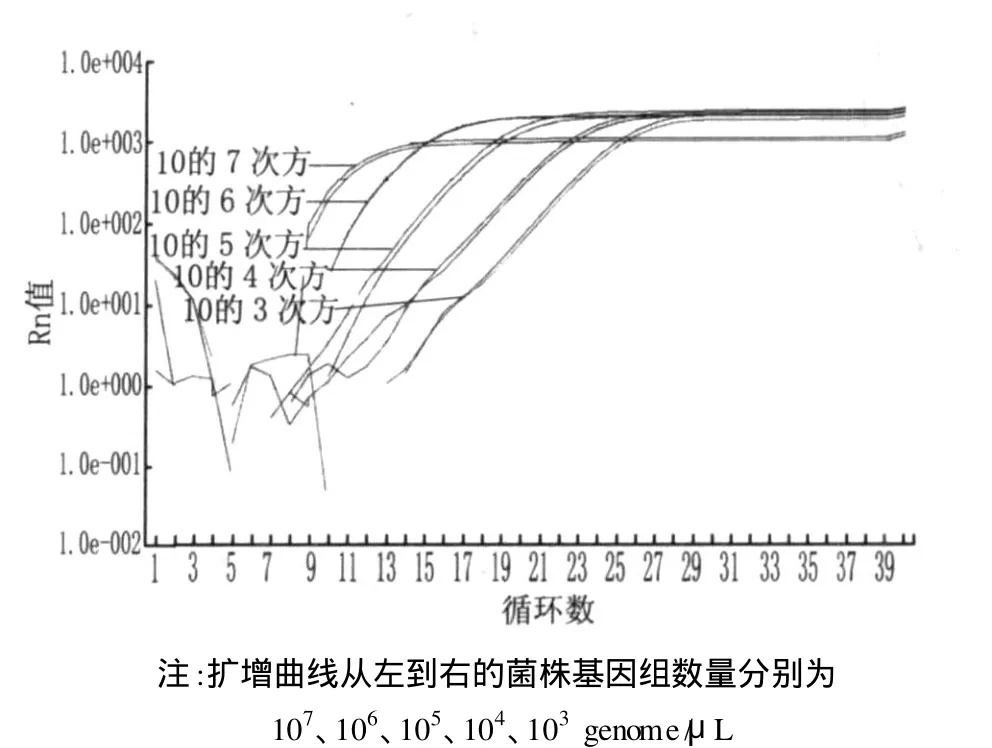
图 3 不同稀释浓度的总菌群的实时定量 PCR扩增曲线图
如图 3扩增曲线和图 4标准曲线表明,循环阈值(Ct值)与标准菌株拷贝数的对数之间存在显著的线性关系 (R2=0.995 1),为待测样品的定量提供了参照标准。从图 5扩增曲线图可以看出,不同处理的样品总菌群数有明显差异,从左到右分别为改进的酚 /氯仿法,Qiagen试剂盒法及 TI ANamp试剂盒法提取的样品。对照标准曲线,经过统计计算得出改进的酚 /氯仿法得到的总菌群数 (log genome/g样品)为 11.51±0.03,Qiagen试剂盒法为 10.85±0.14,TI ANamp试剂盒法为 8.47±0.10。从图 6熔解曲线可以看出,不同方法提取肠道菌群 DNA的 PCR产物特异性好,条带单一,测得的数据可靠。结果表明,改进的酚/氯仿法和 Qiagen试剂盒法能得到较高的菌群数量,其中改良的方法更好一些,而用 TI ANamp试剂盒法得到的菌群数量明显较低,仅是改良酚 /氯仿法的 1/1 000。
2.3.3 DGGE菌群多样性分析
为了进一步考察三种不同方法在菌群分布方面的应用性和可靠性,试验对所提取基因组的 PCR产物进行了 DGGE电泳分析,如图 7所示,7~9泳道的条带最多,说明该法提取的微生物多样性丰富,而 1~3泳道的条带最少,说明微生物多样性相对贫乏。由此可知,改进的酚/氯仿法提取的 DNA有利于后续的微生物多样性分析,可以更全面地揭示菌群特征及其分布,而其它两种试剂盒法 (尤其是 TI ANamp试剂盒法)提取的DNA明显不足以进行 DGGE电泳分析。
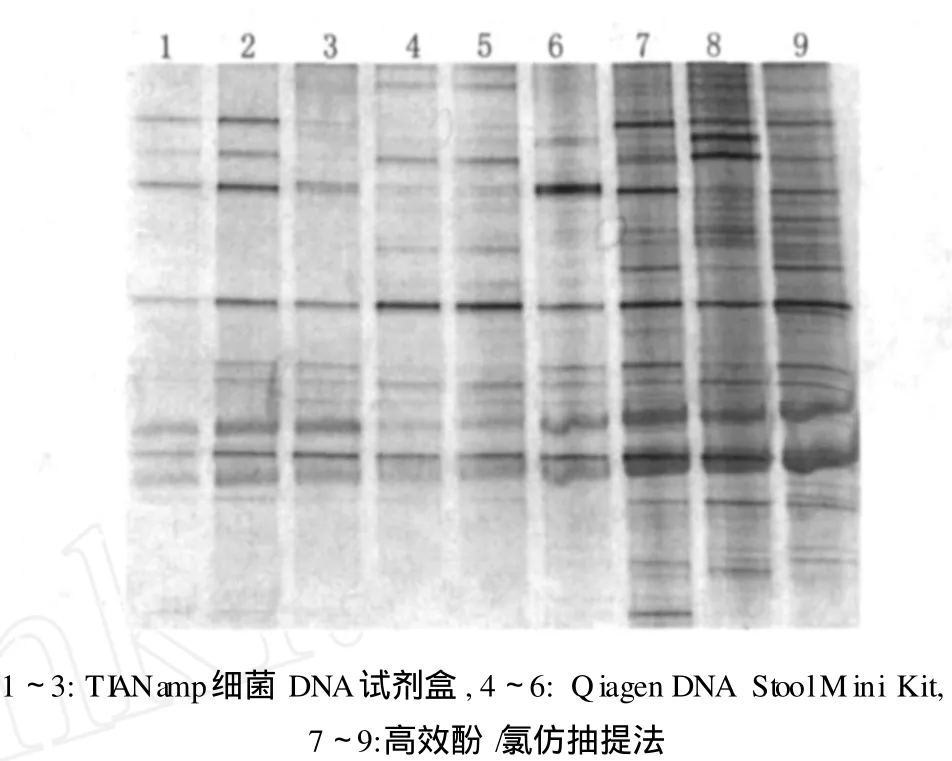
图 7 三种不同方法提取基因组的DGGE图谱
3 讨论
试验结果根据样品保存和提取方法的不同有很大差别[11],首先应该根据样品的特性选择提取DNA的适宜方法[12]。鼠肠道内是一个复杂的环境,不仅寄生着上千种菌群,还有未完全消化的食物残渣,腐殖质,肠壁上脱落的内皮细胞,各种有机物和无机物等[13]。其中存在很多的 PCR抑制因子 (如多糖,脲,腐酸或者血红素),能够轻微抑制或者完全抑制 PCR的扩增,因此对于在进行 DNA提取前对样品进行前处理以去除抑制因子是很有必要的[14]。
试验采用真空冷冻干燥的方法对肠道内容物进行预处理,可以最低限度地减少 DNA在保存期的降解。在前处理时采用液氮研磨样品,方便细胞破碎和DNA的释放,同时在低温环境下可以抑制DNA酶的降解作用。应用 TE缓冲液二次清洗,高速离心后上清液基本澄清,说明已经去除了肠道菌群中的颗粒杂质和色素。尽可能的收集粪便中的肠道菌,使得到的总 DNA能较全面地反映肠道菌群结构的特征。菌体细胞裂解的效率直接影响获取基因组DNA的质量,采用 CTAB和 SDS结合的方法裂解细胞去除蛋白提取 DNA,取得了很好的效果。PCR试验结果说明改进的酚/氯仿抽提法在一定程度上去除了PCR抑制因子的不利影响。通过 DGGE分析,可以看到提取质量高的DNA,菌群丰富度相对较大,有利于后续的分子生物学研究。
综合起来,本试验方法有如下优点:(1)改进的酚 /氯仿提取法 DNA得率更高,且对后续 PCR反应的不利影响最小。(2)节省了大量成本,只用到一些基本的试剂如 Tris-饱和酚,氯仿,SDS,CTAB等,用量很小,且不需要溶菌酶,蛋白酶 K等价格较高的试验药品,很适合处理大批量样品。(3)试验时间短于一般的传统酚/氯仿提取法,如操作熟练,能控制在2 h内完成。
4 结论
4.1 改良后的酚 /氯仿法所提取的肠道菌群基因组数量达 11.51(log genome/g样品),显著高于 TI ANa2 mp试剂盒法得到的菌群数量,同时在成本上要优于Qiagen试剂盒法,是一个提取鼠肠道微生物基因组行之有效的方法,特别适用于处理大批量样品。
4.2 实时荧光定量 PCR是目前常用的分子生物学检测手段之一,能够快速精确地测定菌群基因组拷贝数,在检测过程中 SYBR GreenⅠ荧光染料发出强荧光信号,结合在 DNA双链的小沟部位,其荧光量与扩增产物量成正比,每个循环的延伸阶段收集信号监测 DNA的扩增数量,就可以推出起始模板量[15],而紫外分光光度计在 DNA浓度比较低时,就会测不准甚至测不出来数值。因此实时荧光定量PCR在用于肠道菌群检测的准确性和敏感度上要远远优于传统的紫外分光光度计。
4.3 改良后的酚/氯仿法所提取的肠道菌群 DNA通过DGGE菌群多样性分析,可更全面地揭示鼠肠道微生物群落分布,有利于对整体菌群的变化或针对某一菌种进行检测研究。
[1]Savage D C.Microbial ecology of the gastrointestinal tract[J].Annual Review ofMicrobiology,1977,31:107-133
[2]Nicholson J K,E Holmes,W ilson ID.Gutmicroorganis ms,mammalian metabolism and personalized health care[J].Nature ReviewsMicrobiology,2005,3(5):431-438
[3]W iseM G,Siragusa G R.Quantitative analysisof the intesti2 nal bacterial community in one-to three-week-old com2 mercially reared broiler chickens fed conventional or antibiot2 ic-free vegetable-based diets[J].Journal of Applied Mi2 crobiology,2007,102:1138-1149
[4]Simpson J M,McCracken V J,Gaskin H R.Application of denaturing gradient gel electrophoresis for the analysis of the porcine gastrointestinal microbiota[J].Journal of Microbio2 logicalMethods,1999,(36):167-179
[5]袁志辉,蓝希钳,杨廷,等.家蚕肠道细菌群体调查与分析[J].微生物学报,2006,46(2):285-291
[6]吴天星,金勇丰,曹广力,等.鸡肠道菌基因组 DNA的RAPD分类的研究[J].畜牧兽医学报,2002,33(3):247-249
[7]赵健元,李进华.一种高效的哺乳动物粪便 DNA提取通用方法[J].激光生物学报,2008,17(5):695-700
[8]Thakuria D,Schmidt O,Liliensiek A K.Field preservation and DNA extraction methods for intestinal microbial diversity analysis in earthwor ms[J].Journal ofMicrobiologicalMeth2 ods,2008,10:1-8
[9]杨德君,吴襟,刘毅,等.一种快速提取肠道微生物总DNA的方法[J].中国微生态学杂志,2006,18(2):91-93
[10]Tang J N,Zeng Z G,WangH N.An effectivemethod for i2 solation ofDNA from pig faeces and comparison of five dif2 ferent methods[J].Journal of Microbiological Methods,2008,75:432-436
[11]Zhou X Q,Wang Y F,Cai Y.PCR-DGGE detection of the bacterial community structure in the InnerMongolia steppe with t wo different DNA extraction methods[J].Acta Eco2 logica Sinica,2007,27(5):1684-1689
[12]McOristA L,Jackson M,Bird A R.A comparison of five methods for extraction of bacterial DNA from human faecal samples[J].Journal ofMicrobiologicalMethods,2002,50:131-139
[13]Gillan D C,SpeksnijderA G,Zwart G.Genetic diversity of biofilm covering Montacutaferruginosa (Mollusca,Bi2 valvia)as evaluated by denaturing gradient gel electrophore2 sis analysis and cloning of PCR amplified gene fragment coding for 16S rDNA[J].Applied and EnvironmentalMi2 crobiology,1998,64(9):3464-3472
[14]许文涛,黄昆仑,芦云,等.稻米深加工产品基因组提取方法及其对 PCR的影响[J].农业生物技术学报,2007,15(1):97-101
[15]赵焕英,包金风.实时荧光定量 PCR技术的原理及其应用研究进展[J].中国组织化学与细胞化学杂志,2007,16(4):492-497.
An EffectiveMethod for Genomic DNA Extraction from Rat IntestinalMicroflora
LiLiting1XuWentao1,2Guo Xing2Yao Yecheng1Huang Kunlun1,2
(College of Food Science and Nutrition Engineering,China AgriculturalUniversity1,Beijing 100083)
(Supervision,Inspection and Testing Center of GeneticallyModified Orgini sms,Ministry ofAgriculture2,Beijing 100083)
A modifiedmethod for extractingmicrobial communityDNA from rat intestinal samples to fully reflect the real situation of rat intestinalmicroflora is described in this paper.Combination of CTAB and SDS was used for cell lysis,the traditional DNA extraction method was i mproved,and the whole process was controlled within two hours.Thismodified method and other two DNA extraction kitswere evaluated byUV spectrophotometer,bacterial u2 niversal primer PCR,real-time quantitative PCR and denaturing gradient gel electrophoresis(DGGE).Results show the modified method has a higher yield of DNA and it is simple,rapid and cost-effective compared with the other t wo kits.The PCR and DGGE show that the modified method can reveal the distribution and trait of rat intesti2 nalmicroflora better.
rat intestinal microflora,DNA extraction method,CTAB/SDS,real-time quantitative PCR,DGGE
Q-33
A
1003-0174(2010)01-0122-06
国家高技术研究发展计划 863项目(2006AA10Z440),国家自然科学基金项目(2007-Z8)
2009-01-21
李丽婷,女,1983年出生,硕士,营养与食品安全
黄昆仑,男,1968年出生,副教授,博士生导师,转基因生物安全评价与检测技术

