合成生物系统的组合优化
顾群,李一凡,陈涛
1天津大学化工学院生物工程系,天津 300072
2教育部系统生物工程重点实验室,天津 300072
合成生物学旨在工程学思想的指导下,从头设计并构建新的生物元件、模块和系统,或对现有的、天然的生物系统进行重新设计和改造[1]。利用合成生物学的工程思路,人们不仅建立了模拟物理学的基因电路,包括双稳态开关、振荡器、逻辑门等,还希望通过重新构建生物代谢网络,解决生物化工、生物能源、生物材料、生物医药领域以及关系国计民生的大问题[2]。
对生物元件和系统进行完全理性设计,实现生物系统的标准化一直是合成生物学家多年追求的目标[1,3]。代谢工程最早也致力于利用系统已知计量关系、动力学方程及相关知识对细胞表型进行理性修饰[4]。随着系统生物学和各种组学技术的飞速发展,人们对生物扰动效果的预测能力及对生物系统理性设计的能力都逐渐增强[5]。然而,越来越多的研究也表明,由于生物系统的高度复杂性和非线性的特点,基因线路的完全理性设计通常无法实现功能最优化。于是,一种“半理性”的工程方法——组合工程方法应运而生[6]。
组合优化的工程思路包括以下3个元素:1)通过特定的技术手段构建优化对象 (基因线路,代谢网络或细胞)的文库。2)在文库中筛选获得功能优化的对象。3)检测并分析赋予对象优化功能的要素[6]。组合优化的技术思路与生产菌株的进化相似,但其与传统进化方法相比仍有很大不同。传统进化更偏向于在基因组范围内进行随机突变,而组合优化通常是对系统功能有影响的关键元素进行集中突变或修饰来获取最优表型。这种集中突变的思路既可以更快速高效地获得丰富的基因型或表型,又能同时避免不相关区域的不利突变。换言之,组合优化是介于进化和理性设计之间的优化策略,是一种对进化的理性设计。本文介绍了组合优化方法的工程策略和技术原理,聚焦于设计构建文库多样性的方法,并总结和评述了组合工程方法在工程中的应用及进展 (表 1)。
1 单个元件的微调
生物元件是构成合成基因线路的基本元素[1],因此生物元件的特性和功能直接影响到合成基因线路的功能。一个新合成的基因线路通常需要对其组成元件进行微调,以使线路发挥出最理想的效果。合成基因线路的元件包括复制起始位点、启动子、RBS序列、基因间序列、终止子、功能蛋白等等[7],通过对上述各单个元件的微调可以改善基因线路的性能。
1.1 优化质粒的拷贝数
现今绝大多数功能线路和代谢通路都是在质粒上构建,而质粒拷贝数对基因线路的功能会产生一定影响。高拷贝质粒使功能蛋白基因获得高水平表达的同时,却给细胞带来较大的生理负担。此外,代谢通路中特定酶的高水平表达会对细胞产生毒性物质,阻碍细胞行使某些必需功能。Jones等发现,将表达DXP合酶的基因从高拷贝数的质粒转移到低拷贝数的质粒上,番茄红素的产量可以提高2~3倍[8]。然而如今代谢工程和合成生物学中可供选择的质粒仍然比较有限,且一些质粒之间具有不相容性 (比如pSC101和p15A),因此通过改变质粒拷贝数还无法实现功能元件表达强度的连续性微调。
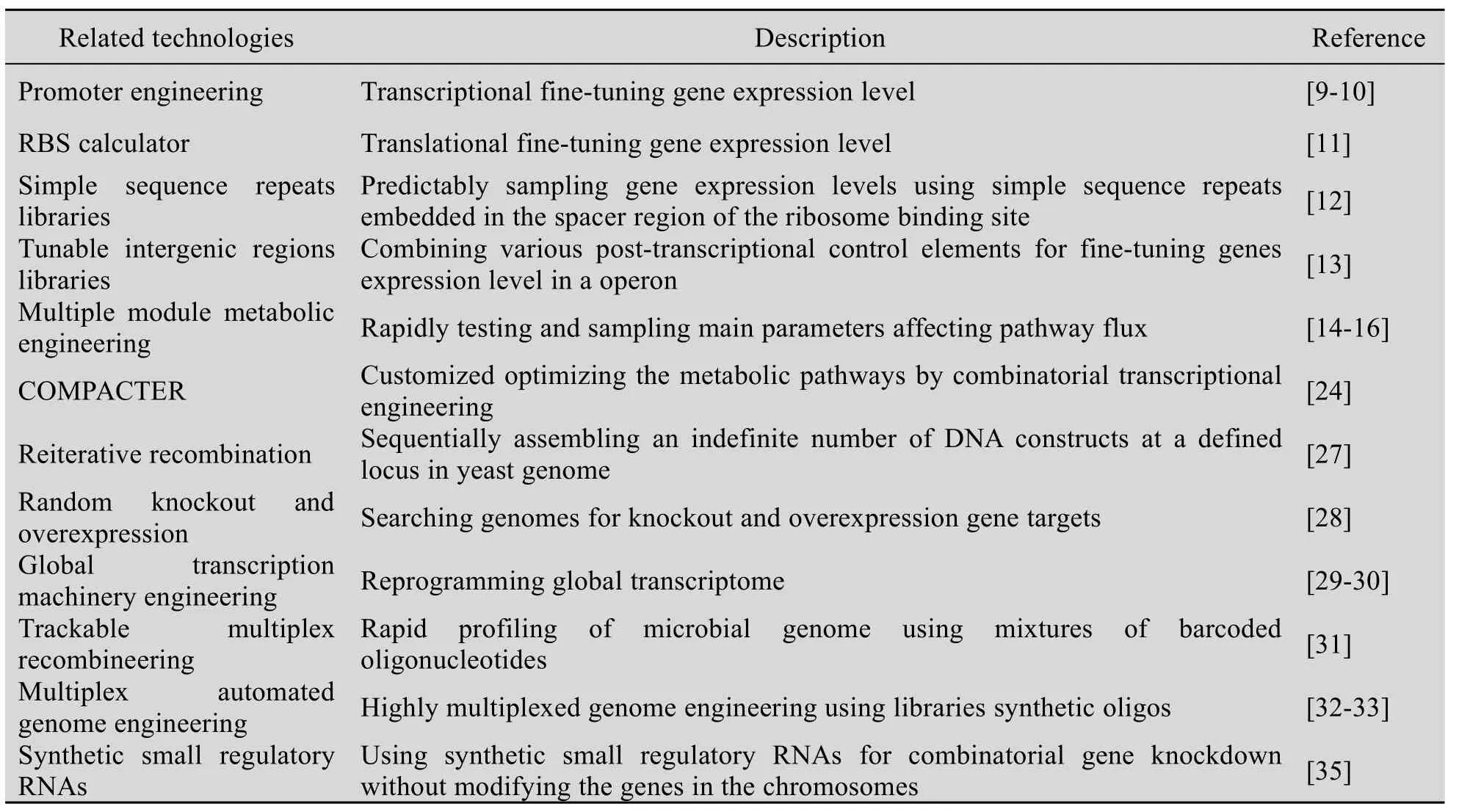
表1 合成生物系统的组合优化方法的研究进展Table 1 Summary of research on combinatorial optimization of synthetic biological systems
1.2 合成启动子文库
启动子是结构基因上游起始转录的一段序列,它的强度决定了结构基因的转录强度,因此启动子成为优化基因线路功能的一个重要对象。Alper等最先使用构建启动子文库的方法来优化代谢通路[9]。他们首先用易错PCR法在高性能天然启动子中引入点突变,随机引入的点突变使文库中的启动子具有不同的强度,由此构建了大肠杆菌PLteto-1启动子文库和酿酒酵母TEF启动子文库。之后,作者利用获得的文库优化了dxs基因的表达强度,以提高番茄红素的产量。研究发现,启动子强度最大时,菌株生产番茄红素的产量并非最高,最高产番茄红素菌株的代谢通路上启动子的强度仅为中等,这一现象说明基因表达强度过高并不能使菌株获得相对优化的产量。Ellis等通过设计简并引物,构建了具有不同表达强度和抑制强度的诱导型启动子文库,这种方法成功应用于前馈环和计时器的微调,最终实现了对酵母沉降时间的控制[10]。此方法并不完全局限于进行非理性微调,而是将非理性的优化与理性的模型预测进行结合,从而降低了优化代谢通路的时间和成本。如今,启动子文库在很多宿主细胞中都能被构建出来,它们能用于优化很多生物系统的功能。
1.3 基于RBS位点的调控工具
RBS位点是调节翻译强度的重要元件。Salis等结合热力学模型开发了RBS计算器 (RBS calculator),可以理性设计所需强度的核糖体结合位点序列[11]。有趣的是,作者发现RBS序列的强度并不是一定的,而会受其所表达基因序列的影响,同样的RBS表达不同的基因序列,其表达强度的差异可达10倍之多,这为理性设计带来了额外的困难,因此有必要通过组合的方法对生物元件进行微调。RBS计算器另一个重要功能是设计RBS的简并序列,生成一个表达强度在所需范围内变化的序列文库。因为RBS序列很短,人工设计的RBS文库可以很方便地通过引物设计和PCR扩增来实现与特定基因的连接,从而对基因表达强度进行微调。
Egbert等开发了一种利用RBS序列中“简单重复序列”(Simple sequence repeats)(SSR)来调节基因表达强度的方法[12]。这种方法有3个优点:一是重复序列的长度与RBS序列的强度具有相关性,这使得该法具有表达强度的可预测性;二是SSR对基因表达强度的调节范围非常广,它们可使基因表达强度最多相差1 000倍;三是这种重复序列不稳定,可以通过PCR法或者组合组装法来对其进行快速突变。为了证明这个方法的实用性,作者用SSR微调了一个双稳态开关系统中两个抑制蛋白的表达强度。值得一提的是,作者发现双稳态开关系统的功能具有菌株特异性,同样的开关,在实验菌株中表现良好,而放到另外一株工业菌中可能会丧失其功能。
1.4 优化可调控基因间序列 (TIGRs)
基因间序列的改变也会对基因表达强度产生重大影响。Pfleger等通过可调控基因间序列(Tunable intergenic regions,TIGRs),实现了在一个操纵子内,对多个基因表达强度的同时调节[13]。TIGRs由多种控制元件组成,它们包括:mRNA二级结构、核糖核酸酶 (RNase)断裂位点及RBS隔离序列,通过改变这些序列可以在转录后水平对基因的表达强度进行调节。作者利用TIGR法对三基因的甲羟戊酸生物合成途径进行微调,将甲羟戊酸的产量提高了7倍。同时也发现当甲羟戊酸产量提高了7倍时,其中两种基因——hmgs和tHMGR的活性是降低的。由此可见这些组合优化策略的确能产生超出预期的有利突变体。
2 代谢通路的系统优化
在合成生物系统的优化过程中,只进行单个元件的调节往往难以实现系统功能的最优化。生物系统的各个部分之间是紧密联系并且互相协调的,单一元件的过度扰动经常会影响其他元件的功能调节,从而导致代谢失衡。大量代谢控制分析方面的研究表明:许多代谢通路的主要通量控制步骤往往分布在多个反应中,代谢通路的优化需要同时对这些反应进行微调以保持代谢平衡。否则,代谢失衡将导致细胞内某些中间代谢物浓度过度波动,从而对细胞的生长或性能造成损害。同时,过度表达的基因也会给细胞带来沉重的代谢负担。另外,不同元件对系统功能的调节作用有时会有协同效应,对单个元件的扰动往往只能对系统功能产生微小的影响,而对多个元件的同时修饰能更好地优化系统的功能。由此可见,合成系统中各个部分的组合优化对获得最优化功能是极为重要的。
2.1 多元模块工程
多元模块工程 (Multiple module engineering)是一种能进行代谢通路组合优化的有效策略[14-15]。其工程思路是将整个代谢通路分成不同的模块,再通过系统地改变各个模块的复制起始位点,启动子或者RBS序列来协调不同模块的表达强度(图1A)。利用多元模块工程策略,只需组合构建少数的模块组合,就可以在大范围内优化代谢通路。
多元模块工程已成功运用于生物产品产量的工程优化,其最成功的例子是Ajikumar等对紫杉二烯代谢通路的优化[14]。作者将整个通路分成两个部分:一为包含内源的MEP通路合成异戊烯焦磷酸的上游模块;二为包含外源的萜类化合物合成途径的下游模块。之后用不同强度的启动子和不同拷贝数的质粒与两个模块组合,或将不同强度的启动子与某个模块组合后整合到基因组上再进行表达。作者共构建了32种不同组合的菌株,并从中成功分离了高产紫杉二烯的菌株,其最高产量能达到1 g/L,是对照菌株的15 000倍。更为有趣的是,作者发现最高产量菌株的上游模块是整合到基因组上的,表达强度很弱,而这恰好平衡了两个模块的代谢通量,降低了胞内吲哚的积累,从而解除了吲哚对细胞生长的抑制。
利用相似工程思路,Xu等将脂肪酸代谢通路分成3个模块:上游的乙酰辅酶A合成模块、中游的乙酰辅酶A激活模块和下游的脂肪酸合成模块。作者通过改变这3个模块的拷贝数,使乙酰辅酶A的合成和丙二酰辅酶A的消耗达到平衡,实现了对多个模块表达强度的组合优化。为了进一步提高产量,作者又将上游模块和下游模块基因的RBS位点进行组合微调,进一步将产量提高了46%[16]。
2.2 基于基因合成技术的组合优化工具
多元模块工程也有其内在的缺点,它通常使用传统的酶切连接技术,构建非常有限的模块组合 (不超过100),而难以进行高通量的系统优化。近几年基因合成技术取得了突飞猛进的发展,许多基于重叠延伸技术和同源重组技术的多片段组装技术如雨后春笋般涌现出来[17-23]。相比传统的酶切连接克隆技术,这些多片段组装技术通常具以下3个优点:一是它们对克隆序列的依赖性较低,不需要酶切位点的辅助;二是能够实现多个片段的同时组装;三是其中某些技术具有高效克隆多片段的能力,能用来生成比较大的文库,因此具有对代谢通路中多个元件进行组合优化的能力[22]。
Shao等基于其实验室早前开发的“DNA组装器 (DNA assembler)[19]”技术,又开发了一种名为“组合转录工程优化法 (COMPACTER)”的技术,此技术通过将不同强度的启动子与不同代谢通路的基因进行组合,之后再运用高通量的筛选方法来构建筛选出最高效的代谢通路,由此实现对代谢通路中多个基因表达强度的组合优化(图1B),该技术已成为将组装技术成功应用于代谢通路组合优化的经典案例[24]。利用此法,Du等优化了酵母细胞中木糖和纤维二糖的利用途径,仅通过一轮的COMPACTER就获得了当时文献报道的最高效的利用途径。这些优化的木糖和纤维二糖利用途径具有菌株特异性,即从实验室菌株和工业菌株出发,各自优化得到的最优代谢通路是不同的,而且同一个代谢通路在实验室菌株和工业菌株中会有截然不同的表现。该技术可以做到量体裁衣,为不同背景的菌株“定制”其特有的最优代谢通路。
Gibson组装技术[23]也可以对基因线路进行组合组装。JCVI实验室的Ramon等利用Gibson组装将3个启动子 (recA,ssb,lacI)和4个物种中的乙酸代谢通路 (ackA,pta)进行了随机的组合组装,一个反应就能得到104的文库。作者用菌落PCR法检测了37个克隆后发现,其中81%含有正确长度的序列,然而遗憾的是作者并没有对文库的质量和多样性做进一步的研究[25]。
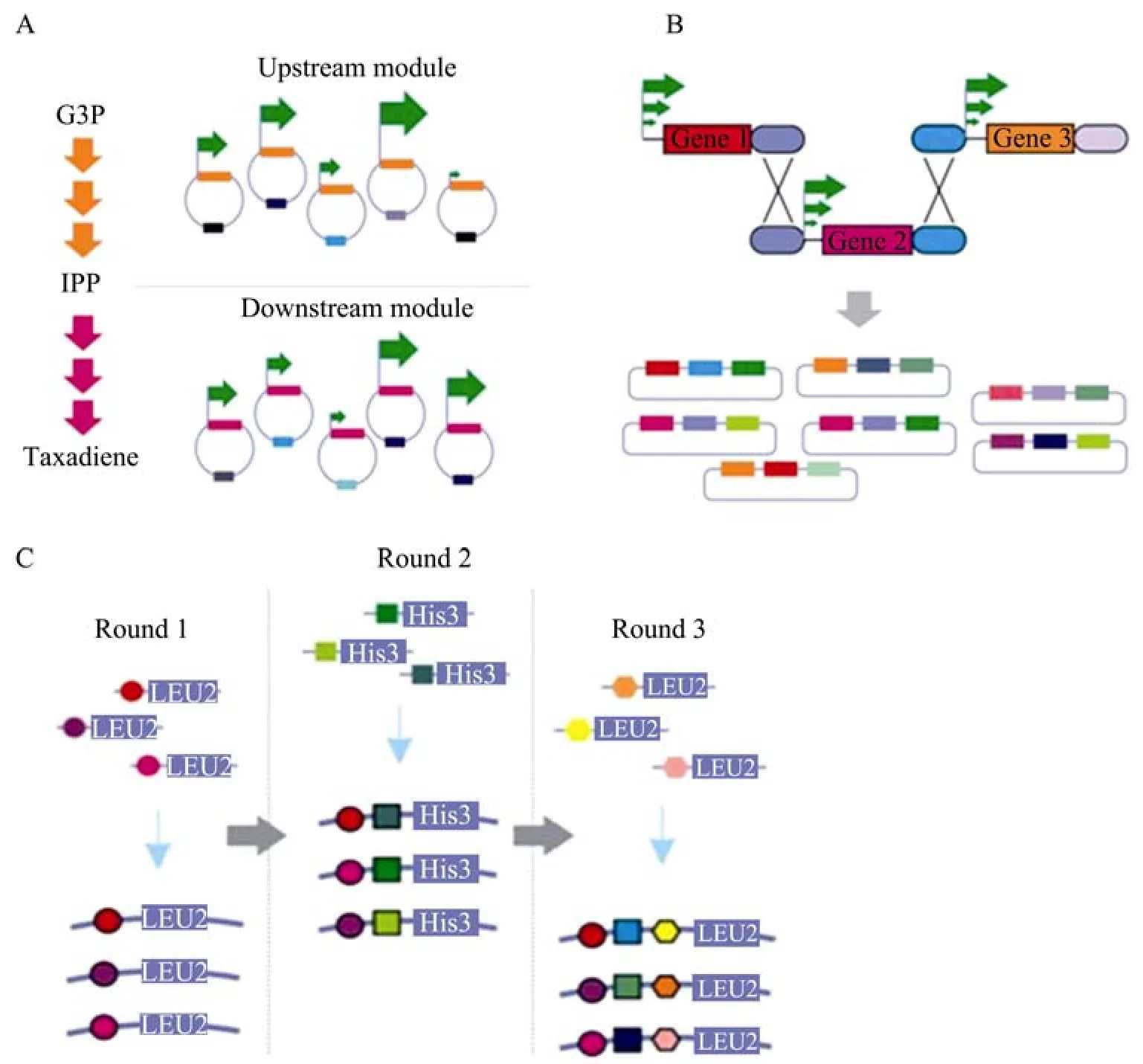
图1 系统优化代谢通路的组合工程工具 (A:多元模块工程;B:组合转录工程优化法;C:迭代重组法)Fig.1 Combinatorial engineering tools for systematically optimization of metabolic pathways.(A)Multiple module engineering.(B)COMPACTER technology.(C)Reiterative recombination.
Quan等开发了一种名为环形聚合酶延伸法(Circular polymerase extension cloning,CPEC) 的技术,此技术能将末端重叠的多个片段和载体一步连接成完整的环状质粒,然后得到的质粒可直接转化入细胞以进行筛选克隆[26]。作者将含1.7 kb的HIV病毒包膜基因gp120分成等长的两个片段,每个片段都含有密码子变体。通过CPEC方法将两个片段和质粒骨架进行组装得到反应产物,转化1 pmol反应产物可以获得2.436×105的克隆,从而证实了其组合组装的强大能力。
另一个特别的构建多基因代谢通路文库的方法是Cornish实验室开发的“迭代重组法(Reiterative recombination)”[27],迭代重组法可以用于构建大于104的代谢通路文库。此方法利用酵母的同源重组系统及几种循环使用筛选标记,可以迭代地在酵母基因组中构建多基因代谢通路 (图1C)。作者用这个技术构建了crtE、crtB、crtI的三基因的番茄红素代谢通路。
2.3 多元质粒工程
本实验室结合之前提到的RBS计算器设计出一系列ssDNA,基于ssDNA重组技术构建了多元质粒工程 (MIPE)。MIPE技术可以同时高效地修改质粒上多个位点。作者利用MIPE技术对核黄素代谢通路的5个基因的表达强度进行了组合优化,仅在1周内就获得了核黄素产量提高2.6倍的克隆。
3 基因组范围内靶点的识别和组合修饰
全局调控因子和一些看似不相关基因的改变都会对生物系统的功能产生影响,从而造就系统的复杂性。因此,为了获得最优的生物学功能,通常需要在全基因组范围内寻找基因靶点,再对其进行组合优化。
3.1 基因组范围内靶点的识别
转座子随机突变是在全基因组范围内搜索目标靶点的有效方法,尽管早前插入序列更倾向于插入某些热点区域,但如今系统经过优化后,插入序列已能高效均匀地插入到整个基因组中。一些能进行基因组基因随机强表达的技术可以与转座子随机敲除技术互补,Jin等将大肠杆菌基因组切割并随机连入到质粒中进行表达,以此来筛选番茄红素高产菌株,最后作者成功发现了对番茄红素的产量有不同程度提高的16个不同位点[28]。
3.2 全转录工程 (gTME)
全转录工程 (Global transcription machinery engineering,gTME)是一种重构转录组的新方法[29]。它通过对菌株的全局转录因子进行突变,以在全局范围内调节基因的表达强度 (图2A)。Alper等通过在大肠杆菌σ因子中引入突变,筛选到一系列最优表型菌株,如具有乙醇、十二烷基钠耐受性且能高产番茄红素的菌株。利用类似方法,对酿酒酵母的TATA盒结合蛋白进行突变,分离了一株具有高度葡萄糖/乙醇耐受性的菌株,其乙醇的体积生产力提高了70%[30]。
3.3 可追踪多元重组工程 (TRMR)与多元自动化基因组工程 (MAGE)
合成生物学的飞速发展催生了一些更加强大的技术,这些技术可以高通量且快速地搜索或修改基因组。Warner等开发了可追踪多元重组工程 (Trackable multiplex recombineering,TRMR),能同时对基因组上千个位点进行分析和修饰[31](图2B)。作者利用TRMR技术成功获得了不同抑制剂存在下大肠杆菌的生长图谱。经过分析发现,一些基因的敲除和强表达都可以使大肠杆菌对不同抑制剂产生耐受性。利用此技术,作者还得到了使细胞耐受纤维素水解液的基因修饰,这一研究成果对工业生物技术领域有着重要的意义。TRMR技术最大的优势是可以在非常短的时间内,只需消耗较低成本就可以获得上千个基因敲除和强表达的细菌,之后辅助微阵列技术就能对这些文库进行简单快速的追踪,使基因功能研究的通量提高了几个数量级。然而在单轮操作中,利用TRMR技术通常只能对单个细胞产生单个基因修饰,因此现今TRMR技术难以用于研究那些需要两个或多个位点同时修饰才能获得的特定细胞表型。
多元自动化基因组工程 (Multiplex automated genome engineering,MAGE)作为另一种强大的高通量修改基因组的工具,能同时瞄准并改造单个细胞基因组上的多个靶点,从而在一个细胞群体中形成组合的基因组多样性[32](图2C)。这种多元化方法是一种基于进化的理性设计法,与传统进化方法相比,此法能使进化更加理性快速地进行。作者又利用MAGE技术循环性和可升级性 (Scalable)的特点,专门为此技术建造了一个自动化机器,进一步实现了多元基因组工程的自动化,此机器能用来快速连续地产生不同基因突变的集合。为了检验MAGE技术的实用性,作者试图对番茄红素生产途径上的24个基因进行同时优化。一天内,借助自动化机器可以产生数十亿个不同基因型的突变株,经过3 d的进化,就能从中挑选到番茄红素生产能力提高5倍的菌株。之后该作者还利用MAGE技术,成功地将大肠杆菌中314个TAG终止密码子定点修改成TAA,由此获得一株不含有TAG终止密码子的菌株[33]。
TRMR技术和MAGE技术一经出现就在相关领域掀起轩然大波,研究者很快发现这两个技术具有互补性,若能将其有效结合将会产生一个更强大的研究策略,即先利用TRMR技术在全基因组范围内寻找基因靶点,再用MAGE技术对发现的基因的表达强度组合微调,Sandoval等很快就进行了这种尝试[34]。他们首先利用TRMR技术去寻找细胞耐受乙酸纤维素裂解液和弱碱环境的基因,发现有27个基因的表达会对菌株的耐受性产生较大影响。之后又通过MAGE技术来修改这27个基因的RBS位点,以实现对其表达强度的组合微调,最终获得了比野生型菌株生长快10%~200%的一系列菌株。
3.4 基于RNA的组合修饰工具
不同于上述基于DNA修饰的技术,Na等又开发了一种利用小型调控RNA对大肠杆菌进行代谢工程操作的技术[35]。它通过表达的sRNA与靶向基因转录翻译区域的结合,来实现对翻译过程的阻遏作用,进而下调相关基因的表达。此技术的优势在于:在不修改基因组的情况下,此技术也能调节基因的表达强度,而且通过将sRNA与DNA结合区域结合,它能实现对基因表达强度的微调。利用这个技术,作者对酪氨酸生物合成途径进行了优化,他们针对14株不同菌株内的4个基因进行了表达强度的组合下调,并最终获得了可以生产2 g/L酪氨酸的菌株。之后,作者又合成了与戊二胺生物合成相关的基因对应的130种sRNA,其中发现murE的弱表达可以使戊二胺产量提高55%。
4 结论与展望
生物系统组合优化已经成为合成生物学和代谢工程必要的组成部分。总体上讲,生物系统组合优化的策略有以下两个发展方向:1)基因修饰和改造的方法会逐渐趋向于多元化,强调多位点的同时修改。2)基因靶点的选择将越来越精确,文库的设计与构建工作也会越来越理性,从而减小对高通量筛选的需求。随着基因合成和修饰技术的快速发展,将会有更高效的工程工具被开发出来。如近期出现的CRISPR-Cas9介导的基因组工程技术,迅速成为研究的热点[36-38]。此技术不仅能进行细菌基因组的高效修饰[36],还通过与RNAi技术相似的方法在不修改基因组的情况下调节基因的强度[39](图2D)。虽然还没有报道暗示这个技术能用于进行组合优化,然而不可否认,此技术具有被开发成一种强大组合优化工程工具的巨大潜力。笔者认为,利用传统的单基因敲除法或强表达法来进行菌株优化的时代已经过去,研究者将会将更多的重点放在组合工程策略上,或选择一种合适高效的组合工程技术,或是将不同技术相结合,以此来优化目标性状,最终为构建生物的新功能提供更多有利条件。
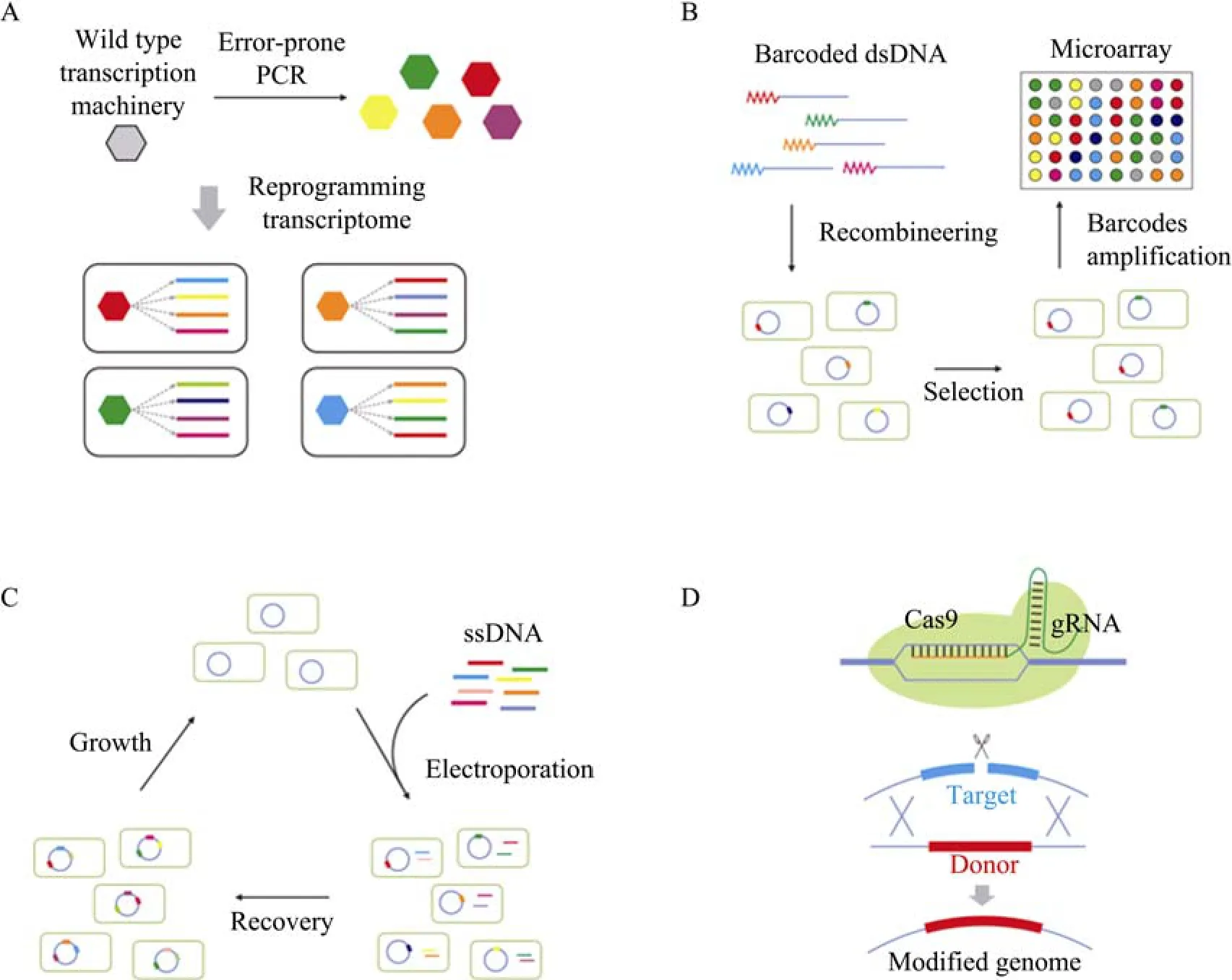
图2 基因组范围内进行组合修饰的组合工程工具及一个具有潜力的基因工程新技术 (A:全转录工程;B:可追踪多元重组工程;C:多元自动化基因组工程;D:CRISPR-Cas9介导的基因组工程技术)Fig.2 Combinatorial engineering tools for introducing combinatorial genome wide modification and a new promising genetic engineering technology.(A)Global transcription machinery engineering(gTME).(B)Trackable multiplex recombineering(TRMR).(C)Multiplex automated genome engineering(MAGE).(D)CRISPR-Cas9 technology.
[1]Endy D.Foundations for engineering biology.Nature,2005,438(7067):449−453.
[2]Khalil AS, Collins JJ. Synthetic biology:applications come of age.Nat Rev Genetics,2010,11(5):367−379.
[3]Prather KL,Martin CH.De novo biosynthetic pathways:rational design of microbial chemical factories.CurrOpin Biotechnol,2008,19(5):468−474.
[4]Stephanopoulos G,Alper H,Moxley J.Exploiting biological complexity for strain improvement through systems biology.Nat Biotechnol,2004,22(10):1261−1267.
[5]Tyo KE,Alper HS,Stephanopoulos GN.Expanding the metabolic engineering toolbox:more options to engineer cells.Trends Biotechnol,2007,25(3):132−137.
[6]Santos CN,Stephanopoulos G.Combinatorial engineering of microbes for optimizing cellular phenotype.Curr Opin Chem Biol,2008,12(2):168−176.
[7]Voigt CA.Genetic parts to program bacteria.Curr Opin Biotechnol,2006,17(5):548−557.
[8]JonesKL,Kim SW,KeaslingJD.Low-copy plasmids can perform as well as or better than high-copy plasmids for metabolic engineering of bacteria.Metab Eng,2000,2(4):328−338.
[9]Alper H,Fischer C,Nevoigt E,et al.Tuning genetic control through promoter engineering.Proc Natl Acad Sci USA,2005,102(36):12678−12683.
[10]Ellis T,Wang X,CollinsJJ.Diversity-based,model-guided construction of synthetic gene networks with predicted functions.Nat Biotechnol,2009,27(5):465−471.
[11]Salis HM,Mirsky EA,Voigt CA.Automated design of synthetic ribosome binding sites to control protein expression.Nat Biotechnol,2009,27(10):946−950.
[12]Egbert RG,Klavins E.Fine-tuning gene networks using simple sequence repeats.Proc Natl Acad Sci USA,2012,109(42):16817−16822.
[13]Pfleger BF,Pitera DJ,Smolke CD, et al.Combinatorial engineering of intergenic regions in operons tunes expression of multiple genes.Nat Biotechnol,2006,24(8):1027−1032.
[14]Ajikumar PK,Xiao WH,Tyo KE,et al.Isoprenoid pathway optimization for Taxol precursor overproduction inEscherichia coli.Science,2010,330(6000):70−74.
[15]Yadav VG,De Mey M,Lim CG,et al.The future of metabolic engineering and synthetic biology:towards a systematic practice.Metab Eng,2012,14(3):233−241.
[16]Xu P,Gu Q,Wang W,et al.Modular optimization of multi-gene pathways for fatty acids production inE.coli.Nat Commun,2013,4:1409.
[17]Geu-Flores F,Nour-Eldin HH,Nielsen MT,et al.USER fusion:a rapid and efficient method for simultaneous fusion and cloning of multiple PCR products.Nucleic Acids Res,2007,35(7):e55.
[18]SleightSC,Bartley BA,LieviantJA,etal.In-Fusion BioBrick assembly and re-engineering.Nucleic Acids Res,2010,38(8):2624−2636.
[19]Shao Z,Zhao H.DNA assembler,anin vivogenetic method forrapid construction ofbiochemical pathways.Nucleic Acids Res,2009,37(2):e16.
[20]Quan J,Tian J.Circular polymerase extension cloning for high-throughput cloning of complex and combinatorial DNA libraries.Nat Protoc,2011,6(2):242−251.
[21]LiMZ,Elledge SJ.Harnessing homologous recombinationin vitroto generate recombinant DNA via SLIC.NatMethods,2007,4(3):251−256.
[22]Ma S,Tang N,Tian J.DNA synthesis,assembly and applications in synthetic biology.Curr Opin Chem Biol,2012.
[23]Gibson DG,Young L,Chuang RY,et al.Enzymatic assembly of DNA molecules up to several hundred kilobases.Nat Methods,2009,6(5):343−345.
[24]Du J,Yuan Y,Si T,et al.Customized optimization of metabolic pathways by combinatorial transcriptional engineering.Nucleic Acids Res,2012,40(18):e142.
[25]Ramon A,Smith HO.Single-step linker-based combinatorialassembly of promoterand gene cassettes for pathway engineering.Biotechnol Lett,2011,33(3):549−555.
[26]Quan J,Tian J.Circular polymerase extension cloning of complex gene libraries and pathways.PLoS ONE,2009,4(7):e6441.
[27]Wingler LM, Cornish VW. Reiterative Recombination for thein vivoassembly of libraries of multigene pathways.Proc Natl Acad Sci USA,2011,108(37):15135−15140.
[28]Jin YS,Stephanopoulos G.Multi-dimensional gene target search for improving lycopene biosynthesis inEscherichiacoli.Metab Eng,2007,9(4):337−347.
[29]Alper H,Stephanopoulos G.Global transcription machinery engineering:A new approach for improving cellular phenotype.Metab Eng,2007,9(3):258−267.
[30]Alper H,Moxley J,Nevoigt E,et al.Engineering yeast transcription machinery for improved ethanol tolerance and production. Science, 2006,314(5805):1565−1568.
[31]Warner JR,Reeder PJ,Karimpour-Fard A,et al.Rapid profiling of a microbial genome using mixtures of barcoded oligonucleotides. Nat Biotechnol,2010,28(8):856−862.
[32]Wang HH,Isaacs FJ,Carr PA,et al.Programming cells by multiplex genome engineering and accelerated evolution.Nature,2009,460(7257):894−898.
[33]Isaacs FJ,Carr PA,Wang HH,et al.Precise manipulation of chromosomes in vivo enables genome-wide codon replacement.Science,2011,333(6040):348−353.
[34]Sandoval NR,Kim JY,Glebes TY,et al.Strategy for directing combinatorial genome engineering inEscherichia coli.Proc Natl Acad Sci USA,2012,109(26):10540−10545.
[35]NaD,Yoo SM,Chung H,etal.Metabolic engineering ofEscherichia coliusing synthetic small regulatory RNAs.Nat Biotechnol,2013,31(2):170−174.
[36]Jiang W,Bikard D,Cox D,et al.RNA-guided editing of bacterial genomes using CRISPR-Cas systems.Nat Biotechnol,2013,31(3):233−239.
[37]Cong L,Ran FA,Cox D,et al.Multiplex genome engineering using CRISPR/Cas systems.Science,2013,339(6121):819−823.
[38]Charpentier E, Doudna JA. Biotechnology:Rewriting a genome.Nature,2013,495(7439):50−51.
[39]Qi LS,Larson MH,Gilbert LA,et al.Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression.Cell,2013,152(5):1173−1183.

