应用合成生物学策略优化光合蓝细菌底盘
巫琴,陈磊,王江新,张卫文
天津大学化工学院 合成微生物学实验室,天津 300072
蓝细菌,又名蓝藻或蓝绿藻,是一类能够进行放氧光合作用的原核生物。它们能够利用太阳能和CO2作为唯一的能源和碳源进行生长,进而经基因工程修饰后生产各种生物燃料以及精细化学品,因而作为“微生物细胞工厂”而在近年来倍受关注[1]。作为一个新兴的基因工程宿主菌,蓝细菌具有以下优势:1)蓝细菌拥有复杂的光合系统,能够吸收广泛波长的太阳光并将其能量直接传递给其他形式的能量载体[2]。同时,蓝细菌光能利用率是陆生植物的数倍 (蓝细菌3%~9%,陆生植物≤0.25%~3%)[3-4]。2)蓝细菌种类多样、分布广泛,培养不需要耕地,甚至可以在极端环境下生长。例如目前实现大规模生产的极大螺旋藻Spirulina maxima就可以在高盐(1.2 mol/L碳酸钠)、碱性 (pH 8.5~11.0)环境下生长[5]。因此,通过蓝细菌合成生物产品能够解决生物质开发中“与粮争地”的问题。3)蓝细菌的生长营养需求低,只需空气 (CO2和N2来源)、水 (电子和还原剂来源)、简单的无机盐和光 (能源)[6],不需要粮食作物为原料,降低了生产成本。同时,利用大气中的温室气体CO2作为碳源,有希望扭转或减缓全球变暖进程。4)蓝细菌生长比植物快,培养周期短,生长密度高。同时,具有相对简单的遗传背景,便于基因操作[7]。伴随着对蓝细菌代谢途径理解的深入以及几十种蓝细菌基因组测序的完成,使得人工改造蓝细菌并将之利用于各种工业产品的规模化生产成为可能。
合成生物学旨在以工程学思想作为指导,从头设计并构建新的生物元件、装置和系统,或对现有的、天然的生物系统进行重新设计和改造[8]。在合成生物学中,宿主细胞被扩展为“底盘”这一全新概念,正如在汽车工业中,同一底盘能够用于多种型号汽车的构建。近年来,合成生物学的研究聚焦于各种模式微生物的底盘,如大肠杆菌和酵母菌等,极大地加深了对这些微生物内分子系统运作机制的理解,也极大地提高了使用这些微生物系统生产可再生能源、合成精细化学品、制造新材料、合成药物、改善环境等的能力和效率。例如,加州大学伯克利Jay Keasling研究组于2003年在大肠杆菌中成功构建了青蒿素合成途径,使其生产成本显著降低,并已投入工业生产[9];加州大学洛杉矶分校James Liao研究组在2009年通过改造酵母细胞使其氨基酸代谢中的碳源转引而合成一系列丁醇的衍生物,走出了合成生物能源的重要一步[10]。相比之下,虽然有超过70种蓝细菌完成了基因组测序,合成生物学在蓝细菌方面的研究和应用仍远远落后于大肠杆菌和酵母菌。鉴于前面提及的蓝细菌作为工程宿主菌的诸多优势,我们相信应用合成生物学的技术和研究策略来优化蓝细菌底盘具有显著的科学研究和工业应用价值。
近年来,研究者在利用光合蓝细菌合成各种生物燃料和精细化学品领域做出很多努力,实现了乙醇[11]、乙烯[12]、正丁醇[13]、异丁醛[14]、丙酮[15]、异戊二烯[16]、游离脂肪酸[17]、脂肪醇[18]、氢气[19]等在光合蓝细菌中的生产。例如,Atsumi等在海生细长聚球藻Synechococcus elongatusPCC 7942中异源表达了缬氨酸合成酶,最终提高异丁醇的前体——异丁醛的产量至1 100 mg/L[14]。虽然这些初步研究证明蓝细菌有作为良好工程底盘的潜能,但目前利用蓝细菌底盘生产工业产品的产量仍远低于其他工业微生物底盘,例如酵母菌利用葡萄糖糖浆合成乙醇的最大产量已达到20%(V/V)[20],而在蓝细菌系统中的乙醇产量仍处于毫克水平,说明对蓝细菌自身的光合效率以及抵抗外界环境能力的提高将会是进一步提高其合成能力的关键。更值得注意的是,很多工业产物对蓝细菌具有毒害性,蓝细菌底盘的低耐受性成为提高产量的一大限制因素。如前所述,Atsumi等对细长聚球藻S.elongatusPCC 7942培养6 d获得1 100 mg/L的异丁醛,同时他们也对产物毒性进行了测试,发现不影响该菌株生长的最大异丁醛浓度为750 mg/L[14]。目前,我们对蓝细菌作为“自养型人工细胞工厂”如何忍耐和适应各种生物产品的分子机制所知甚少,为了进一步开发蓝细菌底盘的工业应用潜力,有必要对光合蓝细菌底盘进行系统优化。以下我们针对底盘优化的方式、蓝细菌光合效率的优化、耐受性蓝细菌底盘的优化、蓝细菌合成产物的外排等方面进行系统地综述。
1 底盘优化的方式
优质底盘应具备以下特质:生长速度快,能量转化效率高;培养条件简单,大规模发酵成本低;遗传背景清楚,技术操作简单;对底物、中间代谢物和目标产品具有较高的耐受性;良好的环境适应能力[21]。为了改善底盘微生物的细胞活力,研究者们提出了一系列优化策略:
1.1 提高底盘的生长速率以及代谢速率
异源蛋白的表达会与宿主竞争细胞资源,这导致菌株生长速率的降低,而通过动态感应回路来调节蛋白表达,能够改善细胞代谢平衡和细胞资源的利用,进而提高生长速率。Liao等在大肠杆菌Escherichia coli中设计了一个调节回路,通过改造Ntr调节子使其能够根据细胞内的葡萄糖水平来控制茄红素合成关键酶的表达,提高了茄红素的产量,同时改善了生长延滞以及代谢失衡[22]。
1.2 改善底盘对底物、中间代谢物、目标产品的耐受性
策略之一是利用系统生物学的方法改造全局转录因子。Alper等利用全局转录工程 (Global Transcription Machinery Engineering,gTME)方法改造酵母菌Saccharomyces cerevisiae中的转录因子Spt15p(一种TATA结合蛋白)和一些其他相关因子,改善了细胞对乙醇的耐受性并提高了乙醇的产量[23]。另外,空间重排和优化途径酶也可以减少有毒中间代谢物的积累。Dueber等通过在E.coli细胞中合成支架从而形成分割区,对乙酰辅酶A硫解酶 (AtoB)、羟甲基戊二酸单酰辅酶A合成酶 (HMGS)、羟甲基戊二酸单酰辅酶A还原酶 (HMGR)进行空间重排,优化了甲羟戊酸的产量[24]。
1.3 改造外排泵以减少胞内的有毒物质
将细胞内合成的工业产品迅速转移到细胞外可以降低对细胞的毒性。Tsukagoshi等通过异源表达AcrAB-TolC外排泵,改善了E.coli对4种烷烃 (己烷、庚烷、辛烷和壬烷)的耐受性[25]。
1.4 提高底盘的环境适应能力
已有多种技术被应用到改善底盘对环境的适应中,包括单基因改造、gTME、进化工程、基因组重排等,针对pH、温度、渗透压、光照、营养等诸多环境因素进行了改造。例如,Shi等通过基因组重排技术改造酵母菌S.cerevisiaeSM-3,使其在45℃时具有最大细胞活力,大大提高了菌株对高温的耐受力[26]。
2 蓝细菌光合效率的优化
光合作用是所有光合微生物生产工业产品的基础,低效率的光合作用和能量转化会使得光合微生物缺乏经济竞争力。尽管蓝细菌的光合效率是植物的2~3倍,但仍处于一个较低的水平 (生长期约为5%~7%,生物反应器中约为3%)[27],90%以上的传递到光合系统的光子能量以热量和荧光形式被耗散[28],这导致基于蓝细菌来生产工业产品目前在经济上仍不具备竞争力,因此,提高光合效率对于进一步应用和发展“自养型人工细胞工厂”有着重要的意义。提高蓝细菌的光合效率可以从光能的捕获以及光能的转化两方面入手:
2.1 光能的捕获
在光合生物的光吸收和初始化学转化阶段,整体光能转化水平仅仅约为50%。提高光能捕获的可能策略包括:
2.1.1 拓宽吸收光谱
对于蓝细菌而言,太阳光谱只有可见光部分(400~700 nm波段)能够被利用。此外,波长短于叶绿素激发光 (680~700 nm)的能量在色素间转化的过程中被缩减,而多余的能量作为热量耗散[29]。同时,光系统Ⅰ和光系统Ⅱ竞争相同波段的太阳光,使得光能转化效率降低了几乎一半[30]。Blankenship等提出将两个光系统之一改造成利用菌绿素吸收太阳光,这种色素主要存在于进行不产氧光合作用的生物中,拥有最大可达~1 100 nm的光吸收。改造后两个光系统将具有互补的吸收光谱 (400~730 nm以及730~1100 nm),将光能捕获效率提高将近一倍[27]。Chen等发现一种叶绿素f能捕获不可见光,这也为拓宽光合微生物的吸收光谱提供了一种新的思路[31]。Gressel等报道了一种在藻类或蓝细菌中表达荧光蛋白的技术,这种荧光蛋白不仅能够吸收有害的紫外线,还能够放射出可被光合系统吸收的光线[32]。
2.1.2 最小化光捕获天线复合体
在自然选择的压力下,微生物趋向于提高繁殖能力而非获得最大生物量或者生物产品产量。在同样的选择压力下,蓝细菌逐渐形成大尺寸的光捕获天线复合体 (Light-harvesting antenna complex,LHC)以适应低光照的环境。然而,在高光照的光生物反应器中,表层细胞过多捕获光子会阻碍下层细胞光吸收,同时大多数吸收的光能以热量和荧光形式耗散来保护细胞不受光损害,这显然不利于生物质和工业产品的生产[33]。近年来,通过最小化光捕获天线复合体来解决这一问题的研究多有报道。一种方式是截短LHC,例如将绿藻模式种株衣藻Chlamydomonas reinhardtii的光捕获天线复合体截短后,得到更高的细胞密度以及氧的释放量[33-34]。此外,利用RNA干扰技术下调LHC的表达,或者通过异源表达LHC翻译抑制剂也能达到同样的效果。Mussgnug等通过RNA干扰下调LHC的表达获得的突变株,在强光处理100 min后,显示出3倍于母株的光合效率[35]。Beckmann等通过异源表达一种LHC翻译抑制剂NAB1的活性持久的变体,使得莱茵衣藻Chlamydomonas reinhardtii的光合效率提高了50%,同时生长速率也提高了50%[36]。
2.2 光能的转化
蓝细菌从各种色素和光捕获天线复合体吸收来的光能,将通过卡尔文循环转化为化学能,同时,外界环境中的碳以CO2的形态进入并以糖的形态离开卡尔文循环,这个过程中能量转化与碳的固定有着紧密联系。因此,提高光能的转化效率与提高碳固定效率密不可分。提高光能转化的可能策略包括:
2.2.1 改善碳固定机制
RuBisCO(1,5-二磷酸核酮糖羧化酶/加氧酶,催化1,5-二磷酸核酮糖与CO2反应,同时也能以O2作为底物)是卡尔文循环中的一个重要的酶[37]。一直以来,人们都在努力提高RuBisCO的催化效率和底物特异性,但研究显示具有更高RuBisCO催化效率的突变株对CO2的选择性则降低[37-38]。到目前为止,在改善RuBisCO功能方面仍没有突破性的进展。羧酶体是存在于一些自养细菌细胞内的多角形或六角形内含体,由以蛋白质为主的单层膜包围,内含RuBisCO和碳酸酐酶 (CA,催化HCO3−向CO2转化)[39]。在进行光合成的过程中,蓝细菌主动将无机碳以HCO3−的形式泵入细胞质中,而具有半渗透膜的功能羧酶体蛋白外壳允许HCO3−的输入并阻挡O2的进入以及CO2的离开,因而碳酸酐酶催化产生的CO2大量聚集在RuBisCO附近而被固定,进而改善由1,5-二磷酸核酮糖与CO2的低反应速率以及RuBisCO的低底物选择性引起的碳固定效率低下问题。在这种机制的基础上,研究者们对蓝细菌碳固定进行了相应的研究与改造。Savage等通过对S.elongatusPCC 7942细胞骨架进行改造从而打乱羧酶体的空间排布,发现分裂出的细胞中羧酶体越多则碳固定速率越大[40]。Atsumi等通过将S.elongatusPCC 6301中编码RuBisCO的rbcLS基因整合入S.elongatusPCC 7942的基因组中,提高了碳固定效率进而提高了异丁醛产量[14]。Price等过表达碳酸氢盐的运载蛋白,同样达到了提高光合效率的效果[41]。另外,引入高活性的碳酸酐酶也不失为一种好方法。
2.2.2 优化卡尔文循环
在提高RuBisCO的催化效率和底物选择性困难的情况下,优化卡尔文循环中的其他酶类或引入其他的固碳方式便成为一个好的选择。Zhu等利用“进化算法”(Evolutionary algorithm)模拟C3光合作用,结果发现碳代谢相关酶之间的有效分配 (Partitioning)能够大大提高光合效率[42]。该模型推测有可能存在这种效应的关键酶之一是景天庚酮糖二磷酸酶 (SBPase),这种酶是二磷酸核酮糖 (RuBP)再生过程中的关键酶,一般处于较低的表达水平。对SBPase的进一步研究证实了这一观点,他们发现过表达SBPase能提高卡尔文循环中间代谢物浓度,从而增加碳的固定[43-44]。另一方面,不依靠RuBisCO固碳而是引入其他固碳方式的假说也被提出。Bar-Even等利用由现有代谢模块重组成的计算机模型,发现一些替代碳固定途径拥有比卡尔文循环更高的动力学速率[45]。例如,将磷酸烯醇式丙酮酸羧化酶与天然的C4碳固定循环核心耦合起来,得到的整体CO2固定速率是卡尔文循环的2~3倍。
3 耐受性蓝细菌底盘的优化
在化学品的生产过程中,蓝细菌会受到产品以及各种环境因素的胁迫,进而影响其细胞活力,导致产量下降、发酵延滞等。为了确保蓝细菌在工业生产中的正常进行,有必要改善蓝细菌的耐受性,包括改善对工业产品的毒性以及严苛外界环境 (pH、温度、渗透压和光照等等)的耐受性。
3.1 产物耐受性的优化
产物毒性是利用微生物生产生物产品中一种常见的问题,特别是对目标产品的产量有较高要求的工业化规模生产。多种生物燃料和精细化学品对蓝细菌都具有毒性,因此,确保蓝细菌生产菌株和目标产品的兼容性十分重要。近年来针对蓝细菌对几种化学品的耐受性已取得了一些研究进展。
3.1.1 乙醇耐受性
乙醇在目前全球可再生能源的供应中占有很高的比例。早在1999年,Deng等就通过在蓝细菌聚球藻Synechococcussp.PCC 7942中异源表达单胞菌Zymomonas mobilis的丙酮酸脱羧酶和乙醇脱氢酶,获得了最大230 mg/L的乙醇产量[11]。最近,Fu等利用集胞藻Synechocystissp.PCC 6803基因组水平的代谢网络模型,获得了最大可达690 mg/L的乙醇产量,同时该研究组还对乙醇毒性进行了测试,发现不影响该菌株生长的最大乙醇浓度为10 g/L[46]。近期,受973项目支持,天津大学合成微生物学实验室在集胞藻对抗乙醇处理的研究中,利用iTRAQ-LC-MS/MS技术,共鉴定到24 877个肽段,对应1 509个蛋白质,覆盖了集胞藻全基因组预测蛋白的42%[47]。使用1.5倍变化以及P<0.05作为阀值,分别鉴定到135个及293个蛋白的表达在24 h及48 h发生了变化。功能分析表明,光合细菌采用了一系列反应包括膜蛋白的修饰、转运蛋白的诱导及移动相关的蛋白等来抵御乙醇毒性。此外,细胞还利用聚羟基脂肪酸积累途径以及乙二醛酶解毒途径来对抗乙醇胁迫。有趣的是,蛋白组分析的结果还表明,多个光合作用相关的蛋白在乙醇处理的样品中表达上调,这与乙醇处理的叶绿素a含量实验结果是一致的。最后,通过对菌株进行基因敲除,验证了可能的目标基因。同时,研究组采用了新一代的测序技术,结合定量反转录聚合酶链反应 (RT-PCR)分析,揭示了蓝细菌的乙醇代谢模型[48]。结果表明,乙醇暴露导致了应激有关蛋白、转运蛋白和细胞表质的修改,光合作用相关基因mRNA水平的上调也进一步证实乙醇耐受在转录水平层面上的调控。最后,通过使用基因敲除方法来验证相关的靶基因在菌株的乙醇耐受性的作用,这一研究给出了一系列改造光合细胞底盘以提高乙醇抗性的潜在靶点。
3.1.2 丁醇耐受性
丁醇既是重要的大宗化工原料,又是继乙醇后的一种极具发展前景的新一代液体燃料。相比于乙醇,丁醇具有更高的能量密度、更低的腐蚀性以及更高的辛烷值。最近,Atsumi等在Synechococcus elongatusPCC 7942中异源表达了缬氨酸合成酶,这种酶能将丙酮酸核心转化成2-酮异戊酸,最终提高异丁醇的前体异丁醛的产量至1 100 mg/L,获得450 mg/L的异丁醇,同时他们也对丁醇毒性进行了测试,发现不影响该菌株生长的最大异丁醛浓度为750 mg/L[14]。目前,针对蓝细菌丁醇耐受机制的研究十分有限,因此,天津大学合成微生物学实验室利用iTRAQLC-MS/MS技术探究了集胞藻对丁醇的响应[49]。共鉴定到25 347个肽段,对应1 452个蛋白质,覆盖了集胞藻全基因组预测的蛋白质的40%。通过蛋白丰度的相对定量,鉴定出303个表达变化的蛋白。功能分析表明,光合细菌采用了一系列反应包括热激蛋白和转运蛋白的感应、膜蛋白的修饰等来抵御丁醇毒性。图1描述了丁醇胁迫下的集胞藻细胞模型,这一模型阐明了推测的对抗丁醇的分子机制。
3.1.3 烷烃耐受性
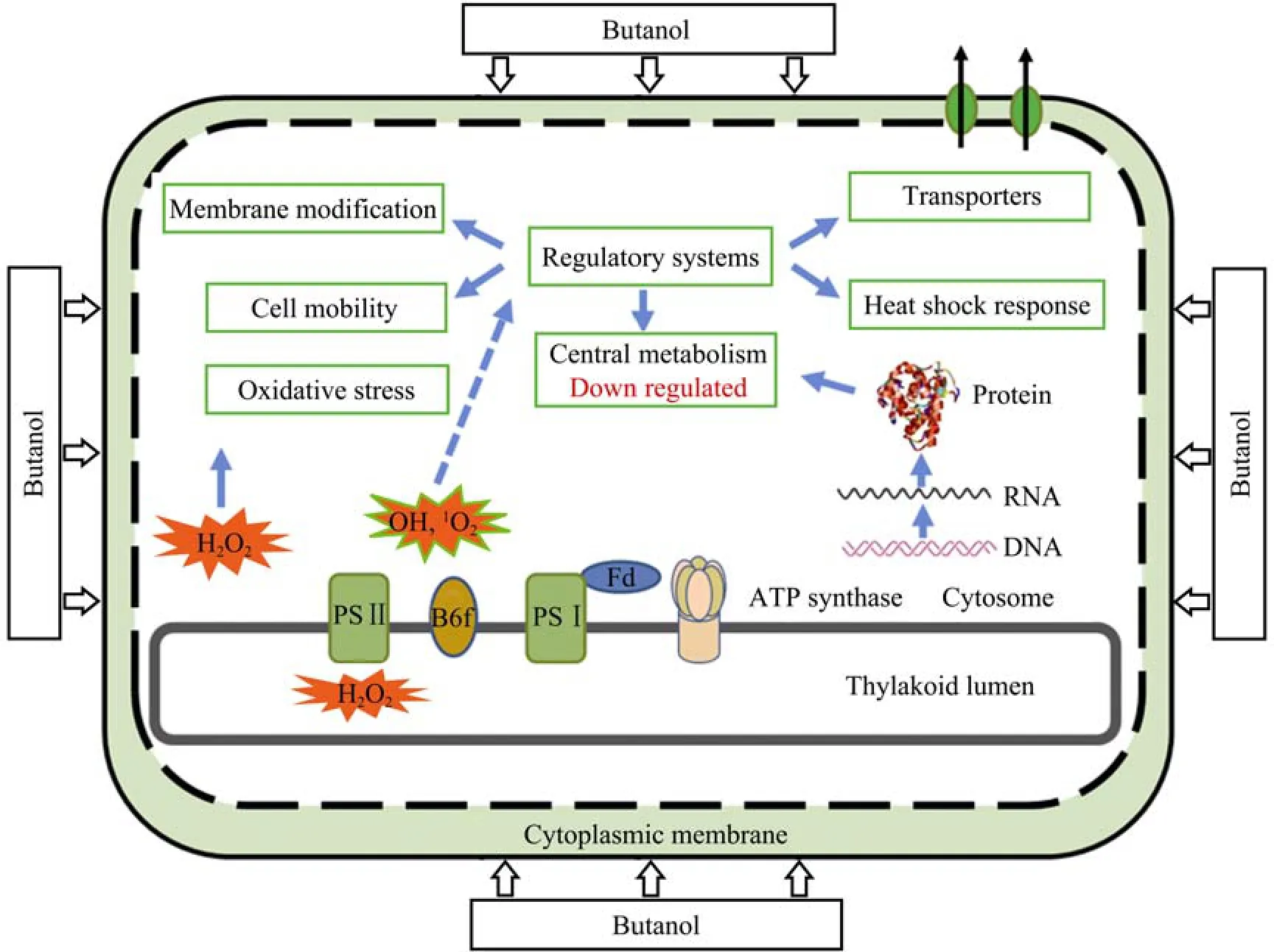
图1 丁醇胁迫下集胞藻Synechocystis PCC 6803的动态响应机制示意图Fig.1 Schematic representation of dynamic responses of Synechocystis PCC 6803 to butanol exposure.
烷烃不仅是燃料的重要来源,而且也是现代化学工业的原料。液态烷烃能被直接应用到内燃机中,作为汽油的替代物具有极大潜力。早在60年代后期,包括光合蓝细菌在内多种微生物就被应用到烷烃的生物合成中,然而天然菌株的烷烃产量一直处于很低的水平。最近,研究者鉴定出蓝细菌中两种与烷烃合成相关的酶——酰基载体蛋白还原酶 (AAR)和乙醛脱羧酶 (AAD),将这两种酶在E.coli中异源表达后细菌合成并分泌了烷烃,培养40 h后C13-C17烷烃混合物产量达到~0.3 g/L[50]。然而,蓝细菌对烷烃的耐受力很低,并且目前针对蓝细菌烷烃耐受机制的研究十分有限。天津大学合成微生物学实验室利用iTRAQ-LC-MS/MS技术在光合细胞对抗己烷处理研究中,共鉴定出1 492个蛋白质,覆盖了集胞藻全基因组预测的蛋白质的42%[51]。其中,己烷处理的样品相对于对照,分别有164个和77个蛋白得到了上调和下调。蛋白功能及代谢途径分析表明,一些常规的逆境反应蛋白的表达都被上调;特别是一些转运蛋白、膜结合蛋白、抗氧化蛋白及一些硫传递系统的蛋白和光合作用相关的蛋白被诱导,表明这些都是细胞针对己烷毒性采用的保护机制。
综合目前的研究进展来看,虽然蓝细菌对产物毒性的耐受力仍保持在一个较低的水平,但蓝细菌对产物毒性采用的复杂分子机制逐渐被揭示,并且报道中也给出了一系列改造光合细胞工厂来提高产物抗性和生产能力的潜在靶点。接下来的工作应将重点放在如何针对这些靶点,利用工程策略改造蓝细菌,例如基因敲除或过量表达特定基因等。
3.2 环境耐受性的优化
环境胁迫 (Environmental stress)是指环境对生物体所处的生存状态产生的压力。蓝细菌的生长以及光合作用受到pH、温度、渗透压、光照、营养等诸多因素的影响,近年来,蓝细菌对环境胁迫的适应机制引起了研究者的广泛光注,原因就在于环境的改变会对菌体的生长和产品的合成造成危害,而具有更高环境胁迫耐受力的底盘能够更好地生长并快速积累生物量,进而获得更高的产物量。
3.2.1 盐胁迫
对自然环境中的光合微生物而言,盐胁迫是最普遍的环境胁迫因素之一[52]。虽然蓝细菌在漫长的进化过程中适应了水生栖息地多变的盐浓度,但高盐浓度会使得水的可用性降低、无机离子浓度增大,加大对细胞的毒性。近来的研究显示,光合蓝细菌对盐胁迫的适应机制主要包括对有毒无机离子的主动外排以及相容性溶质的积累,包括蔗糖、海藻糖、葡萄糖基甘油、甜菜碱等[52-53]。此外,通过动力学分析,蓝细菌对盐的适应过程可以分成了5个连续的阶段:第一阶段细胞收缩[54];第二阶段细胞膨胀且外部的盐 (如Na+和 Cl−)被动流入[55-56];第三阶段把有毒的Na+换成K+,同时激活相容性溶质的合成与光合成[55,57];第四阶段在整个细胞水平上重组基因的表达和活动模式[56,58-59];第五阶段完全适应盐胁迫[52]。为了系统地理解蓝细菌对盐胁迫的响应,近年来已进行了大量的基因组水平的研究,包括对盐短期响应以及长期响应的研究。例如,Kanesaki等利用DNA芯片技术监控集胞藻转录组的响应,在用0.5 mol/L的盐处理30 min后,发现一些和细胞核心代谢相关的基因发生了变化,包括核糖体基因、与PSⅡ反应核心D1蛋白相关的基因、编码热激蛋白和超氧化物歧化酶(SodB)的基因、合成葡萄糖基甘油的基因[60]。Marin等利用DNA芯片对集胞藻盐响应进行了时间序列分析,在用0.684 mol/L的盐处理后,发现整个盐响应过程可被分为两个阶段:盐响应初始阶段和盐适应长期阶段。在盐响应初始阶段,大量的基因表达都会上调,然而仅有少数的基因表现出稳定的响应 (360个中仅有15个),能够在长期响应阶段得以保持,其中包括编码葡萄糖基甘油合成酶的基因[59]。Fulda等利用蛋白组学技术鉴定了适应盐胁迫的集胞藻的细胞周质成分,发现与细胞壁结构有关的酶的表达有极大的提高[61]。Huang等从纯化出的集胞藻质膜中鉴定出25种盐胁迫相关的蛋白,包括调节蛋白GlnB和NrtA、ABC转运蛋白的底物结合蛋白以及一些假设性蛋白质[62]。从以上的研究结果分析,可以从与有毒无机离子的主动外排以及相容性溶质的积累相关的基因入手,改变相关的酶活性、细胞壁结构、类囊体膜蛋白,最终改善蓝细菌的盐耐受能力。
3.2.2 酸胁迫
蓝细菌生长的最佳pH为中性偏碱,当环境pH值偏酸时蓝细菌的生长会受到显著影响。为揭示了蓝细菌应答酸胁迫的代谢机制,Kurian等利用双向电泳、MALDI-MS和LC-MS/MS等技术相结合的方法分析了集胞藻响应酸胁迫的差异表达蛋白质组,鉴定出酸胁迫响应相关蛋白。其中,在pH 7.5、5.5下,对细胞质成分进行分析,鉴定出4种显著变化的蛋白但不包括一些普遍的应激蛋白 (如热激蛋白和伴侣蛋白);在pH 9.0、7.5、6.0、5.5下,对细胞周质成分进行分析,不仅鉴定出草酸脱羧酶、碳酸酐酶两种已知的对pH平衡起作用的酶,还鉴定出一些显著变化的未知功能蛋白,包括14种新型蛋白,为改善蓝细菌细胞耐酸性提供了靶点[63]。Uchiyama等利用DNA芯片技术鉴定了酸胁迫下集胞藻中表达上调的基因,发现其中的一些基因可能是酸胁迫的响应调节子。通过对这些基因进行敲除,发现一种双组分系统中的响应调节子基因slr0081(sphR)的敲除影响酸胁迫下的菌株生长速率,同时通过qRT-PCR技术发现sphR的敲除下调Slr0967和Sll0939(胁迫相关蛋白)的表达,这表明SphR在酸胁迫下6803的生长中具有重要作用[64]。
3.2.3 金属胁迫
相比于其他非光合微生物,光合蓝细菌需要更多的金属元素来辅助光合作用:铁硫簇、细胞色素、非血红素铁中的铁元素;水裂解复合体中的锰元素;质体蓝素中的铜元素;叶绿素中的镁元素。因此,不足或过量的金属元素会导致蓝细菌中的金属胁迫。针对这一问题,已经进行了诸多研究。例如,在铁缺乏响应方面的研究。虽然铁在自然界中大量存在,但是铁的低溶解度导致铁缺乏成为光合自养生长的限制因素。在集胞藻中,铁缺乏加强了isiA、isiB、idiA基因的表达,这3个基因分别编码PSⅠ的一个额外天线系统、黄素氧还蛋白、PSⅡ的一个部分亚基[65-66]。Katoh等鉴定出编码铁离子ABC型转运蛋白的futA1、futA2、futB和futC基因,并发现铁缺乏会诱导fut基因家族的表达[67]。Sin等利用DNA芯片技术来鉴定铁缺乏下转录水平变化的基因[68]。他们对3 165个基因进行了转录组水平的分析,其中,85个基因表现出显著的变化,发现在低铁浓度下编码ATP合成酶和藻胆蛋白的基因下调,同时,叶绿素合成、PSⅠ与PSⅡ的组装、能量代谢相关的基因也表现出明显的变化。总的来看,铁缺乏适应机制主要包括抑制部分光合成基因,调控大量的转运蛋白基因,将离子和其他代谢物运入细胞并诱导形成一种特殊的代谢状态。最近,天津大学研究组选取4种重要的蓝细菌在两种不同金属胁迫条件,包括铁缺乏和高浓度铜处理等条件下的转录组学数据,通过加权基因共表达网络分析 (WGCNA)方法建立了4种蓝细菌的基因调控网络,并进而通过跨种属基因调控网络比较,成功抽提了蓝细菌对环境胁迫的跨种保守性以及物种特异性应对模块及基因,揭示蓝细菌应对环境扰动的生存策略[69]。此外,该项研究首次在微生物中引入跨物种的基因调控网络分析,所发现的蓝细菌环境响应分子机制是从单一物种的数据分析中无法实现的。对于这些新发现的蓝细菌抗逆模块和基因的发掘与认识,将对于进一步构建抗逆境底盘蓝细菌提供了重要的理论指导。
4 蓝细菌合成产物的外排
正如前面提到的,改善菌株的产物毒性耐受水平具有重要价值。传统的基因工程策略 (如突变、进化工程、基因组重排、基因组文库构建等)一般需要较长的时间以及人力物力,以提高乙醇耐受力为例,利用进化工程获得乙醇耐受株需要超过6个月的时间[70]。不同于传统的策略,在细胞上装备外排泵能够将有毒物质驱逐出细胞,直接有效地提高底盘的产物耐受性。
外排泵是与有毒物质外排有关的膜转运蛋白,主要分为两类:ATP结合盒 (ABC)转运蛋白和次级外排泵。其中,ABC转运蛋白是主要的转运蛋白,利用ATP作为能量来源催化底物的转运,包含两个结合化学品并提供转运通道的跨膜结构域以及两个水解ATP的细胞质结构域;次级外排泵利用质子或钠离子梯度作为能量来源,包含3个亚基:一个负责识别化学品和质子交换的细胞质膜蛋白、一个周质连接蛋白、一个外膜通道[70]。利用外排泵来减弱产物毒性以及提高目标产品产量吸引了研究者们的广泛兴趣。Dunlop等通过异源表达来自疏水/两亲外排 (HAE1)家族的外排泵,改善了E.coli对5种代表性生物燃料 (乙酸香叶酯、香叶醇、α-蒎烯、柠檬烯、己酸合欢酯)的耐受性[71]。同时,转入了外排泵的柠檬烯生产菌株表现出1.6倍于母株的柠檬烯产量[72]。Teixeira等过表达了酵母的多抗药性ABC转运蛋白Pdr18p(减弱乙醇引起的质膜透化并降低胞内乙醇浓度),使得乙醇产量提高了17%[73]。
外排泵在提高蓝细菌底盘耐受性方面仍少有报道。针对光合自养蓝细菌缺少输出亲水性初级代谢产物的转运蛋白这一问题,Niederholtmeyer等对Synechococcus elongatesPCC 7942进行了改造,使其表达一种蔗糖酶和一种葡萄糖促扩散蛋白,进而促进葡萄糖和果糖的分泌。类似地,他们还表达了乳酸脱氢酶和乳酸转运蛋白,促进乳酸在胞外的积累。葡萄糖促扩散蛋白和乳酸转运蛋白都属于MFS(Major Facilitator Super-family)家族,MFS转运蛋白包括促扩散转运蛋白和离子耦合转运蛋白,但一般不包括耦合磷酸键水解的转运蛋白。这些结果表明相关转运蛋白对亲水代谢物的分泌是必要的,证明了能够通过工程改造蓝细菌生产和分泌高价值亲水产品[74]。这一策略将来可以应用到提高蓝细菌底盘耐受性方面,通过异源表达产物的转运蛋白,将毒性产物输出到细胞外,从而提高底盘的耐受性以及产品的产量。
5 展望
光合蓝细菌作为合成生物学的底盘具有一系列良好的特质,包括利用太阳能、固定CO2、营养需求低、生长迅速、遗传背景简单等,作为生产可再生燃料和精细化学品的“自养型人工细胞工厂”已倍受关注。在大量蓝细菌基因组测序完成的背景下,近年来,合成生物学在蓝细菌底盘优化方面已经获得了令人振奋的进展,不仅加深了对蓝细菌底盘分子机制的理解,而且为生物燃料和精细化学品的规模化高效生产提供必要的技术支撑。然而,蓝细菌底盘的优化研究仍处于一个初始阶段,在蓝细菌光合效率的优化、耐受性蓝细菌底盘的优化、蓝细菌合成产物的外排等方面仍需要进一步研究。同时,功能基因组学、代谢模型构建等系统生物学手段也有助于蓝细菌代谢网络的改造。可以预见,随着合成生物学的不断发展,更高效经济的光合蓝细菌底盘将成功构建,使得蓝细菌突破实验室研究走向大规模工业化应用成为可能。
[1]Machado IM,Atsumi S.Cyanobacterial biofuel production.J Biotechnol,2012,162(1):50−56.
[2]Wang B,Wang JX,Zhang WW,et al.Application of synthetic biology in cyanobacteria and algae.Front Microbiol,2012,3:344.
[3]Dismukes GC,Carrieri D,Bennette N,et al.Aquatic phototrophs: efficient alternatives to land-based crops for biofuels. Curr Opin Biotechnol,2008,19(3):235−240.
[4]Gupta RS.Molecular signatures for the main phyla of photosynthetic bacteria and their subgroups.Photosynth Res,2010,104(2):357−372.
[5]Ahsan M,Habib B,Parvin M,et al.A review on culture,production and use of spirulina as food for humans and feeds for domestic animals and fish(FAO Fisheries and Aquaculture Circular no.1034).Rome:Food and Agriculture Organization(FAO)of the United Nations,2008.
[6]Hansel A,Lindblad P.Towards optimization of cyanobacteria as biotechnologically relevant producers of molecular hydrogen,a clean and renewable energy source. Appl Microbiol Biotechnol,1998,50(2):153−160.
[7]Koksharova O, Wolk C. Genetic tools for cyanobacteria.Appl Microbiol Biotechnol,2002,58(2):123−137.
[8]Andrianantoandro E,Basu S,Karig DK,et al.Synthetic biology:new engineering rules for an emerging discipline.MolSystBiol,2006,2:2006.0028.
[9]Martin VJ,Pitera DJ,Withers ST,et al.Engineering a mevalonate pathway inEscherichia colifor production of terpenoids.Nat Biotechnol,2003,21:796−802.
[10]Atsumi S,Hanai T,Liao JC.Non-fermentative pathways for synthesis of branched-chain higher alcohols as biofuels.Nature,2008,451:86−89.
[11]Deng MD,Coleman JR.Ethanol synthesis by genetic engineering in cyanobacteria. Appl Microbiol Biotechnol,1999,65:523−528.
[12]Takahama K,Matsuoka M,Nagahama K,et al.Construction and analysis of a recombinant cyanobacterium expressing a chromosomally inserted gene for an ethylene-forming enzyme at thepsbAIlocus.J Biosci Bioeng,2003,95:302−305.
[13]Lan EI,Liao JC.Metabolic engineering of cyanobacteria for 1-butanol production from carbon dioxide.Metab Eng,2011,13:353−363.
[14]Atsumi S, Higashide W, Liao JC. Direct photosynthetic recycling ofcarbon dioxideto isobutyraldehyde. Nat Biotechnol, 2009, 27:1177−1180.
[15]Zhou J,Zhang HF,Zhang YP,et al.Designing and creating a modularized synthetic pathway in cyanobacteriumSynechocystisenables production of acetone from carbon dioxide.Metab Eng,2012,14:394−400.
[16]Lindberg P,Park S,Melis A.Engineering a platform for photosynthetic isoprene production in cyanobacteria,usingSynechocystisas the model organism.Metab Eng,2010,12:70−79.
[17]Liu XY, Fallon S, Sheng J, et al.CO2-limitation-inducible Green Recovery of fatty acids from cyanobacterial biomass.Proc Natl Acad Sci USA,2011,108:6905−6908.
[18]Tan XM,Yao L,Gao QQ,et al.Photosynthesis driven conversion ofcarbon dioxide to fatty alcohols and hydrocarbons in cyanobacteria.Metab Eng,2011,13:169−176.
[19]McNeely K,Xu Y,Bennette N,et al.Redirecting reductantflux into hydrogen productionviametabolic engineering of fermentative carbon metabolism in a cyanobacterium.Appl Microbiol Biotechnol,2010,76:5032−5038.
[20]Antoni D,Zverlov VV,Schwarz WH.Biofuels from microbes.Appl Microbiol Biotechnol,2007,77:23−35.
[21]Attfield PV.Stress tolerance:the key to effective strains of industrial baker's yeast.Nat Biotechnol,1997,15:1351−1357.
[22]Farmer WR, Liao JC. Improving lycopene production inEscherichia coliby engineering metabolic control.NatBiotechnol,2000,18:533−537.
[23]Alper HHS.Developmentof systematic and combinatorial approaches for the metabolic engineering of microorganisms[D].Cambridge:Massachusetts Institute of Technology,2006.
[24]Dueber JE,Wu GC,Malmirchegini GR,et al.Synthetic protein scaffolds provide modular control over metabolic flux.Nat Biotechnol,2009,27:753−759.
[25]Tsukagoshi N,Aono R.Entry into and release of solvents byEscherichiacoliin an Organic-Aqueous Two-Liquid-Phase system and substrate specificity of the AcrAB-TolC Solvent-Extruding pump.J Bacteriol,2000,182:4803−4810.
[26]Shi DJ,Wang CL,Wang KM.Genome shuffling to improve thermotolerance,ethanol tolerance and ethanol productivity ofSaccharomyces cerevisiae.J Ind Microbiol Biot,2009,36:139−147.
[27]Blankenship RE,Tiede DM,Barber J,et al.Comparing photosynthetic and photovoltaic efficiencies and recognizing the potentialfor improvement.Science,2011,332:805−809.
[28]Ort DR,Zhu X,Melis A.Optimizing antenna size to maximize photosynthetic efficiency. Plant Physiol,2011,155:79−85.
[29]DucatDC,Way JC,SilverPA.Engineering cyanobacteria to generate high-value products.Trends Biotechnol,2011,29:95−103.
[30]Hanna M,Nozik A.Solar conversion efficiency of photovoltaic and photoelectrolysis cells with carrier multiplication absorbers.J Applied Physics,2006,100:074510−074510−8.
[31]Chen M,Schliep M,WillowsRD,etal.A red-shifted chlorophyll. Science, 2010, 329:1318−1319.
[32]Gressel J,Eisenstadt D,Schatz D,et al.Use of fluorescent protein in cyanobacteria and algae for improving photosynthesis and preventing cell damage:US,2010/0087006.2010-04-08.
[33]Melis A.Solar energy conversion efficiencies in photosynthesis: minimizing the chlorophyll antennae to maximize efficiency.Plant Sci,2009,177:272−280.
[34]Polle JE,Kanakagiri SD,Melis A.tla1,a DNA insertional transformant of the green algaChlamydomonasreinhardtiiwith a truncated light-harvesting chlorophyll antenna size.Planta,2003,217:49−59.
[35]Mussgnug JH,Thomas-Hall S,Rupprecht J,et al.Engineering photosynthetic light capture:impacts on improved solar energy to biomass conversion.Plant Biotechnol J,2007,5:802−814.
[36]Beckmann J,Lehr F,Finazzi G,et al.Improvement of light to biomass conversion by de-regulation of light-harvesting protein translation inChlamydomonas reinhardtii.J Biotechnol,2009,142:70−77.
[37]Spreitzer RJ,Salvucci ME.Rubisco:structure,regulatory interactions,and possibilities for a better enzyme.Annu Rev Plant Biol,2002,53:449−475.
[38]Zhu XG, Long SP, Ort DR. Improving photosynthetic efficiency for greater yield.Annu Rev Plant Biol,2010,61:235−261.
[39]Espie GS, Kimber MS. Carboxysomes:cyanobacterial RubisCO comes in small packages.Photosynth Res,2011,109:7−20.
[40]Savage DF,Afonso B,Chen AH,et al.Spatially ordered dynamics of the bacterial carbon fixation machinery.Science,2010,327:1258−1261.
[41]Price GD,Badger MR,von Caemmerer S.The prospect of using cyanobacterial bicarbonate transporters to improve leaf photosynthesis in C3 crop plants.Plant Physiol,2011,155:20−26.
[42]Zhu XG,De Sturler E,Long SP.Optimizing the distribution ofresources between enzymes of carbon metabolism can dramatically increase photosynthetic rate:a numerical simulation using an evolutionary algorithm.Plant Physiol,2007,145:513−526.
[43]Miyagawa Y, Tamoi M, Shigeoka S.Overexpression ofa cyanobacterialfructose-1,6-/sedoheptulose-1,7-bisphosphatase in tobacco enhances photosynthesis and growth. Nat Biotechnol,2001,19:965−969.
[44]Tamoi M,Nagaoka M,Miyagawa Y,et al.Contribution of fructose-1,6-bisphosphatase and sedoheptulose-1,7-bisphosphatase to the photosynthetic rate and carbon flow in the Calvin cycle in transgenic plants.Plant Cell Physiol,2006,47:380−390.
[45]Bar-Even A,Noor E,Lewis NE,et al.Design and analysis of synthetic carbon fixation pathways.Proc Natl Acad Sci USA,2010,107:8889−8894.
[46]Fu PC.Genome-scale modeling ofSynechocystissp.PCC 6803 and prediction of pathway insertion.J Chem Technol Biotechnol,2008,84:473−483.
[47]Qiao JJ,Wang JX,Chen L,et al.Quantitative iTRAQ LC-MS/MS proteomics reveals metabolic responsesto biofuelethanolin cyanobacterialSynechocystissp.PCC 6803.J Proteome Res,2012,11:5286−5300.
[48]Wang JX,Chen L,Huang SQ,et al.RNA-seq based identification and mutant validation of gene targets related to ethanolresistancein cyanobacterialSynechocystissp.PCC 6803.Biotechnol Biofuels,2012,5:89.
[49]Tian XX,Chen L,Wang JX,et al.Quantitative proteomics reveals dynamic responses ofSynechocystissp.PCC 6803 to next-generation biofuel butanol.J Proteomics,2012,78:326−345.
[50]Schirmer A,Rude MA,Li X,et al.Microbial biosynthesis of alkanes.Science,2010,329:559−562.
[51]Liu J,Chen L,Wang JX,et al.Proteomic analysis reveals resistance mechanism against biofuel hexane inSynechocystissp.PCC 6803.Biotechnol Biofuels,2012,5:68.
[52]Hagemann M.Molecular biology of cyanobacterial saltacclimation.FEMS MicrobiolRev,2011,35(1):87−123.
[53]Klähn S, Steglich C, Hess WR, et al.Glucosylglycerate:a secondary compatible solute common to marine cyanobacteria from nitrogen-poor environments.Environ Microbiol,2009,12:83−94.
[54]Blumwald E,Mehlhorn RJ,PackerL.Ionic osmoregulation during saltadaptation of the cyanobacteriumSynechococcus6311. Plant Physiol,1983,73:377−380.
[55]Reed RH,WarrSR,Richardson DL,etal.Multiphasic osmotic adjustment in a euryhaline cyanobacterium.FEMS Microbiol Lett,1985,28:225−229.
[56]Fulda S,Mikkat S,Huang F,et al.Proteome analysis of salt stress response in the cyanobacteriumSynechocystissp.strain PCC 6803.Proteomics,2006,6:2733−2745.
[57]Matsuda N,KobayashiH,Katoh H,etal.Na+-dependent K+uptake Ktr system from the cyanobacteriumSynechocystissp.PCC 6803 and its role in the early phases of cell adaptation to hyperosmotic shock.J Biol Chem,2004,279:54952−54962.
[58]Hagemann M,Fulda S,Schubert H.DNA,RNA,and protein synthesis in the cyanobacteriumSynechocystissp.PCC 6803 adapted to different salt concentrations.CurrMicrobiol,1994,28:201−207.
[59]Marin K,Kanesaki Y,Los DA,et al.Gene expression profiling reflects physiological processes in salt acclimation ofSynechocystissp.strain PCC 6803.PlantPhysiol,2004,136:3290−3300.
[60]Kanesaki Y,Suzuki I,Allakhverdiev S,et al.Salt stress and hyperosmotic stress regulate the expression of different sets of genes inSynechocystissp.PCC 6803.Biochem Bioph Res Co,2002,290:339−348.
[61]Fulda S,Huang F,Nilsson F,et al.Proteomics ofSynechocystissp.strain PCC 6803:identification of periplasmic proteins in cells grown at low and high salt concentrations.Eur J Biochem,2000,267:5900−5907.
[62]Huang F,Fulda S,Hagemann M,et al.Proteomic screening of salt-stress-induced changes in plasma membranes ofSynechocystissp.strain PCC 6803.Proteomics,2006,6:910−920.
[63]Kurian D,Phadwal K,Mäenpää P.Proteomic characterization of acid stress response inSynechocystissp.PCC 6803.Proteomics,2006,6:3614−3624.
[64]Uchiyama J,Asakura R,Kimura M,et al.Slr0967 and Sll0939 induced by the SphR response regulatorinSynechocystissp.PCC 6803 are essential for growth under acid stress conditions.Biochim Biophys Acta,2012,1817:1270−1276.
[65]Kunert A,VinnemeierJ,Erdmann N,etal.Repression by Fur is not the main mechanism controlling the iron-inducible isiAB operon in the cyanobacteriumSynechocystissp.PCC 6803.FEMS Microbiol Lett,2006,227:255−262.
[66]Lax J,Arteni A,Boekema E,et al.Structural response of Photosystem 2 to iron deficiency:characterization ofa new photosystem 2-IdiA complex from the cyanobacteriumThermosynechococcus elongatusBP-1.Biochim Biophys Acta,2007,1767:528−534.
[67]Katoh H,Hagino N,Grossman AR,et al.Genes essential to iron transport in the cyanobacteriumSynechocystissp.strain PCC 6803.J Bacteriol,2001,183:2779−2784.
[68]Sen A,Dwivedi K,Rice KA,et al.Growth phase and metal-dependent regulation of the dpsA gene inSynechococcussp. strain PCC 7942. Arch Microbiol,2000,173:352−357.
[69]Wang JX,Wu G,Chen L,et al.Cross-species transcriptional network analysis reveals conservation and variation in responseto metalstressin cyanobacteria.BMC Genomics,2013,14(1):112.
[70]Lo TM,Suong TW,Ling H,et al.Microbial engineering strategies to improve cell viability for biochemical production.Biotechnol Adv,2013.doi:10.1016/j.biotechadv.2013.02.01.
[71]Dunlop MJ,DossaniZY,SzmidtHL,etal.Engineering microbial biofuel tolerance and export using efflux pumps.Mol Sys Biol,2011,7:487.
[72]Dunlop MJ.Engineering microbes for tolerance to next-generation biofuels. Biotechnol Biofuels,2011,4:32.
[73]Teixeira MC,Godinho CP,Cabrito TR,et al.Increased expression of the yeast multidrug resistance ABC transporter Pdr18 leads to increased ethanol tolerance and ethanol production in high gravity alcoholic fermentation.Microb Cell Fact,2012,11:98.
[74]Niederholtmeyer H,Wolfstädter BT,Savage DF,et al.Engineering cyanobacteria to synthesize and export hydrophilic products. Appl Microbiol Biotechnol,2010,76:3462−3466.

