DNA组装新方法的研究进展
李雷,芦银华,姜卫红
中国科学院上海生命科学研究院植物生理生态研究所 合成生物学重点实验室,上海 200032
2000年,以生物振荡器 (Synthetic oscillator)[1]和拨动开关 (Toggle switch)[2]为代表的人造功能器件的创造,宣告合成生物学作为一门新学科的正式诞生[3-4]。十年后,J.Craig Venter实验室完成了蕈状支原体Mycoplasma mycoides“Synthia”的人工构建[5],合成生物学研究再次掀起了新的热潮[6]。事实上,人造生命得以成功产生,主要得益于DNA大片段组装方法的突破。近几年来,利用大肠杆菌、酵母与芽胞杆菌等菌种的高效同源重组能力,各种体内DNA多片段、大尺度的组装方法相继被开发,其代表有DNA装配器[7]、“Inchworm method”[8]等。同时,为解决体外获取DNA大片段的局限性,以非典型酶切连接或序列非依赖的chew-back组装作为新理念,各种灵巧的体外组装方法也应运而生,常见有 BiobrickBglbrick[9-10]、SLIC[11]与 Gibson 等温一步法[12]等。这些体内、体外DNA组装方法的相继诞生,加速了合成生物学功能元件库[13]、生物合成途径[7]乃至酵母染色体[14]的人工构建,促进了代谢工程[15]、基因组编辑[16-17]、诱导性多功能干细胞[18]等领域的迅速发展。
为了更详细地阐述这些最新进展,本文基于DNA组装基本理念,综述了近几年来不同尺度DNA组装新方法,以期为不同类型的合成生物学功能器件以及生物合成途径的构建提供有效的操作工具。
1 同源重组依赖的体内DNA大片段组装
同源重组 (Homologous recombination)是指含有同源序列的DNA分子之间或分子内部的重新组合,其原理已广泛应用于基因克隆、定点突变和基因敲除等分子生物学研究。当操作的DNA分子超过100 kb时,DNA体外组装会十分困难,同源重组的优势将变得更加突出。现在已被开发用于体内大片段DNA组装的菌种有:大肠杆菌、酿酒酵母与枯草芽胞杆菌等。这些菌种DNA重组能力的再开发,为后期进行真核生物染色体与大尺度基因合成环路 (如光合作用系统、生物固氮系统或神经环路等)的构建奠定了坚实的基础[14,19]。
不过,任何方法都不是万能的,体内重组也存在一些问题,比如酵母可能对大肠杆菌复制子进行修饰,导致组装后的载体无法工作;又如当组装片段存在较高同源性时,可能引发不正确的组装。因此,在方法的选择与使用上,还需要仔细考量。下面将重点介绍几种酵母及枯草芽胞杆菌体内的大片段DNA拼接方法。
1.1 酵母介导的转化耦联重组
作为一种模式生物,酿酒酵母是第一个完成测序的真核生物[20]。由于高效的DNA同源重组能力和操作的简便性,由Larionov等提出的酵母转化耦联重组 (Transformation-associated recombination,TAR)理念已发展为多种DNA体内拼接方法[21],实现了启动子库、目标产物合成途径甚至小尺度基因组 (如线粒体、叶绿体基因组)的快速构建[19]。
1.1.1 Gibson体内一步组装法
2008年,Science杂志报道了Gibson及其同事人工构建生殖道支原体Mycoplasma genitalium基因组 (全长580 kb)的突破性进展[22]。他们首先采用自创的体外变温两步组装法 (后改进为Gibson等温一步法[12],详见下文),获得了支原体1/4基因组,但无法实现更大片段的组装。不过,后来采用等温一步法成功组装了生殖道支原体的完整基因组,所以该研究中提及的、大片段DNA(>200 kb)在大肠杆菌中的不稳定性及细菌人工染色体携载能力限制的原因,实际上并不成立。
回过来讲,当时在未改进体外组装方法时,是怎么实现完整基因组的拼接呢?按照上述作者提及的“主要原因”,就不得不更换宿主和载体。已知酵母人工染色体 (YAC)最大能携载2 Mb的DNA分子,以及现存已成熟的转化耦联重组理念,作者转向了酵母的体内拼接。
如图1所示,作者将BAC与YAC的功能元件都整合到pTARBAC3载体上,与另外5个DNA片段一并转入酿酒酵母以实现组装 (片段重叠区60~260 bp不等)。通过后续的脉冲场电泳鉴定与碱基错误纠正 (详见下文“碱基错误控制”一栏),实现了整个生殖道支原体基因组的正确组装。事实上,2010年人造生命“Synthia”的诞生,就是在这样的基础上,组合人工细胞膜,演绎了上帝的角色。
后来,Gibson又在酵母中一次性组装了38个寡核苷酸序列 (片段重叠区 20~200 bp不等)[23],充分展现了酵母组装多片段DNA的潜能,为后续蓝藻Synechococcus elongatus染色体片段的组装 (最大至450 kb)做好了准备[24]。同年,赵惠民研究组也开发出类似的酵母体内组装方法——DNA装配器 (DNA assembler),并成功应用于D-木糖代谢途径 (9 kb)、玉米黄质生物合成途径 (11 kb)以及两者的组合途径 (19 kb)的快速组装[25],使天然产物的组合生物合成研究变得更为简便、高效,加快了廉价原料利用和高价值目标产物的生产。
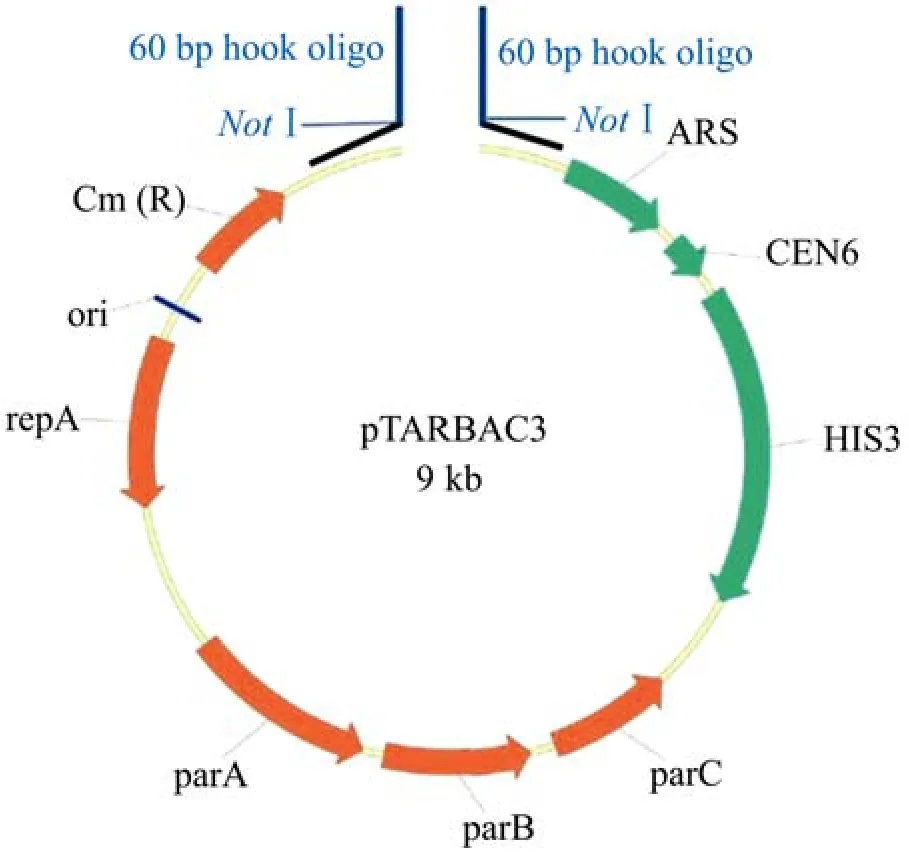
图1 用于酵母体内基因组水平DNA组装的载体(pTARBAC3)Fig.1 The vector used for DNA assembly at the genome-scale in yeast contains both BAC(shown in orange)and YAC(shown in green)functional elements.The “hooks” are added to the pTARBAC3 vector by PCR.
1.1.2 内切酶诱导的迭代拼接
多基因环路的组装,首先想到的方式是逐级(Step-by-step)添加。虽然操作较为繁琐,但方法稳定,尤其当组装片段之间存在较高同源性时,上述提及的多片段体内拼接,有可能导致错误的组装。2011年,Laura等在酵母体内开发了一种体内迭代拼接法[26],较好实现了逐级添加组装多基因环路的理念。
作者通过两套不同内切酶与选择标记 (如HO/LacZ与SceI/HIS3)的交叉使用,可将多个基因逐步添加到酵母染色体上,实现了番茄红素生物合成途径的组装。由于导入的基因都是整合在酵母染色体上,因此原则上来说可以实现“无限”长度的DNA分子组装,前提是酵母染色体能够容纳。
1.2 枯草芽胞杆菌介导的基因组大片段拼接
2005年,Itaya等采用 “inchworm method”(类似于酵母中发展的内切酶诱导的迭代拼接)[8],成功将集胞藻Synechocystis基因组 (3.5 Mb)导入到枯草芽胞杆菌中,构建了1个7.7 Mb的集成基因组。然而,该方法需要100 kb以上的DNA片段作为模板,其应用受到了限制。于是,作者相继开发了一种 “Domino method”[19],借助芽胞杆菌基因组载体 (BGM vector),由小片段 (4~6 kb)出发,利用同源重组原理与逐级添加模式,成功构建了小鼠线粒体基因组 (16.3 kb)和水稻叶绿体基因组 (134.5 kb)。但由于比较耗时,组装过程需长的同源臂,目前还未得到普遍使用。
事实上,当DNA分子超过100 kb后,DNA的提取与转化等常规操作将变得十分困难。2010年,Itaya及其同事们在实验中发现,含有大质粒(大于100 kb)的大肠杆菌被噬菌体侵染后,其中的大质粒仍能够稳定存在。当直接混合大肠杆菌裂解液与芽胞杆菌感受态,可以将DNA大片段稳定整合到芽胞杆菌的基因组上。这种利用天然转化方式,实现DNA大片段直接克隆的模式,被 Itaya 称之为 “culture mix method”[27]。
综上,从几 kb到上百kb组装的 “Domino method”,以及上百kb直接克隆的 “Culture mix method”,最后到几 Mb水平拼接的“Inchwormmethod”,Itaya在芽胞杆菌中建立了一套较为完善的不同尺度DNA组装体系,其应用值得期待。
2 不同尺度的DNA体外组装新方法
DNA化学合成,作为组装DNA分子的起点之一,目前普遍使用固相亚磷酰胺三酯合成法(Phosphoramidite chemistry)[28]。由于合成长度在100 bp之内,该方法一般仅用于引物等寡核苷酸的合成。如果进一步采用连接酶拼接法 (Ligase chain reaction,LCR)或聚合酶拼接法 (Polymerase cycling assembly,PCA),可以实现完整基因的合成[29]。至于更大的DNA片段拼接,上述方法已无法胜任。最近几年,各种尺度的DNA体外组装新方法相继产生,克服了传统酶切连接的局限性,使得无论在组装的DNA片段数目还是尺度上都变得更为高效。
虽然体外DNA组装方法琳琅满目,但都是由DNA拼接的基本理念衍生而来,也即两分子衔接思想 (DNA joining)与多片段组装模式(Assembly organization)的巧妙组合[30],那么这些方法具体是如何产生的呢?其次,随着组装尺度的增加,碱基突变率将逐级扩大,又如何控制碱基突变,获得具有生理功能的DNA产物呢?最后,哪些体外组装方法操作更为简便、通用呢?下面就针对这些问题进行简要阐述。
2.1 DNA组装的基本理念
DNA组装基本理念包括了两分子衔接思想与多片段组装模式。DNA分子的衔接,如同建造房屋时砖块间的堆砌。不言而喻,狭义上的“分子水泥”即是DNA连接酶,而广义上则是指创造两分子间的重叠区,通过连接或聚合的思想(图2),实现分子间拼接。同时,为获取DNA大片段,我们不得不将多个不同小片段加以组装,这就涉及到多片段的组装模式。
在上文体内拼接时,我们已介绍了基于“逐级添加模式”的迭代拼接法与 “Domino method”,也涉及到了几十个片段一次性拼接的Gibson体内“一步组装模式”。实际上,体外组装通常还采用一种等级化 (Hierarchical)组装模式 (图2),它使得多基因环路的构建过程变得更为高效、快捷。通过将多片段组装模式与两分子衔接思想进行组合,便产生了各种各样的DNA体外组装新方法,如基于连接思想与等级化模式的Gibson等温一步法,环式聚合酶延伸克隆(Circular polymerase extension cloning,CPEC)则是聚合思想和一步组装模式的产物[31]。为系统说明DNA组装方法的产生方式,此处一并将基于重组思想的体内拼接方法囊括进来,总结于表1中。
2.2 碱基错误的控制
当构建的DNA尺度很大时,碱基突变频率将逐级增加,此时就不得不特殊对待。其实,在合成任何长度的DNA分子时,碱基突变都必须仔细考虑。虽然更大尺度DNA组装的碱基错误控制与一般基因合成大同小异,都必须经过测序、筛选与修复的过程,但为了获得具有预期功能的组装产物,还需注意以下两点:1)保证出发片段的准确性:采用化学合成或最高保真性DNA聚合酶获得出发片段;2)组装过程中需对片段间连接处进行测序鉴定。
2.3 DNA体外组装的常见方法
2.3.1 序列依赖的非典型酶切连接
随着组装DNA片段大小或数目的增加,基于常规限制性内切酶的分子克隆,将变得举步维艰。于是人们开始寻找一些独特的工具酶,包括同尾酶、识别位点与切割位点不一致的Ⅱ型限制性内切酶等,以延伸序列依赖的DNA拼接模式的应用,建立新的非典型酶切连接方法。
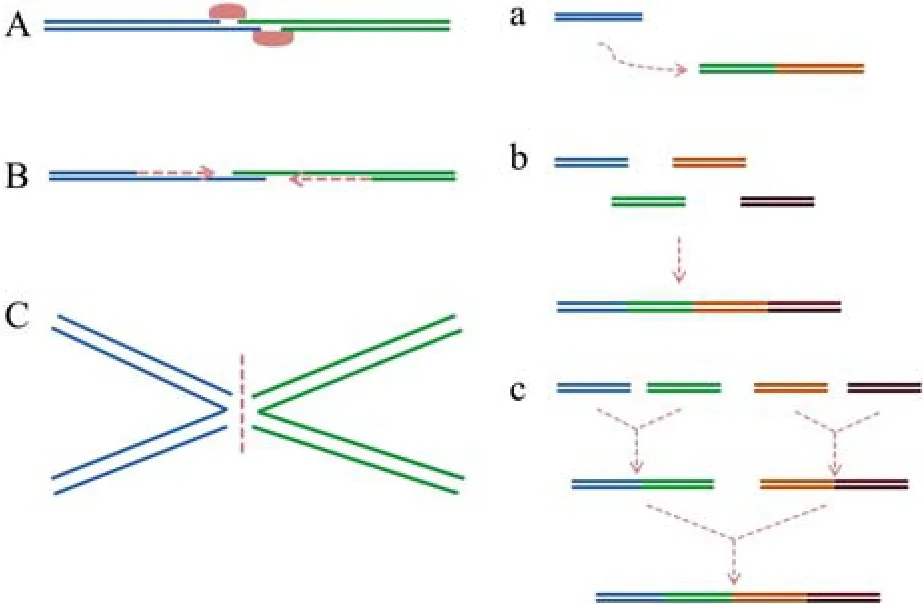
图2 DNA组装的基本理念 (左图为两分子衔接思想,A、B、C分别代表连接、聚合与重组;右图为多片段组装模式,a、b、c分别代表逐级添加、一次性拼接与等级化组装)Fig.2 Fundamental concepts of DNA assembly.In general,two DNA molecules can be joined in one of three ways:ligation (A),polymerization (B)and recombination(C).Small DNA fragments assembled into larger fragments are achieved by three organizational schemes,including assembly in series(a),by hierarchy(b),or by pooling(c).
1)Golden gate cloning:有一种特殊的Ⅱ型限制性内切酶 (ⅡS型酶,如BsaⅠ和AarⅠ),能够在其DNA识别处的相邻位置进行剪切。如果人为设计识别位点不同的相邻序列,就可利用同种限制酶产生不同的粘性末端,从而一次组装多个片段,克服传统多片段组装时限制酶种类的限制,而且还可进行无缝连接。Engler等利用上述原理结合基因改组技术 (Gene shuffling),发展了 Golden gate shuffling 方法[32],一次实现9个小片段与载体的拼接,构建了理论上可达19 683种、不同的重组胰蛋白酶原基因,以筛选高效表达的胰蛋白酶突变体。同时,这种方法还可用于重复序列的组装,如最近兴起的一种强大的基因组编辑工具——转录激活样效应子核酶 (Transcription activator-like effector nucleases,TALENs),由于该蛋白的 DNA结合结构域是由序列差异很小的多个模块组成,当进行人工设计组装时,Golden gate cloning正好满足此要求[16,33]。虽然该方法也依赖于粘性末端,但由于可产生更长的重叠区,也可用于多基因环路的构建。比如,陈威华等借助一种可识别甲基化序列——(m)CNNR(R=A或G)——的ⅡS型内切酶MspJI,发展了一种MASTER(Methylation-assisted tailorable ends rational)DNA组装方法,并利用该方法成功克隆了天蓝色链霉菌放线紫红素生物合成基因簇 (29 kb)[34]。
2)同尾酶依赖的BiobrickBglbrick:为了构建可预测的人工生物系统,合成生物学的主要任务之一就是获得标准化的生物元件 (Biobrick)。当采用如易错PCR、DNA改组或化学随机合成获得的不同元件,包括启动子、核糖体结合位点、开放阅读框和终止子等[3],如何将其快速组建起来获得所需的功能器件,除了上述提及的Golden gate cloning,还有一种依赖于同尾酶的BiobrickBglbrick 方法[9-10]。
同尾酶 (如BglⅡ和BamHⅠ、XbaⅠ和SpeⅠ等)是指一类识别DNA分子中不同核苷酸序列,但能产生相同粘性末端的限制性内切酶。当酶切后的两片段相连时,原来的酶切位点将不复存在,也就不能被原有的限制酶所识别,达到“焊死”状态。因此,该种方法仅利用一对同尾酶,就可以实现DNA片段的多次组装。虽然Biobrick与Bglbrick两种方法在连接处均会产生疤痕,但后者疤痕处翻译后为甘氨酸与丝氨酸,对于融合蛋白表达无影响,所以推荐选用Bglbrick方法。
2.3.2 序列非依赖型的Chew-back组装
在上述提及的非典型酶切连接中,由于限制性内切酶一般只能产生不超过10个碱基的粘性末端 (重叠区),无法满足更大片段 (几十kb以上)的拼接。那么是否可以跳出限制酶的使用,利用DNA外切酶产生更长的突出末端呢?实际上,这就是所谓的Chew-back组装。最早实现该理念的是连接酶非依赖型克隆 (Ligationindependent cloning,LIC),在此基础上又相继发展了SLIC与Gibson等温一步法等。
1)SLIC(Sequence and ligation-independent cloning)及其改进版
LIC主要利用T4 DNA聚合酶独特性能,在一种碱基存在的反应中,能够特定停在某一位置,产生独特的互补末端,从而进行片段间的衔接。但LIC只能组装两个片段 (载体与目标基因),为了一次组装多个DNA分子,Li与Elledge开发了一种序列与连接酶非依赖型克隆[11]。这种方法主要不同点是利用T4 DNA聚合酶外切酶活性非特异的chew-back,产生不同的单链DNA末端。然后采用单链退火或RecA介导的体外重组,从而实现1次反应拼接5个或10个片段。
SLIC方法虽然一次可组装超过10个片段,但需要很长的DNA重叠区,如组装4个片段需要至少40 bp长的重叠区。为了减少重叠区的DNA长度,降低引物合成费用,Schmid-Burgk等利用等级化组装模式,开发了1次只需20 bp重叠区的DNA组装新方法,我们称之为SLIC改进版[18]。在该研究中,作者将非特异的单链末端设计为独特的ID序列 (仅含3种碱基),通过反复使用这些ID与上述介绍的特殊Ⅱ型限制酶,就可以实现多片段中等尺度DNA(小于100 kb)的等级化组装。他们将这种方法利用于产生诱导性多功能干细胞 (iPS)的基因环路构建,虽未进行功能鉴定,但方法灵活简便,应用前景值得期待。后来,作者又巧妙地将SLIC改进版用于上文提及的转录激活样效应子核酶(TALENs)的重构,相比Golden gate cloning方法,组装的数目与效率都有所提高,同时满足了TALENs高通量建库筛选的要求[17]。
综上所述,等级化组装模式很好规避了逐级添加的耗时耗力与一次性组装可能不正确的拼接。事实上,最早采用这种模式的是Gibson组装方法,如上文提到利用此方法成功体外构建了生殖道支原体基因组 (580 kb)[22],下面就重点介绍其中一种具有代表性的Gibson组装方法——Gibson等温一步法。
2)Gibson等温一步法
虽然SLIC或改进版仅使用了一种十分便宜的T4 DNA聚合酶,但只能进行中等尺度的DNA组装,而且连接处将产生疤痕。因此,我们十分推荐Gibson等开发的等温一步法[12],因为该方法不仅可实现无缝拼接,而且组装尺度也十分可观,现成功组装最大的DNA分子大小为1.08 Mb。Gibson等温一步法依旧采用优秀的等级化组装模式,最大的不同是将T4 DNA聚合酶(同时具有外切酶与聚合酶活性)划分为T5外切酶和Phusion聚合酶,同时添加了Taq连接酶。
如图3,T5外切酶具有热不稳定性,在50℃孵育时将逐渐失活。通过反应时间的控制,T5外切酶会切割DNA使其产生一定长度的3′单链末端,在Phusion聚合酶与Taq连接酶介导下进行退火,可1次实现多个片段的拼接。然后采用NotⅠ酶切获得一级组装产物,从而可进行下一级组装。这样操作的优势是避免了单链粘性末端只能含有3种碱基的特殊限制,成功实现了大尺度DNA分子的无痕拼接。
3 总结与展望
随着合成生物学的快速发展,不同尺度的DNA组装方法 (体内或体外)都相继诞生。虽然这些方法均有各自的优点,但如何选择最适合的方法,主要基于三点:一是组装的DNA分子尺度,二是能否容忍DNA片段间连接处疤痕的存在,三是组装序列的同源性程度。等级化组装模式与酵母体内一次性多片段组装的能力,由于自身突出的优势,将是未来更优秀的DNA组装方法开发的基点。同时,作为合成生物学不可或缺的一部分,各种DNA组装方法除在代谢工程中的广泛应用外,将在更多领域发挥作用,如正开展的酵母真核染色体构建、癌症治疗等[38-39]。
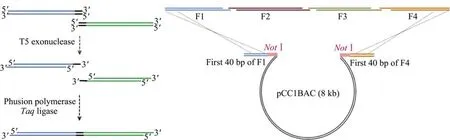
图3 Gibson等温一步法 (左图为两DNA分子衔接方法,右图为多片段组装策略)Fig.3 Gibson one-step isothermal assembly method.Two DNA fragments can be seamlessly joined into a molecule due to the overlapping end in a one-step isothermal process,involving three enzymes,including T5 exonuclease,Phusion polymerase and Taq ligase.Four DNA fragments(F1-F4)are assembled simultaneously using the cloning vector(pCC1BAC)and can be recovered by digestion with NotⅠfor the next level assembly.
[1]Elowitz MB,Leibler S.A synthetic oscillatory network of transcriptional regulators.Nature,2000,403(6767):335−338.
[2]Gardner TS,Cantor CR,Collins JJ.Construction of a genetic toggle switch inEscherichia coli.Nature,2000,403(6767):339−342.
[3]Endy D.Foundations for engineering biology.Nature,2005,438(7067):449−453.
[4]Benner SA,Sismour AM.Synthetic biology.Nat Rev Genet,2005,6(7):533−543.
[5]Gibson DG,Glass JI,Lartigue C,et al.Creation of a bacterialcell controlled by a chemically synthesized genome.Science,2010,329(5987):52−56.
[6]Cheng AA,Lu TK.Synthetic biology:an emerging engineering discipline.Annu Rev Biomed Eng,2012,14(1):155−178.
[7]Shao Z,Zhao H.DNA assembler,anin vivogenetic method forrapid construction ofbiochemical pathways.Nucleic Acids Res,2009,37(2):e16.
[8]Itaya M,Tsuge K,Koizumi M,et al.Combining two genomes in one cell:stable cloning of theSynechocystisPCC6803 genome in theBacillussubtilis168 genome.Proc Natl Acad Sci USA,2005,102(44):15971−15976.
[9]Anderson JC,Dueber JE,Leguia M,etal.BglBricks:a flexible standard for biological part assembly.J Biol Eng,2010,4(1):1−12.
[10]Shetty RP,Endy D,KnightTF.Engineering BioBrick vectors from BioBrick parts.J Biol Eng,2008,2(5):1−12.
[11]LiMZ,Elledge SJ.Harnessing homologous recombinationin vitroto generate recombinant DNA via SLIC.Nat Methods,2007,4(3):251−256.
[12]Gibson DG,Young L,Chuang RY,et al.Enzymatic assembly of DNA molecules up to several hundred kilobases.Nat Methods,2009,6(5):343−345.
[13]Ramon A,Smith HO.Single-step linker-based combinatorial assembly of promoter and gene cassettes for pathway engineering.Biotechnol Lett,2011,33(3):549−555.
[14]Dymond JS,Richardson SM,Coombes CE,et al.Synthetic chromosome arms function in yeast and generate phenotypic diversity by design.Nature,2011,477(7365):471−476.
[15]Keasling JD. Synthetic biology and the development of tools for metabolic engineering.Metab Eng,2012,14(3):189−195.
[16]Cermak T,Doyle EL,Christian M,et al.Efficient design and assembly of custom TALEN and other TAL effector-based constructs for DNA targeting.Nucleic Acids Res,2011,39(12):e82.
[17]Schmid-Burgk JL,Schmidt T,Kaiser V,et al.A ligation-independent cloning technique for high-throughput assembly of transcription activator-like effector genes.Nat Biotechnol,2013,31(1):76−81.
[18]Schmid-Burgk JL,Xie Z,Frank S,et al.Rapid hierarchical assembly of medium-size DNA cassettes.Nucleic Acids Res,2012,40(12):e92.
[19]Itaya M,Fujita K,Kuroki A,et al.Bottom-up genome assembly using theBacillussubtilisgenome vector.Nat Methods,2008,5(1):41−43.
[20]Ostergaard S,Olsson L,Nielsen J.Metabolic engineering ofSaccharomycescerevisiae.Microbiol Mol Biol Rev,2000,64(1):34−50.
[21]Noskov V,Kouprina N,Leem SH,et al.A genetic system for direct selection of gene-positive clones during recombinational cloning in yeast.Nucleic Acids Res,2002,30(2):e8.
[22]Gibson DG,Benders GA,Pfannkoch CA,et al.Complete chemical synthesis, assembly, and cloning ofaMycoplasmagenitaliumgenome.Science,2008,319(5867):1215−1220.
[23]Gibson DG.Synthesis of DNA fragments in yeast by one-step assembly of overlapping oligonucleotides.Nucleic Acids Res,2009,37(20):6984−6990.
[24]Noskov VN,Karas BJ,Young L,et al.Assembly of large,high G+C bacterial DNA fragments in yeast.ACS Synth Biol,2012,1(7):267−273.
[25]Shao Z,Luo Y,Zhao H.Rapid characterization and engineering of natural product biosynthetic pathways via DNA assembler.Mol Biosyst,2011,7(4):1056−1059.
[26]Wingler LM, Cornish VW. Reiterative recombination for thein vivoassembly of libraries of multigene pathways.Proc Natl Acad Sci USA,2011,108(37):15135−15140.
[27]Itaya M, Kaneko S. Integration of stable extracellular DNA released fromEscherichia coliinto theBacillus subtilisgenome vector by culture mix method.Nucleic Acids Res,2010,38(8):2551−2557.
[28]Heinonen P, Lonnberg H. Synthesis of phosphate-branched oligonucleotides.Bioconjug Chem,2004,15(6):1158−1160.
[29]Caruthers MH.Genesynthesismachines–DNA chemistry and its uses.Science,1985,230(4723):281−285.
[30]Carr PA,Church GM.Genome engineering.Nat Biotechnol,2009,27(12):1151−1162.
[31]Quan J,Tian J.Circular polymerase extension cloning of complex gene libraries and pathways.PLoS ONE,2009,4(7):e6441.
[32]Engler C,Marillonnet S.Generation of families of construct variants using golden gate shuffling.Methods Mol Biol,2011,729:167−181.
[33]Weber E,Gruetzner R,Werner S,et al.Assembly of designer TAL effectors by golden gate cloning.PLoS ONE,2011,6(5):e19722.
[34]Chen WH,Qin ZJ,Wang J,et al.The MASTER(methylation-assisted tailorable ends rational)ligation method forseamlessDNA assembly.Nucleic Acids Res,2013,41(8):e93.
[35]Engler C,Gruetzner R,Kandziaet R,et al.Golden gate shuffling:a one-pot DNA shuffling method based on Type IIs restriction enzymes.PLoS ONE,2009,4(5):e5553.
[36]Zhang Y,Werling U,Edelmann W.SLiCE:a novel bacterial cell extract-based DNA cloning method.Nucleic Acids Res,2012,40(8):e55.
[37]Zhang L,Zhao G,Ding X.Tandem assembly of the epothilone biosynthetic gene cluster byin vitrosite-specific recombination.SciRep,2011,1:141−145.
[38]Weber W,Fussenegger M.Emerging biomedical applications of synthetic biology.Nat Rev Genet,2012,13(1):21−35.
[39]Nandagopal N,Elowitz MB.Synthetic biology:integrated gene circuits.Science,2011,333(6047):1244−1248.

