一种改进的普洱茶固态发酵过程微生物DNA提取方法
吕昌勇,陈朝银,徐致远
(1.光明乳业股份有限公司乳业生物技术国家重点实验室,上海200436;2.昆明理工大学生命科学与技术学院,昆明650500)
一种改进的普洱茶固态发酵过程微生物DNA提取方法
吕昌勇1,2,陈朝银2,徐致远1
(1.光明乳业股份有限公司乳业生物技术国家重点实验室,上海200436;2.昆明理工大学生命科学与技术学院,昆明650500)
为开发一种改进的普洱茶固态发酵过程微生物DNA提取方法,首先配制含有吐温-20、聚乙烯吡咯烷酮(PVP)、乙二胺四乙酸二钠(Na2EDTA)等组分的洗涤缓冲液,引入超声波振荡、涡旋、65 ℃水浴、反复洗涤等手段,把普洱茶发酵微生物较高质量的收集起来,再使用十六烷基三甲基溴化铵(CTAB)法提取微生物总DNA。按照本文所述方法所得到的菌体沉淀接近白色,后续提取出的DNA总量高、色度低、杂质少,适用于PCR及基因组学等研究。
普洱茶;固态发酵;提取DNA;发酵食品
渥堆发酵是现代普洱茶发酵的主要手段[1],即利用湿热原理、微生物及其代谢作用加速普洱茶发酵过程[2-3]。通常环境中可培养的微生物不足1%[4-6],利用常规方法对环境中微生物真实群落结构了解的非常有限[7-8],因此,采用现代分子生物学技术对传统发酵工艺中的微生物进行研究是提高普洱茶质量的有效途径。但是,普洱茶茶叶自身生长过程中就代谢有非常复杂的次生代谢产物,加之在渥堆发酵过程中茶叶成分的分解转化更为复杂[9-10],这些都影响着普洱茶渥堆发酵过程微生物总DNA的高质量提取,进而影响着分子生物学在普洱茶研究中的应用。
普洱茶发酵微生物的收集好坏,直接影响着后面微生物DNA提取质量的好坏,如果收集到的菌体本身表面附着太多色素及多酚类等次级代谢产物,或者菌体里面混杂着植物叶片茶渣,在提取DNA裂解细胞的时候,这些次级代谢产物很容易与释放出来的DNA结合交缠在一块,一旦这些物质交缠在一块,就很难再分开,进而严重影响DNA的提取质量。
为此笔者在总结了最近有关普洱茶微生物DNA提取的论文所用的微生物收集方法的基础上,结合在实验过程中发明总结的方法并对这些方法收集后的菌体进行提取DNA效果比较,以期得到一种能较为科学高效的普洱茶发酵微生物收集方法及建立其DNA提取体系。
1 材料与方法
1.1 材料
实验所用的普洱茶样品采自云南省勐海县鹏程茶厂正在渥堆发酵的普洱茶,样品在干冰环境下运输,回到实验室后置于-70 ℃冰箱内保存。
主要试剂: NaCl,北京市化学试剂三厂;十六烷基三甲基溴化铵(CTAB)、乙二胺四乙酸(EDTA)、聚乙烯吡咯烷酮(PVP),Solarbio公司;三羟甲基氨基甲烷(Tris),Novon公司;冰乙酸,天津化学试剂三厂;巯基乙醇,Amresco公司; Taq酶、PCR试剂盒,TaKaRa 公司;Tween-20,Sigma公司。
主要仪器:核酸电泳槽,Wealtec公司; X-22低温离心机,Beckman Coulter公司; HHS-S恒温水浴锅,上海比朗仪器有限公司; 2100 Pro紫外分光光度计,GE公司; SW-CJ-2D型无菌操作台,苏州净化设备有限公司;VGT-1613T超声波清洗机,深圳威固特清洗设备有限公司。
1.2 实验方法
1.2.1 普洱茶表面微生物的收集
1) 无菌水浸泡分离(方法1)
参考Chen等[11]提取分离普洱茶微生物的方法,具体操作如下:① 取3 g茶叶样品加入装有30 mL无菌水中;② 3 000 r/min 1 min去除茶叶残渣,收集悬浮液;③ 15 000 r/min 10 min收集菌体沉淀。
2) 磷酸盐缓冲液浸泡分离(方法2)
参考张阳等[12]分离普洱茶微生物的方法,具体操作如下:① 取3 g茶叶样品加入装有30 mL无菌的磷酸盐缓冲液(pH 7.4)中;② 用超声波振荡(40 kHz,500 W) 7 min;③ 在摇床摇动30 min(200 r/min);④ 后低速(500 r/min)离心7 min去除掉叶片残留样品,9 000 r/min离心8 min,收集菌体沉淀。
3)Tween-NaCl 缓冲液分离(方法3)
参考孙婷婷等[13]分离普洱茶微生物的方法,具体操作如下:① 取3 g茶叶样品加入装有30 mL无菌的Tween-NaCl 缓冲液(10 g/L Tween-20,1.5 mol/L NaCl,pH 7.5~8.0);② 静置30 min;③ 超声波振荡(40 kHz,500 W)10 min;④ 离心(12 000 r/min,10 min)的菌体收集。
4) 笔者改进的方法(方法4)
① 称取2~3 g普洱茶样品,剪碎,放到50 mL离心管中;② 按照10 mL/g茶叶的比例加入配置的洗涤缓冲液(含有1.5 mol/L NaCl、100 mmol/L Tris-HCl、50 mmol/L Na2EDTA、0.6 g/L Tween-20、质量分数2% PVP、pH为8.0的溶液),茶叶浸泡其中,静置15 min;③ 在超声波清洗仪(40 kHz,500 W)环境中振荡10 min,涡旋45 s,上清液转入新的离心管中;④ 65 ℃水浴6 min,低速离心去除残渣,高速离心收集沉淀;⑤ 加入适当的洗涤缓冲液(组分同上),涡旋振荡混匀,65 ℃水浴6 min,高速离心收集(10 000 r/min,10 min)菌体,完成一次洗涤过程;步骤⑤洗涤重复多次,直到离心收集菌体的上清液接近透明为止。笔者改进的方向主要为洗涤缓冲液的配方调整及组合优化洗涤方法。
1.2.2 DNA的粗提
1)向步骤1.2.1中所得到的沉淀用液氮充分研磨,以1.0 mL/g茶叶的用量加入DNA提取液(成分为10~20 g/L CTAB、20~50 mmol/L Na2EDTA、80~100 mmol/L Tris-HCl、1~1.5 mol/L NaCl、10~20 mL/L 巯基乙醇);
2)加入0.1 mg/mL蛋白酶K,55 ℃环境水浴30 min,接着65 ℃水浴1 h;
3)加入等体积氯仿-异戊醇混合液(两者体积比为24∶ 1),混匀后10 000g离心10 min,上层的水相回收于新的离心管中,重复此步骤1次;
4)加入等体积预冷的异丙醇,1/10体积3 mol/L醋酸钠,混匀,置于-20 ℃静置45 min;
5)15 000g离心20 min,所得沉淀用75%(体积分数)乙醇洗涤,自然风干,加入100 μL双蒸水,此为粗提的总DNA。
1.2.3 粗提DNA的纯化
1)粗提的DNA中加入0.5 mg/ mL RNAse,37 ℃水浴1 min以上;
2)加双蒸水补足至500 μL,加等体积的Tris饱和酚,混匀,10 000g离心10 min,上层水相回收于新离心管中;
3)加等体积的氯仿-异戊醇混合液,混匀,10 000g离心10 min,上层水相回收于新离心管中;
4)加2倍体积的无水乙醇,1/10体积3 mol/L醋酸钠,混匀,-20 ℃静止30 min;
5)15 000g离心20 min,所得沉淀自然风干后,加100 μL双蒸水,此为纯化后的总DNA。
1.2.4 DNA提取效率及提取质量的检测
1)提取的DNA通过琼脂糖电泳后在凝胶成像系统下观察,以此判断DNA提取效果及完整性;
2)选择16S rDNA通用引物27F:(5′-A ̄G ̄A ̄G ̄T ̄T ̄T ̄G ̄A ̄T ̄C ̄C ̄T ̄G ̄G ̄C ̄T ̄C ̄A ̄G-3′)和1492R:(5′-G ̄G ̄T ̄T ̄A ̄C ̄C ̄T ̄T ̄G ̄T ̄T ̄A ̄C ̄G ̄A ̄C ̄T ̄T-3′)进行细菌PCR扩增,间隔约1.5 kb; PCR反应体系20 μL,成分为0.5 μL未经纯化的DNA模版,250 μmol/L dNTP(每种),0.1 μmol/L引物,10×Buffer(含MgCl2)2 μL,TaqDNA聚合酶1 U,其余为去离子水,扩增过程先94 ℃ 5 min,94 ℃ 30 s,53 ℃ 35 s,72 ℃ 1.5 min,设定32个循环,最后72 ℃ 7 min。
选择18S rDNA通用引物ITS3:(5′-G ̄C ̄A ̄T ̄C ̄G ̄A ̄T ̄G ̄A ̄C ̄G ̄C ̄A ̄G ̄C-3′)和ITS4:(5′-T ̄C ̄C ̄T ̄C ̄C ̄G ̄C ̄T ̄T ̄A ̄T ̄T ̄G ̄A ̄T ̄A ̄T ̄G ̄C-3′)进行真菌PCR扩增,间隔约300 bp;PCR反应体系同上,扩增过程先94 ℃ 5 min,94 ℃ 30 s,55 ℃ 35 s,72 ℃ 30 s,设定32个循环,最后72 ℃ 7 min。
3)利用紫外分光光度计对纯化后的DNA吸光度进行测量,计算OD260/OD280,并根据吸光度值估算提取DNA的浓度及产率。
2 结果与讨论
2.1 洗涤后所得菌体的颜色及粗提DNA所得沉淀颜色
对已经报道的4种收集菌体的方法进行比较,分析比较洗涤所得到的微生物沉淀的颜色及醇沉DNA后沉淀颜色结果见表1。由表1可知:无菌水、磷酸盐、Tween-NaCl缓冲液所分离出来的菌体均有较深的颜色,这些颜色很有可能是由于得到的沉淀菌体表面附着大量次级代谢产物,从而导致粗提得到的DNA醇沉后有较深的颜色,DNA沉淀的颜色较深从侧面说明含有的杂质较多。从一个侧面说明笔者所用的洗涤缓冲液及收集微生物方法在一定程度上优于其他普洱茶发酵微生物的收集方法。
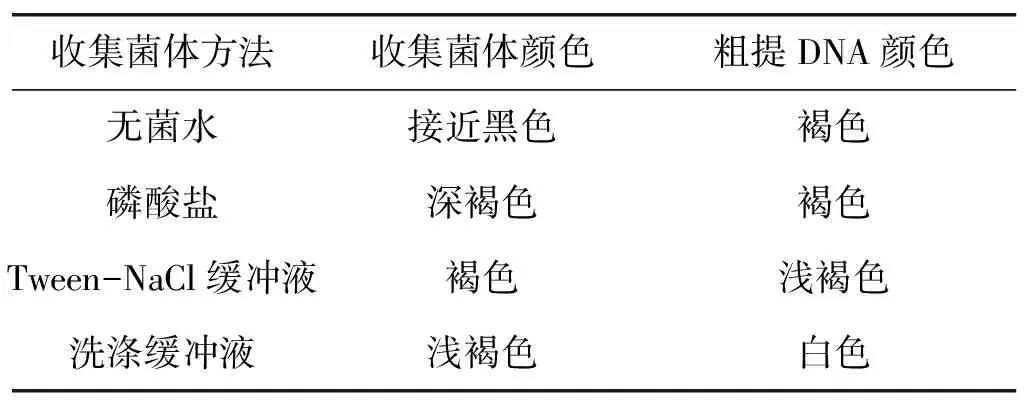
表1 4种方法收集菌体颜色及粗提DNA颜色对比
2.2 粗提DNA电泳图
图1为4种方法分离后菌体粗提微生物总DNA的电泳图。由图1可知:利用方法1和2分离微生物后提取的DNA条带非常不明显,表明提出的DNA含量极少,质量很差,可能原因是洗涤过程中,菌体收集质量太差,含有大量杂质,影响了微生物DNA的提取;方法3所得到的DNA隐约有条带,提取量高于方法1和2,表明所用的收集菌体的缓冲液在一定程度上可以达到保护DNA的效果,但是提取率相对较少;方法4所得的DNA主条带清晰,片段较大且亮度较高,初步确定为最好的DNA分离菌体方法。
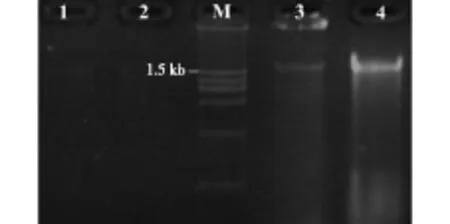
M—标准DNA;1~4— 4种方法收集后提取微生物DNA图1 4种方法粗提DNA效果比较Fig.1 Comparison of four methods to extract DNA
2.3 粗提DNA PCR
图2和图3分别为普洱茶渥堆发酵过程中总微生物DNA 16S rRNA基因、18S rRNA基因PCR凝胶电泳图。由图2和图3可以看出:只有方法4在1.5 kb和300 bp左右处有明显主条带,片段大小符合细菌和真菌的特征条带大小,表明该方法提取出的微生物总DNA既包括细菌的又包括真菌的DNA,且可以直接用于PCR扩增。
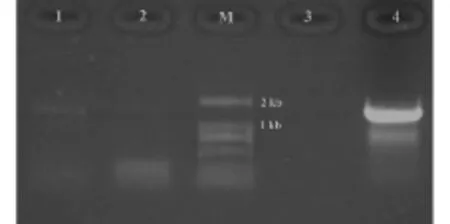
M—标准DNA;1~4—4种方法收集后微生物图2 普洱茶渥堆发酵过程中总微生物 DNA 16S rRNA基因PCRFig.2 16S rRNA gene PCR of extraction of total microbial DNA of Puer tea during pile-fermentation

M—标准DNA;1~4—4种方法收集后微生物图3 普洱茶渥堆发酵过程中总微生物 DNA ITS3-ITS4基因PCRFig.3 ITS3-ITS4 gene PCR of extraction of total microbial DNA of Puer tea during pile-fermentation
2.4 提取效率
对纯化后所提取的DNA,使用紫外分光光度计进行纯度检测并进行提取率的计算。提取DNA所用样品为3 g,溶解DNA为100 μL,稀释12倍。所得数据如表2所示。
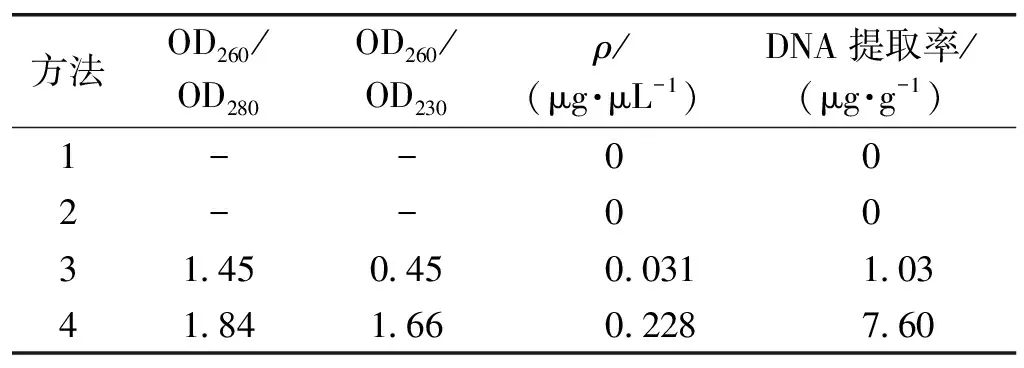
表2 DNA紫外分光光度计检测结果
注:“-”表示比值无比较意义。
由表2可知:方法1和方法2提取率非常小,几乎可以忽略其提取率,方法3和方法4的提取率分别为1.03、7.60 μg/g普洱茶。
结合用OD260和OD280的比值估计核酸的纯度,OD260和OD230比值估计去盐的程度,可以明显发现,方法3所得到的DNA纯度较差,难以满足后续分子生物学的要求。而方法4收集菌体后提取的DNA OD260/OD280=1.84,OD260/OD230=1.66,能比较好地满足后续分子生物学研究的要求,同时具有较高的提取率,可以认为方法4优于方法1、2和3,较好地收集菌体后对高质量地提取DNA具有关键的作用。
值得强调的是,本文所用的提取DNA方法是较为普遍的CTAB法。虽然如参考文献所述方法没有对收集的微生物进行一定的处理,但是依旧取得了一定的实验结果,笔者认为这是由于他们在提取DNA过程中采用了一定的补救措施。而本文中所述的提取DNA的方法所需试剂等均为常规试剂,在菌体分离后,没有用到DNA提取试剂盒、溶菌酶破壁酶等试剂等,操作简单,目的就是直观地说明微生物收集及其预处理对发酵微生物DNA提取的重要性。
对比之前报道,Chen等[11]对发酵普洱茶微生物总DNA进行提取,然后用真菌DGGE分析,最终在整个发酵过程只得到3种真菌的信息;张阳等[12]和孙婷婷等[13]的方法提取出来的DNA必须经过一定的稀释(100~1 000倍)才可以用于扩增出细菌16S rDNA和真菌β-微管蛋白基因,表明所提取出来的DNA含有较多杂质,多酚等代谢产物残存量都没有被完全去除,所以必须将粗提总DNA样品进行稀释后才能作为模板;表明这些文献所述的方法均有一定的局限性,其中的原因很有可能就与没有进行恰当科学的微生物收集和洗涤有关,
本文在实验的基础上配制专门用于洗涤用的缓冲液,其包含的Tween-20是一种乳化剂,有去污功能;PVP能有效去除多糖多酚类物质;Na2EDTA可以螯合普洱茶叶表重金属离子,这些物质的配合使用,可以有效去除微生物表面的次级代谢产物;同时在收集微生物时摒弃了用恒温振荡器等较为温和的方法,因为在微生物分离耗时越长,微生物在分离出茶叶表面的同时次生代谢产物也会分离出来,进而与微生物表面二次黏附,本文首先对茶叶短时间浸泡,使其吸水膨胀,增加表水接触面积,然后快速激烈地分离微生物,尽量减少次生代谢物的浸出;收集菌体后又采取较高温水浴、振荡等方法反复洗涤以使菌体表面残存的色素、多酚类等次生代谢产物等去除。本文所提取的DNA已经应用于普洱茶发酵微生物高通量测序的研究,说明了本文所述的收集微生物及后续的提取DNA可以用于后续分子生物学研究。
3 结 论
按照本文的方法,所得到的菌体沉淀和DNA沉淀颜色均有较大的改观,所得到的菌体沉淀接近白色,后续沉淀出的DNA为白色,此方法可以比较有效地去除次生代谢物质对提取DNA的干扰。因此在后续的提取DNA过程中不需要昂贵的实验仪器、试剂盒及复杂的提取手段,依然能得到较高质量的DNA。所提取出的DNA条带完整,RNA有效去除,提取率为7.60 μg/g以上,OD260/OD280=1.84,OD260/OD230=1.66,经过细菌及真菌PCR均能扩增出符合特征大小的条带。
目前,关于普洱茶的分子生物学研究开展较少,而本研究方法的体系为普洱茶发酵分子生物学的研究奠定了一定的基础,进而从分子水平揭露普洱茶发酵微生物中关键菌群及其代谢过程,为普洱茶产业的科技化提供了理论依据。
[1] 杨瑞娟,吕杰,严亮,等.普洱茶渥堆发酵中嗜热真菌的分离和鉴定[J].茶叶科学,2011,31(4):371-378.
[2] 陈可可,朱宏涛,王东,等.普洱熟茶后发酵加工过程中曲霉菌的分离和鉴定[J].云南植物研究,2006,28(2):123-126.
[3] Yang D J,Hwang L S.Study on the conversion of three natural statins from lactone forms to their corresponding hydroxy acid forms and their determination in Pu-erh tea[J].J Chromatogr A,2006,1119(1):277-284.
[4] Vaz-Moreira I,Egas C,Nunes O C,et al.Culture-dependent and culture-independent diversity surveys target different bacteria:a case study in a freshwater sample[J].Antonie Van Leeuwenhoek,2011,100(2):245-257.
[5] Lewis K,Epstein S,D′Onofrio A,et al.Uncultured microorganisms as a source of secondary metabolites[J].J Antibiot (Tokyo),2010,63(8):468-476.
[6] 吕昌勇,陈朝银,葛锋,等.微生物分子生态学研究方法的新进展[J].中国生物工程杂志,2012,32(8):111-118.
[7] Chen C,Ye Y,Kong X,et al.A primary study on the thermophilic fungi from pile-fermentation of Pu-er tea[C]// IT in Medicine and Education (ITME).2011 International Symposium on IEEE,2011(2):696-699.
[8] Chen C,Zhang W,Lu C,et al.Primary study on thermophiles during pile-fermentation of Pu-er tea[C]//IT in Medicine and Education (ITME).2011 International Symposium on IEEE,2011(2):691-695.
[9] Alcazar A,Ballesteros O,Jurado J M,et al.Differentiation of green,white,black,Oolong,and Pu-erh teas according to their free amino acids content[J].J Agr Food Chem,2007,55(15):5960-5965.
[10] Horanni R,Engelhardt U H.Determination of amino acids in white,green,black,Oolong,Pu-erh teas and tea products[J].J Food Comp Anal,2013,31(1):91-100.
[11] Chen Y S,Liu B L,Chang Y N.Bioactivities and sensory evaluation of Pu-erh teas made from three tea leaves in an improved pile fermentation process[J].J Biosci Bioeng,2010,109(6):557-563.
[12] 张阳,赵树欣,李巍,等.普洱茶发酵过程中微生物总DNA提取方法的比较[J].食品工业科技,2012,33(12):230-232.
[13] 孙婷婷,赵明,李亚莉,等.普洱茶发酵样品细菌和真菌DNA同时提取方法研究[J].中国农学通报,2011,27(15):249-253.
(责任编辑 荀志金)
Modified extraction total microbial DNA in Puer tea solid-state fermention
LYU Changyong1,2,CHEN Chaoyin2,XU Zhiyuan1
(1.State Key Laboratory of Dairy Biotechnology,Bright Dairy & Food Co.Ltd.,Shanghai 200436,China; 2.Faculty of Life Science and Technology,Kunming University of Science and Technology,Kunming 650500,China)
Isolation high purity,large fragment DNA is important in microbial molecular research and while collecting microbial cells properly plays key roles to isolate total DNA.We developed a modified method to extract total microbial DNA in Puer tea fermentation.First,microbes tea was collected by washing samples several times with a buffer containing with Tween-20,polyvinylpyrrolidone(PVP),and ethylenediaminetetraacetic acid disodium (Na2EDTA),in combination with ultrasonic oscillations and vortex at 65 ℃ until microbial cells with nearly white color.Then,microbial total DNA was extracted by using hexadecyltrimethylammouium bromide(CTAB) approach to obtain an integral,light color and pure microbial DNA suitable for PCR and genomics research.
Puer tea;solid-state fermentation;DNA extraction;fermented food
10.3969/j.issn.1672-3678.2014.06.010
2013-08-18
国家科技支撑计划(2013BAD18B01);国家高技术研究发展计划(863计划)(2011AA100901)
吕昌勇(1987—),男,河南固始人,硕士,助理工程师,研究方向:微生物工程与酶工程,E-mail:abcd1000efgh2000@163.com
Q93
A
1672-3678(2014)06-0052-05

