大豆GmUGD基因家族生物信息学分析
朱延明,朱毅,端木慧子,肖佳雷,于洋,刘艾林
(东北农业大学农业生物功能基因重点实验室,哈尔滨150030)
大豆GmUGD基因家族生物信息学分析
朱延明,朱毅,端木慧子,肖佳雷,于洋,刘艾林
(东北农业大学农业生物功能基因重点实验室,哈尔滨150030)
尿苷二磷酸葡萄糖脱氢酶(UGD)是多糖合成中的一个关键酶,广泛参与植物生长发育与非生物胁迫响应过程。采用生物信息学方法,在全基因组水平预测出10个大豆GmUGD基因;序列分析结果显示,GmUGD基因均具有UDP-葡萄糖/GDP-甘露糖脱氢酶家族的3个典型结构域;基因复制分析表明,80%GmUGD基因存在复制事件;系统发生分析结果显示,GmUGD基因分为三类,分类结果与基因复制分析结果基本吻合;基于UniGene数据库与GEO数据库的表达模式分析显示,根中GmUGD基因的ESTs最多,上胚轴中最少;另外,GmUGD基因能够响应盐碱胁迫诱导,在大豆根和叶中表达模式不同。结果可为进一步研究大豆GmUGD基因功能提供理论依据。
GmUGD基因;大豆;生物信息学;序列分析;表达分析
尿苷二磷酸-葡萄糖6-脱氢酶(UDP-glucose 6-dehydrogenase,UGD)是一种以NAD+或NADP+为受体、作用于供体CH-OH基团上的氧化还原酶,参与多种糖代谢过程。UGD能催化产生多糖合成过程中的关键前体物质——UDP-葡萄糖醛酸,因此在多糖合成过程中起重要作用[1]。Samac等研究将大豆UGD基因的cDNA转入到紫花苜蓿中,转基因植株中UGD活性和非转基因植株相比显著增强,且木质素和木糖含量明显增高[2]。在玉米中,UGD基因突变导致酶活性显著下降,抑制下游多糖合成[3]。半纤维素是植物细胞壁主要组成部分,半纤维素中大部分糖来自于UGD催化产生的UDP-葡萄糖醛酸,因此UGD与植物细胞壁形成密切相关[4-5]。研究表明UGD基因在植物非生物胁迫应答中起重要作用。贺顺姬等发现,高盐胁迫培养条件对盐藻DsUGD基因在mRNA水平上的表达具有明显诱导作用[6];Plomion等证明白杨中UGD蛋白响应干旱胁迫[7];Lata等研究指出粟米UGD蛋白在脱水胁迫中上调表达[8]。
大豆是我国主要粮食作物之一,是植物蛋白和植物油主要来源,主产于我国东北地区。大豆产量和品质易受低温、盐碱等非生物胁迫影响。因此,研究UGD基因对研究大豆生长发育和非生物胁迫响应机制具有重要意义。
目前在基因组水平上大豆GmUGD基因家族的生物信息学分析及功能研究尚未见报道。本研究利用已公布的大豆基因组序列信息,采用生物信息学方法,在基因组水平上对GmUGD基因进行预测,通过序列分析、进化分析、组织表达特异性分析及盐碱胁迫下表达模式分析等,以期为进一步研究GmUGD基因功能提供参考及理论依据。
1 材料与方法
1.1GmUGD基因筛选与序列分析
根据GmUGD基因Glyma18g50000蛋白保守结构域的氨基酸序列,利用BLASTP程序在大豆基因组注释数据库Phytozome(http://www.phytozome.net/)中进行序列相似性搜索。利用ClustalX2.0[9]软件对GmUGD同源蛋白进行序列比对,利用蛋白质家族数据库PFAM(http://pfam.xfam.org/)分析蛋白保守结构域,利用MEME(http://meme.nbcr.net/meme/cgibin/meme.cgi)对其进行序列基序(Motif)分析,所用参数为软件默认值。
1.2GmUGD基因复制分析
基因染色体定位信息在大豆基因组数据库(www.phytozome.com)中通过BLASTP程序比对确认,染色体物理定位图使用MapInspect绘制。根据Yang等方法判断旁系同源基因的复制事件[10],判定标准如下:①在不同染色体上,两个基因序列比对部分长度占较长序列的80%以上;②比对部分相似性大于70%。
1.3GmUGD基因系统发生分析
根据序列比对结果,利用MEGA5.0软件[11]中的邻接法(Neighbor-Joining,NJ)构建GmUGD基因家族系统发生树,随机检验参数Bootstrap设为1 000次,其他参数为软件默认值。
1.4GmUGD基因表达特性分析
GmUGD基因组织表达EST数据下载自NCBI上的UniGene(http://www.ncbi.nlm.nih.gov/unigene)数据库;大豆根、叶GmUGD基因盐碱胁迫表达谱下载自NCBI上的基因表达仓库GEO(http://www.ncbi.nlm. nih.gov/geo/)数据库,数据编号分别为GSE17883[12]和GSE20323[13],并利用MATLAB软件分别对其进行层次聚类及热图绘制。
2 结果与分析
2.1GmUGD基因获取与序列分析
2.1.1GmUGD基因获取
通过序列相似性搜索,获得12个GmUGD基因,剔除相同基因的不同转录本,最终获得10个GmUGD基因,分别为Glyma01g06970、Glyma02g 12870、Glyma05g00590、Glyma08g26520、Glyma13g 06050、Glyma14g14161、Glyma17g08491、Glyma18g 50000、Glyma19g03500和Glyma20g03221。
2.1.2GmUGD基因保守结构域分析
下载大豆UGD蛋白序列,利用ClustalX进行比对分析,发现这些蛋白序列均具有3个高度保守的蛋白结构域,分属于PFAM数据库中UDP-葡萄糖/ GDP-甘露糖脱氢酶家族的3个典型区域:N端NAD结合区UDPG-MGDP-dh-N(Pfam:03721)、C端UDP结合区UDPG-MGDP-dh-C(Pfam:03720)及中间部分的UDP-葡萄糖/GDP-甘露糖脱氢酶家族区UDPG-MGDP-dh(Pfam:00984)(见图1)。
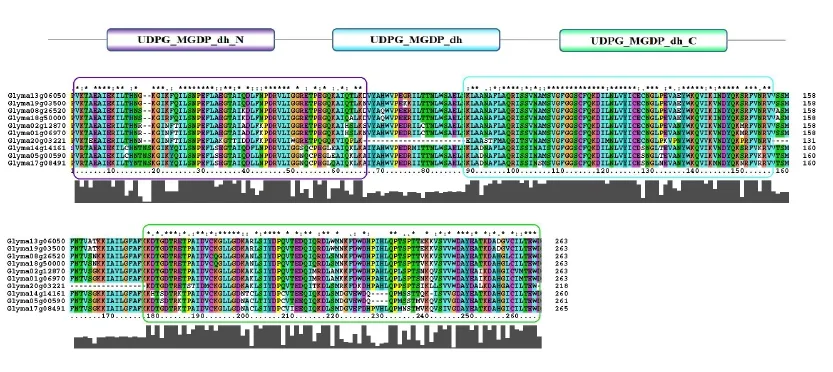
图1GmUGD家族蛋白的保守结构域Fig.1Conserved domain of GmUGD proteins
结果证实这10个基因均属于UDP-葡萄糖脱氢酶家族基因,而3个保守结构域可能是GmUGD基因参与糖代谢过程中主要功能区域。
2.1.3GmUGD基因的motif分析
利用MEME网站进行蛋白motif预测。结果见图2,除Glyma14g14161和Glyma20g03221,其他GmUGD基因具有相同的motif。在预测所得的10个motif中,motif7和motif10功能未知,其他8个motif均为UDP-葡萄糖/GDP-甘露糖脱氢酶家族的3个典型保守结构域的组成部分,进一步证实GmUGD基因的保守性。
2.2GmUGD基因的复制分析及系统发生分析
2.2.1GmUGD基因的复制分析
GmUGD基因序列具有高度保守性,为研究进化关系,对其进行旁系同源基因复制判定。结果显示,除Glyma14g14161和Glyma20g03221,其他8个基因两两之间均满足基因复制的判定标准。因此,推测GmUGD基因进化中存在多次基因复制现象。为直观显示GmUGD基因复制事件,对其进行染色体定位,标注复制事件(见图3)。
由图3可知,基因间实线表示基因间序列相似性大于90%,虚线表示基因间相似性在80%~90%。结果显示,Glyma01g06970和Glyma02g12870,Glyma05g00590和Glyma17g08491,Glyma08g26520、Glyma13g06050、Glyma18g50000和Glyma19g03500序列高度相似,可能存在复制事件,具有较近进化关系。
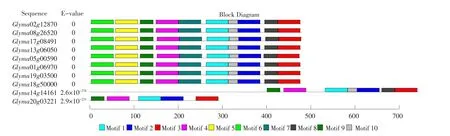
图2GmUGD家族蛋白基序分布Fig.2Distribution of GmUGD proteins motifs
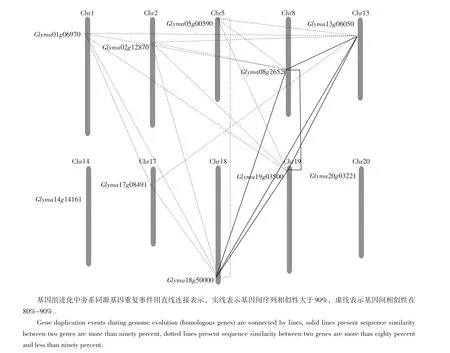
图3GmUGD基因染色体分布及基因复制事件Fig.3Chromosome distribution and gene duplications of GmUGD genes
2.2.2GmUGD基因系统发生分析
为进一步研究GmUGD基因进化关系,采用邻近法(NJ)构建GmUGD基因家族系统发生树。结果表明,所构建系统发生树中基因间随机检验(Bootstrap)得分均>80,即系统发生树中代表进化关系可靠。GmUGD基因家族系统发生树显示,GmUGD基因分三类:Glyma08g26520、Glyma13g06050、Glyma18g50000和Glyma19g03500为一类,且基因间遗传距离较近;Glyma01g06970、Glyma02g12 870和Glyma20g03221为一类,其中Glyma01g06970和Glyma02g12870间遗传距离较近,而Glyma20g03 221与其遗传距离较远;其余3个基因聚为一类,基因间遗传距离相对较远(见图4)。这个结果与基因复制分析结果基本吻合。由于蛋白序列与其功能具有一定相关性。进化关系较近基因如Glyma08g26520和Glyma18g50000、Glyma13g 06050和Glyma19g03500可能具有相同或相似功能。
2.3GmUGD基因表达模式分析
2.3.1GmUGD基因组织表达特异性分析
表达序列标签EST是研究基因表达模式有效方法之一。为研究GmUGD基因在不同组织中表达模式,利用NCBI的UniGene数据库筛选GmUGD基因在不同组织和器官中的ESTs。结果显示,有6个GmUGD基因(Glyma01g06970、Glyma02g12870、Glyma08g26520、Glyma13g06050、Glyma18g50000和Glyma19g03500)在根、茎、叶等不同组织器官中具有ESTs(见表1)。其中,根中ESTs最多,其次是下胚轴与叶;上胚轴中ESTs最少,子叶、豆荚中次之。在具有ESTs的GmUGD基因中,Glyma18g 50000的ESTs最多,其次是Glyma08g26520。
2.3.2GmUGD基因盐碱胁迫表达模式分析
为进一步研究GmUGD基因组织表达特异性及其是否响应盐碱胁迫,本研究从GEO数据库中下载大豆根、叶盐碱胁迫(NaHCO3处理)基因表达谱数据,从中获得GmUGD基因家族表达谱并进行聚类分析。由图5可知,有4个GmUGD基因在盐碱胁迫下表达发生改变,分别为Glyma01g 06970、Glyma08g26520、Glyma18g50000和Glyma 19g03500。盐碱胁迫下,这些基因在根和叶中的表达模式存在差异,如Glyma01g06970在盐碱胁迫的大豆叶中表达下调,而在根中表达上调;Glyma 19g03500在盐碱胁迫的大豆叶中表达始终呈上调趋势,而在根中表达水平先降低后升高;Glyma08g26520和Glyma18g50000在根中显著下调表达,表达量变化显著高于叶中。结果说明GmUGD基因在碱胁迫下的表达模式具有组织特异性。另外,和其他GmUGD基因相比,Glyma08g26520和Glyma18g50000在盐碱胁迫各时间点的表达变化显著,表达模式相似,说明其可能在大豆盐碱胁迫响应过程中发挥重要作用,行使相同或相似功能。与之前系统发生分析和组织表达分析结果相符。
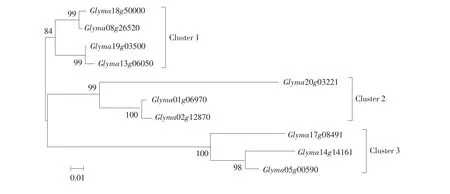
图4GmUGD基因家族系统发生树Fig.4Phylogenetic tree of GmUGD genes
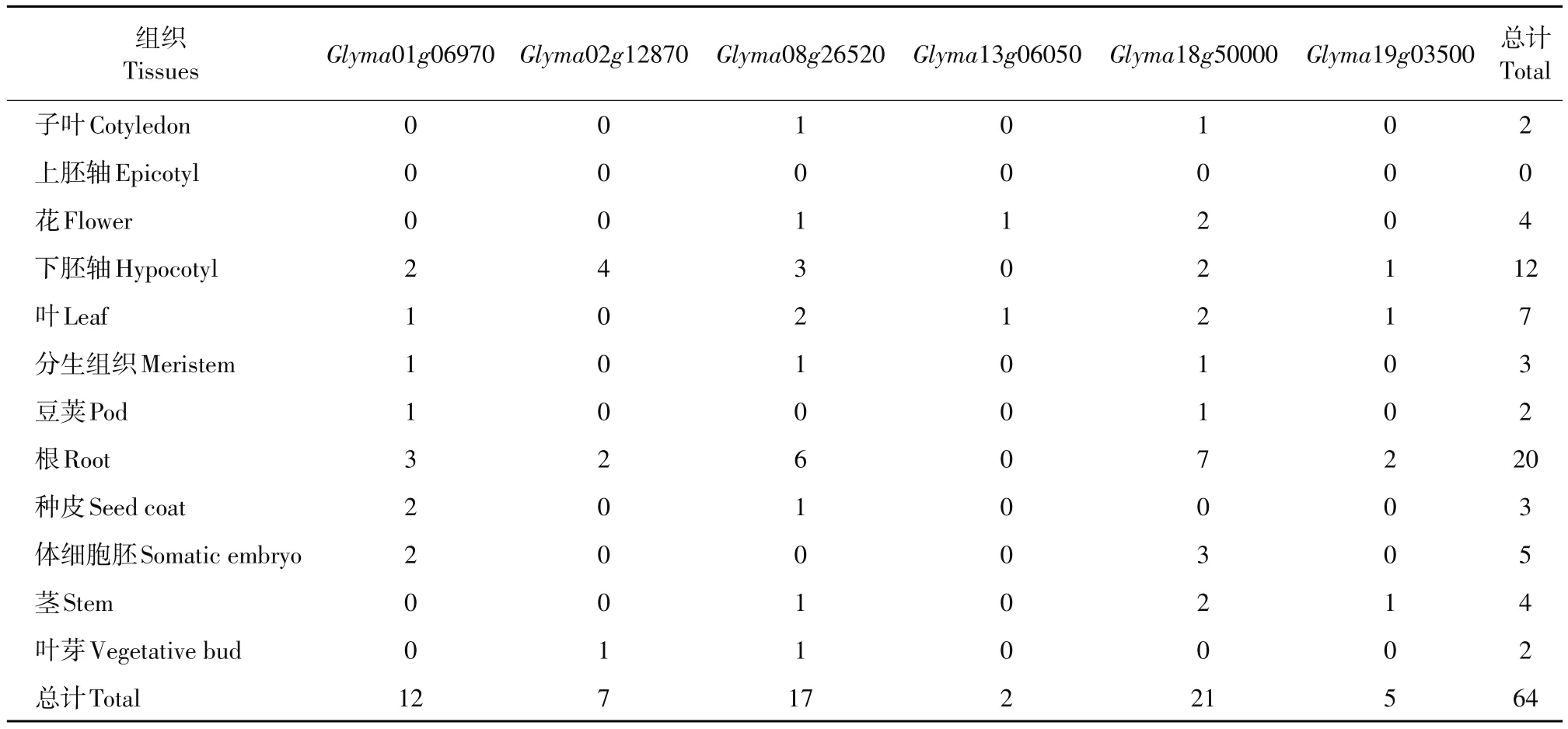
表1GmUGD基因组织表达特异性Table 1Expression patterns of GmUGD genes in tissues
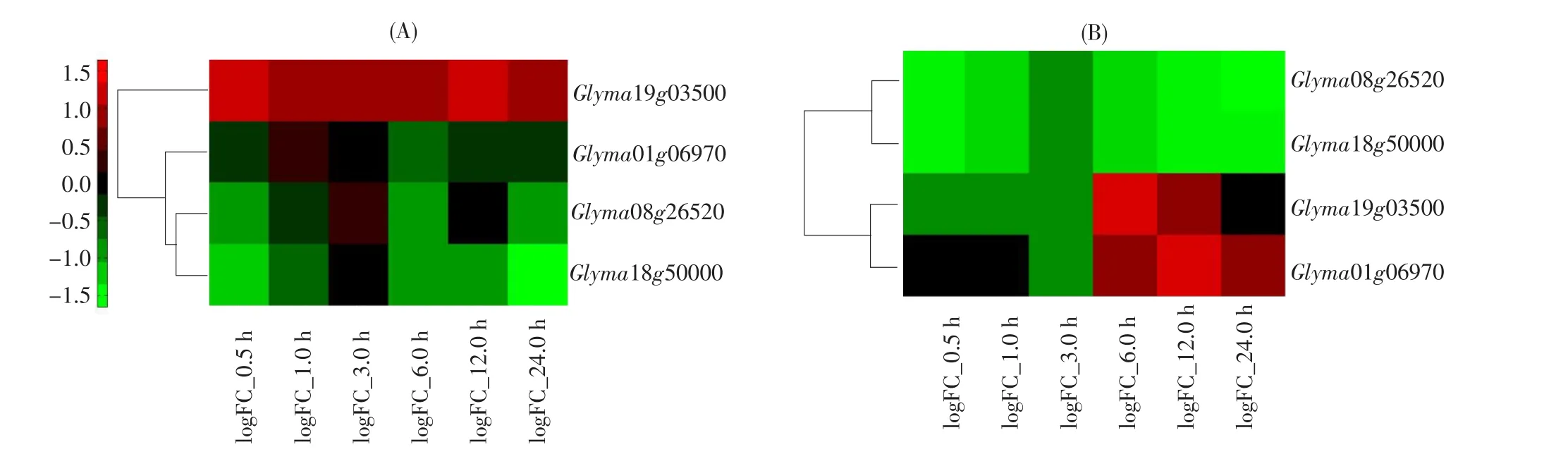
图5GmUGD家族基因表达模式聚类Fig.5Hierarchical cluster of expression pattern of GmUGD genes
3 讨论与结论
大豆全基因测序完成,为研究人员从全基因组水平研究基因功能奠定基础。生物信息学是全基因组数据分析的有效工具,已广泛用于多个基因家族鉴定、基因结构分析与功能预测[14-16]。但是,大豆中UGD家族基因及其功能研究尚未见报道。
本研究利用生物信息学方法,从大豆基因组中鉴定10个GmUGD家族基因。通过氨基酸序列分析,发现大部分GmUGD基因序列高度保守,均具有UDP-葡萄糖/GDP-甘露糖脱氢酶家族的3个典型区域:N端的NAD结合区、C端的UDP结合区及中间部分的UDP-葡萄糖/GDP-甘露糖脱氢酶家族区,说明这些高度保守的结构域可能是GmUGD基因参与糖代谢过程的主要功能区域。
基因复制是基因组扩增的原因之一。GmUGD家族基因80%基因之间存在基因复制事件,系统发生分析结果与基因复制分析结果相吻合:存在复制事件基因在系统发生树中处于同一分枝。这些基因可能在植物生长发育或胁迫应答过程中行使相同或相似功能。
研究表明,UGD基因在不同植物中组织表达情况不同。在拟南芥幼苗期,UGD基因主要在根部表达,而成熟期主要在维管束中表达[17-18]。而在苎麻中,UGD基因在茎中表达量最高,其次是种皮、叶和根[19]。本研究显示,GmUGD基因主要在根部表达,其次是下胚轴、叶等营养器官,在豆荚和叶芽中数量较少,此结果与之前研究结果一致[5]。因此,推测GmUGD基因在不同植物生长发育过程中发挥不同功能。
GmUGD基因盐碱胁迫表达模式分析同样显示其在不同组织表达具有较大差异。在盐碱胁迫下,4个GmUGD基因表达水平发生显著变化,说明这些GmUGD基因可能在大豆盐碱胁迫响应过程中发挥作用。但是,其在碱胁迫下的具体功能和响应模式还需进一步验证。
本研究利用生物信息学方法,从基因组水平系统筛选大豆GmUGD,并对其进行保守结构域分析、序列motif分析、基因复制分析、系统发生分析、组织表达模式分析及盐碱胁迫表达模式分析。从大豆基因组中共预测10个GmUGD家族基因,这些GmUGD基因高度保守,均具有UDP-葡萄糖/GDP-甘露糖脱氢酶家族典型区域。GmUGD家族基因间存在大量基因复制事件,存在复制事件的GmUGD基因间具有较近进化距离。GmUGD基因在根、茎、叶等不同组织器官中均有表达,其中,根中ESTs最多,上胚轴中ESTs最少。GmUGD基因在盐碱胁迫下表达水平发生显著变化,在盐碱胁迫下的表达模式具有组织特异性。本研究阐明了GmUGD基因基本特点,可为后期该家族基因功能分析提供参考与理论依据。
[1]Lei L M,Song L R.Cloning and characterization of the gene for UDPGlc dehydrogenase from the cyanobacterium,Microcystis aeruginosa FACHB 905[J].Acta Botanica Sinica,2004,46(11):1373-1382.
[2]Samac D A,Litterer L,Temple G,et al.Expression of UDP-glu-cose dehydrogenase reduces cell-wall polysaccharide concentration and increases xylose content in alfalfa stems[J].Appl Biochem Biotechnol,2004,113-116:1167-1182.
[3]Karkonen A,Murigneux A,Martinant J P,et al.UDP-glucose dehydrogenases of maize:A role in cell wall pentose biosynthesis[J]. Biochem J,2005,391(Pt 2):409-415.
[4]Johansson H,Sterky F,Amini B,et al.Molecular cloning and characterization of a cDNA encoding poplar UDP-glucose dehydrogenase,a key gene of hemicellulose/pectin formation[J].Biochim Biophys Acta,2002,1576(1-2):53-58.
[5]Tenhaken R,Thulke O.Cloning of an enzyme that synthesizes a key nucleotide-sugar precursor of hemicellulose biosynthesis from soybean:UDP-glucose dehydrogenase[J].Plant Physiol, 1996,112(3):1127-1134.
[6]贺顺姬,赵向丽,韩锐,等.盐藻多糖合成关键酶—尿苷二磷酸葡萄糖脱氢酶(DsUGD)基因分析及耐盐相关性研究[J].四川大学学报,2006,43(3):713-716.
[7]Plomion C,Lalanne C,Claverol S,et al.Mapping the proteome of poplar and application to the discovery of drought-stress responsive proteins[J].Proteomics,2006,6(24):6509-6527.
[8]Lata C,Sahu P P,Prasad M.Comparative transcriptome analysis of differentially expressed genes in foxtail millet(Setaria italica L.)during dehydration stress[J].Biochem Biophys Res Commun, 2010,393(4):720-727.
[9]Larkin M A,Blackshields G,Brown N P,et al.Clustal W and ClustalXversion2.0[J].Bioinformatics,2007,23(21):2947-2948.
[10]Yang S,Zhang X,Yue J X,et al.Recent duplications dominate NBS-encoding gene expansion in two woody species[J].Mol Genet Genomics,2008,280(3):187-198.
[11]Tamura K,Peterson D,Peterson N,et al.MEGA5:Molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods[J].Mol Biol Evol, 2011,28(10):2731-2739.
[12]Ge Y,Li Y,Zhu Y M,et al.Global transcriptome profiling of wild soybean(Glycine soja)roots under NaHCO3treatment[J].BMC Plant Biol,2010(10):153.
[13]Ge Y,Li Y,Lv D K,et al.Alkaline-stress response in Glycine soja leaf identifies specific transcription factors and ABA-mediated signalingfactors[J].FunctIntegrGenomics,2011,11(2):369-379.
[14]王志坤,常健敏,李丹丹,等.大豆GmWRI1a基因克隆及生物信息学分析[J].东北农业大学学报,2013,44(7):11-16.
[15]朱红霞,胡利宗,邓小莉,等.大豆SBP基因家族的序列特征、表达及进化分析[J].东北农业大学学报,2012,43(7):26-33.
[16]罗晓,曹蕾,王明超,等.野生大豆盐碱胁迫响应基因GsZFP1的克隆及序列分析[J].东北农业大学学报,2012,43(4):20-26.
[17]Seitz B,Klos C,Wurm M,et al.Matrix polysaccharide precursors in Arabidopsis cell walls are synthesized by alternate pathways with organ-specific expression patterns[J].Plant J,2000,21(6):537-546.
[18]Kanter U,Usadel B,Guerineau F,et al.The inositol oxygenase gene family of Arabidopsis is involved in the biosynthesis of nucleotide sugar precursors for cell-wall matrix polysaccharides[J]. Planta,2005,221(2):243-254.
[19]刘峰,黄妤,郭清泉,等.苎麻UDPGDH基因的cDNA克隆及表达分析[J].中国农业科学,2008(11):3542-3548.
Bioinformatics analysis ofGmUGDgene family in soybean genome/
ZHU Yanming,ZHU Yi,DUANMU Huizi,XIAO Jialei,YU Yang,LIU Ailin
(Key Laboratory of Agricultural Biological Functional Genes,NortheastAgricultural University,Harbin 150030,China)
UDP-glucose 6-dehydrogenase(UGD)is the key enzyme of polysaccharide biosynthesis, which plays important roles in the development and response to abiotic stresses of plant.A genome-wide analysis was conducted using bioinformatics and tenGmUGDgenes of soybean were identified.Sequence analysis showed that all theGmUGDgenes had three conserved domains which were typical in UDP-glucose/GDP-mannose dehydrogenase family.Gene duplication analysis indicated eighty percent of GmUGDgenes were duplicated.Phylogenetic analysis showed thatGmUGDgenes were classified into three clusters,and the result was similar with gene duplication analysis.Expression pattern analysis which based on UniGene and GEO database indicated that more ESTs ofGmUGDswere found in root than other tissues and none were found in epicotyls.In addition,theGmUGDgenes responded to salinity-alkalinity stress and showed differential expression pattern under salinity-alkalinity stress.These results provide theoretical basis for further research ofGmUGDgenes function in soybean.
GmUGDgenes;soybean;bioinformatics;sequence analysis;expression analysis
Q945.78;S565.1
A
1005-9369(2015)09-0023-07
时间2015-9-23 10:43:33[URL]http://www.cnki.net/kcms/detail/23.1391.S.20150923.1043.028.html
朱延明,朱毅,端木慧子,等.大豆GmUGD基因家族生物信息学分析[J].东北农业大学学报,2015,46(9):23-29.
Zhu Yanming,Zhu Yi,Duanmu Huizi,et al.Bioinformatics analysis ofGmUGDgene family in soybean genome[J].Journal of Northeast Agricultural University,2015,46(9):23-29.(in Chinese with English abstract)
2015-03-08
国家自然科学基金项目(31171578);转基因生物新品种培育重大专项(2011ZX08004-002);黑龙江省高校科技创新团队建设计划(2011TD055)
朱延明(1955-),男,教授,博士,博士生导师,研究方向为分子生物学与基因工程。E-mail:ymzhu2001@neau.edu.cn

