TAIL—PCR研究进展及其在畜牧兽医中的应用
马玲 王志强 覃绍敏 吴健敏
摘要:熱不对称交错PCR(TAIL-PCR)是一种以PCR为基础的染色体步移,主要用于分离已知基因的侧翼序列,具有简便、快速、高效、特异性强等特点。文章从TAIL-PCR的原理、优势及局限性、反应体系优化等方面对TAIL-PCR的研究进展进行综述,其中在畜牧兽医中主要用于已知基因侧翼克隆和转基因动物模型外源基因插入位点确定。但由于基因组DNA的庞大和复杂性,使得从基因组中克隆获得目的基因仍存在诸多限制因素,如特异性引物错配和随机引物结合位点不确定,易造成非特异性产物的产生;模板GC含量异常或存在高度重复序列时,即使使用多种简并引物也难以获得阳性产物。因此,应根据实际情况对TAIL-PCR进行灵活调整。当模板较复杂时,可借鉴连接介导PCR原理,给引物加上接头,提高扩增反应的特异性;当模板较简单或序列较清晰时,可根据模板特点(如高GC含量)或已知模板序列设计引物、调整反应条件,增加操作的简便性;还可参考Suppression-PCR原理,选择引物和反应条件,获得较长的扩增片段。此外,TAIL-PCR可与多种技术联合使用,促使其在畜牧兽医中获得更广泛深入的应用。
关键词: TAIL-PCR;染色体步移;研究进展;应用
中图分类号: Q813 文献标志码:A 文章编号:2095-1191(2017)05-0926-07
Advances in TAIL-PCR and its application in animal
husbandry and veterinary
MA Ling 1, WANG Zhi-qiang 2,3, QIN Shao-min1, WU Jian-min1 *
(1 Guangxi Veterinary Research Institute / Guangxi Key Laboratory of Animal Epidemic Etiology and Diagnostic, Nanning 530001, China; 2 State Key Laboratory for Conservation and Utilization of Subtropical Agro-Bioresearches, Nanning 530004, China; 3 College of Life Science and Technology, Guangxi University, Nanning 530004, China)
Abstract:Simple, rapid, high efficient and specific, thermal asymmetric interlaced PCR(TAIL-PCR), a kind of chromosome walking based on PCR, was mainly used to isolate the flanking sequences of target DNA segments. The principle, advantage, disadvantage and reaction system optimization of TAIL-PCR as well as some critical problem were summarized. In animal husbandry and veterinary medicine, TAIL-PCR was mainly used to isolate exogenous gene insertion sites and clone flanking sequences of the known genes in transgenic animals. Being limited by the complexity of genome DNA, there were so many limited factors preventing cloning target genes from genome, such as amplification of nonspecific products caused by mismatch of specific primers and arbitrary primers, and the difficulties in obtaining positive products even using multiple degenerate primers caused by abnormal template GC content and highly repeated sequences. So the adjustments should be made depend on actual situation. When the templates were complex, adapter could be added to the primers to improve the specificity based on the principle of LM-PCR; while when the template were simple or the sequence were clear, primers could be designed and reaction conditions could be set based on the characters of the templates(high GC content) or known template sequence, which made it easier to operate; and the suitable primers and reaction conditions could be chosen to get longer amplification products based on the principle of Suppression-PCR. Moreover, TAIL-PCR could be used with other technologies to facilitate its application in animal husbandry and veterinary.
Key words: TAIL-PCR; chromosome walking; research progress; application
0 引言
获得较完整基因序列是研究基因结构、功能和表达调控的必要条件,确定外源基因的插入位点是建立转基因动植物模型和插入突变体后的首要步骤,掌握反转录病毒科病毒在宿主基因组中的整合位点是研究病毒致病机理的根本前提,而开展这些研究均需要从已知基因序列出发,通过染色体步移逐步获得其上、下游未知序列或与其有线性关系的目标序列。近年来,PCR和生物信息学的发展促使染色体步移变得越来越简单、快捷,相继出现了多种以PCR为基础的染色体步移技术,包括反向PCR、连接介导PCR、抑制PCR、锅柄PCR、高通量染色体步移等(李付鹏等,2010;雷明等,2016;彭雷等,2016),但这些方法大多存在操作繁琐、试验周期较长的缺陷,而且有的依赖于酶切和连接,受酶切位点、连接效率影响较明显,有的则因基因组DNA结构限制了反应效率。热不对称交错PCR(Thermal asymmetric interlaced PCR,TAIL-
PCR)具有简便、快速、高效、特异性强等特点,已广泛应用于多个学科研究领域(Liu and Whittier,1995;郑岑等,2009;陈笑芸等,2013),成功获得多种动植物和微生物基因启动子序列、基因全长,并用于转基因动植物外源基因插入位点侧翼序列的克隆。为此,本文基于TAIL-PCR的原理和特点,总结近年来该方法的研究进展,以期为TAIL-PCR的推广应用提供参考。
1 TAIL-PCR原理
1. 1 基本原理
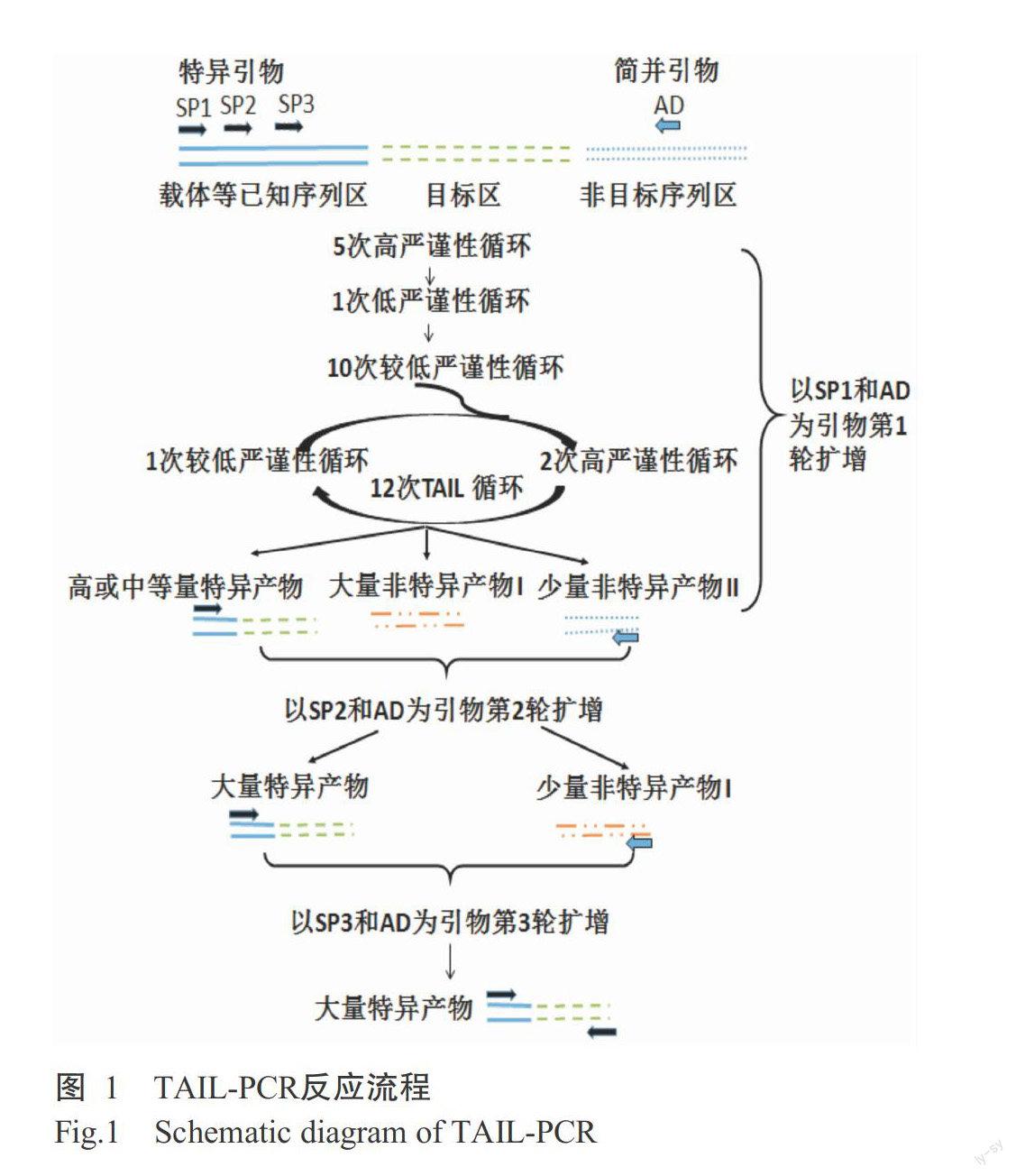
TAIL-PCR原理是根据已知序列和物种保守氨基酸设计3个高DNA熔解温度(Melting temperature,Tm)的嵌套式特异性引物(Specific primer,SP1/2/3)和多个低Tm的随机简并引物(Arbitrary primer,AD),然后以基因组DNA为模板,设计3轮半巢式PCR扩增反应(李付鹏等,2010)。由于引物对的Tm存在明显差别,TAIL-PCR通常被设计成热不对称的多级反应(Liu and Whittier,1995)。其中,第1轮扩增包括5个使SP与已知序列基因退火并延伸的高严谨性循环、1个使AD与未知序列模板退火并延伸的低严谨性循环及促使2种引物退火并延伸的10个较低严谨性循环和12个热不对称交错循环(图1);第2和3轮扩增则分别包括10个热不对称交错循环和20个较低严谨性循环。
第1轮扩增以SP1和AD为引物,可获得三类产物:SP1和AD(I类)、SP1(II类)、AD(III类)扩增产物。第2轮扩增以SP2和AD为引物,以1000倍稀释的第1轮扩增产物为模板,起始的高严谨性循环更有利于I类产物的大量扩增;在随后的循环中,由于缺少SP2结合位点和模板量较少,II類、III类产物也相对较少。第3轮扩增以SP3和AD为引物,20个较低严谨性循环虽然有利于2种引物退火,但不足以使模板量较少的II类、III类产物扩增到可见程度,从而进一步特异性扩增I类产物。通过上述反应,特异性扩增产物占有绝对比例,可通过胶回收或直接测序获得目的基因的序列而获得已知基因的侧翼序列。
1. 2 引物设计
根据TAIL-PCR的原理可发现,引物错配可直接导致非特异性(II类、III类)产物的扩增。因此,引物设计是TAIL-PCR的关键,要求即使在低严谨性循环中也不能产生引物二聚体。
除Tm较高(58~68 ℃)外,3个SP间应相距100 bp以上,以便于通过电泳区分各级反应产物,进而初步判断反应特异性。SP错配可导致II类产物扩增(郑岑等,2009),尤其当SP的3'端GC含量较高时,因此设计SP时需注意3'端GC含量。通过TAIL-PCR的反应流程(图1)可知,第1轮扩增对整个流程影响较大,是控制特异性扩增的关键,如TAIL-PCR最终未能获得满意结果,可重新设计SP1用于试验。
AD的Tm需比SP低10 ℃以上,且使用5个简并碱基(简并度128~256),同时设4种3'序列:AGA/AGG/
GAA/GGA,以增加AD在已知基因侧翼序列中的结合位点,便于目的基因的扩增。一般情况下,TAIL-PCR的成功率为60.0%~80.0%,扩增片段0.3~1.0 kb,甚至2.0 kb,若仅获得250 bp以下的扩增产物,则需更换AD。此外,不同AD在不同物种中的应用效果也有差异,可同时采用多个AD进行扩增筛选(陈笑芸等,2013)。
1. 3 反应条件
在TAIL-PCR中,高、较低和低严谨性循环的退火温度分别设为63、44和30 ℃。其中,高严谨性循环中的退火温度可比SP的Tm高1~5 ℃,以提高反应特异性。为减少错配,可使用较低浓度的SP(0.15~0.20 μmol/L),但AD使用浓度宜高(3.00~5.00 μmol/L),以满足结合效率;使用含有次黄嘌呤的AD时,用量减半。此外,如需直接测序TAIL-PCR扩增产物,第2、3轮扩增中添加25 μmol/L dNTP可满足扩增要求。在TAIL-PCR中,低严谨性循环和简并引物的使用可提高引物正确结合的概率。
2 TAIL-PCR优势及局限性
与反向PCR、连接介导PCR等相比,TAIL-PCR无需进行酶切、连接、加尾等模板前期处理,也不需要Southern blotting、引物标记等后期处理,操作简便,同时避免了酶切、连接对反应效率的影响和连接过程可能出现的嵌合体;对模板的含量和纯度也没有更高要求,操作过程基本与PCR类似,可实现大量样品的自动化加样、扩增;扩增产物特异性较强,可直接用于杂交探针和序列测定(郑岑等,2009;Chen et al.,2016)。在适用性方面,TAIL-PCR可用于包括哺乳动物基因组在内各种复杂、简单样品的检测,整个反应流程可在24 h内完成,极大节省了时间,成功率在60.0%~
80.0%。
TAIL-PCR由三级连续扩增反应组成,第1轮扩增的产物对后续反应有较大影响,因此试验前须优化各反应条件,且TAIL-PCR的引物设计和反应条件相对复杂,给其推广应用带来了困难。如同一简并引物在不同物种中的扩增效果明显不同,需同时采用多个通用引物平行扩增以获得理想的结果;而第1轮扩增中II、III类产物可能会导致扩增失败或仅获得小片段的扩增产物(Liu and Chen,2007;陈笑芸等,2013)。分子生物学的发展对染色体步移技术提出了更高要求,包括希望获得更长的侧翼基因序列、进一步缩短反应时间、从含量较低或较高GC含量样品中扩增出目的基因等,因此需要围绕TAIL-PCR开展多方面的优化设计,通过与生物信息学等方法联用,提高反应成功率,扩大推广应用面。
3 TAIL-PCR反應体系优化
3. 1 引物筛选优化
为提高反应效率、获得更长的基因片段,Liu和Chen(2007)改进优化TAIL-PCR反应体系,以LAD代替AD,并根据LAD的5'端序列合成引物AC1,同时参照Suppression-PCR原理抑制500 bp以下片段的扩增,最终建立了高效TAIL-PCR(High-efficiency thermal asymmetric interlaced PCR,hiTAIL-PCR)。hiTAIL-PCR的扩增产物以1.0~3.0 kb为主,远大于TAIL-PCR的0.3~1.0 kb。Liu和Chen(2007)研究证实,hiTAIL-PCR的成功率可达93.3%,且大多数目的基因通过两轮PCR即可获得,有效节省了反应时间。此外,在第1轮扩增中如将两种LAD混合使用,可使成功率提高到97.4%。
与hiTAIL-PCR相似,Wang等(2011b)在TAIL-PCR基础上,于AD的5'端添加31 bp基因片段(Fusion primer),以此作为第1轮PCR扩增的引物,相当于给扩增产物连上“接头”,在第2、3轮扩增中分别根据“接头”序列设计FSP1、FSP2引物与特异性引物配对扩增,增强反应特异性,成功率超过85.0%。此外,该方法在反应条件上也进行优化,仅在第1轮扩增中使用超级循环,使整个反应可在4 h内完成,极大节省了反应时间,并获得2.0 kb以上的片段。优化后的TAIL-PCR通用性良好,可从7种蔷薇科植物中扩增获得21个目的基因片段、从玫瑰中扩增获得4个MYB基因片段、从矮牵牛的MADS-Box转录因子家族中扩增获得3个启动子序列,以及日本落叶松TCTP基因的5'侧翼启动子区序列(Zhang et al.,2013)和沙雷氏菌ChrT基因5'和3'侧翼序列(Deng et al.,2015)。
TAIL-PCR扩增产物通常需要克隆才能获得目的基因序列,为控制克隆时扩增产物插入载体的方向,Tan等(2005)建立了SiteFinding-PCR,即设计2种SiteFinder引物(5'端为含Not I酶切位点和6个N的基因片段,3'端分别为GCCT/GCGC),并根据SiteFinder 5'端设计部分序列重复的巢式引物SFP1和SFP2。在其反应程序中,首先采用SiteFinder进行1个反应的单引物扩增,然后在体系中加入特异性引物和SFP1,进行30个循环的二温式PCR扩增,两轮扩增结束后以Not I酶切扩增产物,连接至经Not I和EcoR V双酶切的pBlueScript SK(+)载体上,最后以M13R和特异性引物配对,PCR筛选目标产物。采用优化后的TAIL-PCR,Tan等(2005)在噬蓝藻体中扩增获得4617 bp的目的片段、从15种拟南芥的变异体中扩增获得14个T-DNA插入位点(未优化的TAIL-PCR仅扩增获得9个)。该方法首先根据Suppression-PCR原理,使非目的基因扩增产物形成茎环结构而抑制其扩增;其次是进一步简化PCR反应条件,缩短反应时间,并通过酶切位点控制基因片段插入载体的方向,以便于PCR鉴定,是一种简便、快捷扩增大片段侧翼序列的方法。
但SiterFinder中6个N的使用增加了自我配对的风险,为此,Luo等(2011)设计了11条CLP简并引物,仅使用4个N,并将3'端的碱基增加到7个。这7个碱基中,有的含有6 bp的酶切位点,有的是2个3 bp的重复序列,有的则是4 bp的酶切位点及1个嘌呤碱和1个嘧啶碱。此外,在第1轮扩增中只添加特异性引物进行30个循环的普通反应,快速冷却后加入CLP,进行1个变性温度80 ℃、退火温度43 ℃的循环,此举可增加CLP结合到新合成单链DNA上的机会;在第2轮扩增中,以特异性引物和根据CLP 5'端序列合成的引物RL进行二温式PCR扩增。采用优化后的TAIL-PCR,分别扩增获得创伤弧菌rpoB基因的3044和4055 bp片段、溶藻弧菌转座酶基因的3820 bp片段、霍乱弧菌sto基因的1632 bp片段。该方法较TAIL-PCR和SiteFinding-PCR更简便快速,且特异性强,但由于没有任何一条CLP可同时扩增出上述基因片段,即要求在扩增不同样品基因时需分别设计引物。此外,Wu等(2015)设计22条简并引物从T-DNA插入突变体库中扩增T-DNA插入位点,结果证实使用较多的AD可将TAIL-PCR成功率从39.0%提升至69.0%,对于仍难以扩增的样品,可结合高通量测序获得其插入位点的侧翼序列。
可见,TAIL-PCR引物的筛选优化主要是通过设计较长或较多的简并引物,但简并引物的设计需要参考生物密码子的偏好性。Terauchi和Kahl(2000)、Qiu等(2008)使用商品化的RAPD为简并引物,通过调整反应条件,成功分离获得山药Pal和Pgi的5'区域及小麦X基因5'端未知1077 bp的侧翼序列。TAIL-PCR中AD的3'端是3~4个确定序列的碱基,是决定扩增能否进行的关键。在已知基因序列中寻找与AD 3'端3~4个碱基完全相同的基因序列不难,李道恒等(2013)利用该原则,以已知基因序列为基础,设计可同时作为特异引物和简并引物的引物,并通过单引物扩增获得转基因水稻的2个插入位点,同时将反应条件修改为5个低特异性反应及30个较高特异性反应,极大简化了引物设计、节省了反应时间。 此外,在简并引物设计方面,Hecht等(2010)、Teixeira等(2011)、Guimaro等(2014)依据克氏锥虫感染人体后基因组主要插入人类染色体还原转座酶LINE-1的特点,分别针对插入基因和被插入基因的保守区域设计特异引物取代AD,有效减少了简并碱基的使用,且将第3轮扩增简化为普通PCR。
3. 2 反应条件优化
虽然经典TAIL-PCR可满足许多样品的扩增要求,但特定条件下,优化反应条件可获得更理想的效果。在Liu和Whittier(1995)建立的TAIL-PCR中,低严谨性循环的退火温度为30 ℃,保持3 min后缓慢升至72 ℃,但在实际应用中较多选用25 ℃为退火温度(王宝维等,2010;Wada et al.,2014),也有研究将其调整为28 ℃(孙科军等,2012),或选择快速升温至72 ℃(孔庆然等,2009;Huang et al.,2010)。潘华奇等(2009)在克隆鸭肠炎病毒时,为充分变性而将变性温度升高至98 ℃。He等(2010)在扩增猪APC10基因时,去除第1轮扩增中的10个低特异性循环。可见,各种反应条件的优化均与物种基因组的复杂性、引物的简并和特异性等有关。
TAIL-PCR的AD中GC含量低于50%,且Tm较低,对扩增高GC含量的样品非常不利。为此,Zhou等(2010)建立了GC TAIL-PCR,即根据放线菌属GC含量在51%~70%及其木聚糖酶CDS中密码子第3位通常是G/C的特点,设计12条GC含量在50%~85%的GCAD及6条特异性引物;其扩增程序则根据GCAD Tm较高的特点,分别将高、低和较低特异性反应中退火温度升高至70、30和50 ℃,并使用GC Buffer,扩增出常规AD无法扩增的木聚糖酶1089 bp的基因片段和4个高GC含量的基因片段。这为扩增高GC含量的基因片段提供了技术支撑。
为提高反应特异性,Lin等(2014)通过分析已知基因和基因组的序列特征,选用EcoR I和Hind III酶切基因组为模板,优化后的TAIL-PCR能从转基因山羊中扩增获得4个IGF-1的插入位点,与经典TAIL-PCR相比,其成功率明显提高。在从大量、复杂的环境样品中扩增含量极低的基因时,Huang等(2010)将第1轮扩增拆分为两步:先用特异性引物进行80个循环的单引物扩增,增加目的基因的拷贝数,然后加入简并引物完成后续扩增,通过此法成功从波尔山羊和荷斯坦奶牛的瘤胃液及纸浆厂的废水中扩增获得GH10、GH11和PTP-like植酸酶基因。
3. 3 与其他方法联合使用
TAIL-PCR可通过已知基因扩增其未知的侧翼序列,因此已知基因序列获得是TAIL-PCR的基础。Wang等(2011a)在扩增冬虫夏草的微卫星标记时,先应用ISSR-PCR扩增获得基因序列,再据此设计引物,通过TAIL-PCR获得完整基因序列,从48份样品中扩增出30个微卫星标记,可用于冬虫夏草群体遗传学和保护生物学研究。Bushley等(2013)先通过简并PCR扩增冬虫夏草DNA裂解酶基因、MAT1-1-1的α-box和MAT1-1-3的HMG-box,然后通过TAIL-PCR和Long-rang PCR扩增获得各基因间的未知区域序列,并将基因片段连接起来,最终获得冬虫夏草MAT1-1的基因座。Wada等(2014)通过Exon Array在急性T淋巴细胞白血病细胞系T-ALL中查找到3个融合基因,然后结合EST数据库在24个乳腺癌细胞系和20个胰腺癌细胞系中搜索到7个可能的融合基因,最终通过TAIL-PCR进行验证,证实了其中3个融合基因。
在扩增双拷贝基因及其侧翼序列时,Zhang等(2012)先用Southern blotting选择EcoR I酶切基因组,然后以反向PCR将双拷贝的侧翼序列区分开,根据反向PCR结果,采用TAIL-PCR扩增未知的侧翼序列,成功获得棉花可育胞质和不可育胞质线粒体双拷贝atpA基因的所有EcoR I(2.2~5.1 kb)和Hind III(8.5~
11.7 kb)限制片段。在进行较长片段的染色体步移时,Chen等(2016)、Zha等(2016)采用3'-RACE获得目的基因的部分序列,结合EST或GenBank数据库设计SP,分别通过多次TAIL-PCR、hiTAIL-PCR向5端方向进行染色体步移,直至获得武昌鱼PHD1、PHD3的5'端启动子和露珠杜鹃花蜜中几丁质酶Rhchi2和Rhchi3的cDNA全长序列。
综上所述,生物信息学和分子生物技术的发展使得PCR扩增未知基因变得越来越高效、简便。根据基因保守区采用简并PCR、基因数据库查找及其他方法可快速获得基因片段,用作TAIL-PCR的基础。
4 TAIL-PCR在畜牧兽医中的应用现状及发展前景
4. 1 TAIL-PCR在畜牧兽医中的应用现状
目前,TAIL-PCR在畜牧兽医中主要用于已知基因侧翼克隆和转基因动物模型外源基因插入位点确定。潘华奇等(2009)采用包括TAIL-PCR在内的多种PCR克隆鸭肠炎病毒gB基因;刘峰源等(2009)根据GenBank中鴨肠炎病毒UL45基因设计特异性引物,以TAIL-PCR扩增获得gC基因全长和UL43基因部分序列,并进一步证实鸭肠炎病毒的系统发育分类。在病原基因分离方面,马春骥等(2011)利用同源克隆和TAIL-PCR扩增获得绵羊肺炎支原体Y98株热休克蛋白Hsp70长2481 bp的基因序列,其中包含1815 bp的开放阅读框,为绵羊肺炎支原体的抗原、Hsp70功能研究打下了基础。在微生物基因克隆方面,王宝维等(2010)采用TAIL-PCR扩增获得鹅源草酸青霉F64 CBHI基因序列全长,且从草酸青霉中克隆出一种新的膨胀素样蛋白POSWOI全长基因(Kang et al.,2013),为纤维素类生物质的高效生物转化开辟了新途径。
动物体内的某些基因通常与疾病发生和机体生长发育有密切关系,尤其是基因的启动子区域。如甘露聚糖结合凝集素基因是机体天然免疫系统的重要组成,通过激活补体系统参与构成抗感染第一道防线。余鹏等(2011)利用TAIL-PCR扩增获得绵羊甘露聚糖结合凝集素基因启动子区,为研究该基因的表达调控和启动子区甲基化提供了支持;孙科军等(2012)通过TAIL-PCR和PCR克隆获得的斑鳜肌肉生长抑制素基因及其启动子序列,为研究斑鳜肌肉生长和发育调控打下了基础;Zhou等(2014)克隆获得S100A6启动子区的NF-κB结合位点,并证实猪繁殖与呼吸综合征病毒感染细胞后可通过多种途径激活NF-κB通路。
转基因动物中,外源基因定位和拷贝数的确定与基因表达、基因稳定性、生产性能和安全评价等存在直接关系,也是建立转基因动物模型后的首要检测步骤之一。孔庆然等(2009)、Lin等(2014)采用qPCR和TAIL-PCR代替传统的Southern blotting分别鉴定外源基因在IGF-1转基因羊和绿色荧光蛋白转基因猪中的拷贝数和整合位点,成功建立了转基因动物鉴定体系。Wang等(2012)利用TAIL-PCR检测肝脏特异性表达分泌型IgD转基因小鼠中目的基因插入位点,但发现外源基因的插入并未破坏位于小鼠5号染色体上的甲羟戊酸激酶(MVK)基因,即高IgD综合征发病机理尚有待进一步探究。除转基因动物外源基因定位外,也有通过TAIL-PCR确定外源基因在宿主基因组中插入位点的研究。Bi等(2013)和王毅(2013)分别制备链霉菌属噬菌体PhiC31整合酶表达载体,转染猪肾PK15细胞和山羊胎儿成纤维细胞,然后通过TAIL-PCR筛选定向整合到宿主基因的Pseudo attP位点;Huang等(2014)为鉴定构建的通用型细胞永生化载体元件功能,将细胞永生化载体转染至HEK293细胞,并通过TAIL-PCR确定该载体是以转座整合的方式插入宿主基因组中。
4. 2 TAIL-PCR在畜牧兽医中的发展前景
近年来,畜牧兽医业快速发展,动物品种改良、动物微生物学和病原学、食品安全检测及利用转基因动物生产珍贵蛋白、进行基因治疗和建立疾病模型等均涉及染色体步移。相对于其他以PCR为基础的染色体步移技术,TAIL-PCR对DNA的质量和用量要求相对较低,同时克服了连接、酶切等的限制,且随着方法的不断改进,其操作更简便、步移长度更长、成功率更高,因此TAIL-PCR在畜牧兽医中的应用将进一步扩大。但由于基因组DNA的庞大和复杂性,使得从基因组中克隆目的基因仍存在诸多限制因素,如特异性引物错配和随机引物结合位点不确定,易造成非特异性产物的产生;模板GC含量异常或存在高度重复序列时,即使使用多种简并引物也难以获得阳性产物;此外,复杂的反应条件给TAIL-PCR优化也带来一定困难。因此,应根据实际情况对TAIL-PCR进行灵活调整。当模板较复杂时,可借鉴连接介导PCR原理,给引物加上接头,提高反应的特异性;当模板较简单或序列较清晰时,可根据模板特点(如高GC含量)或已知模板序列设计引物、调整反应条件,增加操作的简便性;还可参考Suppression-PCR原理,选择引物和反应条件,获得较长的扩增片段。此外,随着生物信息学的发展及分子生物学技术的改进和新技术的出现,TAIL-PCR可与多种技术联合使用,发挥各自优势,更快速、高效地完成检测目标,进而促使TAIL-PCR在畜牧兽医中获得更广泛深入的应用。
参考文献:
陈笑芸,汪小福,刘仁虎,张小明,周育,缪青梅,徐俊锋. 2013. 不同TAIL-PCR通用引物在转基因大豆、水稻、油菜侧翼序列分离中的应用评价[J]. 核农学报,27(7):922-928. [Chen X Y,Wang X F,Liu R H,Zhang X M,Zhou Y,Miu Q M,Xu J F. 2013. Evaluate of different TAIL-PCR aribitrary degenerate on analysis of transgenic soybean and transgenic rices flanking sequence[J]. Journal of Nuclear Agriculture Science,27(7):922-928.]
孔庆然,武美玲,朱江,格日乐其木格,郇延军,尹智,牟彦双,刘忠华. 2009. 转基因猪中外源基因拷贝数和整合位点的研究[J]. 生物化学与生物物理进展,36(12):1617-1625. [Kong Q R,Wu M L,Zhu J,Gerelchimeg,Xun Y J,Yin Z,Mu Y S,Liu Z H. 2009. Transgene copy number and integration site analysis in transgenic pig[J]. Progress in Biochemistry and Biophysics,36(12):1617-1625.]
雷明,王加宾,丛汉卿,李志英,徐立. 2016. 蜻蜓凤梨AfWIN1基因的克隆及表达特性[J]. 贵州农业科学,44(11):17-23. [Lei M,Wang J B,Cong H Q,Li Z Y,Xu L. 2016. Cloning and expression characteristics of AfWIN1 in Aechemia fasciata[J]. Guizhou Agricultural Sciences,44(11):17-23.]
李道恒,马建,王云鹏,马景勇. 2013. 应用单引物PCR获得以Bar基因为锚定基因的转基因水稻侧翼序列[J]. 中国水稻科学,27(3):312-324. [Li D H,Ma J,Wang Y P,Ma J Y. 2013. Cloning flanking sequence of Bar gene by single-primer PCR in transgenic rice[J]. Chinese Journal of Rice Science,27(3):312-324.]
李付鹏,伍宝朵,马朝芝,傅廷栋. 2010. 基于PCR的染色體步移技术研究进展[J]. 中国生物工程杂志,30(12):87-94. [Li F P,Wu B D,Ma C Z,Fu T D. 2010. Progress of chromosome walking by PCR amplification techniques[J]. China Biotechnology,30(12):87-94.]
劉峰源,马波,张杨,赵妍,王君伟. 2009. 鸭肠炎病毒糖蛋白gC基因克隆与序列分析[J]. 中国兽医杂志,45(2):5-7. [Liu F Y,Ma B,Zhang Y,Zhao Y,Wang J W. 2009. Cloning,sequence analysis of glycoprotein C gene of duck enteritis virus[J]. Chinese Journal of Veterinary Medicine,45(2):5-7.]
马春骥,李敏,赵东,王玉炯. 2011. 绵羊肺炎支原体Y98株未知基因Hsp70(DnaK)的克隆[J]. 中国兽医学报,31(12):1733-1737. [Ma C J,Li M,Zhao D,Wang Y J. 2011. Using tail-PCR technology to clone the unknown gene Hsp70(DnaK) of Mycoplasma ovipneumoniae Y98[J]. Chinese Journal of Veterinary Science,31(12):1733-1737.]
潘华奇,曹瑞兵,陈溥言,刘磊,王书锦,潘光炎,胡江春. 2009. 几种PCR方法在克隆鸭肠炎病毒未知基因中的应用[J]. 微生物学通报,36(12):1842-1848. [Pan H Q,Cao R B,Chen P Y,Liu L,Wang S J,Pan G Y,Hu C J. 2009. Application of several PCR methods in cloning unknown genes of duck enteritis virus[J]. Microbiology,36(12):1842-1848.]
彭雷,赵艳,马银花. 2016. 褐飞虱看家基因启动子克隆与序列分析[J]. 贵州农业科学,44(9):4-7. [Peng L,Zhao Y,Ma Y H. 2016. Cloning and sequence analysis of brown planthopper house-keeping gene promoter[J]. Guizhou Agricultural Sciences,44(9):4-7.]
孙科军,刘希良,王开卓,陈敦学,褚武英,陈韬,张建社,成嘉. 2012. 斑鳜myostatin基因及其启动子的克隆与序列分析[J]. 基因组学与应用生物学,31(2):133-140. [Sun K J,Liu X L,Wang K Z,Chen D X,Chu W Y,Chen T,Zhang J S,Cheng J. 2012. Cloning and sequence analysis of the myostatin gene and its promoter in golden mandarin fish (Siniperca scherzeri)[J]. Genomics and Applied Biology,31(2):133-140.]
王宝维,张倩,葛文华,张名爱,岳斌,黄国清. 2010. 鹅源草酸青霉CBH I基因克隆及真核表达载体构建[J]. 中国农业科学,43(7):1464-1472. [Wang B W,Zhang Q,Ge W H,Zhang M A,Yue B,Huang G Q. 2010. Cloning of the CBH I gene of goose-origin Penicillium oxalicum Currie & Thom and its eukaryotic expression vector construction[J]. Scientia Agricultura Sinica,43(7):1464-1472.]
王毅. 2013. 检测山羊基因组中链霉菌属噬菌体φC31整合酶作用的pseudo attp位点[D]. 杨凌:西北农林科技大学. [Wang Y. 2013. Identification of pseudo attp sites for the streptomyces phage φC31 integrase in goat genome[D]. Yang-
ling:Northwest A & F University.]
余鹏,王遵宝,吴洪宾,班谦,赵宗胜. 2011. 绵羊甘露聚糖结合凝集素基因启动子区的克隆与序列分析[J]. 中国畜牧兽医,38(3):87-92. [Yu P,Wang Z B,Wu H B,Ban Q,Zhao Z S. 2011. Cloning and sequence analysis of the promoter region of sheep mannan-binding lectin gene[J]. China Animal Husbandry & Veterinary Medicine,38(3):87-92.]
郑岑,张立平,唐忠辉,赵昌平,苑少华. 2009. TAIL-PCR技术及其在植物基因中的克隆[J]. 基因组学与应用生物学,28(3):544-548. [Zheng C,Zhang L P,Tang Z H,Zhao C P,Yuan S H. 2009. TAIL-PCR and its cloning in plant gene[J]. Genomics and Applied Biology,28(3):544-548.]
Bi Y Z,Liu X M,Zhang L,Shao C W,Ma Z,Hua Z D,Zhang L P,Li L,Hua W J,Xiao H W,Wei Q X,Zheng X M. 2013. Pseudo attP sites in favor of transgene integration and expression in cultured porcine cells identified by Streptomyces phage phiC31 integrase[J]. BMC Molecular Biology,14:20.
Bushley K E,Li Y,Wang W J,Wang X L,Jiao L,Spatafora J W,Yao Y J. 2013. Isolation of the MAT1-1 mating type idio-
morph and evidence for selfing in the Chinese medicinal fungus Ophiocordyceps sinensis[J]. Fungal Biology,117(9):599-610.
Chen N,Wang W M,Wang H L. 2016. An efficient full-length cDNA amplification strategy based on bioinformatics technology and multiplexed PCR methods[J]. Scientific Reports,5:1-9.
Deng P,Tan X,Wu Y,Bai Q,Jia Y,Xiao H. 2015. Cloning and sequence analysis demonstrate the chromate reduction abili-
ty of a novel chromate reductase gene from Serratia sp.[J]. Experimental and Therapeutic Medicine,9(3):795-800.
Guimaro M C,Alves R M,Rose E,Sousa A O,de Cássia Rosa A,Hecht M M,Sousa M V,Andrade R R,Vital T,Plachy J,Nitz N,Hejnar J,Gomes C C,L Teixeira A R. 2014. Inhibition of autoimmune Chagas-like heart disease by bone ma-
rrow transplantation[J]. PLoS Neglected Tropical Diseases,8(12):e3384.
He W B,Wang Z W,Li Y,Tang Z L,Yang S L,Mu Y L,Peng K M,Li K. 2010. Sequence characterization and promoter identification of porcine APC10 gene[J]. Molecular Biology Reports,37(8):3841-3849.
Hecht M M,Nitz N,Araujo P F,Sousa A O,Rosa Ade C,Gomes D A,Leonardecz E,Teixeira A R. 2010. Inheritance of DNA transferred from American trypanosomes to human hosts[J]. PloS One,5(2):e9181.
Huang H,Hu G,Kang J,Qing S Z,Zhang Y. 2014. Construction of a general piggy Bac transposon inducible cell immortali-
zation vector and verification of its basic properties[J]. Chinese Journal of Biotechnology,30(8):1182-1192.
Huang H,Wang G,Zhao Y Y,Shi P J,Luo H Y,Yao B. 2010. Direct and efficient cloning of full-length genes from environmental DNA by RT-qPCR and modified TAIL-PCR[J]. Applied Microbiology and Biotechnology,87(3):1141-1149.
Kang K,Wang S W,Lai G H,Liu G,Xing M. 2013. Characterization of a novel swollenin from Penicillium oxalicum in facilitating enzymatic saccharification of cellulose[J]. BMC Biotechnology,13:42.
Lin J,Zhang Q,Zhu L Q,Yu Q H,Yang Q. 2014. The copy number and integration site analysis of IGF-1 transgenic goat[J]. International Journal of Molecular Medicine,34(3):900-910.
Liu Y G,Chen Y. 2007. High-efficiency thermal asymmetric interlaced PCR for amplification of unknown flanking sequences[J]. BioTechniques,43(5):649-650.
Liu Y G,Whittier R F. 1995. Thermal asymmetric interlaced PCR:Automatable amplification and sequencing of insert end fragments from P1 and YAC clones for chromosome walking[J]. Genomics,25(3):674-681.
Luo P,Su T,Hu C,Ren C. 2011. A novel and simple PCR wal-
king method for rapid acquisition of long DNA sequence flanking a known site in microbial genome[J]. Molecular Biotechnology,47(3):220-228.
Qiu Y,Tian J,Ge R,Zhao B,Shen Y,Huang Z. 2008. A modified TAIL-PCR and its application in isolating gene promoter of wheat[J]. Chinese Journal of Biotechnology,24(4):695-699.
Tan G,Gao Y,Shi M,Zhang X Y,He S P,Chen Z H,An C C. 2005. SiteFinding-PCR:A simple and efficient PCR method for chromosome walking[J]. Nucleic Acids Research,33(13):e122.
Teixeira A R,Gomes C,Nitz N,Sousa A O,Alves R M,Guimaro M C,Cordeiro C,Bernal F M,Rosa A C,Hejnar J,Leonardecz E,Hecht M M. 2011. Trypanosoma cruzi in the chicken model:Chagas-like heart disease in the absence of parasitism[J]. PLoS Neglected Tropical Diseases,5(3):e1000.
Terauchi R,Kahl G. 2000. Rapid isolation of promoter sequences by TAIL-PCR:The 5'-flanking regions of Pal and Pgi genes from yams(Dioscorea)[J]. Molecular & General Genetics,263(3):554-560.
Wada Y,Matsuura M,Sugawara M,Sugawara M,Ushijima M,Miyata S,Nagasaki K,Noda T,Miki Y. 2014. Development of detection method for novel fusion gene using GeneChip exon array[J]. Journal of Clinical Bioinformatics,4(1):3.
Wang P,Wei Z,Yan B,Huang T,Gou K,Dai Y,Zheng M,Wang M,Cheng X,Wang X,Xu C,Sun Y. 2012. Establishment of a transgenic mouse model with liver-specific expression of secretory immunoglobulin D[J]. Science China Life Sciences,55(3):219-227.
Wang X L,Yang R H,Yao Y J. 2011a. Development of micro-
satellite markers for Ophiocordyceps sinensis(Ophiocordycipitaceae) using an ISSR-TAIL-PCR method[J]. American Journal of Botany,98(12):e391-e394.
Wang Z,Ye S,Li J,Zheng B,Bao M,Ning G. 2011b. Fusion primer and nested integrated PCR(FPNI-PCR):A new high-efficiency strategy for rapid chromosome walking or flanking sequence cloning[J]. BMC Biotechnology,11:109.
Wu L,Di D W,Zhang D,Song B,Luo P,Guo G Q. 2015. Frequent problems and their resolutions by using thermal asymmetric interlaced PCR(TAIL-PCR) to clone genes in Arabidopsis T-DNA tagged mutants[J]. Biotechnology & Biotechnological Equipment,29(2):260-267.
Zha H G,Milne R I,Zhou H X,Chen X Y,Sun H. 2016. Identification and cloning of class II and III chitinases from alkaline floral nectar of Rhododendron irroratum,Ericaceae[J]. Planta,244(4):805-818.
Zhang L F,Li W F,Han S Y,Yang W H,Qi L W. 2013. cDNA cloning, genomic organization and expression analysis du-
ring somatic embryogenesis of the translationally controlled tumor protein(TCTP) gene from Japanese larch(Larix leptolepis)[J]. Gene,529(1):150-158.
Zhang X,Zhang R,Sun G,Shi J,Meng Z,Zhou T,Hou S,Liang C,Yu Y,Guo S. 2012. High efficiency genome walking method for flanking sequences of cotton mitochondrial double-copy atpA gene based on optimized inverse PCR and TAIL-PCR[J]. Chinese Journal of Biotechnology,28(1):104-115.
Zhou J,Huang H,Meng K,Shi P,Wang Y,Luo H,Yang P,Bai Y,Yao B. 2010. Cloning of a new xylanase gene from Streptomyces sp. TN119 using a modified thermal asymme-
tric interlaced-PCR specific for GC-rich genes and bioche-
mical characterization[J]. Applied Biochemistry and Biote-
chnology,160(5):1277-1292.
Zhou X,Wang P,Michal J J,Wang Y,Zhao J,Jiang Z,Liu B. 2014. Molecular characterization of the porcine S100A6 gene and analysis of its expression in pigs infected with highly pathogenic porcine reproductive and respiratory syndrome virus(HP-PRRSV)[J]. Journal of Applied Genetics,56(3):355-363.
(責任编辑 兰宗宝)

