茶树细胞色素P450基因CYP710A1的克隆及差异表达特征分析
单睿阳,陈常颂,钟秋生,林郑和,陈志辉,游小妹
(福建省农业科学院茶叶研究所,福建 福安 355015)
植物细胞色素P450(简称P450,Cyt P450)是一类含有血红素和疏基的氧化酶系,该酶系能与植物细胞内的质体、内质网和高尔基体等细胞器膜结合且具有复杂的生物学功能,因而近年来成为学术界的研究热点[1]。植物P450基因的命名一般以氨基酸序列的相似性作为依据,把同源性大于40%划归为同一家族成员,把同源性大于55%的视为同一家族的另一亚家族成员。随着转录组测序的发展,越来越多的植物P450被鉴定出来,目前已在拟南芥、玉米、大豆、水稻、番茄和蓖麻等模式农作物中发现了大量P450基因[2]。
P450在植物体中担当着重要的生物学功能,按照前人的研究结果,这些功能大致可分为2类,第一类是具有代谢解毒功能,它可催化一些外来物质的降解,使植物对一些除草剂、杀虫剂和环境毒物等产生抗性。例如水稻能通过P450的脱甲基(demethylation)作用降解苄嘧磺隆(bensulfuron-methyl,BSM)[3]。小麦能通过P450的环甲基羟基化作用(cyclomethylhydroxylation)降解绿麦隆(chlortoluron)[4];第二类是能催化植物体的一些内源性物质转化为次生代谢产物,使植物抵抗一些外源性物质如昆虫、细菌的侵扰,增强植物的逆境生存能力。如植物P450在生物碱(alkaloid)、萜类(terpene)、植物激素类(phytohormone)、木质素(lignin)和黄酮类(flavonoid)等的生物合成中起到重要的催化作用,这些物质在植物抗虫、抗病过程中起到重要的作用[5]。
茶树是我国重要的经济作物,而选育具有抗虫、抗病性强的茶树新品种是育种工作者的重要目标之一,因此深入挖掘并研究茶树P450的功能对今后茶树的育种工作具有重要的指导意义。基于此,本研究从NCBI中下载获得了茶树全转录组数据库(Taxonomy ID:4442),从该数据库中选取了茶树CYP710A1的基因片段,采用RT-PCR技术对该基因片段进行了碱基校正,并通过RACE技术获得了基因全长。通过相关分子生物学分析软件,分析了该基因的氨基酸结构、蛋白质理化性质、系统进化树及蛋白功能域。最后,通过qRT-PCR技术检测了该基因在茶树不同叶片中的表达特征。
1 材料与方法
1.1 试验材料及主要试剂
本研究以常规品种铁观音为试验材料,采样部位为单芽、一叶、二叶、三叶、四叶、五叶。样品采摘于福安市社口镇福建省农业科学院茶叶研究所2号山品种园(东经119°32′11″~119°38′之间,北纬27°8′~27°13′40″)。构建cDNA模板所用的植物总RNA提取试剂盒RNAprep pure plant kit (dp441)及反转录试剂盒FastKing RT Kit(With gDNase)购于Invitrogen公司;cDNA全长克隆所用的SMARTer®RACE 5′/3′Kit试剂盒以及荧光定量所用的FastFire qPCR PreMix(SYBR Green)试剂盒购于TaKaRa公司;普通分子试剂Taq DNA聚合酶、dNTPs、DNase/RNase Free Water、DL2000 Marker及T4 DNA连接酶等均购于根生化科技(北京)有限公司。
1.2 总RNA提取及cDNA模板的合成
长势良好、叶片生长均匀的铁观音一芽一叶,用液氮冷冻后将其研磨成粉,参照植物总RNA提取试剂盒说明书,提取铁观音的总RNA,使用1.5%的琼脂糖凝胶检测所提取RNA的完整性,再用NanoDrop 2000分光光度计检测其纯度。最后,通过RNA反转录试剂盒,以提取的RNA为模板,将其反转录成克隆所用的cDNA模板。
1.3 茶树CYP710A1基因的中间片段克隆
在NCBI的物种分类学数据库中找到茶树的转录组基因信息(Taxonomy ID:4442),通过下载获得了已登入的茶树转录组mRNA。分析发现,该转录组共有34万多条不完整mRNA信息,但无完整的基因序列命名。本研究通过建立本地Blast数据库(方法参照华大基因“windows系统下本地Blast的实现”),在windows系统下重新建立单机版的茶树转录组数据库,通过比对分析获得茶树CYP710A1基因的序列片段。
根据已有的茶树CYP71A26序列信息,通过Primer 5.0软件设计一对简并引物710A1-F1/R1(表1),并以已构建的茶树cDNA为模板,通过RT-PCR技术进行扩增。RT-PCR反应程序为:94℃总变性3 min;94℃预变性30 s,59℃退火60 s,72℃延伸1.5 min,共33个循环;最后,72℃延伸15 min后,4℃保存备用。紧接着,将目的条带进行双酶切与载体pMD19-T进行连接,并进行转化、蓝白斑挑选后送往测序公司(上海铂尚生物技术有限公司)委托测序。
1.4 茶树CYP710A1基因的5′及3′ RACE扩增
获得茶树CYP710A1的中间序列后,在头部5′端设计一对特异性引物710A1-R2-outer与710A1-R3-inner(表1)进行5′ RACE扩增。紧接着,在尾部3′端设计另外一对特异性引物710A1-F2-outer与710A1-F3-inner(表1)进行3′ RACE扩增。5′与3′RACE反应程序参照步骤1.3。最后,使用DNAstar 8.0软件,将三段序列的重复序列去除,并拼接获得茶树CYP710A1的全长序列。
1.5 茶树CYP710A1基因的生物信息学分析
茶树CYP710A1基因的氨基酸结构、蛋白质理化性质、三级结构、蛋白功能域及系统进化树的分析方法及分析的网站参见单睿阳等[2]。
1.6 茶树CYP710A1基因的表达检测
试验茶样取自于长势良好、叶片生长均匀的铁观音茶园,并于新梢的芽头、一芽1~5叶共6个叶外分别取样,置于-80℃冰箱中保存备用。参照步骤1.2,将所取样品反转录成cDNA模板,用于检测茶树CYP710A1基因在不同叶片中的表达含量。根据已获得的茶树CYP710A1基因全长,采用Primer 5.0软件设计一对特异性的荧光定量引物710A1-F4/R4(表1)及一对荧光定量引物GAPDH-F1/R1(表1),参照荧光定量试剂盒的操作步骤,配制每样20 μL的总反应体系:正反引物各1.0 μL;样品cDNA 1.0 μL;SYBR Green 10.0 μL;最后补ddH2O 7.0 μL。qRT-PCR 反应程序为:95℃总变性30s;95℃预变性5s,60℃退火30s,72℃延伸30 s,共33个循环;最后,采用比较CT的计算方法(2-ΔΔCT)计算茶树CYP710A1基因在不同叶片中的表达含量。

表1 设计的引物
注:表中的F为基因的正向引物,R为基因的反向引物,inner为基因的内引物,outer为基因的外引物。
1.7 数据统计与分析
qRT-PCR每组样品设3个重复,并采用means±SE取均值的计算方法,对每组数据于Microsoft Office Excel软件中进行作图,差异性显著分析由SPSS 13.0软件中的邓肯氏(Duncan’s multiple range test)单因素方差(one-way ANOVA)方法分析完成(P<0.05)。
2 结果与分析
2.1 茶树CYP710A1基因的全长克隆
首先,通过RT-PCR技术克隆获得了1 022 bp(图1-条带2)的CYP710A1中间序列。然后,通过5′ RACE技术克隆得了281 bp(图1-条带1)的CYP710A1头部序列,通过3′ RACE技术克隆得了610 bp(图1-条带3)的尾部序列。最后,拼接得到1 911 bp的CYP710A1基因全长。
2.2 茶树CYP710A1基因的生物信息学分析
2.2.1 蛋白质理化性质分析 通过ProtParam网站对CYP710A1基因全长进行了分析,结果表明CYP710A1含有1 506 bp的编码序列(ORF),能够编码501个氨基酸(图2),所编码蛋白的分子式为C1772H2803N481O526S12,分子量大小为57.06 kDa,理论等电点为7.64,总的亲水性平均系数-0.092。
2.2.2 系统进化树分析 在NCBI比对了茶树CYP710A1基因的同源性,获得了与该基因同源性较高的博落回Macleayacordata、莲花Nelumbonucifera、胡桃Juglansregia、栓皮栎Quercussuber、葡萄Vitisvinifera、黄花蒿Artemisiaannua、莴苣Lactucasativa、向日葵Helianthusannuus、风铃椒Capsicumbaccatum和芝麻Sesamumindicum的CYP710A1基因序列。将以上10个基因序列与茶树CYP710A1基因制成fasta格式文档,导入至MEGA 4.0软件中,利用邻位相连法(neighbor-joining,NJ)的1 000次多重比对方法,构建了茶树CYP710A1基因的系统进化树(图3)。从该进化树可以看出,茶树CYP710A1基因与风铃椒C.baccatum亲缘关系较近,而与博落回M.cordata亲缘关系较远。

图1茶树CYP710A1基因的全长克隆
Fig.1FulllengthofCYP710A1geneclonedfromC.sinensis
注:M1-M3条带为DNA Marker 2000,1-3条带分别
为CYP710A1基因的中间、头部和尾部序列。

图2 茶树 CYP710A1基因编码的氨基酸序列Fig.2 Sequence of ORF from CYP710A1 gene of C.sinensis

图3 茶树 CYP710A1基因的系统进化树Fig.3 Phylogenetic tree of CYP710A1 gene from C.sinensis 注:树中左侧包含的数字代表不同物种间亲缘关系的远近。
2.2.3 三级结构预测 进一步分析了CYP710A1蛋白的三级结构,结果显示该模型主要由α螺旋和β折叠组成,该模型的GMQE(Global Model Quality Estimation,模型质量估测)值为0.65(值越接近1,表明建模质量越好),表明模型可信度较高。
2.2.4 蛋白功能域及亚细胞定位分析 通过SMART网站,进一步分析了茶树CYP710A1蛋白的功能结构域(functional domain structure)。结果表明,CYP710A1具有一个P450蛋白结构域(Pfam domain),位于编码序列的第34~475(图5-黑色部分)位点上。另外,蛋白含有一个由“WLTLAPYLFSLVLLLVLLEQIFY”组成的跨膜结构域(transmembrane structure)(图5-蓝色部分),位于编码序列的第5~27位点上。
2.3 茶树CYP71A26基因在不同叶片间的差异表达分析
研究了CYP710A1基因在不同叶片间的表达差异。结果表明,CYP710A1基因在芽头的表达量最低,在一叶、二叶、三叶中的表达量依次升高,而后在四叶、五叶中的表达量依次回落。其中,在三叶中的最高表达量是芽头的38.17倍。
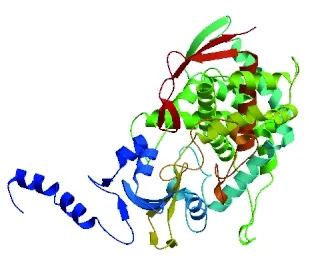
图4 茶树 CYP710A1蛋白的三级结构Fig.4 Tertiary structure of CYP710A1 of C.sinensis

图5 茶树 CYP710A1的蛋白功能域Fig.5 Functional domain of CYP710A1 protein of C.sinensis注:图下方的数字代表编码的氨基酸顺序。

图6 茶树CYP710A1基因的在不同叶片间的表达量Fig.6 Expression of CYP710A1 gene in leaves at different positions on a C.sinensis plant
3 讨论与结论
本研究利用NCBI中的茶树转录组数据库,获得了茶树CYP710A1的部分片段。通过RT-PCR及RACE技术克隆了该基因的全长。该基因全长含有1 506 bp的编码序列(ORF),能够编码502个氨基酸,具有一段较长的P450蛋白结构域(Pfam domain),从而说明该基因为典型的茶树P450基因。同时,该蛋白具有能与细胞膜结合的小段跨膜结构域(transmembrane structure),与前人所阐述的P450多数为跨膜蛋白,且多数位于细胞内的如质体、内质网和高尔基体等细胞器膜上相符[2]。
植物在自然生长过程中,不可避免的会受到外界病虫害的侵扰,而P450在抵抗侵染过程中发挥着重要的作用[6]。如Wang等[7]研究表明,当蚜虫Myzuspersicae为害烟草叶片后,烟草叶片中的腺毛能激发细胞色素P450(CYP51)的特异表达,从而提高对蚜虫的抵抗能力。以及P450所催化的内源性物质代谢,能将苯丙烷类物质转化成有毒的花椒毒素(xanthotoxin),提高棉花对棉铃虫Helicoverpaarmigera的抵抗能力[8]。郭萧等[9]研究了取食茶树不同成熟度叶片对茶尺蠖Ectropisoblique生长发育的影响。结果表明,嫩叶有利于茶尺蠖的生长,而老叶则不利于茶尺蠖种群的发生。本研究对CYP710A1在不同叶片的表达含量做了相应的检测,结果表明CYP710A1基因在芽头的表达量最低,在一叶、二叶、三叶中的表达量依次升高,而后在四叶、五叶中的表达量依次回落,这是否与不同成熟度的叶片对病虫害具有不同的抵抗能力相关,后续仍需做进一步研究。

